É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Caracterização de uma cepa patogênica de Escherichia coli derivada de Oreochromis spp. Fazendas usando sequenciamento de genoma completo
Neste Artigo
Resumo
A viabilidade de estratégias de sequenciamento do genoma completo (WGS) usando instrumentos de bancada simplificou a interrogação do genoma de cada micróbio de relevância para a saúde pública em um ambiente de laboratório. Uma adaptação metodológica do fluxo de trabalho para WGS bacteriano é descrita e um pipeline de bioinformática para análise também é apresentado.
Resumo
A aquicultura é um dos setores produtores de alimentos que mais crescem em todo o mundo e o cultivo de tilápia (Oreochromis spp.) constitui a principal variedade de peixes de água doce cultivada. Como as práticas de aquicultura são suscetíveis à contaminação microbiana derivada de fontes antropogênicas, é necessário o uso extensivo de antibióticos, levando os sistemas de aquicultura a se tornarem uma importante fonte de bactérias patogênicas e resistentes a antibióticos de relevância clínica, como a Escherichia coli (E. coli). Aqui, as características de resistência antimicrobiana, virulência e mobiloma de uma cepa patogênica de E. coli , recuperada de Oreochromis spp. cultivada no interior, foram elucidadas por meio de sequenciamento de genoma completo (WGS) e análise in silico . Foram realizados testes de suscetibilidade a antimicrobianos (AST) e WGS. Além disso, o grupo filogenético, o sorotipo, a tipagem de sequência multilocus (MLST), a resistência antimicrobiana adquirida, a virulência, o plasmídeo e o conteúdo de prófagos foram determinados usando diversas ferramentas web disponíveis. O isolado de E. coli apresentou apenas suscetibilidade intermediária à ampicilina e foi caracterizado como cepa ONT:H21-B1-ST40 por tipagem baseada em WGS. Embora apenas um único gene relacionado à resistência antimicrobiana tenha sido detectado [mdf(A)], vários genes associados à virulência (VAGs) do patótipo enteropatogênico atípico de E. coli (aEPEC) foram identificados. Além disso, a carga de replicões plasmídicos de grandes grupos plasmídicos e 18 regiões associadas a prófagos foram detectadas. Em conclusão, a caracterização do WGS de um isolado de aEPEC, recuperado de uma piscicultura em Sinaloa, México, permite insights sobre seu potencial patogênico e o possível risco para a saúde humana do consumo de produtos aquícolas crus. É necessário explorar técnicas de sequenciamento de próxima geração (NGS) para estudar microrganismos ambientais e adotar uma estrutura de saúde única para aprender como os problemas de saúde se originam.
Introdução
A aquicultura é um dos setores produtores de alimentos que mais crescem em todo o mundo, e suas práticas de produção destinam-se a satisfazer a crescente demanda de alimentos para consumo humano. A produção aquícola global triplicou de 34 milhões de toneladas (Mt) em 1997 para 112 Mt em 20171. Os principais grupos de espécies, contribuindo para quase 75% da produção, foram algas marinhas, carpas, bivalves, bagres e tilápias (Oreochromis spp.) 1. No entanto, o aparecimento de doenças causadas por entidades microbianas é inevitável devido à piscicultura intensiva, levando a potenciais perdas econômicas2.
O uso de antibióticos em práticas de piscicultura é bem conhecido por prevenir e tratar infecções bacterianas, principal fator limitante da produtividade 3,4. No entanto, os antibióticos residuais se acumulam nos sedimentos e na água da aquicultura, exercendo pressão seletiva e modificando as comunidades bacterianas associadas aos peixes e residentes 5,6,7,8. Consequentemente, o ambiente de aquicultura serve como um reservatório para genes de resistência antimicrobiana (ARGs) e o surgimento e disseminação de bactérias resistentes a antibióticos (ARB) no meio circundante9. Além dos patógenos bacterianos comumente observados que afetam as práticas de piscicultura, membros da família Enterobacteriaceae são frequentemente encontrados, incluindo cepas patogênicas humanas de Enterobacter spp., Escherichia coli, Klebsiella spp. e Salmonella spp.10. E. coli é o microrganismo mais comum isolado da farinha de peixe e da água na piscicultura 11,12,13,14,15.
E. coli é uma bactéria gram-negativa versátil que habita o trato gastrointestinal de mamíferos e aves como um membro comensal de sua microbiota intestinal. No entanto, E. coli possui uma capacidade altamente adaptativa de colonizar e persistir em diferentes nichos ambientais, incluindo solo, sedimentos, alimentos e água16. Devido ao ganho e perda de genes através do fenômeno da transferência horizontal de genes (HGT), a E. coli evoluiu rapidamente para um patógeno resistente a antibióticos bem adaptado, capaz de causar um amplo espectro de doenças em humanos e animais17,18. Com base na origem do isolamento, as variantes patogênicas são definidas como E. coli patogênica intestinal (InPEC) ou E. coli patogênica extra-intestinal (ExPEC). Além disso, o InPEC e o ExPEC são subclassificados em patótipos bem definidos de acordo com a manifestação da doença, antecedentes genéticos, características fenotípicas e fatores de virulência (FV)16,17,19.
A cultura tradicional e as técnicas moleculares para cepas patogênicas de E. coli têm permitido a rápida detecção e identificação de diferentes patotipos. No entanto, podem ser demorados, trabalhosos e frequentemente requerem alto treinamento técnico19. Além disso, nenhum método único pode ser usado para estudar de forma confiável todas as variantes patogênicas de E. coli devido à complexidade de seu histórico genético. Atualmente, essas desvantagens foram superadas com o advento das tecnologias de sequenciamento de alto rendimento (HTS). Abordagens de sequenciamento de genoma completo (SGB) e ferramentas de bioinformática melhoraram a exploração do DNA microbiano de forma acessível e em larga escala, facilitando a caracterização aprofundada de micróbios em uma única execução, incluindo variantes patogênicas intimamente relacionadas20,21,22. Dependendo das questões biológicas, várias ferramentas de bioinformática, algoritmos e bancos de dados podem ser usados para realizar a análise de dados. Por exemplo, se o objetivo principal é avaliar a presença de ARGs, VFs e plasmídeos, ferramentas como ResFinder, VirulenceFinder e PlasmidFinder, juntamente com seus bancos de dados associados, podem ser um bom ponto de partida. Carriço et al.22 forneceram uma visão detalhada dos diferentes softwares de bioinformática e bases de dados relacionadas aplicadas para análise microbiana WGS, desde o pré-processamento de dados brutos até a inferência filogenética.
Vários estudos têm demonstrado a ampla utilidade do WGS para a interrogação do genoma em relação aos atributos de resistência antimicrobiana, potencial patogênico e rastreamento do surgimento e relações evolutivas de variantes clinicamente relevantes de E. coli provenientes de diversas origens23,24,25,26 . O WGS permitiu a identificação de mecanismos moleculares subjacentes à resistência fenotípica aos antimicrobianos, incluindo os mecanismos de resistência raros ou complexos. Isso ocorre por meio da detecção de variantes ARG adquiridas, novas mutações em genes-alvo de drogas ou regiões promotoras27,28. Além disso, o WGS oferece o potencial de inferir perfis de resistência antimicrobiana sem a necessidade de conhecimento prévio sobre o fenótipo de resistência de uma cepa bacteriana29. Alternativamente, o WGS permitiu a caracterização dos elementos genéticos móveis (MGEs) portadores de características de resistência antimicrobiana e virulência, o que impulsionou a evolução do genoma bacteriano dos patógenos existentes. Por exemplo, a aplicação de WGS durante a investigação do surto alemão de E. coli em 2011 resultou na descoberta das características genômicas únicas de um patótipo de E. coli aparentemente novo; Curiosamente, essas cepas de surto originaram-se do grupo enteroagregativo E. coli (EAEC), que adquiriu o prófago que codifica a toxina Shiga do patótipo30 entero-hemorrágico de E. coli (EHEC).
Este trabalho apresenta uma adaptação metodológica do fluxo de trabalho para SGB bacterianos utilizando um sequenciador de bancada. Além disso, um pipeline de bioinformática é fornecido usando ferramentas baseadas na web para analisar as sequências resultantes e apoiar ainda mais os pesquisadores com experiência limitada ou nenhuma em bioinformática. Os métodos descritos permitiram a elucidação das características de resistência antimicrobiana, virulência e mobiloma de uma cepa patogênica de E. coli ACM5, isolada em 2011 de Oreochromis spp. cultivada no interior em Sinaloa, México12.
Protocolo
NOTA: A cepa ACM5 de E. coli foi recuperada por meio do processamento e cultivo da amostra de peixes para determinação de coliformes fecais (CF)12. Durante a amostragem dos peixes, os peixes não apresentaram sinais clínicos de doença, infecção bacteriana ou fúngica, prevalecendo uma temperatura média de 22,3 °C. Após o isolamento, o isolado de E. coli foi submetido a testes bioquímicos e criopreservado em caldo de infusão cardíaca cerebral (BHI) com DMSO (8% v/v) como agente crioprotetor.
1. Reativação da cultura de estoque de E. coli ACM5 congelada
- A partir do estoque bacteriano congelado, abra o tubo e use um laço estéril, ponta de pipeta ou palito de dente para raspar a superfície da cultura bacteriana congelada.
- Estice as bactérias numa placa de ágar Luria-Bertani (LB) e incube a 37 ± 2 °C durante 24 h.
2. Determinação da suscetibilidade antibacteriana
NOTA: O teste de suscetibilidade antimicrobiana aqui descrito corresponde ao método de difusão em disco baseado nas diretrizes do Clinical and Laboratory Standards Institute (CLSI) (M02 Ed13:2018)31. E. coli A cepa ATCC 25922 é necessária para fins de controle de qualidade.
- Preparação de inóculo pelo método da suspensão de colónias
- A partir da placa LB incubada na etapa 1.2, riscar uma ou duas colônias em uma placa de ágar Mueller-Hinton (MH) e incubar a 37 ± 2 °C por 18-24 h.
- Use um laço estéril para colher duas ou três colônias do isolado de E. coli recém-subcultivado na placa de ágar MH e ressussuscitá-las em 3 mL de caldo de MH estéril ou solução salina a 0,85% (p/v), misturando completamente por vórtice para obter uma suspensão bacteriana uniforme.
- Usando um espectrofotômetro UV-visível (ver Tabela de Materiais), ajuste a suspensão bacteriana à turbidez comparável a um padrão MacFarland de 0,5 (equivalente a aproximadamente 1-2 x 108 células/mL). Se a suspensão bacteriana for muito leve ou muito pesada, adicione mais colônias de E. coli ou caldo de MH, conforme apropriado. Use o inóculo preparado dentro de 15 minutos após a preparação.
- Inoculação das placas de ágar de ensaio
- Mergulhe um cotonete estéril na suspensão bacteriana ajustada. Gire o cotonete várias vezes e pressione-o firmemente na parede interna do tubo para remover o excesso de líquido do cotonete.
- Inocule uma placa de ágar MH espalhando o cotonete 3x sobre toda a superfície do ágar, girando a placa 60° de cada vez. Esfregue a borda do ágar também para garantir uma distribuição uniforme do inóculo.
- Deixe a tampa da placa de Petri aberta por 3-5 minutos para permitir que a umidade evapore.
- Aplicação do disco antimicrobiano em placas de ágar inoculadas
- Distribua uniformemente e pressione cada disco antimicrobiano (ver Tabela de Materiais) para baixo na superfície das placas de ágar inoculadas para garantir o contato completo. Inverter as placas no prazo de 15 min após a aplicação do disco e incubar a 35 ± 2 °C durante 16-18 h.
NOTA: Devem ser utilizados um máximo de 12 e seis discos em placas de Petri de 150 mm e 100 mm, respectivamente.
- Distribua uniformemente e pressione cada disco antimicrobiano (ver Tabela de Materiais) para baixo na superfície das placas de ágar inoculadas para garantir o contato completo. Inverter as placas no prazo de 15 min após a aplicação do disco e incubar a 35 ± 2 °C durante 16-18 h.
- Após a incubação, medir a zona de tamanhos de inibição usando um paquímetro de Vernier e interpretar os diâmetros resultantes de acordo com os critérios de ponto de interrupção do CLSI (M100 - Tabela 2A)32.
3. Extração e quantificação do DNA genômico (gDNA)
- Extração de DNA genômico
- Ressuspender uma alça cheia de colónias de E. coli recém-cultivadas em 5 ml de caldo LB e incubar durante a noite numa incubadora agitada (180 rpm) a 37 ± 2 °C.
- Centrifugar a suspensão bacteriana a 3.500 x g durante 5 min e eliminar cuidadosamente o sobrenadante.
- Extraia gDNA seguindo as diretrizes do kit de extração de DNA (consulte Tabela de Materiais).
- Verifique a pureza do gDNA medindo a densidade óptica em 260/280 nm (proporção: >1,8) e 260/230 nm (proporção: 2,0-2,2) usando um espectrofotômetro UV-visível. Realizar eletroforese em gel de agarose a 0,8% para verificar a integridade do gDNA.
- Quantificação do DNA genômico
NOTA: Use apenas tubos de PCR de parede fina, transparentes e de 0,5 mL aprovados pelo fabricante do kit de ensaio de fluorescência (consulte Tabela de materiais).- Configure o número necessário de tubos para amostras e padrões de ensaio. Prepare a solução de ensaio de trabalho misturando o componente A e o componente B fornecido no kit, seguindo as diretrizes do fabricante.
- Preparar as normas de ensaio adicionando 190 μL da solução de trabalho e 10 μL de cada norma ao tubo apropriado (são necessárias duas normas).
- Adicionar 198 μL da solução de trabalho e 2 μL da amostra de ADN ao tubo adequado. Misture vigorosamente por vórtice todos os tubos por 5 s. Tenha cuidado para não criar bolhas.
- Incubar todos os tubos durante 2 min à temperatura ambiente, protegidos da luz. Meça a fluorescência de todos os tubos com base nas diretrizes do fabricante usando o fluorômetro (consulte Tabela de materiais).
- Ajustar adequadamente a concentração da amostra de gDNA para o processo de sequenciamento.
4. Preparação da biblioteca de ADN
NOTA: A preparação e o sequenciamento da biblioteca de DNA foram realizados seguindo as diretrizes e protocolos do fabricante (consulte Tabela de Materiais). A concentração inicial de gDNA é de 4,0 ng.
- Marcação, amplificação por PCR e indexação
- Em um tubo de 0,2 mL, adicione 2,5 μL de tampão de marcação e 2 μL de gDNA de entrada (2,0 ng/μL). Misture suavemente por pipetagem.
- Adicione 1 μL de tampão de amplificação e misture suavemente por pipetagem. Gire para baixo a 280 x g por 1 min à temperatura ambiente.
- Colocar as amostras num termociclador (ver Tabela de Materiais) e executar o seguinte programa de PCR: 55 °C durante 5 min, e depois manter a 10 °C. Quando as amostras atingirem 10 °C, proceder imediatamente à neutralização da reacção.
- Adicionar 1 μL de tampão neutralizante aos tubos de amostra e misturar suavemente por pipetagem. Gire a 280 x g por 1 min e incube por 5 min à temperatura ambiente.
- Adicione 1,7 μL de cada adaptador de índice (ou seja, índices i7 e i5) e 3 μL de indexação PCR master mix. Misture suavemente por pipetagem. Gire para baixo a 280 x g por 1 min à temperatura ambiente.
- Colocar as amostras no termociclador e efectuar uma segunda reacção de PCR da seguinte forma: 72 °C durante 3 min, 95 °C durante 30 s, 18 ciclos de 95 °C durante 10 s, 55 °C durante 30 s, 72 °C durante 30 s, 72 °C durante 5 min e manter a 10 °C.
- Limpeza amplificada da biblioteca
- Misture por vórtice da solução comercial de grânulos magnéticos (consulte Tabela de materiais) com base nas diretrizes do fabricante.
- Transfira a amostra de gDNA marcada/indexada para um novo tubo de 1,5 mL e adicione 0,6 μL de esferas magnéticas para cada μL do volume final da amostra de gDNA (≈13 μL). Misture suavemente por pipetagem e incube por 5 min à temperatura ambiente.
- Coloque os tubos de amostra num suporte magnético (ver Tabela de Materiais) durante 2 minutos até que o sobrenadante se dissipe. Retire e descarte o sobrenadante com cuidado sem perturbar as contas.
- Adicionar 200 μL de etanol a 80% preparado na hora sem misturar. Incubar por 30 s até que o eluado limpe e remova cuidadosamente e descarte o sobrenadante sem perturbar as contas.
- Execute uma segunda etapa de lavagem. Adicione 200 μL de etanol a 80% às contas e incube por 30 s. Depois que o eluído clarear, remova e descarte o sobrenadante e seque as contas ao ar livre por 10 min.
- Adicione 15 μL de tampão tris de 10 mM (pH 8) às contas e misture suavemente por pipetagem. Incubar durante 2 min à temperatura ambiente. Coloque os tubos de amostra novamente no rack magnético por 2 min, permitindo que o sobrenadante se dissipe.
- Transfira cuidadosamente 14 μL do sobrenadante do tubo de amostra para um novo tubo de 0,2 mL. Esta é a biblioteca limpa.
NOTA: Neste ponto, a biblioteca limpa pode ser armazenada a -20 °C por até 7 dias.
- Normalização da biblioteca
- Prossiga para quantificar as bibliotecas limpas por ensaio de fluorescência, conforme descrito na etapa 3.2, exceto na etapa 3.2.5.
- Visualize as bibliotecas limpas em uma eletroforese em gel de agarose a 1% para determinar os tamanhos médios dos fragmentos.
- Calcule o valor de molaridade da biblioteca usando a seguinte equação:
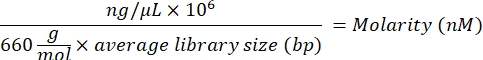
- Usando o valor de molaridade, calcule os volumes adequados de buffer de ressuspensão (RSB) e bibliotecas limpas para diluir a biblioteca individual em uma concentração inicial de 10 nM.
5. Agrupamento de bibliotecas, desnaturalização e início do sequenciador
- Descongele o cartucho reagente seguindo as instruções do fabricante. Extrair a célula de fluxo do frigorífico (4 °C) e levá-la à temperatura ambiente antes da sequenciação.
- Agrupe 5 μL de cada biblioteca normalizada (10 nM) em um tubo de baixa ligação de 1,5 mL. Dilua a biblioteca agrupada a 4 nM com o volume adequado de RSB.
- Adicione 5 μL da biblioteca agrupada (4 nM) e 5 μL de NaOH 0,2 N em um novo tubo de baixa ligação de 1,5 mL. Misture brevemente por vórtice, gire a 280 x g por 1 min e incube por 5 min à temperatura ambiente para desnaturar a biblioteca agrupada em fios únicos.
- Misture suavemente 10 μL de biblioteca agrupada desnaturada e 990 μL de tampão de hibridização pré-resfriado por pipetagem e coloque no gelo até que a diluição final para o carregamento do sequenciador seja realizada. A concentração da biblioteca desnaturada agrupada é às 20 horas.
- Descongelar e preparar uma biblioteca de controlo (ver Tabela de Materiais) a uma concentração de 4 nM misturando 2 μL de biblioteca de controlo (10 nM) e 3 μL de água isenta de nuclease.
- Misture 5 μL da biblioteca de controle (4 nM) e 5 μL de NaOH 0,2 N recém-preparado. Misture brevemente por vórtice, gire a 280 x g por 1 min e incube por 5 min à temperatura ambiente para desnaturar a biblioteca de controle em fios simples.
- Misture suavemente 10 μL de biblioteca de controle desnaturada e 990 μL de tampão de hibridização pré-resfriado por pipetagem e coloque no gelo até que a diluição final para o carregamento do sequenciador seja feita. A concentração da biblioteca de controle desnaturada é às 20 horas.
- Em um novo tubo de 1,5 mL de baixa ligação, combine 594 μL da biblioteca agrupada desnaturada a 20 pM e 6 μL da biblioteca de controle desnaturada às 20 pM. Misture corretamente.
- Diluir a mistura final da biblioteca (20 pM) para uma concentração de carga final de 1,2 pM num volume de 600 μL utilizando o tampão de hibridização pré-refrigerado.
- Antes de carregar a mistura final da biblioteca no cartucho reagente, execute um pré-tratamento térmico adicional para obter uma carga eficiente na célula de fluxo. Incubar a mistura final da biblioteca durante 2 min a 96 °C. Inverta o tubo para misturar e coloque o tubo no gelo por 5 min.
- Carregue 500 μL da mistura final da biblioteca no reservatório projetado no cartucho do reagente. Inicie o sequenciamento seguindo as diretrizes. Carregue a célula de fluxo e o cartucho do reagente e configure a execução de sequenciamento.
6. Análise de dados de sequência
NOTA: Verifique o Arquivo Suplementar 1 para obter uma descrição mais detalhada do pré-processamento geral de dados do WGS, software, configurações de parâmetros e análise de sequência do genoma de E. coli.
- Conecte-se ao servidor de dados de sequenciamento e baixe os arquivos FASTQ.
- Avalie a qualidade inicial dos dados brutos de sequência com software de terceiros. Remova sequências de adaptadores remanescentes, bases de baixa qualidade (Arquivo Suplementar 1).
- Monte os dados de sequenciamento com verificação de qualidade em nível de contig ou scaffold usando software de terceiros (consulte Arquivo Suplementar 1).
- Execute a anotação do genoma enviando o arquivo FASTA contendo o genoma montado para o servidor RAST (https://rast.nmpdr.org/).
- Carregue o arquivo FASTA nas plataformas da Web Center for Genome Epidemiology (CGE) (http://www.genomicepidemiology.org/services/) e ClermonTyping (http://clermontyping.iame-research.center/index.php) para identificar características epidemiológicas, ARGs, VAGs e plasmídeos (consulte o Arquivo Suplementar 1).
- Carregue o arquivo FASTA no servidor PHASTER para identificar sequências de prófagos (https://phaster.ca/).
Resultados
A suscetibilidade aos antimicrobianos foi determinada pelo método de difusão em disco e interpretada pelos critérios de ponto de interrupção do CLSI para 12 antibióticos abrangendo seis classes antimicrobianas distintas, ou seja, aminoglicosídeos, β-lactâmicos, fluoroquinolonas, nitrofuranos, fenicol e antagonistas da via do folato. A E. coli ACM5 apresentou sensibilidade a todos os antibióticos, exceto a um fármaco β-lactâmico. Quatro drogas β-lactâmicas foram testadas: ampicilina, carbenicilina,...
Discussão
Este estudo apresenta uma adaptação do fluxo de trabalho do WGS bacteriano utilizando um sequenciador de bancada e um pipeline para caracterização genômica de uma variante patogênica de E. coli. Dependendo da plataforma de sequenciamento utilizada, os tempos de resposta (TATs) para procedimentos laboratoriais úmidos (cultivo bacteriano, extração de gDNA, preparação de biblioteca e sequenciamento) e análise de sequência podem variar, particularmente se bactérias de crescimento lento forem estudadas....
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Ao Conselho Nacional de Ciência e Tecnologia do México (CONACyT) pela bolsa de doutorado concedida a José Antonio Magaña-Lizárraga [No. 481143].
Materiais
| Name | Company | Catalog Number | Comments |
| Accublock Mini digital dry bath | Labnet | D0100 | Dry bath for incubation of tubes |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Magnetic beads in solution for DNA library purification |
| DeNovix DS-11 | DeNovix Inc. | UV-Vis spectophotometer to check the quality of the gDNA extracted | |
| DNA LoBind Tubes | Eppendorf | 0030108418 | 1.5 mL PCR tubes for DNA library pooling |
| DynaMag-2 Magnet | Invitrogen, Thermo Fisher Scientific | 12321D | Magnetic microtube rack used during magnetic beads-based DNA purification |
| Gram-negative Multibac I.D. | Diagnostic reseach (Mexico) | PT-35 | Commercial standard antibiotic disks for antimicrobial susceptibility testing |
| MiniSeq Mid Output Kit (300-cycles) | Illumina | FC-420-1004 | Reagent cartdrige for paired-end sequencing (2x150) |
| MiniSeq System Instrument | Illumina | SY-420-1001 | Benchtop sequencer used for Next-generation sequencing |
| MiniSpin centrifuge | Eppendorf | 5452000816 | Standard centrifuge for tubes |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1024 | Reagents to perform DNA libraries for sequencing. Includes Box 1 and Box 2 reagents for 24 samples |
| Nextera XT Index Kit v2 | Illumina | FC-131-2001, FC-131-2002, FC-131-2003, FC-131-2004 | Index set A, B, C, D |
| PhiX Control v3 | Illumina | FC-110-3001 | DNA library control for sequencing |
| Precision waterbath | LabCare America | 51221081 | Water bath shaker used for bacterial culture |
| Qubit 1X dsDNA HS Assay Kit | Invitrogen, Thermo Fisher Scientific | Q33231 | Reagents for fluorescence-based DNA quantification assay |
| Qubit 2.0 Fluorometer | Invitrogen, Thermo Fisher Scientific | Q32866 | Fluorometer used for fluorescence assay |
| Qubit Assay tubes | Invitrogen, Thermo Fisher Scientific | Q32856 | 0.5 mL PCR tubes for fluorescence-based DNA quantification assay |
| SimpliAmp Thermal Cycler | Applied Biosystems, Thermo Fisher Scientific | A24811 | Thermocycler used for DNA library amplification |
| Spectronic GENESYS 10 Vis | Thermo | 335900 | Spectophotometer used for bacterial suspension in antimicrobial susceptibility testing |
| ZymoBIOMICS DNA Miniprep Kit | Zymo Research Inc. | D4300 | Kit for genomic DNA extraction (50 preps) |
Referências
- Naylor, R. L., et al. A 20-year retrospective review of global aquaculture. Nature. 591 (7851), 551-563 (2021).
- Quesada, S. P., Paschoal, J. A. R., Reyes, F. G. R. Considerations on the aquaculture development and on the use of veterinary drugs: special issue for fluoroquinolones-a review. Journal of Food Science. 78 (9), 1321-1333 (2013).
- Defoirdt, T., Sorgeloos, P., Bossier, P. Alternatives to antibiotics for the control of bacterial disease in aquaculture. Current Opinion in Microbiology. 14 (3), 251-258 (2011).
- Stentiford, G. D., et al. New paradigms to help solve the global aquaculture disease crisis. PLOS Pathogens. 13 (2), 1006160 (2017).
- Chen, H., et al. Tissue distribution, bioaccumulation characteristics and health risk of antibiotics in cultured fish from a typical aquaculture area. Journal of Hazardous Materials. 343, 140-148 (2018).
- Zhou, M., et al. Antibiotics control in aquaculture requires more than antibiotic-free feeds: A Tilapia farming case. Environmental Pollution. 268, 115854 (2021).
- Feng, Y., et al. Ecological effects of antibiotics on aquaculture ecosystems based on microbial community in sediments. Ocean & Coastal Management. 224, 106173 (2022).
- Shen, X., Jin, G., Zhao, Y., Shao, X. Prevalence and distribution analysis of antibiotic resistance genes in a large-scale aquaculture environment. Science of The Total Environment. 711, 134626 (2020).
- Su, H., et al. Contamination of antibiotic resistance genes (ARGs) in a typical marine aquaculture farm: source tracking of ARGs in reared aquatic organisms. Journal of Environmental Science and Health, Part B. 55 (3), 220-229 (2020).
- Oliveira, R. V., Oliveira, M. C., Pelli, A. Disease infection by Enterobacteriaceae family in fishes: a review. Journal of Microbiology & Experimentation. 4 (5), 00128 (2017).
- Barbosa, M. M. C., et al. Sorologia e suscetibilidade antimicrobiana em isolados de Escherichia coli de pesque-pagues. Arquivos do Instituto Biológico. 81 (1), 43-48 (2014).
- Valenzuela-Armenta, J. A., et al. Microbiological analysis of Tilapia and water in aquaculture farms from Sinaloa. Biotecnia. 20 (1), 20-26 (2018).
- Reza, R. H., Shipa, S. A., Naser, M. N., Miah, M. F. Surveillance of Escherichia coli in a fish farm of Sylhet, Bangladesh. Bangladesh Journal of Zoology. 48 (2), 335-346 (2021).
- Liao, C. -. Y., et al. Antimicrobial resistance of Escherichia coli From aquaculture farms and their environment in Zhanjiang, China. Frontiers in Veterinary Science. 8, 806653 (2021).
- Dewi, R. R., et al. Prevalence and antimicrobial resistance of Escherichia coli, Salmonella and Vibrio derived from farm-raised Red Hybrid Tilapia (Oreochromis spp.) and Asian Sea Bass (Lates calcarifer, Bloch 1970) on the west coast of Peninsular Malaysia. Antibiotics. 11 (2), 136 (2022).
- Leimbach, A., Hacker, J., Dobrindt, U. E. coli as an all-rounder: the thin line between commensalism and pathogenicity. Current Topics in Microbiology and Immunology. 358, 3-32 (2013).
- Kaper, J. B., Nataro, J. P., Mobley, H. L. T. Pathogenic Escherichia coli. Nature Reviews Microbiology. 2 (2), 123-140 (2004).
- Croxen, M. A., Finlay, B. B. Molecular mechanisms of Escherichia coli pathogenicity. Nature Reviews Microbiology. 8 (1), 26-38 (2010).
- Croxen, M. A., et al. Recent advances in understanding enteric pathogenic Escherichia coli. Clinical Microbiology Reviews. 26 (4), 822-880 (2013).
- Bertelli, C., Greub, G. Rapid bacterial genome sequencing: methods and applications in clinical microbiology. Clinical Microbiology and Infection. 19 (9), 803-813 (2013).
- Lynch, T., Petkau, A., Knox, N., Graham, M., Van Domselaar, G. A primer on infectious disease bacterial genomics. Clinical Microbiology Reviews. 29 (4), 881-913 (2016).
- Carriço, J. A., Rossi, M., Moran-Gilad, J., Van Domselaar, G., Ramirez, M. A primer on microbial bioinformatics for nonbioinformaticians. Clinical Microbiology and Infection. 24 (4), 342-349 (2018).
- Magaña-Lizárraga, J. A., et al. Draft genome sequence of Escherichia coli M51-3: a multidrug-resistant strain assigned as ST131-H30 recovered from infant diarrheal infection in Mexico. Journal of Global Antimicrobial Resistance. 19, 311-312 (2019).
- Pérez-Vázquez, M., et al. Emergence of NDM-producing Klebsiella pneumoniae and Escherichia coli in Spain: phylogeny, resistome, virulence and plasmids encoding blaNDM-like genes as determined by WGS. Journal of Antimicrobial Chemotherapy. 74 (12), 3489-3496 (2019).
- Massella, E., et al. Snapshot study of whole genome sequences of Escherichia coli from healthy companion animals, livestock, wildlife, humans and food in Italy. Antibiotics. 9 (11), 782 (2020).
- Magaña-Lizárraga, J. A., et al. Genomic profiling of antibiotic-resistant Escherichia coli isolates from surface water of agricultural drainage in north-western Mexico: detection of the international high-risk lineages ST410 and ST617. Microorganisms. 10 (3), 662 (2022).
- Saracino, I. M., et al. Next Generation sequencing for the prediction of the antibiotic resistance in Helicobacter pylori: a literature review. Antibiotics. 10 (4), 437 (2021).
- Ghosh, A., Saha, S. Survey of drug resistance associated gene mutations in Mycobacterium tuberculosis, ESKAPE and other bacterial species. Scientific Reports. 10 (1), 8957 (2020).
- Su, M., Satola, S. W., Read, T. D. Genome-based prediction of bacterial antibiotic resistance. Journal of Clinical Microbiology. 57 (3), 01405-01418 (2019).
- Brzuszkiewicz, E., et al. Genome sequence analyses of two isolates from the recent Escherichia coli outbreak in Germany reveal the emergence of a new pathotype: Entero-Aggregative-Haemorrhagic Escherichia coli (EAHEC). Archives of Microbiology. 193 (12), 883-891 (2011).
- . CLSI Performance Standards for Antimicrobial Disk Susceptibility Tests. 13th ed. CLSI standard M02. Wayne, PA: Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m02/ (2018)
- CLSI Performance Standards for Antimicrobial Susceptibility Testing. 31st ed. CLSI supplement M100. Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m100/ (2021)
- Ewing, B., Green, P. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Research. 8 (3), 186-194 (1998).
- Quainoo, S., et al. Whole-genome sequencing of bacterial pathogens: the future of nosocomial outbreak analysis. Clinical Microbiology Reviews. 30 (4), 1015-1063 (2017).
- Desai, A., et al. Identification of optimum sequencing depth especially for de novo genome assembly of small genomes using next generation sequencing data. PLoS ONE. 8 (4), 60204 (2013).
- Nishino, K., Yamada, J., Hirakawa, H., Hirata, T., Yamaguchi, A. Roles of TolC-dependent multidrug transporters of Escherichia coli in resistance to β-lactams. Antimicrobial Agents and Chemotherapy. 47 (9), 3030-3033 (2003).
- Li, M., et al. The resistance mechanism of Escherichia coli induced by ampicillin in laboratory. Infection and Drug Resistance. 12, 2853-2863 (2019).
- Ménard, L. -. P., Dubreuil, J. D. Enteroaggregative Escherichia coli heat-stable enterotoxin 1 (EAST1): a new toxin with an old twist. Critical Reviews in Microbiology. 28 (1), 43-60 (2002).
- Dubreuil, J. D. EAST1 toxin: An enigmatic molecule associated with sporadic episodes of diarrhea in humans and animals. Journal of Microbiology. 57 (7), 541-549 (2019).
- Goldstein, S., Beka, L., Graf, J., Klassen, J. L. Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing. BMC Genomics. 20 (1), 23 (2019).
- Guerrero, A., Gomez-Gil, B., Lizarraga-Partida, M. L. Genomic stability among O3:K6 V. parahaemolyticus pandemic strains isolated between 1996 to 2012 in American countries. BMC Genomic Data. 22 (1), 38 (2021).
- FAO Applications of Whole Genome Sequencing (WGS) in food safety management. Food and Agriculture Organization of the United Nations Available from: https://www.fao.org/documents/card/es/c/61e44b34-b328-4239-b59c-a9e926e327b4/ (2016)
- Rantsiou, K., et al. Next generation microbiological risk assessment: opportunities of whole genome sequencing (WGS) for foodborne pathogen surveillance, source tracking and risk assessment. International Journal of Food Microbiology. 287, 3-9 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados