Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Характеристика патогенного штамма Escherichia coli , полученного из Oreochromis spp. Фермы с использованием секвенирования всего генома
В этой статье
Резюме
Осуществимость стратегий секвенирования всего генома (WGS) с использованием настольных инструментов упростила опрос генома каждого микроба, имеющего отношение к общественному здравоохранению в лабораторных условиях. Описана методологическая адаптация рабочего процесса для бактериальных WGS и представлен конвейер биоинформатики для анализа.
Аннотация
Аквакультура является одним из самых быстрорастущих секторов производства продуктов питания во всем мире, а выращивание тилапии (Oreochromis spp.) представляет собой основной культивируемый сорт пресноводных рыб. Поскольку практика аквакультуры восприимчива к микробному загрязнению, полученному из антропогенных источников, необходимо широкое использование антибиотиков, что приводит к тому, что системы аквакультуры становятся важным источником устойчивых к антибиотикам и патогенных бактерий, имеющих клиническое значение, таких как Escherichia coli (E. coli). Здесь устойчивость к противомикробным препаратам, вирулентность и мобиломные особенности патогенного штамма E. coli , извлеченного из внутреннего выращивания Oreochromis spp., были выяснены с помощью секвенирования всего генома (WGS) и анализа in silico . Было проведено тестирование чувствительности к противомикробным препаратам (AST) и WGS. Кроме того, филогенетическая группа, серотип, типирование многолокусных последовательностей (MLST), приобретенная устойчивость к противомикробным препаратам, вирулентность, содержание плазмиды и профагов были определены с использованием различных доступных веб-инструментов. Изолят E. coli проявлял только промежуточную восприимчивость к ампициллину и характеризовался как штамм ONT:H21-B1-ST40 путем типирования на основе WGS. Хотя был обнаружен только один ген, связанный с устойчивостью к противомикробным препаратам [mdf(A)], было идентифицировано несколько вирулентно-ассоциированных генов (VAG) из атипичного энтеропатогенного патотипа E. coli (aEPEC). Кроме того, был обнаружен груз плазмидных репликонов из крупных плазмидных групп и 18 профаго-ассоциированных областей. В заключение, характеристика WGS изолята aEPEC, извлеченного с рыбной фермы в Синалоа, Мексика, позволяет понять его патогенный потенциал и возможный риск для здоровья человека, связанный с потреблением сырых аквакультурных продуктов. Необходимо использовать методы секвенирования следующего поколения (NGS) для изучения микроорганизмов окружающей среды и принять единую систему здравоохранения, чтобы узнать, как возникают проблемы со здоровьем.
Введение
Аквакультура является одним из самых быстрорастущих секторов производства продуктов питания во всем мире, и ее производственная практика предназначена для удовлетворения растущего спроса на продовольствие для потребления человеком. Мировое производство аквакультуры утроилось с 34 миллионов тонн в 1997 году до 112 тонн в 2017году1. Основными группами видов, на которые приходилось почти 75% производства, были морские водоросли, карпы, двустворчатые моллюски, сом и тилапия (Oreochromis spp.) 1. Однако появление заболеваний, вызванных микробными образованиями, неизбежно из-за интенсивного рыбоводства, что приводит к потенциальным экономическим потерям2.
Использование антибиотиков в рыбоводстве хорошо известно профилактикой и лечением бактериальных инфекций, что является основным ограничивающим фактором продуктивности 3,4. Тем не менее, остаточные антибиотики накапливаются в отложениях аквакультуры и воде, оказывая селективное давление и модифицируя связанные с рыбой и проживающие бактериальные сообщества 5,6,7,8. Следовательно, среда аквакультуры служит резервуаром для генов устойчивости к противомикробным препаратам (ARG) и дальнейшего появления и распространения устойчивых к антибиотикам бактерий (ARB) в окружающей среде9. В дополнение к бактериальным патогенам, обычно наблюдаемым, влияющим на методы рыбоводства, часто встречаются члены семейства Enterobacteriaceae, включая штаммы патогенов человека Enterobacter spp., Escherichia coli, Klebsiella spp. и Salmonella spp.10. Кишечная палочка является наиболее распространенным микроорганизмом, выделенным из рыбной муки и воды в рыбоводстве 11,12,13,14,15.
Кишечная палочка является универсальной грамотрицательной бактерией, населяющей желудочно-кишечный тракт млекопитающих и птиц как комменсальный член их кишечной микробиоты. Тем не менее, e. coli обладает высокой адаптивной способностью колонизировать и сохраняться в различных экологических нишах, включая почву, отложения, пищу и воду16. Из-за усиления и потери генов в результате явления горизонтального переноса генов (HGT) e. coli быстро превратилась в хорошо адаптированный устойчивый к антибиотикам патоген, способный вызывать широкий спектр заболеваний у людей и животных17,18. Исходя из происхождения выделения, патогенные варианты определяются как кишечная патогенная Кишечная палочка (InPEC) или внекишечная патогенная E. coli (ExPEC). Кроме того, InPEC и ExPEC подразделяются на четко определенные патотипы в соответствии с проявлением заболевания, генетическим фоном, фенотипическими признаками и факторами вирулентности (VFs)16,17,19.
Традиционная культура и молекулярные методы для патогенных штаммов E. coli позволили быстро обнаружить и идентифицировать различные патотипы. Однако они могут быть трудоемкими, трудоемкими и часто требуют высокой технической подготовки19. Кроме того, ни один метод не может быть использован для достоверного изучения всех патогенных вариантов кишечной палочки из-за сложности их генетического фона. В настоящее время эти недостатки были преодолены с появлением технологий высокопроизводительного секвенирования (HTS). Подходы к секвенированию всего генома (WGS) и биоинформационные инструменты улучшили исследование микробной ДНК по доступной цене и в больших масштабах, облегчая углубленную характеристику микробов за один прогон, включая тесно связанные патогенные варианты 20,21,22. В зависимости от биологических вопросов для выполнения анализа данных можно использовать несколько инструментов, алгоритмов и баз данных биоинформатики. Например, если основной целью является оценка наличия ARG, VF и плазмид, такие инструменты, как ResFinder, VirulenceFinder и PlasmidFinder, вместе с связанными с ними базами данных, могут быть хорошей отправной точкой. Carriço et al.22 представили подробный обзор различных программ биоинформатики и связанных с ними баз данных, применяемых для анализа микробных WGS, от предварительной обработки необработанных данных до филогенетического вывода.
Несколько исследований продемонстрировали широкую полезность WGS для опроса генома в отношении признаков устойчивости к противомикробным препаратам, патогенного потенциала и отслеживания возникновения и эволюционных отношений клинически значимых вариантов E. coli, полученных из различных источников 23,24,25,26 . WGS позволила идентифицировать молекулярные механизмы, лежащие в основе фенотипической устойчивости к противомикробным препаратам, включая редкие или сложные механизмы резистентности. Это происходит путем обнаружения приобретенных вариантов ARG, новых мутаций в генах-мишенях лекарств или промоторных областях27,28. Кроме того, WGS предлагает потенциал для вывода профилей устойчивости к противомикробным препаратам, не требуя предварительных знаний о фенотипе резистентности бактериального штамма29. В качестве альтернативы, WGS позволила охарактеризовать мобильные генетические элементы (MME), несущие как устойчивость к противомикробным препаратам, так и признаки вирулентности, что привело к эволюции бактериального генома существующих патогенов. Например, применение WGS во время расследования вспышки кишечной палочки в Германии в 2011 году привело к раскрытию уникальных геномных особенностей явно нового патотипа E. coli; Интересно, что эти штаммы вспышки произошли из группы энтерогрегации E. coli (EAEC), которая приобрела профаг, кодирующий токсин Шига, из энтерогеморрагического патотипа E. coli (EHEC)30.
В данной работе представлена методологическая адаптация рабочего процесса для бактериальных WGS с использованием настольного секвенсора. Кроме того, конвейер биоинформатики предоставляется с использованием веб-инструментов для анализа полученных последовательностей и дальнейшей поддержки исследователей с ограниченным опытом или вообще без опыта в области биоинформатики. Описанные методы позволили выяснить антимикробную устойчивость, вирулентность и мобиломные особенности патогенного штамма E. coli ACM5, выделенного в 2011 году из внутреннего выращивания Oreochromis spp. в Синалоа, Мексика12.
протокол
ПРИМЕЧАНИЕ: Штамм E. coli ACM5 был извлечен путем обработки и культивирования образца рыбы для определения фекальной кишечной палочки (FC)12. Во время отбора проб рыбы у рыбы не было клинических признаков заболевания, бактериальной или грибковой инфекции, и преобладала средняя температура 22,3 ° C. После выделения изолят E. coli подвергали биохимическому тестированию и криоконсервировали в отваре для инфузии сердца мозга (BHI) с ДМСО (8% v/v) в качестве криопротекторного средства.
1. Реактивация замороженной культуры E. coli ACM5
- Из замороженного бактериального бульона откройте трубку и используйте стерильную петлю, кончик пипетки или зубочистку, чтобы соскоблить поверхность замороженной бактериальной культуры.
- Нанесите бактерии на агаровую пластину Luria-Bertani (LB) и инкубируйте при 37 ± 2 °C в течение 24 ч.
2. Определение антибактериальной восприимчивости
ПРИМЕЧАНИЕ: Описанное здесь тестирование чувствительности к противомикробным препаратам соответствует методу диффузии диска, основанному на руководящих принципах Института клинических и лабораторных стандартов (CLSI) (M02 Ed13: 2018)31. Кишечная палочка Штамм ATCC 25922 необходим для контроля качества.
- Приготовление инокулята методом колониальной суспензии
- Из пластины LB, инкубированной на стадии 1.2, проведите одну или две колонии на агаровую пластину Мюллера-Хинтона (MH) и инкубируйте при 37 ± 2 °C в течение 18-24 ч.
- Используйте стерильную петлю, чтобы выбрать две или три колонии из свежевыращенного изолята E. coli на агаровой пластине MH и повторно суспендировать их в 3 мл стерильного бульона MH или 0,85% (мас./об.) физиологического раствора, тщательно перемешивая путем вихря для получения однородной бактериальной суспензии.
- Используя УФ-видимый спектрофотометр (см. Таблицу материалов), отрегулируйте бактериальную суспензию до мутности, сопоставимой со стандартом 0,5 Макфарланда (что эквивалентно примерно 1-2 х 108 клеток / мл). Если бактериальная суспензия слишком легкая или слишком тяжелая, добавьте больше колоний E. coli или бульона MH в зависимости от обстоятельств. Используйте приготовленный инокулятор в течение 15 мин после приготовления.
- Инокуляция тестовых агаровых пластин
- Окуните стерильный ватный тампон в скорректированную бактериальную суспензию. Поверните тампон несколько раз и плотно прижмите его к внутренней стенке трубки, чтобы удалить лишнюю жидкость из тампона.
- Инокулируйте агаровую пластину MH, распределив тампон 3x по всей поверхности агара, каждый раз поворачивая пластину на 60°. Смажьте также ободок агара, чтобы обеспечить равномерное распределение инокулята.
- Оставьте крышку чашки Петри открытой на 3-5 минут, чтобы влага испарилась.
- Нанесение антимикробного диска на инокулированные агаровые пластины
- Равномерно распределите и прижмите каждый антимикробный диск (см. Таблицу материалов) к поверхности привитых агаровых пластин, чтобы обеспечить полный контакт. Инвертировать пластины в течение 15 мин после нанесения на диск и инкубировать при 35 ± 2 °C в течение 16-18 ч.
ПРИМЕЧАНИЕ: Максимум 12 и шесть дисков должны использоваться в чашках Петри диаметром 150 мм и 100 мм соответственно.
- Равномерно распределите и прижмите каждый антимикробный диск (см. Таблицу материалов) к поверхности привитых агаровых пластин, чтобы обеспечить полный контакт. Инвертировать пластины в течение 15 мин после нанесения на диск и инкубировать при 35 ± 2 °C в течение 16-18 ч.
- После инкубации измерьте зону размеров ингибирования с помощью суппорта Вернье и интерпретируйте полученные диаметры в соответствии с критериями точки останова CLSI (M100 - таблица 2A)32.
3. Экстракция и количественная оценка геномной ДНК (гДНК)
- Геномная экстракция ДНК
- Повторно суспендировать петлю свежевыращенных колоний кишечной палочки в 5 мл бульона LB и инкубировать в течение ночи в встряхивающем инкубаторе (180 об/мин) при 37 ± 2 °C.
- Центрифугируйте бактериальную суспензию при 3 500 х г в течение 5 мин и осторожно выбросьте супернатант.
- Экстрагируйте гДНК в соответствии с рекомендациями по набору для экстракции ДНК (см. Таблицу материалов).
- Проверьте чистоту гДНК, измерив оптическую плотность при 260/280 нм (соотношение: >1,8) и 260/230 нм (отношение: 2,0-2,2) с помощью УФ-видимого спектрофотометра. Выполните электрофорез 0,8% агарозного геля для проверки целостности гДНК.
- Количественная оценка геномной ДНК
ПРИМЕЧАНИЕ: Используйте только тонкостенные прозрачные ПЦР-трубки объемом 0,5 мл, одобренные производителем флуоресцентного набора для анализа (см. Таблицу материалов).- Установите необходимое количество пробирок для образцов и стандартов анализа. Подготовьте рабочий раствор для анализа, смешивая компонент А и компонент В, поставляемые в комплекте, следуя рекомендациям производителя.
- Подготовьте стандарты анализа, добавив 190 мкл рабочего раствора и 10 мкл каждого стандарта в соответствующую трубку (требуется два стандарта).
- Добавьте 198 мкл рабочего раствора и 2 мкл образца ДНК в соответствующую пробирку. Энергично перемешайте, вихря все трубки в течение 5 с. Будьте осторожны, чтобы не создавать пузырьки.
- Инкубировать все трубки в течение 2 мин при комнатной температуре, защищенной от света. Измерьте флуоресценцию всех трубок на основе рекомендаций производителя с помощью флуорометра (см. Таблицу материалов).
- Правильно отрегулируйте концентрацию образца гДНК для продолжения секвенирования.
4. Подготовка библиотеки ДНК
ПРИМЕЧАНИЕ: Подготовка и секвенирование библиотеки ДНК осуществлялись в соответствии с руководящими принципами и протоколами производителя (см. Таблицу материалов). Начальная концентрация гДНК составляет 4,0 нг.
- Маркировка, ПЦР-амплификация и индексация
- В пробирку объемом 0,2 мл добавьте 2,5 мкл буфера тегментации и 2 мкл входной гДНК (2,0 нг/мкл). Аккуратно перемешайте путем пипетки.
- Добавьте 1 мкл буфера амплификации и аккуратно перемешайте путем пипетирования. Открутите при 280 х г в течение 1 мин при комнатной температуре.
- Поместите образцы в термоциклер (см. Таблицу материалов) и запустите следующую программу ПЦР: 55 °C в течение 5 мин, затем подержите при 10 °C. Когда образцы достигнут 10 °C, немедленно приступайте к нейтрализации реакции.
- Добавьте 1 мкл нейтрализующего буфера в пробирки для образцов и аккуратно перемешайте путем пипетки. Открутите при 280 х г в течение 1 мин и инкубируйте в течение 5 мин при комнатной температуре.
- Добавьте 1,7 мкл каждого индексного адаптера (т.е. индексов i7 и i5) и 3 мкл индексирующей основной смеси ПЦР. Аккуратно перемешайте путем пипетки. Открутите при 280 х г в течение 1 мин при комнатной температуре.
- Поместите образцы в термоциклер и выполните вторую реакцию ПЦР следующим образом: 72 °C в течение 3 мин, 95 °C в течение 30 с, 18 циклов 95 °C в течение 10 с, 55 °C в течение 30 с, 72 °C в течение 30 с, 72 °C в течение 5 мин и держите при 10 °C.
- Усиленная очистка библиотеки
- Смешивание путем вихря коммерческого раствора магнитных шариков (см. Таблицу материалов) на основе рекомендаций производителя.
- Перенесите маркированный/индексированный образец гДНК в новую пробирку объемом 1,5 мл и добавьте 0,6 мкл магнитных шариков на каждый мкл конечного объема образца гДНК (≈13 мкл). Аккуратно перемешайте путем пипетки и инкубируйте в течение 5 мин при комнатной температуре.
- Поместите пробоины на магнитную стойку (см. Таблицу материалов) на 2 мин до очистки супернатанта. Аккуратно удалите и выбросьте супернатант, не потревожив бусины.
- Добавьте 200 мкл свежеприготовленного 80% этанола без смешивания. Насиживайте в течение 30 с, пока элюат не очистится, аккуратно удалите и выбросьте супернатант, не нарушая бусины.
- Выполните второй шаг стирки. Добавьте 200 мкл 80% этанола в шарики и инкубируйте в течение 30 с. После того, как элюат очистится, удалите и выбросьте супернатант и высушите бусины на воздухе в течение 10 минут.
- Добавьте к шарикам 15 мкл 10 мМ трис-буфера (рН 8) и аккуратно перемешайте путем пипетки. Инкубировать в течение 2 мин при комнатной температуре. Снова поместите пробирки для образцов на магнитную стойку на 2 мин, чтобы супернатант очистился.
- Осторожно перенесите 14 мкл супернатанта из пробирки в новую пробирку объемом 0,2 мл. Это очищенная библиотека.
ПРИМЕЧАНИЕ: На этом этапе очищенная библиотека может храниться при -20 °C в течение 7 дней.
- Нормализация библиотек
- Приступайте к количественной оценке очищенных библиотек с помощью флуоресцентного анализа, как описано на этапе 3.2, за исключением этапа 3.2.5.
- Визуализируйте очищенные библиотеки на электрофорезе 1% агарозного геля для определения средних размеров фрагментов.
- Вычислите значение молярности библиотеки, используя следующее уравнение:
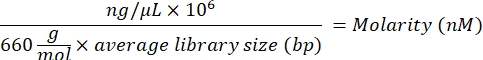
- Используя значение молярности, рассчитайте соответствующие объемы буфера повторного суспензии (RSB) и очищенных библиотек для разбавления отдельной библиотеки при начальной концентрации 10 нМ.
5. Объединение библиотек, денатурализация и инициализация секвенсора
- Разморозьте картридж с реагентами в соответствии с указаниями производителя. Извлеките проточную ячейку из холодильника (4 °C) и доведите ее до комнатной температуры перед секвенированием.
- Пул 5 мкл каждой нормализованной библиотеки (10 нМ) в трубку с низким связыванием 1,5 мл. Разбавьте объединенную библиотеку на 4 нМ с соответствующим объемом RSB.
- Добавьте 5 мкл объединенной библиотеки (4 нМ) и 5 мкл 0,2 Н NaOH в новую трубку с низким связыванием 1,5 мл. Кратковременно перемешайте путем вихря, открутите при 280 х г в течение 1 мин и инкубируйте в течение 5 мин при комнатной температуре, чтобы денатурировать объединенную библиотеку на отдельные нити.
- Аккуратно смешайте 10 мкл денатурированной объединенной библиотеки и 990 мкл предварительно охлажденного гибридизационного буфера путем пипетирования и поместите на лед до тех пор, пока не будет выполнено окончательное разбавление для загрузки секвенсора. Концентрация денатурированной объединенной библиотеки составляет 20 пМ.
- Разморозить и подготовить контрольную библиотеку (см. Таблицу материалов) в концентрации 4 нМ путем смешивания 2 мкл контрольной библиотеки (10 нМ) и 3 мкл безнуклеазной воды.
- Смешайте 5 мкл контрольной библиотеки (4 нМ) и 5 мкл свежеприготовленного 0,2 Н NaOH. Кратковременно перемешайте путем вихря, открутите при 280 х г в течение 1 мин и инкубируйте в течение 5 мин при комнатной температуре, чтобы денатурировать контрольную библиотеку на отдельные нити.
- Аккуратно смешайте 10 мкл денатурированной контрольной библиотеки и 990 мкл предварительно охлажденного буфера гибридизации путем пипетирования и поместите на лед до окончательного разбавления для загрузки секвенсора. Концентрация денатурированной контрольной библиотеки составляет 20 пМ.
- В новой трубке с низким уровнем связывания 1,5 мл объедините 594 мкл денатурированной объединенной библиотеки при 20 пМ и 6 мкл денатурированной контрольной библиотеки при 20 пМ. Правильно перемешать.
- Разбавляют конечную библиотечную смесь (20 пМ) до конечной нагрузочной концентрации 1,2 пМ в объеме 600 мкл с помощью предварительно охлажденного буфера гибридизации.
- Перед загрузкой окончательной библиотечной смеси на картридж с реагентами выполните дополнительную термическую предварительную обработку для достижения эффективной загрузки в проточную ячейку. Инкубируйте окончательную библиотечную смесь в течение 2 мин при 96 °C. Переверните трубку, чтобы перемешать, и поместите трубку на лед на 5 минут.
- Загрузите 500 мкл конечной библиотечной смеси в проектируемый резервуар на реагентном картридже. Инициируйте виртуализацию в соответствии с рекомендациями. Загрузите проточную ячейку и картридж реагента и настройте цикл последовательности.
6. Анализ данных последовательности
ПРИМЕЧАНИЕ: Проверьте Дополнительный файл 1 для дальнейшего описания общей предварительной обработки данных WGS, программного обеспечения, настроек параметров и анализа последовательностей генома E. coli .
- Подключитесь к серверу данных виртуализации и загрузите файлы FASTQ.
- Оцените исходное качество необработанных данных последовательностей с помощью стороннего программного обеспечения. Удалите остаточные последовательности адаптеров, низкокачественные базы (Дополнительный файл 1).
- Соберите данные секвенирования, проверенные на качество, на смежный уровень или на уровне шаблона с помощью стороннего программного обеспечения (см. Дополнительный файл 1).
- Выполните аннотацию генома, отправив файл FASTA, содержащий собранный геном, на сервер RAST (https://rast.nmpdr.org/).
- Загрузите файл FASTA в веб-платформы Центра геномной эпидемиологии (CGE) (http://www.genomicepidemiology.org/services/) и ClermonTyping (http://clermontyping.iame-research.center/index.php) для идентификации эпидемиологических особенностей, ARG, VAG и плазмид (см. Дополнительный файл 1).
- Загрузите файл FASTA на сервер PHASTER для идентификации последовательностей профагов (https://phaster.ca/).
Результаты
Антимикробную восприимчивость определяли методом дисковой диффузии и интерпретировали по критериям точки разрыва CLSI для 12 антибиотиков, охватывающих шесть различных классов противомикробных препаратов, то есть аминогликозиды, β-лактамы, фторхинолоны, нитрофураны, фениколы и антаго?...
Обсуждение
В этом исследовании представлена адаптация рабочего процесса бактериальной WGS с использованием настольного секвенсора и конвейера для геномной характеристики патогенного варианта E. coli . В зависимости от используемой платформы секвенирования время выполнения (TATs) для влажных ла?...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Национальному совету по науке и технике Мексики (CONACyT по его аббревиатуре на испанском языке) за докторскую стипендию, присужденную Хосе Антонио Маганья-Лисарраге [No 481143].
Материалы
| Name | Company | Catalog Number | Comments |
| Accublock Mini digital dry bath | Labnet | D0100 | Dry bath for incubation of tubes |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Magnetic beads in solution for DNA library purification |
| DeNovix DS-11 | DeNovix Inc. | UV-Vis spectophotometer to check the quality of the gDNA extracted | |
| DNA LoBind Tubes | Eppendorf | 0030108418 | 1.5 mL PCR tubes for DNA library pooling |
| DynaMag-2 Magnet | Invitrogen, Thermo Fisher Scientific | 12321D | Magnetic microtube rack used during magnetic beads-based DNA purification |
| Gram-negative Multibac I.D. | Diagnostic reseach (Mexico) | PT-35 | Commercial standard antibiotic disks for antimicrobial susceptibility testing |
| MiniSeq Mid Output Kit (300-cycles) | Illumina | FC-420-1004 | Reagent cartdrige for paired-end sequencing (2x150) |
| MiniSeq System Instrument | Illumina | SY-420-1001 | Benchtop sequencer used for Next-generation sequencing |
| MiniSpin centrifuge | Eppendorf | 5452000816 | Standard centrifuge for tubes |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1024 | Reagents to perform DNA libraries for sequencing. Includes Box 1 and Box 2 reagents for 24 samples |
| Nextera XT Index Kit v2 | Illumina | FC-131-2001, FC-131-2002, FC-131-2003, FC-131-2004 | Index set A, B, C, D |
| PhiX Control v3 | Illumina | FC-110-3001 | DNA library control for sequencing |
| Precision waterbath | LabCare America | 51221081 | Water bath shaker used for bacterial culture |
| Qubit 1X dsDNA HS Assay Kit | Invitrogen, Thermo Fisher Scientific | Q33231 | Reagents for fluorescence-based DNA quantification assay |
| Qubit 2.0 Fluorometer | Invitrogen, Thermo Fisher Scientific | Q32866 | Fluorometer used for fluorescence assay |
| Qubit Assay tubes | Invitrogen, Thermo Fisher Scientific | Q32856 | 0.5 mL PCR tubes for fluorescence-based DNA quantification assay |
| SimpliAmp Thermal Cycler | Applied Biosystems, Thermo Fisher Scientific | A24811 | Thermocycler used for DNA library amplification |
| Spectronic GENESYS 10 Vis | Thermo | 335900 | Spectophotometer used for bacterial suspension in antimicrobial susceptibility testing |
| ZymoBIOMICS DNA Miniprep Kit | Zymo Research Inc. | D4300 | Kit for genomic DNA extraction (50 preps) |
Ссылки
- Naylor, R. L., et al. A 20-year retrospective review of global aquaculture. Nature. 591 (7851), 551-563 (2021).
- Quesada, S. P., Paschoal, J. A. R., Reyes, F. G. R. Considerations on the aquaculture development and on the use of veterinary drugs: special issue for fluoroquinolones-a review. Journal of Food Science. 78 (9), 1321-1333 (2013).
- Defoirdt, T., Sorgeloos, P., Bossier, P. Alternatives to antibiotics for the control of bacterial disease in aquaculture. Current Opinion in Microbiology. 14 (3), 251-258 (2011).
- Stentiford, G. D., et al. New paradigms to help solve the global aquaculture disease crisis. PLOS Pathogens. 13 (2), 1006160 (2017).
- Chen, H., et al. Tissue distribution, bioaccumulation characteristics and health risk of antibiotics in cultured fish from a typical aquaculture area. Journal of Hazardous Materials. 343, 140-148 (2018).
- Zhou, M., et al. Antibiotics control in aquaculture requires more than antibiotic-free feeds: A Tilapia farming case. Environmental Pollution. 268, 115854 (2021).
- Feng, Y., et al. Ecological effects of antibiotics on aquaculture ecosystems based on microbial community in sediments. Ocean & Coastal Management. 224, 106173 (2022).
- Shen, X., Jin, G., Zhao, Y., Shao, X. Prevalence and distribution analysis of antibiotic resistance genes in a large-scale aquaculture environment. Science of The Total Environment. 711, 134626 (2020).
- Su, H., et al. Contamination of antibiotic resistance genes (ARGs) in a typical marine aquaculture farm: source tracking of ARGs in reared aquatic organisms. Journal of Environmental Science and Health, Part B. 55 (3), 220-229 (2020).
- Oliveira, R. V., Oliveira, M. C., Pelli, A. Disease infection by Enterobacteriaceae family in fishes: a review. Journal of Microbiology & Experimentation. 4 (5), 00128 (2017).
- Barbosa, M. M. C., et al. Sorologia e suscetibilidade antimicrobiana em isolados de Escherichia coli de pesque-pagues. Arquivos do Instituto Biológico. 81 (1), 43-48 (2014).
- Valenzuela-Armenta, J. A., et al. Microbiological analysis of Tilapia and water in aquaculture farms from Sinaloa. Biotecnia. 20 (1), 20-26 (2018).
- Reza, R. H., Shipa, S. A., Naser, M. N., Miah, M. F. Surveillance of Escherichia coli in a fish farm of Sylhet, Bangladesh. Bangladesh Journal of Zoology. 48 (2), 335-346 (2021).
- Liao, C. -. Y., et al. Antimicrobial resistance of Escherichia coli From aquaculture farms and their environment in Zhanjiang, China. Frontiers in Veterinary Science. 8, 806653 (2021).
- Dewi, R. R., et al. Prevalence and antimicrobial resistance of Escherichia coli, Salmonella and Vibrio derived from farm-raised Red Hybrid Tilapia (Oreochromis spp.) and Asian Sea Bass (Lates calcarifer, Bloch 1970) on the west coast of Peninsular Malaysia. Antibiotics. 11 (2), 136 (2022).
- Leimbach, A., Hacker, J., Dobrindt, U. E. coli as an all-rounder: the thin line between commensalism and pathogenicity. Current Topics in Microbiology and Immunology. 358, 3-32 (2013).
- Kaper, J. B., Nataro, J. P., Mobley, H. L. T. Pathogenic Escherichia coli. Nature Reviews Microbiology. 2 (2), 123-140 (2004).
- Croxen, M. A., Finlay, B. B. Molecular mechanisms of Escherichia coli pathogenicity. Nature Reviews Microbiology. 8 (1), 26-38 (2010).
- Croxen, M. A., et al. Recent advances in understanding enteric pathogenic Escherichia coli. Clinical Microbiology Reviews. 26 (4), 822-880 (2013).
- Bertelli, C., Greub, G. Rapid bacterial genome sequencing: methods and applications in clinical microbiology. Clinical Microbiology and Infection. 19 (9), 803-813 (2013).
- Lynch, T., Petkau, A., Knox, N., Graham, M., Van Domselaar, G. A primer on infectious disease bacterial genomics. Clinical Microbiology Reviews. 29 (4), 881-913 (2016).
- Carriço, J. A., Rossi, M., Moran-Gilad, J., Van Domselaar, G., Ramirez, M. A primer on microbial bioinformatics for nonbioinformaticians. Clinical Microbiology and Infection. 24 (4), 342-349 (2018).
- Magaña-Lizárraga, J. A., et al. Draft genome sequence of Escherichia coli M51-3: a multidrug-resistant strain assigned as ST131-H30 recovered from infant diarrheal infection in Mexico. Journal of Global Antimicrobial Resistance. 19, 311-312 (2019).
- Pérez-Vázquez, M., et al. Emergence of NDM-producing Klebsiella pneumoniae and Escherichia coli in Spain: phylogeny, resistome, virulence and plasmids encoding blaNDM-like genes as determined by WGS. Journal of Antimicrobial Chemotherapy. 74 (12), 3489-3496 (2019).
- Massella, E., et al. Snapshot study of whole genome sequences of Escherichia coli from healthy companion animals, livestock, wildlife, humans and food in Italy. Antibiotics. 9 (11), 782 (2020).
- Magaña-Lizárraga, J. A., et al. Genomic profiling of antibiotic-resistant Escherichia coli isolates from surface water of agricultural drainage in north-western Mexico: detection of the international high-risk lineages ST410 and ST617. Microorganisms. 10 (3), 662 (2022).
- Saracino, I. M., et al. Next Generation sequencing for the prediction of the antibiotic resistance in Helicobacter pylori: a literature review. Antibiotics. 10 (4), 437 (2021).
- Ghosh, A., Saha, S. Survey of drug resistance associated gene mutations in Mycobacterium tuberculosis, ESKAPE and other bacterial species. Scientific Reports. 10 (1), 8957 (2020).
- Su, M., Satola, S. W., Read, T. D. Genome-based prediction of bacterial antibiotic resistance. Journal of Clinical Microbiology. 57 (3), 01405-01418 (2019).
- Brzuszkiewicz, E., et al. Genome sequence analyses of two isolates from the recent Escherichia coli outbreak in Germany reveal the emergence of a new pathotype: Entero-Aggregative-Haemorrhagic Escherichia coli (EAHEC). Archives of Microbiology. 193 (12), 883-891 (2011).
- . CLSI Performance Standards for Antimicrobial Disk Susceptibility Tests. 13th ed. CLSI standard M02. Wayne, PA: Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m02/ (2018)
- CLSI Performance Standards for Antimicrobial Susceptibility Testing. 31st ed. CLSI supplement M100. Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m100/ (2021)
- Ewing, B., Green, P. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Research. 8 (3), 186-194 (1998).
- Quainoo, S., et al. Whole-genome sequencing of bacterial pathogens: the future of nosocomial outbreak analysis. Clinical Microbiology Reviews. 30 (4), 1015-1063 (2017).
- Desai, A., et al. Identification of optimum sequencing depth especially for de novo genome assembly of small genomes using next generation sequencing data. PLoS ONE. 8 (4), 60204 (2013).
- Nishino, K., Yamada, J., Hirakawa, H., Hirata, T., Yamaguchi, A. Roles of TolC-dependent multidrug transporters of Escherichia coli in resistance to β-lactams. Antimicrobial Agents and Chemotherapy. 47 (9), 3030-3033 (2003).
- Li, M., et al. The resistance mechanism of Escherichia coli induced by ampicillin in laboratory. Infection and Drug Resistance. 12, 2853-2863 (2019).
- Ménard, L. -. P., Dubreuil, J. D. Enteroaggregative Escherichia coli heat-stable enterotoxin 1 (EAST1): a new toxin with an old twist. Critical Reviews in Microbiology. 28 (1), 43-60 (2002).
- Dubreuil, J. D. EAST1 toxin: An enigmatic molecule associated with sporadic episodes of diarrhea in humans and animals. Journal of Microbiology. 57 (7), 541-549 (2019).
- Goldstein, S., Beka, L., Graf, J., Klassen, J. L. Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing. BMC Genomics. 20 (1), 23 (2019).
- Guerrero, A., Gomez-Gil, B., Lizarraga-Partida, M. L. Genomic stability among O3:K6 V. parahaemolyticus pandemic strains isolated between 1996 to 2012 in American countries. BMC Genomic Data. 22 (1), 38 (2021).
- FAO Applications of Whole Genome Sequencing (WGS) in food safety management. Food and Agriculture Organization of the United Nations Available from: https://www.fao.org/documents/card/es/c/61e44b34-b328-4239-b59c-a9e926e327b4/ (2016)
- Rantsiou, K., et al. Next generation microbiological risk assessment: opportunities of whole genome sequencing (WGS) for foodborne pathogen surveillance, source tracking and risk assessment. International Journal of Food Microbiology. 287, 3-9 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены