Method Article
Étudier l’initiation de la mort cellulaire à l’aide d’un microscope numérique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons ici un protocole d’étude du taux d’initiation de la mort cellulaire programmée par imagerie en continu des feuilles inoculées après l’induction de la mort cellulaire.
Résumé
La résistance conférée par la réponse hypersensible (HR) est une réponse de défense efficace qui peut être déterminée par les gènes de résistance à l’azote . La HR se manifeste par la formation de zones de mort cellulaire sur les feuilles inoculées. Ici, un protocole d’étude du taux d’initiation de la mort cellulaire par imagerie des feuilles inoculées dans le temps entre l’initiation de la mort cellulaire et l’apparition de la mort cellulaire à l’aide d’un microscope numérique est présenté. Le microscope numérique permet un processus d’imagerie continu aux intervalles souhaités, ce qui permet une détermination précise du taux d’initiation de la mort cellulaire jusqu’à quelques minutes exactement, par opposition à des heures dans les méthodes traditionnelles. L’imagerie au microscope numérique est également indépendante de la lumière et peut donc être utilisée de jour comme de nuit sans perturber le rythme circadien de la plante. Différents pathosystèmes entraînant le développement de la mort cellulaire programmée pourraient être étudiés à l’aide de ce protocole avec des modifications mineures. Dans l’ensemble, le protocole permet ainsi une identification simple, précise et peu coûteuse du taux d’initiation de la mort cellulaire.
Introduction
La pomme de terre est l’une des cultures vivrières les plus cultivées au monde, à la quatrième place derrière le riz, le blé et le maïs. Cependant, la production de pommes de terre peut être gravement affectée par le virus Y de la pomme de terre (PVY), qui est actuellement considéré comme son agent pathogène viral le plus important 1,2. Dans les plants de pommes de terre cv. Rywal, plusieurs souches de virus Y (y compris la souche N-Wilga du virus Y) déclenchent une résistance conférée par la réponse hypersensible (HR), où la restriction de l’agent pathogène au site d’infection se manifeste par des lésions nécrotiques sur les feuilles inoculées3. Dans ce pathosystème, la HR est médiée par le gène de résistance Ny-1, qui dépend de la température, car les plantes cultivées à des températures plus basses développent efficacement des lésions nécrotiques, tandis que chez les plantes cultivées de manière constitutive à une température élevée (28 °C), l’avortement de la résistance est démontré par l’absence de formation de lésions et la propagation systémique du virus 3,4. Lorsque les plantes sont transférées à une température plus basse (22 °C), la mort cellulaire est initiée, qui peut être exploitée pour suivre le taux d’initiation de la mort cellulaire en imageant les feuilles inoculées dans le temps entre l’initiation de la mort cellulaire et l’apparition de la mort cellulaire.
Ce protocole démontre une méthode simple pour déterminer le taux d’initiation de la mort cellulaire à l’aide d’un microscope numérique. En imageant les feuilles inoculées après le transfert de la plante de 28 °C à 22 °C, un microscope numérique permet d’observer en continu la feuille aux intervalles souhaités. Contrairement à l’utilisation d’autres méthodes (par exemple, la microscopie confocale ou l’observation de la formation des lésions à l’œil nu), cela permet de déterminer le moment exact de la formation des lésions et, par conséquent, le taux d’initiation de la mort cellulaire jusqu’à quelques minutes exactement, par opposition aux heures dans les méthodes susmentionnées 5,6. L’utilisation du microscope numérique est également indépendante de la lumière et peut donc être utilisée de jour comme de nuit. Ce protocole peut également être utilisé pour identifier les composants impliqués dans l’initiation de la mort cellulaire ou pour déterminer les effets de différents composants sur le taux d’initiation de la mort cellulaire si les plantes utilisées sont transgéniques et ont des niveaux modifiés de composants d’intérêt.
Protocole
NOTA : Les sections 1 et 2 décrivent un protocole modifié pour la préparation du matériel végétal fondé sur les méthodes décrites par Lukan et al.7. Plus précisément, certaines modifications ont été apportées aux conditions environnementales contrôlées et à la préparation de l’inoculum.
1. Cultiver des plants de pommes de terre
- Cultivez des pommes de terre saines. Plantes Rywal en culture tissulaire de noeud de tige8.
- Après 6 à 8 semaines, coupez dix explants de 1 cm de long contenant des nœuds de plants de pommes de terre sur du papier stérile dans des conditions stériles à l’aide d’une pince à épiler stérile et d’un couteau.
- Transférez-les dans des boîtes en plastique avec du milieu Murashige et Skoog (MS30) (Figure 1A) et cultivez-les dans des conditions environnementales contrôlées (22 °C à la lumière et 19 °C à l’obscurité avec un rayonnement de 250 μE ou μmol/m2/s et une photopériode de 16 h, à une humidité relative de 55 % ± 5 %).
- Transférez-les dans le sol 2 semaines après la micropropagation.
- Remplissez les pots (r = 10 cm) de terre. Utilisez un doigt pour créer un trou de 3 à 4 cm de profondeur dans le sol au milieu d’un pot, remplissez le trou d’eau et attendez qu’elle soit absorbée.
- Placez une plante par pot dans le trou, en laissant les feuilles au-dessus de la surface. Recouvrez délicatement les racines de terre (figure 1B).
REMARQUE : Certaines plantes peuvent ne pas développer de racines. Excluez ces plantes de l’analyse.
- Cultivez les plantes en terre dans une chambre de culture dans des conditions environnementales contrôlées (22 °C à la lumière et 19 °C à l’obscurité, à une humidité relative de 55 % ± 5 %, avec un rayonnement de 250 μmol/m2/s et une photopériode de 16 h).
- Après 3-4 semaines, les plantes sont prêtes. Utilisez ces plantes pour vous faire inoculer.
REMARQUE : Les plantes doivent avoir au moins 3 ou 4 feuilles complètement développées avec des folioles visibles (figure 1C). Ils doivent avoir l’air en bonne santé, sans symptômes visibles (feuilles jaunes ou brunes), qui pourraient être confondus avec des lésions causées par le virus Y. Pour obtenir des résultats comparables, les plantes doivent être inoculées en même temps (par exemple, à 9 heures) dans toutes les expériences afin d’éviter les effets possibles du rythme circadien sur la réponse immunitaire des plantes et la formation de lésions 9,10,11.
2. Préparation de l’inoculum et inoculation de la pomme de terre
- Inoculer la plante avec la souche N-WILGA (PVYN-Wi ; no d’accession). EF558545) l’inoculum tel qu’indiqué ci-dessous.
REMARQUE : Plus de plantes peuvent être inoculées en parallèle si plus d’un microscope numérique est disponible. Les plantes doivent avoir au moins trois feuilles complètement développées avant l’inoculation (figure 1C). Le PVYN-Wi est multiplié et maintenu dans le cv de la pomme de terre Pentland.- Préparer le tampon phosphaté, complété par du diéthyldithiocarbamate de sodium (DIECA), pour l’inoculation : Mélanger 1,3 mL de 0,2 M NaH 2 PO4, 8,7 mL de 0,2 M Na2HPO4 et 0,225 g de DIECA. Augmentez le volume à 100 mL en utilisant ddH20. Ajustez le pH à 7,6 en ajoutant 1 M de NaOH ou 1 M de solution HCl.
- Récoltez des pommes de terre infectées par le virusY N-Wi âgées de 6 à 8 semaines. Pentland plantes issues de la culture tissulaire dans des sacs d’extraction avec filet filtrant (0,5 g pour 6 plantes) et ajouter un tampon phosphate complété par DIECA (4x masse de matière végétale, rapport masse/volume). Utilisez un homogénéisateur manuel pendant 1 à 2 min pour obtenir une solution homogène.
- Saupoudrer légèrement les trois premières feuilles du bas complètement développées des plants de pommes de terre âgés de 3 à 4 semaines (à partir de l’étape 1.6) avec de la poudre de carborundum.
REMARQUE : Ajustez la quantité de poudre de carborundum. Une trop grande quantité peut causer des dommages, tandis qu’une trop faible quantité peut entraîner une infection inefficace. Idéalement, on utilise 0,1 mg/cm de poudre de carborundum, soit environ 1,5 mg de poudre par feuille de taille moyenne (environ 15 cm2). - Frottez doucement les feuilles avec l’inoculum (~100 μL par feuille). Après 10 min, lavez soigneusement les feuilles à l’eau du robinet.
- Veillez à ne pas endommager les feuilles. Ne pas dépasser le temps d’incubation. Ajustez la quantité d’inoculum en fonction de la taille des feuilles - 6,5 μL/cm, soit environ 100 μL par feuille de taille moyenne (environ 15 cm2).
- Transférez les plantes dans une chambre de culture et cultivez-les dans des conditions environnementales contrôlées (28 °C à la lumière et 28 °C à l’obscurité à une humidité relative de 55 % ± 5 %, avec un rayonnement de 250 μmol/m2/s et une photopériode de 16 h) pendant 3 jours.
3. Préparation de la plante et utilisation du microscope numérique pour l’enregistrement du développement des lésions
- Transférer les plantes inoculées de la chambre de culture maintenue à 28 °C dans une chambre de culture à 22 °C 3 jours après l’inoculation, avec les autres conditions décrites à l’étape 1.5.
- Sélectionnez une plante à observer. Immobiliser la deuxième feuille inoculée à l’aide de ruban adhésif (Figure 1D). Assurez-vous que la feuille souhaitée est complètement immobilisée avant de commencer l’imagerie (Figure 1D).
- Installez une application logicielle pour la capture d’images sur l’ordinateur portable. Connectez le microscope numérique à l’ordinateur. Ouvrez le logiciel.
REMARQUE : Assurez-vous que l’usine, le microscope numérique et l’ordinateur portable sont positionnés sur l’étagère près de la prise pour brancher l’ordinateur. La décision sur le choix des feuilles à surveiller peut dépendre de la question biologique étudiée, par exemple, le type de processus qui conduit à la formation d’une mort cellulaire programmée limitée. - Ajustez le microscope au-dessus de la feuille immobilisée. Faites la mise au point à l’aide de la molette du microscope numérique (illustrée à la Figure 1E).
- Assurez-vous que la ligne de visée est aussi large que possible pour augmenter les chances de capturer la formation de la lésion, mais qu’elle est suffisamment agrandie pour repérer l’apparence de la lésion (généralement, un grossissement de 25x est utilisé).
- Définissez les paramètres de l’appareil photo. Cliquez sur le bouton Paramètres dans le coin supérieur droit de l’image (Figure 2A, partie 1 encerclée) et ajustez les paramètres de l’appareil photo : Luminosité (60-70), Contraste (10-15), Teinte (0), Balance des blancs, Saturation (45-50), Netteté (0), Gamma (5) (Figure 2B). Les paramètres utilisés dans cette étude étaient les suivants : Luminosité (64), Contraste (14), Teinte (0), Saturation (47), Netteté (0), Gamma (5).
REMARQUE : Les paramètres de l’appareil photo doivent être ajustés à la lumière extérieure pour garantir la meilleure qualité d’image. Les réglages peuvent être adaptés en fonction des conditions de l’utilisateur dans la chambre de culture. - Définissez les paramètres de capture d’image en cliquant sur l’icône de capture d’image (Figure 2C, partie 2 encerclée). Cliquez sur le bouton Time-Lapsed Video (Vidéo accélérée ) et réglez Image Capture (Capture d’image ) toutes les 15 minutes pendant 24 h (Figure 2C).
REMARQUE : L’intervalle entre les images prises et la durée de l’imagerie sont entièrement flexibles et peuvent être adaptés en fonction des besoins de l’expérience. Entre la capture de l’image, la lumière LED s’éteint. La lumière LED s’allume automatiquement pendant la capture d’image. - Lancez la capture d’image en cliquant sur le bouton START (Figure 2C).
REMARQUE : Après avoir été transférés de 28 °C à 22 °C, les plantes devraient commencer à développer des lésions dans les 24 heures. Si ce n’est pas le cas, cela pourrait être le signe d’une inoculation inefficace. Ajustez la quantité de poudre de carborundum, assurez-vous que le moment de l’incubation de l’inoculum sur les feuilles était correct et déterminez l’abondance du virus dans l’inoculum par qPCR. - Pour enregistrer des images, sélectionnez toutes les images et cliquez sur l’icône Enregistrer (Figure 2B, partie 3 encerclée). Définissez les options d’exportation. Réglez la résolution au maximum (300) (Figure 2D). Après l’enregistrement, supprimez toutes les images du programme.
- Pour l’analyse d’images, n’importe quel programme de visualisation/édition d’images est suffisant. L’utilisation d’un logiciel gratuit de retouche d’images, ImageJ, est expliquée ci-dessous.
- Importez la séquence temporelle d’images à partir d’un seul champ de vision (dans le coin supérieur gauche, cliquez sur Fichier, sélectionnez Importer, puis Séquence d’images). Collez le chemin du répertoire des images enregistrées et appuyez sur le bouton OK pour lancer la conversion.
- Après la conversion, le logiciel ouvre automatiquement un lecteur vidéo interne affichant la vidéo finale. Exportez le fichier vidéo en cliquant sur l’option Fichier > Enregistrer sous et en sélectionnant Format AVI. Une petite fenêtre s’ouvre. Réglez la fréquence d’images sur 0,3 ips et appuyez sur OK pour enregistrer la vidéo en tant que fichier vidéo AVI.
REMARQUE : Le moment du développement de la lésion peut également être déterminé en vérifiant manuellement toutes les images et en trouvant une image où la lésion apparaît.
Résultats
Cette étude démontre un protocole étape par étape pour étudier l’initiation de la mort cellulaire par l’apparition de lésions sur le CV de la pomme de terre. Rywal, avec un microscope numérique. Cela permet de déterminer l’heure exacte de l’initiation programmée de la mort cellulaire.
Les plantes qui ont développé des racines ont été mises en terre 2 semaines après la pomme de terre. Micropropagation de Rywal (Figure 1A,B). Après 3 à 4 semaines de croissance dans les conditions décrites, des plantes avec au moins 3 à 4 feuilles complètement développées avec des folioles visibles qui semblaient saines, sans aucun signe d’abscission, ont été utilisées pour une analyse plus approfondie (Figure 1C). À l’aide d’un microscope numérique tel que décrit dans ce protocole, nous avons observé la même zone sur la feuille inoculée à des intervalles de 15 minutes et déterminé l’apparition et l’expansion de la lésion dans le temps (Figure 3). La lésion est survenue à 15 h 30 min (Figure 3).

Figure 1 : Préparation de la plante pour l’analyse au microscope numérique. (A) Une boîte en plastique avec un milieu MS 30 et un cv de pommes de terre. Explants de plantes Rywal contenant des nœuds. (B) Pomme de terre cv. Rywal plante dans le sol (2 semaines après la micropropagation). (C) Pomme de terre cv. Plante Rywal, prête à être inoculée (4 semaines après avoir été mise en terre), ayant au moins trois feuilles complètement développées. (D) Deuxième feuille inoculée (flèche) de la pomme de terre cv. Plante Rywal positionnée et immobilisée (flèche) avec du ruban adhésif. (E) Plante positionnée sous le microscope numérique avec la flèche pointant vers le cadran utilisé pour la mise au point. Veuillez cliquer ici pour voir une version agrandie de cette figure.
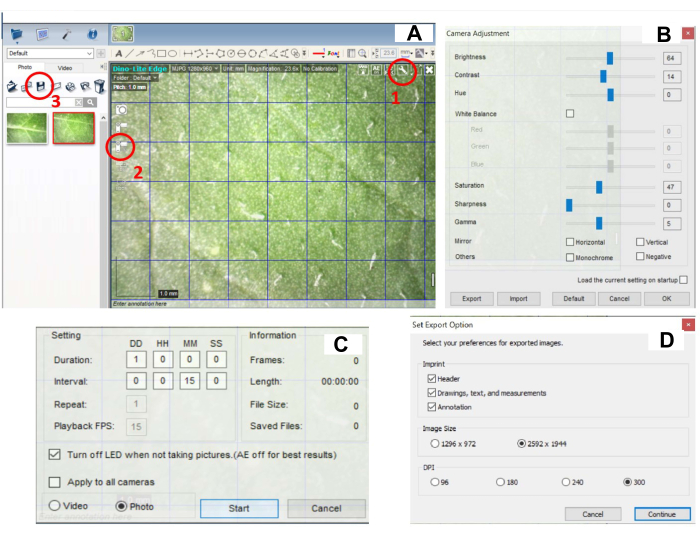
Figure 2 : Réglage du logiciel numérique pour l’enregistrement de l’évolution des lésions. (A) Interface logicielle - entourées de rouge se trouvent les options du bouton pour (1) les paramètres de l’appareil photo, (2) les paramètres de capture d’image et (3) l’enregistrement des images. (B) Fenêtre avec les paramètres de l’appareil photo, qui s’ouvre par un clic sur (1) dans le panneau A. La luminosité, le contraste, la saturation, la netteté et le gamma doivent être correctement réglés. (C) Fenêtre avec les paramètres de capture d’image, qui s’ouvre en cliquant sur (2) indiqué dans le panneau A. (D) Fenêtre avec les paramètres d’enregistrement d’image, qui s’ouvre en cliquant sur (3) indiqué dans le panneau A. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Formation de lésions sur la feuille inoculée observée au microscope numérique. Images de la partie centrale de la feuille de pomme de terre inoculée par le virus Y à un grossissement de 23,6x, vues au microscope numérique, prises à des intervalles de 5 min. Les plantes inoculées ont été placées à 28 °C pendant 3 jours, et le troisième jour, l’observation au microscope numérique à 22 °C a commencé à 7h00. (A) À 21h02, la lésion n’est pas encore visible, (B) 90 min plus tard, à 22h32, la lésion est visible. (C) L’expansion de la lésion a été observée à 01h02 et (D) à 07h32 le lendemain matin. L’expérience a été répétée deux fois, et les lésions sont survenues 8 h 15 min et 12 h après le début de la mort cellulaire, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Le protocole démontré permet à l’utilisateur de déterminer avec précision le taux d’initiation de la mort cellulaire en imageant en continu les feuilles inoculées dans le temps entre l’initiation de la mort cellulaire et l’apparition de la mort cellulaire à l’aide d’un microscope numérique. Même s’il existe de nombreuses façons de surveiller l’apparition des lésions et des maladies des plantes12,13,14,15, ce protocole présente l’avantage d’une mesure indépendante de la lumière sans perturber le rythme circadien de la plante, car la lumière est éteinte entre les mesures.
Après l’inoculation, les plantes doivent pousser à 28 °C pendant 3 jours. Le gène de résistance Ny-1 , qui induit une réponse d’hypersensibilité, est dépendant de la température et, chez les plantes cultivées à des températures plus élevées, conduit à l’avortement de la résistance, qui se manifeste par l’absence de formation de lésions et la propagation systémique du virus3. Après le transfert des plantes à 22 °C, la mort cellulaire est initiée, donc pour des résultats précis, l’observation au microscope numérique doit commencer dès que possible après ce transfert. Une autre étape cruciale dans la préparation de la plante pour l’imagerie est l’immobilisation de la feuille (Figure 1D), car la plante continuera à croître pendant l’imagerie, ce qui pourrait déplacer la feuille observée hors de la mise au point, ou une telle configuration ne donnera pas les résultats souhaités.
Si le protocole décrit est utilisé sur des plantes transgéniques présentant des composants d’intérêt altérés, supposés être impliqués dans l’initiation de la mort cellulaire, le protocole permet à l’utilisateur de déterminer si la diminution du niveau d’un composant étudié affecte le taux d’initiation de la mort cellulaire. Grâce à cela, les composants impliqués dans l’initiation de la mort cellulaire peuvent être identifiés dans les systèmes pathopathiques, où la mort cellulaire programmée se produit, à l’aide de ce protocole. D’autres méthodes d’identification de ces composants sont, par exemple, l’analyse transcriptomique telle que le séquençage de l’ARN ou diverses formes de microscopie, qui peuvent être coûteuses et prendre du temps16. La méthode décrite dans ce protocole permet d’identifier facilement et à moindre coût les composants impliqués dans l’initiation de la mort cellulaire en observant les différences dans les taux d’initiation de la mort cellulaire entre les plantes transgéniques et témoins. Idéalement, dans une telle configuration, deux caméras numériques doivent être utilisées, car une plante transgénique doit être analysée en parallèle avec une usine témoin au sein de la même expérience.
Dans ce protocole, la souche N-Wilga du virus Y a été utilisée ; cependant, d’autres souches de ce virus, par exemple le virus Y marqué par la GFP (PVY-N605(123)-GFP)7, pourraient également être utilisées. De plus, d’autres pathosystèmes, qui entraînent le développement de la mort cellulaire programmée, pourraient être étudiés à l’aide de ce protocole avec des modifications mineures.
Déclarations de divulgation
Les auteurs déclarent n’avoir aucun conflit d’intérêts.
Remerciements
Nous remercions Barbara Jaklič pour son assistance technique. Cette recherche a été financée par l’Agence slovène pour la recherche et l’innovation (financement de base de recherche n° P4-0165 et projet Z4-3217 : Déchiffrer l’interconnexion de signalisation liée à l’oxydoréduction dans la résistance de la pomme de terre aux virus).
matériels
| Name | Company | Catalog Number | Comments |
| Alcohol burner | Mikro+Polo | SH-234002455 | For tweezers and scalpel sterilization |
| Autoclave A-21 CAV | Kambi | N/A | |
| Bacto Agar | Becton, Dickinson and Company | 214010 | |
| Carborundum powder | VWR Chemicals | 22505297 | |
| DinoCapture 2.0 | Dino-Lite | Version 2.0 | software for digital microscope |
| Dino-Lite Edge AM7915MZTL digital microscope | AnMo Electronics Corporation | AM7915MZTL | |
| Ethanol, 70% | Stella Tech | P94000 | For tweezers and scalpel sterilization |
| Extraction bags | Bioreba | 420100 | |
| Growth chamber FS-WI | Photon Systems Insturments | N/A | |
| Hand homogenizer | Bioreba | 400010 | |
| Hawita Special Substrate | HAWITA Gruppe | 2000000071701 | Ready to use substrate, made using peat (H4-H6 and H6-H8) |
| Hydrochloric acid (HCl) | Merck | 109057 | |
| Label tape | Sigma | L8144-5EA | |
| Laptop computer with installed DinoCapture 2.0 | HP | Z2V77EA#BED | Computer needs to be transferable as experiment takes part in a growth chamber |
| Murashige and Skoog medium | Duchefa Biochemie | M02220100 | |
| Na2HPO4 | Emsure | 1065860500 | |
| NaH2PO4 | Emsure | 1064700250 | |
| Pasteur pipette 0.5 mL | Brand | 21500209 | |
| pH-meter | Mettler Toledo | ML1601 | |
| Plastic boxes | Cvetlice Dornig | VCG10.5 | Radius = 10.5 cm |
| Plastic pots | Lab Associates | DIS40003 | Radius = 11.5 cm (top), Radius = 9.8 cm (bottom) |
| Saccharose | Kemika d.d. | 1800408 | |
| Sodium Diethyldithiocarbamate (DIECA) | Sigma-Aldeich | 228680 | Sodium diethyldithiocarbamate trihydrate, ACS reagent |
| Sodium hydroxide (NaOH) | Merck | 106462 | |
| Sterile surgical blades | Braun | 4511733633 | |
| Tweezers | Braun | BD033R |
Références
- Karasev, A. V., Gray, S. M. Continuous and emerging challenges of potato virus y in potato. Annual Review of Phytopathology. 51, 571-586 (2013).
- Quenouille, J., Vassilakos, N., Moury, B. Potato virus Y: A major crop pathogen that has provided major insights into the evolution of viral pathogenicity. Molecular Plant Pathology. 14 (5), 439-452 (2013).
- Szajko, K., et al. The novel gene Ny-1 on potato chromosome IX confers hypersensitive resistance to Potato virus Y and is an alternative to Ry genes in potato breeding for PVY resistance. Theoretical and Applied Genetics. 116 (2), 297-303 (2008).
- Szajko, K., Strzelczyk-Żyta, D., Marczewski, W. Ny-1 and Ny-2 genes conferring hypersensitive response to potato virus Y (PVY) in cultivated potatoes: Mapping and marker-assisted selection validation for PVY resistance in potato breeding. Molecular Breeding. 34 (1), 267-271 (2014).
- Lukan, T., et al. Cell death is not sufficient for the restriction of potato virus Y spread in hypersensitive response-conferred resistance in potato. Frontiers in Plant Science. 9, 168 (2018).
- Baebler, &. #. 3. 5. 2. ;., et al. Salicylic acid is an indispensable component of the Ny-1 resistance-gene-mediated response against Potato virus y infection in potato. Journal of Experimental Botany. 65 (4), 1095-1109 (2014).
- Lukan, T., Coll, A., Baebler, &. #. 3. 5. 2. ;., Gruden, K. Analysis of virus spread around the cell death zone at spatiotemporal resolution using confocal microscopy. Methods in Molecular Biology. 2447, 261-270 (2022).
- Vinterhalter, D., Dragiüeviü, I., Vinterhalter, B. Potato in vitro culture techniques and biotechnology. Fruit, Vegetable and Cereal Science and Biotechnology. 2, 16-45 (2008).
- Wang, W., et al. Timing of plant immune responses by a central circadian regulator). Nature. 470, 110-115 (2011).
- Roden, L. C., Ingle, R. A. Lights, rhythms, infection: The role of light and the circadian clock in determining the outcome of plant-pathogen interactions. Plant Cell. 21 (9), 2546-2552 (2009).
- Srivastava, D., et al. Role of circadian rhythm in plant system: An update from development to stress response. Environmental and Experimental Botany. 162, 256-271 (2019).
- Mulaosmanovic, E., et al. High-throughput method for detection and quantification of lesions on leaf scale based on trypan blue staining and digital image analysis. Plant Methods. 16, 62 (2020).
- Martinelli, F., et al. Advanced methods of plant disease detection. A review. Agronomy for Sustainable Development. 35, 1-25 (2015).
- Ali, M., Bachik, N., Muhadi, N. A., Tuan Yusof, T. N., Gomes, C. Non-destructive techniques of detecting plant diseases: A review. Physiological and Molecular Plant Pathology. 108, 101426 (2019).
- Sankaran, S., Mishra, A., Ehsani, R., Davis, C. A review of advanced techniques for detecting plant diseases. Computers and Electronics in Agriculture. 72 (1), 1-13 (2010).
- Rowarth, N. M., et al. RNA-Seq analysis reveals potential regulators of programmed cell death and leaf remodelling in lace plant (Aponogeton madagascariensis). BMC Plant Biology. 21 (1), 375 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon