Method Article
Différenciation in vitro de lymphocytes T CD4+ naïfs en lymphocytes Th17 pathogènes chez la souris
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit la différenciation des lymphocytes T CD4+ naïfs en lymphocytes Th17 pathogènes in vitro. Plus précisément, lorsqu’il est combiné à une approche basée sur la cytométrie en flux multiparamétrique, une pureté de 90 % de cellules Th17 pathogènes peut être obtenue à partir de cellules T CD4+ naïves en utilisant cette méthode de différenciation.
Résumé
In vitro Les techniques de différenciation des lymphocytes T sont essentielles pour l’étude fonctionnelle et mécaniste des lymphocytes T CD4+ . Les cellules Th17 pathogènes ont été associées à un large éventail de maladies ces derniers temps, notamment la sclérose en plaques (SEP), la polyarthrite rhumatoïde, le syndrome de détresse respiratoire aiguë (SDRA), la septicémie et d’autres troubles auto-immuns. Cependant, les protocoles de différenciation in vitro actuellement connus ont du mal à atteindre une grande pureté des cellules Th17 pathogènes, avec une efficacité d’induction souvent inférieure à 50%, ce qui est un défi clé dans les expériences in vitro . Dans ce protocole, nous proposons un protocole amélioré de culture et de différenciation in vitro pour les cellules Th17 pathogènes, qui est utilisé pour différencier directement les cellules T CD4+ naïves isolées de la rate de souris en cellules Th17 pathogènes. Ce protocole fournit des instructions détaillées sur l’isolement des splénocytes, la purification des lymphocytes T CD4+ naïfs et la différenciation des lymphocytes Th17 pathogènes. Grâce à ce protocole, nous pouvons atteindre une pureté de différenciation d’environ 90% pour les cellules Th17 pathogènes, ce qui répond aux besoins fondamentaux de nombreuses expériences cellulaires.
Introduction
Après avoir quitté le thymus, les lymphocytes T CD4+ naïfs traversent les organes lymphoïdes secondaires. Les cellules présentatrices d’antigènes qui transmettent des antigènes homologues aux lymphocytes T CD4+ naïfs les activent, déclenchant une série de programmes de différenciation qui aboutissent finalement à la production de lignées de cellules T auxiliaires (Th) hautement spécialisées1. La production d’interleukine 17 (IL-17) caractérise les cellules Th17, une sous-populationde cellules Th 2 pro-inflammatoires. Les cellules Th17 jouent un rôle dans la défense de l’hôte contre les agents pathogènes extracellulaires et dans la pathogenèse de nombreuses maladies auto-immunes, telles que l’uvéite auto-immune et la sclérose en plaques. Les signaux des récepteurs et des cytokines des lymphocytes T IL-6 et du facteur de croissance transformant β (TGF-β) induisent la différenciation des lymphocytes T naïfs en lymphocytes Th17 par la phosphorylation du transducteur de signal et activateur de transcription (STAT)33. STAT3 est encore amplifié dans une boucle de rétroaction positive par la signalisation médiée par l’IL-23 et l’IL-21 4,5. La phosphorylation de STAT3 peut induire l’expression des facteurs de transcription RORγt et RORα, qui agissent comme des interrupteurs maîtres régulant le profil cytokinique de l’IL-17A, de l’IL-17F, de l’IL-21 et de l’IL-22 dans les cellules Th176. Cependant, il a été rapporté que les cellules Th17 induites par l’IL-6 et le TGF-β sont insuffisantes pour déclencher des maladies auto-immunes, qui nécessitent une co-stimulation par l’IL-23 ou une co-stimulation distincte de l’IL-6, de l’IL-1β et de l’IL-23 en l’absence de TGF-β 7,8.
Les sous-ensembles Th17 qui ne peuvent pas induire efficacement une encéphalomyélite auto-immune expérimentale (EAE) sont parfois appelés Th17 non pathogènes, tandis que les sous-ensembles Th17 qui peuvent induire l’EAE sont connus sous le nom de Th179 pathogènes. Des études récentes ont montré que, bien que le Th17 pathogène et le Th17 non pathogène co-expriment le facteur de transcription de base RORγt, il existe de grandes différences dans la capacité à produire de l’IL-17A et des propriétés pro-inflammatoires et anti-inflammatoires10. En plus de l’expression élevée de RORγt, CCR6, STAT4 et RUNX4, qui sont des facteurs de transcription caractéristiques communs de Th17, les cellules pathogènes Th17 présentent également des caractéristiques d’expression de signal génique supplémentaires liées à la maladie, telles que TBX21, IFN-γ et CXCR3, qui ont les caractéristiques des sous-ensembles de cellules Th1. Les cellules Th17 pathogènes peuvent sécréter des niveaux élevés de facteur de stimulation des colonies de granulocytes-macrophages (GM-CSF), d’IFN-γ, de TNF-α et d’autres cytokines11,12. Le phénotype des cellules Th17 non pathogènes est instable, et ce n’est que sous la stimulation de CD3 et de la cytokine IL-2 que certaines de ces cellules peuvent se différencier en cellules Th17 pathogènes. Par conséquent, dans les modèles cliniques courants tels que la polyarthrite rhumatoïde, la sclérose en plaques et le syndrome de détresse respiratoire aiguë, les cellules Th17 pathogènes exercent principalement des effets pathogènes.
Les cellules Th17 pathogènes et non pathogènes peuvent se différencier in vitro sous l’influence de différentes cytokines. Ces dernières années, plusieurs études ont proposé des méthodes pour induire la différenciation des cellules Th17 à l’aide de différents types et concentrations de cytokines. Les cellules Th17 sont stimulées par une combinaison d’IL-6, d’IL-1β et d’IL-23 13,14,15,16. Il a été prouvé que l’IL-6 et le TGF-β, deux cytokines nécessaires à la différenciation des cellules Th17, régulent de manière synergique l’expression des cellules RORγt et Th17 en interagissant avec deux séquences d’ADN non codantes conservées différentes au locus17 du gène Rorc. La phase stable des cellules pathogènes Th17 est principalement maintenue par l’IL-23 18,19. L’IL-23 se lie à son récepteur et active la voie de signalisation JAK-STAT20, provoquant ainsi la phosphorylation de Jak2 et Tyk2 et favorisant la phosphorylation de STAT1, STAT3, STAT4 et STAT5. L’IL-4 et l’IFN-γ sont des régulateurs négatifs de cette voie. Cependant, des études ont montré que l’IL-1β peut réguler positivement la transcription de Rorα et Rorγt par la voie mTOR pour maintenir la stabilité du phénotype21 de la cellule Th17.
En raison de l’hétérogénéité de nombreuses études, nous avons choisi les protocoles d’induction pour les cellules Th17 pathogènes et non pathogènes des dernières recherches comme témoins22. Les résultats indiquent que, en supposant que tout se déroule selon ce protocole, après 5 jours de culture dans les conditions de génération de Th17 pathogène, plus de 90% des cellules survivantes peuvent être des cellules Th17 pathogènes.
Protocole
Le Comité d’examen institutionnel des études animales de l’Université du Sud-Est a approuvé toutes les études animales détaillées dans cette étude, qui ont été réalisées conformément aux normes des bureaux locaux et institutionnels. Des échantillons de rate ont été prélevés sur des souris C57BL6/J. Des souris femelles et mâles, âgées de 5 à 8 semaines, ont été incluses dans cette étude. Le milieu de culture et le tampon ont été conservés à 4 °C pendant 1 mois maximum. Les instruments chirurgicaux ont été autoclavés avant d’être utilisés. Portez des gants et des masques en latex pour éviter de contaminer la peau, les yeux et les vêtements avec des réactifs ; Utilisez beaucoup d’eau ou de rince-bouche salin pour la peau et les yeux.
1. Pré-revêtement de la plaque de culture tissulaire à 24 puits avec un anti-CD3
- Diluer l’anti-CD3 à une concentration finale de 1 μg/mL dans une solution saline stérile tamponnée au phosphate (PBS) ou un milieu RPMI 1640.
- Remplissez bien une plaque de culture tissulaire de 24 puits avec 1 mL de solution anti-CD3 (1 μg/mL) ; Ensuite, couvrez le puits avec du parafilm.
- Placez la plaque revêtue d’un anti-CD3 au réfrigérateur (2 °C-8 °C) pendant 16 h ou conservez-la à 37 °C dans un incubateur cellulaire pendant 2-3 h.
2. Isolement de la rate de souris et préparation d’une suspension unicellulaire de la rate
- Induire une anesthésie complète dans le C57BL/6J avec 3 % d’isoflurane et euthanasier en inhalant 100 % de dioxyde de carbone pendant 2 min. Immédiatement après la mort des souris, faites-les tremper dans de l’éthanol à 75 % pour les désinfecter afin d’éviter toute contamination.
- Placez la souris en position couchée sur le banc propre, ouvrez-la le long de la ligne médiane de l’abdomen et séparez les structures abdominales couche par couche. Ouvrez doucement le grand épiploon et l’estomac à l’aide d’une pince à épiler, retirez doucement la rate avec le ligament gastro-splénique et séparez brusquement la rate des tissus et ligaments environnants pour obtenir une rate intacte (veillez à éviter l’écrasement ou la rupture de la rate).
REMARQUE : Pour maintenir un état stérile, l’opération suivante est effectuée dans un banc stérile super propre. - Placez un filtre cellulaire de 70 μm dans une boîte de culture stérile de 100 mm, ajoutez la rate dans la crépine cellulaire et écrasez-la avec un piston de seringue. En même temps, ajoutez 5 à 8 ml de liquide de rinçage homogéné pour pousser toutes les cellules à travers le filtre dans la boîte de culture.
REMARQUE : La composition du kit de liquide de séparation des lymphocytes peut être consultée dans la table des matériaux. - Centrifuger le filtrat à 450 × g pendant 5 min à température ambiante. Jetez le surnageant.
- Remettre en suspension la pastille cellulaire avec le diluant de l’échantillon fourni avec le kit de séparation des lymphocytes ou avec le milieu PBS ou RPMI 1640. Ajuster la concentration cellulaire de la suspension cellulaire à 2 × 108-1 × 109 cellules/mL.
REMARQUE : En règle générale, 4 à 6 ml de diluant de l’échantillon sont nécessaires pour une rate. - Dans un tube à centrifuger, prendre la même quantité de solution de séparation des lymphocytes que la suspension unicellulaire tissulaire. Pipeter soigneusement la suspension unicellulaire sur la surface de la solution de séparation des lymphocytes et centrifuger à 800 × g, 25 °C pendant 30 min. Réglez l’accélération et la décélération de la centrifugeuse sur une plage appropriée (par exemple, réglée sur 3 s’il y a les vitesses habituelles de 1 à 9).
REMARQUE : La solution de séparation des lymphocytes ne doit pas être inférieure à 3 ml. - Après la centrifugation, observez les quatre couches du tube de centrifugation de haut en bas. La première couche correspond au diluant de l’échantillon, suivie de la couche annulaire de lymphocytes blanc laiteux. La troisième couche est le liquide de séparation, et la quatrième couche est constituée de globules rouges.
- À l’aide d’une pipette, aspirez soigneusement la deuxième couche de lymphocytes annulaires blanc laiteux dans un autre tube à centrifuger et ajoutez 10 ml de solution de nettoyage dans le tube à centrifuger pour mélanger les cellules.
- Centrifuger à 250 × g pendant 5 min à température ambiante. Jetez le surnageant. Utilisez 10 ml de milieu RPMI 1640 pour remettre en suspension la pastille de cellule pour le comptage des cellules.
3. Purification de lymphocytes T CD4+ naïfs par sélection négative de billes magnétiques
REMARQUE : Isoler des lymphocytes T CD4+ naïfs intacts et hautement purifiés (CD4+CD44lowCD62Lhigh) à partir de splénocytes de souris par sélection immunomagnétique négative.
- Préparez l’échantillon pour qu’il contienne 1 × 108 cellules/mL dans la plage de volume de 0,1 à 2 mL.
- Ajouter 50 μL/mL de sérum de rat et transférer l’échantillon dans un tube en polystyrène à fond rond de 5 mL.
REMARQUE : La composition du kit de tri des lymphocytes T CD4 naïf peut être consultée dans la table des matériaux. - Ajouter 50 μL/mL de cocktail d’isolement à l’échantillon, bien mélanger et incuber pendant 7,5 minutes à température ambiante.
- Ajouter 50 μL/mL de Depletion Cocktail à l’échantillon, bien mélanger et incuber pendant 2,5 min à température ambiante.
- Vortex les billes magnétiques pour assurer une dispersion uniforme. Ajouter 75 μL/mL de billes magnétiques à l’échantillon, bien mélanger et incuber pendant 2,5 minutes à température ambiante.
REMARQUE : Les particules doivent apparaître uniformément dispersées ; Le temps de vortex ne doit pas être inférieur à 30 s. - Complétez l’échantillon avec du RPMI 1640 medium jusqu’à un volume final de 2,5 ml. Mélangez plusieurs fois en pipetant doucement de haut en bas.
- Placez le tube (sans le couvercle) dans l’aimant et incubez pendant 2,5 min à température ambiante.
- En prenant l’aimant, retournez l’aimant et le tube en un mouvement continu et versez la suspension cellulaire enrichie dans un nouveau tube.
REMARQUE : Gardez le tube et l’aimant inversés pendant 2-3 s avant de les retourner à la verticale. Ne dérangez pas les gouttes qui pourraient rester sur l’embouchure du tube. - Déterminez le nombre de cellules à l’aide d’un hémocytomètre.
- Effectuer une analyse par cytométrie en flux des lymphocytes T CD4+ naïfs avant et après l’isolement (figure 1).
- Transférez les cellules dans un nouveau tube à centrifuger, centrifugez à 400 × g pendant 5 min à température ambiante et jetez le surnageant.
- Remettre les cellules en suspension dans 200 à 500 μL de milieu RPMI 1640 (complété par 10 % de sérum de veau fœtal). Pour chaque 1 mL de culture cellulaire (p. ex., ~106 cellules/mL), ajoutez 2 μL de cocktail d’activation des leucocytes et mélangez-le. Cultivez les cellules dans un incubateur au CO2 à 37 °C et à une humidité saturée pendant 4 à 6 h.
REMARQUE : Les cellules ont été vortex une fois toutes les 1 à 2 h. - Centrifuger les cellules à 400 × g, 4 °C pendant 5 min. Jetez soigneusement le surnageant et remettez les cellules en suspension dans 200 μL de PBS. Ajouter 1 μL d’inhibiteur Fc dans les cellules (par exemple, ~106 cellules/mL). Incuber les cellules pendant 10 min à 4 °C.
- Laver une fois les cellules avec 1 mL de PBS par centrifugation à 400 × g pendant 5 min à 4 °C. Jetez le surnageant et remettez les cellules en suspension dans 200 μL de PBS.
- Ajoutez un cocktail d’anticorps (Fixable Viability Dye, 1:1 000 ; anti-CD4, 1:200 ; anti-CD44, 1:200 ; anti-CD62L, 1:200) et incubez les cellules pendant 15 à 20 minutes dans l’obscurité à 4 °C.
- Laver les cellules avec 2 x 1 mL de PBS, puis centrifuger à 400 × g pendant 5 min à 4 °C.
- Jetez le surnageant, remettez les cellules en suspension dans 250 μL de tampon fixe et incubez-les pendant 40 à 60 min dans l’obscurité à 4 °C.
REMARQUE : Le tampon fixe est fourni avec le tampon de coloration Foxp3/facteur de transcription, qui est une solution mère 4x qui doit être diluée avec un diluant de perméabilisation. - Ajouter 1 mL de 1x tampon de perméabilisation pour laver les cellules 2x en centrifugant les cellules à 300 × g, 4 °C pendant 5 min.
- Jetez le surnageant, remettez les cellules en suspension dans 200 μL de tampon de perméabilisation et ajoutez l’anticorps (Foxp3 [nucléaire], 1:200) aux cellules. Après la fixation et la rupture de la membrane, incuber les mélanges cellules-anticorps dans l’obscurité pendant 40 à 60 minutes, en agitant de temps en temps pendant de courtes périodes à 4 °C.
REMARQUE : La solution de perméabilisation/lavage est une solution mère 10x qui doit être diluée avant d’être utilisée avec PBS.
4. Induction de cellules Th17 pathogènes in vitro
- Retirez le PBS stérile des plaques pré-enduites à 24 puits avant de l’utiliser.
- Utilisez un milieu de culture cellulaire Th17 (tableau 1) et un produit de contraste pour la culture, y compris le milieu de culture cellulaire Th0 (tableau 1), le milieu de culture Th17 non pathogène classique (tableau 1) et le milieu de culture pathogène Th17 classique (tableau 1). Remettre en suspension les lymphocytes T CD4 + T naïfs enrichis et les distribuer dans différents puits dans la même plaque de 24 puits (pré-recouverte d’anti-CD3), en ajustant la concentration à 4 × 105 cellules/mL, avec 1 mL de milieu dans chaque puits.
- Ajouter 1 μL/mL de solution anti-CD28 (concentration finale : diluée avec du milieu à 2 μg/mL) dans chaque puits. Cultivez les cellules dans un incubateur à 5 % de CO2 à 37 °C pendant 5 jours.
- Remplacez le milieu surnageant de culture cellulaire par un milieu de culture (Th0, Th17 non pathogène, Th17 pathogène classique et Th17 objectif) en pipetant soigneusement pour répartir uniformément les cellules, jetez la moitié du milieu contenant les cellules, puis ajoutez un nouveau milieu égal au volume de rejet le jour 2. Répétez l’opération le jour 4.
- Observez les cellules au microscope optique le jour 5 (Figure 2). Collectez les surnageants cellulaires de chaque groupe et cryoconservez-les à -80 °C.
5. Analyse cytométrique en flux pour la différenciation des cellules pathogènes Th17 et Th0
- Récoltez toutes les cellules (par puits) après 5 jours d’initiation de la culture. Effectuez les étapes 3.12 à 3.15.
- Ajoutez un cocktail d’anticorps (Fixable Viability Dye, 1:1 000 ; anti-CD4, 1:200) et incubez les cellules pendant 15 à 20 min dans l’obscurité à 4 °C.
- Effectuez les étapes 3.17 à 3.19.
- Jeter le surnageant, remettre les cellules en suspension dans 200 μL de tampon de perméabilisation et ajouter le cocktail d’anticorps (IL-17A [intracellulaire], 1:200 ; RORγT [nucléaire], 1:200) aux cellules. Après la fixation et la rupture de la membrane, incuber les mélanges cellules-anticorps dans l’obscurité pendant 40 à 60 minutes, en agitant de temps en temps pendant de courtes périodes à 4 °C.
- Ajouter 1 mL de 1x solution de perméabilisation/lavage pour laver les cellules 2x, en centrifugeant les cellules à 400 × g pendant 5 min à 4 °C ; Ensuite, jetez le surnageant.
- Examinez les cellules sur un cytomètre en flux et analysez les données à l’aide du logiciel (Figure 3).
- Allumez le cytomètre en flux et activez le processus de nettoyage et d’étalonnage à démarrage automatique.
- Sélectionnez le canal laser utilisé dans cette expérience et configurez un tube vierge, des tubes monocolores et des tubes d’échantillons.
- Cliquez sur l’option de tube vierge et testez sur la machine ; Ajustez la tension de manière à ce que l’événement de la cellule soit, dans la mesure du possible, au centre de la boîte de collecte.
- Prélever des cellules tubulaires monocolorées et effectuer une détection spectrale des canaux correspondants pour confirmer que l’anticorps fluorescent est correctement ajouté.
- Cliquez sur le bouton Annuler le mixage et laissez la machine ajuster automatiquement la compensation spectrale. Avec d’autres instruments de cytométrie en flux, ajustez normalement la compensation de fluorescence.
- Collectez chaque cellule de tube d’échantillon, enregistrez le format des données sous forme de fichiers FCS et exportez-les. Analysez et tracez dans le logiciel d’analyse de données de cytométrie en flux approprié.
- Encerclez la population cellulaire principale et éliminez les éventuelles adhésions cellulaires à travers les portes circulaires diagonales de FSC-H et FSC-A.
- Encerclez les cellules vivantes en fonction de la zone du cercle négatif FVS ; ensuite, encerclez la population de cellules CD4+.
- Construire la porte transversale avec l’axe horizontal comme IL-17 A et l’axe vertical comme RORγT ; la sélection double positive dans la zone Q2 est la population de cellules Th17.
6. Test immuno-enzymatique (ELISA) pour la détection de la sécrétion d’IL-17A induite par différents milieux
- Décongeler les surnageants cellulaires congelés recueillis à la section 4.5.
- Ajouter 100 μL de l’étalon dilué, du blanc et de l’échantillon dans les puits désignés. Utilisez la soudeuse pour couvrir la plaque et incuber pendant 90 min à 37 °C.
REMARQUE : Ajoutez bien les solutions au fond de la plaque micro-ELISA sans toucher la paroi intérieure ni provoquer de mousse. - Décantez le liquide de chaque puits et ajoutez immédiatement 100 μL de solution de travail d’anticorps de détection biotinylée. Utilisez une nouvelle soudeuse pour couvrir la plaque et incubez pendant 1 h à 37 °C.
- Décantez la solution et ajoutez 350 μL de tampon de lavage dans chaque puits. Après 1 min, aspirez ou décantez la solution de chaque puits et séchez-la contre du papier absorbant propre. Répétez cette étape de lavage 3 fois.
- Ajouter 100 μL de la solution de travail conjuguée de peroxydase de raifort dans chaque puits. Utilisez une nouvelle soudeuse pour couvrir la plaque et incubez pendant 30 min à 37 °C.
- Décantez la solution de chaque puits et répétez le processus de lavage 5 fois comme décrit à l’étape 6.4.
- Ajoutez 90 μL de réactif de substrat dans chaque puits et incubez pendant environ 15 min à 37 °C avec un nouveau scellant, en protégeant la plaque de la lumière.
REMARQUE : Le temps de réaction peut être modifié en fonction du changement de couleur réel, mais ne doit pas dépasser 30 min. Laissez le lecteur de microplaques se réchauffer pendant ~15 min avant la mesure de la DO. - Ajouter 50 μL/puits de solution d’arrêt dans le même ordre que la solution de substrat.
- Déterminez la densité optique (valeur OD) de chaque puits à la fois à l’aide d’un lecteur de microplaques réglé sur 450 nm.
- Obtenez les valeurs de DO pour les échantillons standard et répliquez les puits des échantillons, puis soustrayez les valeurs de DO des puits à blanc pour obtenir les valeurs corrigées. Ensuite, effectuez un ajustement linéaire avec la concentration comme abscisse et la valeur OD comme ordonnée. À l’aide de l’équation ajustée, calculer les valeurs de concentration d’IL-17A pour les puits d’échantillonnage.
REMARQUE : Après 4 jours de différenciation, la teneur en IL-17A dans le surnageant de chaque groupe est illustrée à la figure 4. La méthode d’ajustement peut faire référence au logiciel Orgin21. La courbe standard obtenue à partir de cette expérience a une valeur R2 de 0,9946.
7. Différenciation des lymphocytes T par des tests d’expression génique de signature via PCR quantitative (qPCR)
REMARQUE : Pour exclure la possibilité d’une instabilité de la cytométrie en flux due aux effets des colorants et de la fixation/rupture, nous avons détecté l’expression de gènes caractéristiques par qPCR afin d’élucider l’effet de différenciation du Th17 pathogène au niveau transcriptionnel.
- Mesurez le nombre de cellules à la fin de la culture de lymphocytes T. Prélever les cellules dans des tubes à centrifuger de 1,5 mL et centrifuger à 400 × g pendant 5 min. Jetez soigneusement tous les surnageants.
- Extraction de l’ARN
REMARQUE : Le processus d’extraction doit être effectué dans une hotte ou un banc ultra-propre pour éviter la contamination par les enzymes ARN.- Ajoutez 500 μL de tampon de lyse (par exemple, à 1-2 × 106 cellules) dans le tube d’échantillon (étape 7.1), agitez et mélangez immédiatement jusqu’à ce qu’il n’y ait plus de masse cellulaire, et laissez reposer pendant 1 min.
- Ajouter le mélange dans la colonne d’adsorption placée dans un tube de collecte, centrifuger à 13 400 × g pendant 30 s et jeter le filtrat.
- Ajoutez la quantité spécifiée d’éthanol absolu dans la bouteille de tampon de lavage avant de l’utiliser pour la première fois. Ajouter 500 μL de tampon de lavage dans la colonne d’adsorption, centrifuger à 13 400 × g pendant 30 s et jeter le filtrat.
- Répétez le lavage à l’étape 7.2.3.
- Placez la colonne d’adsorption RA dans le tube de collecte vide et centrifugez à 13 400 × g pendant 2 minutes pour éliminer la solution de rinçage.
REMARQUE : La solution de rinçage doit être retirée pour éviter que l’éthanol résiduel dans la solution de rinçage n’inhibe la réaction en aval. - Retirez la colonne d’adsorption RA et placez-la dans un tube à centrifuger propre et exempt de RNase. Ajoutez 50 μL de H2O sans RNase au milieu de la membrane d’adsorption, laissez reposer 1 min à température ambiante et centrifugez à 9 600 × g pendant 1 min.
- Mesurez la pureté et la concentration de l’ARN.
- Synthétisez le premier brin d’ADNc en suivant les instructions du fabricant du kit.
- Supprimer l’ADN génomique. Prélever 500 ng de l’ARN matrice extrait dans un tube à centrifuger sans RNase ; ajouter un mélange de ddH2O sans RNase et un mélange d’ADNg 4x pour former un mélange. Mélangez délicatement à l’aide d’une pipette et placez dans un bain-marie à 42 °C pendant 2 min.
REMARQUE : La quantité du mélange réactionnel et la quantité ajoutée sont liées à la concentration en ARN ; reportez-vous aux instructions en détail. Le système de réaction de 20 μL a été utilisé dans cette expérience. - Ajoutez directement un système de réaction de transcription inverse 5x dans le tube de réaction de l’étape précédente. Prélever 4 μL du système de réaction de transcription inverse et 16 μL du mélange de l’étape précédente et mélanger délicatement à l’aide d’une pipette.
REMARQUE : Le système de transcription inverse comprend toute la transcriptase inverse requise, qui est un système de transcription inverse directement configuré. - Réglez la réaction de transcription inverse : 37 °C 15 min, 85 °C, 5 s, et enfin refroidissez à 4 °C.
- Supprimer l’ADN génomique. Prélever 500 ng de l’ARN matrice extrait dans un tube à centrifuger sans RNase ; ajouter un mélange de ddH2O sans RNase et un mélange d’ADNg 4x pour former un mélange. Mélangez délicatement à l’aide d’une pipette et placez dans un bain-marie à 42 °C pendant 2 min.
- Configurez la réaction PCR en suivant les instructions du fabricant.
- Ajoutez 10 μL de mélange d’enzymes de réaction 2x PCR dans le tube qPCR, 0,4 μL d’amorce 1, 0,4 μL d’amorce 2, 0,4 μL de 50x de colorant de référence ROX 1, 2 μL d’ADNc matrice et 6,8 μL de ddH2O pour préparer un mélange de 20 μL.
- Effectuez une PCR dans l’instrument de PCR quantitative à fluorescence en temps réel en utilisant les paramètres suivants : stade 1, 95 °C, 30 s, Rep x 1 ; stade 2, d’abord 95 °C, 10 s, puis 60 °C, 30 s, Rep x 40 ; stade 3, d’abord 95 °C, 15 s, suivant 60 °C, 60 s, enfin 95 °C, 15 s, Rep x 1.
REMARQUE : Après 4 jours de différenciation, les niveaux d’expression relatifs d’Il17a, d’Il17f, de Rora et d’Il23r dans les lymphocytes T CD4+ naïfs sont illustrés à la figure 5.
Résultats
Notre protocole a été développé sur la base de recherches antérieures sur la différenciation des cellules Th17 pathogènes. La première étape de l’expérience consiste à détecter la pureté des lymphocytes T CD4+ naïfs isolés de la rate par tri magnétique des billes, qui est à la base du succès de notre différenciation ultérieure des lymphocytes Th17 pathogènes. La pureté des lymphocytes T CD4+ naïfs a été détectée à l’aide des marqueurs de surface CD62L23 et CD4424 , tandis que FOXP325 a été utilisé comme marqueur des cellules Treg. Nous avons constaté que le contenu des cellules Treg était significativement réduit après le tri, et que la pureté des lymphocytes T CD4+ naïfs pouvait atteindre au moins 95 % (Figure 1). Pour comparer la différenciation des cellules Th17 pathogènes, des lymphocytes T CD4+ naïfs ont été cultivés avec un milieu Th0 (tableau 1) et un milieu de différenciation Th17 (tableau 1) pendant un total de 5 jours. Il a été constaté que les lymphocytes T présentaient une croissance en grappes dans les milieux Th0 et Th17 (Figure 2).
Ensuite, les cellules ont été fixées, perméabilisées et marquées avec des anticorps contre plusieurs cytokines dans des lymphocytes T CD4+ différenciés basés sur la cytométrie en flux. Nous avons examiné IL17A26 et RORγT27 en tant que cytokines caractéristiques de la différenciation des cellules Th17 et avons constaté que 90 % des lymphocytes T CD4+ naïfs se sont différenciés avec succès en cellules Th17 pathogènes sous la stimulation d’un nouveau milieu de culture cellulaire Th17 (Figure 3). La figure 5 montre les gènes de signature des cellules Th17 détectées par PCR, ce qui a prouvé que les cellules pathogènes Th17 que nous avons obtenues par différenciation s’exprimaient de manière stable.
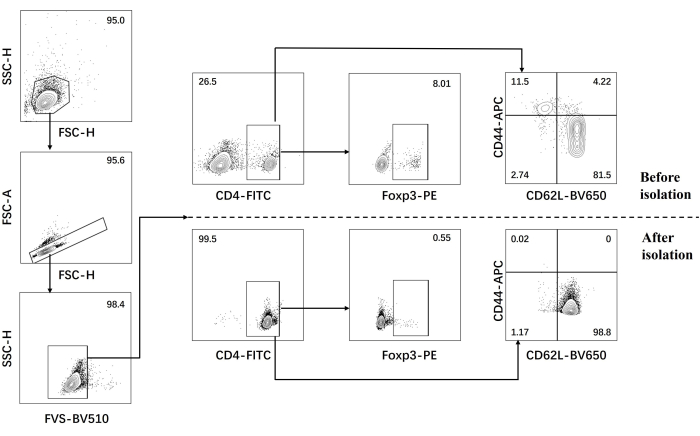
Figure 1 : Stratégie de contrôle pour l’analyse des cytokines de signature chez la souris C57BL/6J avant et après l’isolement naïf des lymphocytes T CD4+. Abréviations : FSC-H = hauteur de crête de diffusion vers l’avant ; SSC-H = hauteur de dispersion latérale ; FSC-A = aire de crête de diffusion vers l’avant ; FITC = isothiocyanate de fluorescéine ; PE = phycoérythrine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
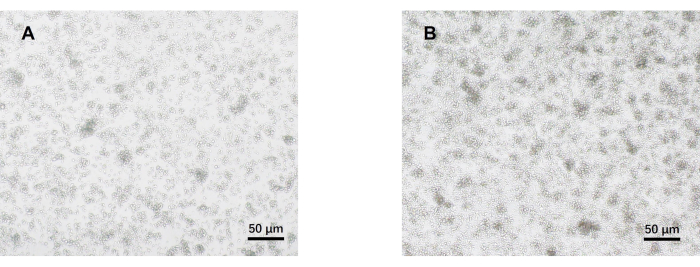
Figure 2 : Images représentatives de lymphocytes T CD4+ n’ayant jamais été cultivés dans des conditions pathogènes Th17 et Th0 pendant 5 jours. (A) cellules Th0 ; (B) cellules Th17 pathogènes. Barres d’échelle = 50 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
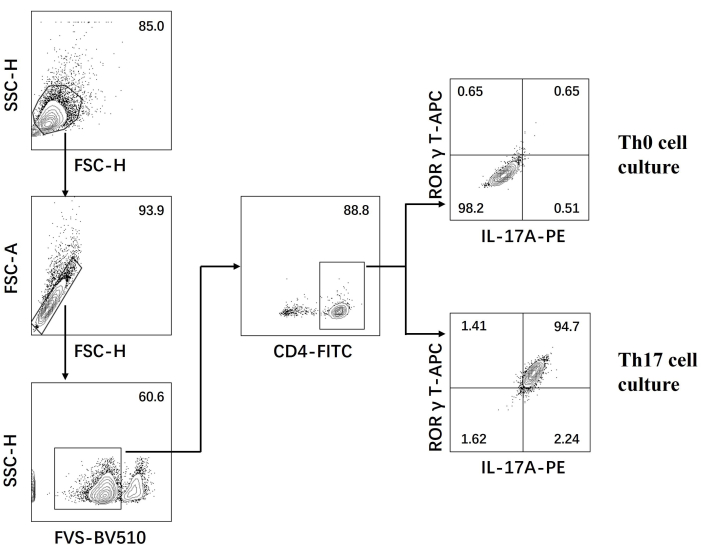
Figure 3 : Analyse par cytométrie en flux après différenciation induite par le milieu de culture cellulaire Th0 et le milieu de culture cellulaire Th17. Abréviations : FSC-H = hauteur de crête de diffusion vers l’avant ; SSC-H = hauteur de dispersion latérale ; FSC-A = aire de crête de diffusion vers l’avant ; FITC = isothiocyanate de fluorescéine ; PE = phycoérythrine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
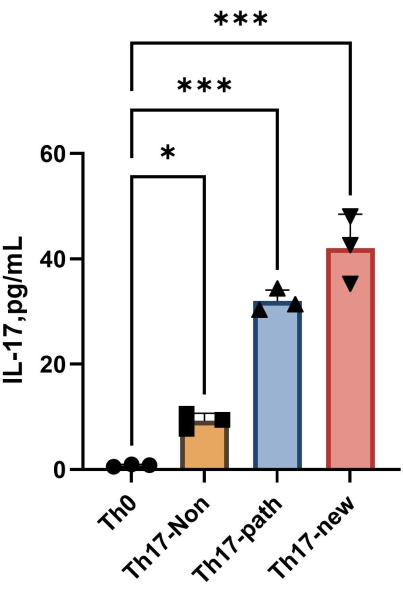
Figure 4 : La teneur en IL-17A dans le surnageant du milieu de culture cellulaire Th0 et du milieu de culture cellulaire Th17 après induction de la différenciation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
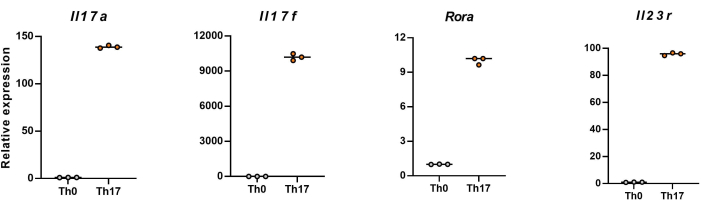
Figure 5 : Résultats représentatifs des niveaux d’expression des cytokines de signature dans les lymphocytes T CD4+ différenciés de souris C57BL/6J. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Milieu de culture de cellules Th17 pathogène cible | Milieu de culture cellulaire Th0 | Milieu de culture cellulaire Th17 classique non pathogène | Milieu de culture cellulaire pathogène classique Th17. | |||||||||||
| Réactif | Concentration finale | Quantité | Concentration finale | Quantité | Concentration finale | Quantité | Concentration finale | Quantité | ||||||
| Pénicilline-streptomycine (100x) | 1x | 500 μL | 1x | 500 μL | 1x | 500 μL | 1x | 500 μL | ||||||
| Sérum fœtal bovin | 10% | 5 mL | 10% | 5 mL | 10% | 5 mL | 10% | 5 mL | ||||||
| β-mercaptoéthanol | 50 μM | 50 μL | 50 μM | 50 μL | 50 μM | 50 μL | 50 μM | 50 μL | ||||||
| Supplément GlutaMAXMC (100x) | 1x | 500 μL | 1x | 500 μL | 1x | 500 μL | 1x | 500 μL | ||||||
| Solution de pyruvate de sodium (100x) | 1 million d’euros | 500 μL | 1 million d’euros | 500 μL | 1 million d’euros | 500 μL | 1 million d’euros | 500 μL | ||||||
| IFN gamma anti-souris (1 mg/mL) | 5 μg/mL | 250 μL | 10 μg/mL | 500 μL | 10 μg/mL | 500 μL | ||||||||
| IL-4 anti-souris (2 mg/mL) | 5 μg/mL | 125 μL | 5 μg/mL | 250 μL | 10 μg/mL | 250 μL | ||||||||
| Souris rIL-1 bêta (20 μg) | 20 ng/mL | NA | ||||||||||||
| Souris rIL-6 (20 μg) | 20 ng/mL | NA | 50 ng/mL | NA | 50 ng/mL | NA | ||||||||
| Souris rIL-23 (50 μg) | 50 ng/mL | NA | 10 ng/mL | NA | ||||||||||
| TGF bêta de souris (100 μg) | 3 ng/mL | NA | 1 ng/mL | NA | 1 ng/mL | NA | ||||||||
| IL-2 murine (5 μg) | 20 ng/mL | NA | ||||||||||||
| RPMI 1640 | NA | Jusqu’à 50 mL | NA | Jusqu’à 50 mL | NA | Jusqu’à 50 mL | ||||||||
| Total | NA | 50 ml | NA | 50 ml | NA | 50 ml | ||||||||
Tableau 1 : Culture cellulaire pathogène cible Th17, culture cellulaire Th0, milieu de culture cellulaire Th17 non pathogène classique et pathogène.
Discussion
Cette procédure a offert un moyen productif d’augmenter le nombre de lymphocytes T CD4+ naïfs spléniques de souris pour la production in vitro de cellules Th17 pathogènes. Bien que nous utilisions plus de cytokines que les autres milieux de culture de cellules Th17 signalés, nous nous engageons à optimiser les conditions de croissance des cellules Th17 pathogènes. Nous envisageons d’optimiser davantage notre protocole de différenciation induite.
Ici, nous avons simplement utilisé la cytométrie en flux et la qPCR pour examiner la production de cytokines caractéristiques. Avec quelques modifications mineures, cette approche peut également être utilisée pour d’autres tests de fonction, tels que la prolifération cellulaire.
Nous avons utilisé un kit d’isolement de lymphocytes produit en Chine pour isoler les lymphocytes de la rate de souris, car il est efficace et permet de gagner du temps. Les solutions de séparation des lymphocytes basées sur d’autres marques peuvent également atteindre l’objectif de séparer les lymphocytes de la rate de souris à travers différentes étapes. Une autre méthode consiste à lyser directement les globules rouges de la suspension de cellules de rate obtenue ; Cependant, nous avons constaté que les globules rouges de la rate ne peuvent souvent pas être lysés en une seule fois.
Certains problèmes peuvent survenir lors de l’exécution de ce protocole. Premièrement, le nombre de lymphocytes T CD4+ naïfs obtenus par tri par billes magnétiques peut être très faible (étape 3 du protocole). Cela pourrait être attribué au fait que le processus d’écrasement des organes est insuffisant. Il est important de s’assurer que l’organe est correctement homogénéisé. L’augmentation de la fréquence des rinçages pendant le processus d’homogénéisation améliorera le taux de récupération. Pour obtenir un plus grand nombre de lymphocytes T CD4+ naïfs spléniques, nous suggérons d’utiliser des souris plus jeunes (âgées de 6 à 10 semaines). Il existe différentes méthodes pour séparer les lymphocytes de la rate, et le rendement peut varier en fonction du liquide de séparation utilisé. Il est recommandé d’utiliser un liquide de séparation universellement certifié et d’essayer d’extraire la couche lymphocytaire autant que possible.
Deuxièmement, la proportion de lymphocytes T CD4+ en cytométrie en flux peut être de <80 % (étape 5 du protocole). L’une des causes possibles de ce problème pourrait être un nombre de splénocytes imprécis, ce qui entraînerait un nombre de cellules supérieur à celui du cocktail d’anticorps supplémentaire et des billes magnétiques. Pour augmenter l’efficacité de la purification des lymphocytes T CD4+ naïfs, la numération cellulaire doit être précise. De plus, 10 % plus de cocktail d’anticorps et de billes magnétiques peuvent être utilisés par rapport à ce que ce protocole recommande. Enfin, la cytométrie en flux peut être effectuée immédiatement après le tri des lymphocytes T CD4+ naïfs.
Troisièmement, il se peut qu’il n’y ait pas beaucoup d’amas de lymphocytes T formés pendant la culture, et que la plupart des cellules soient mortes pendant la différenciation des lymphocytes T (étape 4 du protocole). La raison possible de ce problème pourrait être la détermination inexacte du nombre de cellules T CD4+ naïves avant l’ensemencement, ce qui entraîne une faible densité cellulaire. Il est recommandé d’adopter une méthode de comptage plus précise pour obtenir la densité cellulaire souhaitée d’environ 4 × 105 cellules/mL pour chaque puits dans une plaque de 48 puits. Une autre cause possible pourrait être des problèmes techniques avec l’incubateur de CO2 , tels qu’une température ou une concentration de CO2 incorrecte. Enfin, une force excessive lors du changement de milieu de culture cellulaire pourrait potentiellement provoquer la mort cellulaire.
Quatrièmement, les niveaux d’expression relatifs des gènes de signature des cytokines peuvent être faibles (étape 7 du protocole). Pour garantir l’authenticité de l’ARN extrait, il est recommandé d’utiliser une concentration de détection de nanogouttelettes supérieure à 100 ng/mL. La raison potentielle de la diminution de la concentration peut être la nature malsaine des cellules cultivées, par exemple qu’une grande partie des cellules collectées sont mortes ou en cours de mort. Pour obtenir la véritable concentration d’ARN, il est nécessaire de résoudre la situation qui peut conduire à une mauvaise croissance pendant la culture cellulaire. Une autre raison de cette préoccupation pourrait être le nombre de cellules finales excessivement faible obtenu lors de l’extraction de l’ARN, peut-être en raison d’une perte de cellules par inadvertance lors de l’élimination du surnageant. L’utilisation de techniques d’extraction d’ARN avancées, telles que les kits d’extraction d’ARN en une étape, pourrait s’avérer avantageuse. Le rapport idéal OD260/OD280, tel que mesuré par Nanodrop, doit se situer dans la plage de 1,9 à 2,1. En cas de rapport excessivement faible, la contamination par les protéines devient une possibilité. L’augmentation de la fréquence du lavage des tampons sans RNase peut aider à atténuer ce problème. À l’inverse, un rapport inhabituellement faible implique une dégradation potentielle de l’ARN. Pour contrer ce problème, il est recommandé d’utiliser de l’eau sans RNase et d’utiliser des tubes de 1,5 mL à des fins de décontamination de la RNase.
En conclusion, le protocole actuel décrit l’utilisation d’un nouveau milieu de culture cellulaire pour induire directement les lymphocytes T CD4+ naïfs à se différencier en cellules Th17 pathogènes in vitro. Par rapport à la séparation directe, il ne fait aucun doute que cette méthode est plus directe, peu coûteuse et plus efficace. La configuration du milieu est également très simple, de sorte que les cellules Th17 construites peuvent être utilisées de manière plus intuitive pour les expériences ultérieures, fournissant un très bon modèle cellulaire pour l’étude de nombreuses maladies.
Déclarations de divulgation
Les auteurs déclarent qu’il n’y a pas de conflit d’intérêts concernant la publication de cet article.
Remerciements
Les travaux ont été soutenus par le Programme national de R&D clé de la Chine (n° 2022YFC2304604), la Fondation nationale des sciences naturelles de Chine (n° 81971812), la Fondation nationale des sciences naturelles de Chine (n° 82272235), la Fondation des sciences de la Commission de la santé de la province du Jiangsu (n° 8272235), la Fondation scientifique de la Commission de la santé de la province du Jiangsu (n°. ZDB2020009), Programme clé de recherche, de développement de la province du Jiangsu (développement social) Projet spécial (n° BE2021734), Programme national clé de R & D du ministère des Sciences et de la Technologie (n° 2020YFC083700), Laboratoire clé de médecine de soins intensifs de la province du Jiangsu (BM2020004), Projet clé de la Fondation nationale des sciences naturelles de Chine (81930058), Fondation nationale des sciences naturelles de Chine (82171717), Fonds de recherche fondamentale des universités centrales (2242022K4007), et le projet général de la Fondation provinciale des sciences naturelles du Jiangsu (BK20211170).
matériels
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| Rat anti-mouse CD62L, BV650, clone MEL-14, 1:200 dilution | BD | Cat# 564108; RRID: AB_2738597 | |
| Rat monoclonal anti-CD4, FITC, clone RM4-5, 1:200 dilution | BioLegend | Cat#100509; RRID: AB_312712 | |
| Rat monoclonal anti-IL-17A, PE, clone TC11-18H10.1, 1:200 dilution | BioLegend | Cat#506903; RRID: AB_ 315463 | |
| Rat monoclonal anti mouse/human CD44, APC, clone IM&, 1:200 dilution | BioLegend | Cat#103012; RRID: AB_312963 | |
| Rat monoclonal anti-RORγT, APC, clone B2D, 1:200 dilution | Invitrogen | Cat#17-6981-80; RRID: AB_2573253 | |
| Rat monoclonal FOXP3 antibody, PE, clone FJK-16s, 1:200 dilution | Invitrogen | Cat#12-5773-82; RRID: AB_465936 | |
| Chemicals, peptides, and recombinant proteins | |||
| Anti-Mouse CD3 SAFIRE purified | biogems | Cat#05112-25 | |
| Anti-Mouse CD28 SAFIRE purified | biogems | Cat#10312-25 | |
| Anti-Mouse IFN gamma | biogems | Cat#80822-25 | |
| Anti-Mouse IL-4 | biogems | Cat#81112-25 | |
| Ethanol | Xilong scientific | Cat#64-17-5 | |
| Fetal bovine serum | Gibco | Cat#10437-028 | |
| FcR Blocking reagent | Miltenyi Biotec | Cat#130-092-575 | |
| GlutaMAX supplement | gibco | Cat#35050079 | |
| Mouse rIL-1 beta | Sino Biological | Cat#50101-MNAE | |
| Mouse rIL-6 | Sino Biological | Cat#50136-MNAE | |
| Mouse rIL-23 | Sino Biological | Cat#CT028-M08H | |
| Mouse TGF beta 1 | Sino Biological | Cat#50698-M08H | |
| PBS | Procell | Cat#PB180327 | |
| Recombinant Murine IL-2 | peprotech | Cat#212-12 | |
| RPMI 1640 with L-glutamine | Gibco | Cat#11875-119 | |
| Penicillin-streptomycin solution | Gibco | Cat#15070063 | |
| β-mercaptoethanol | Sigma-Aldrich | Cat#M6250-100ML | |
| Critical commercial assays | |||
| Animal Organ Lymphocyte Separation Solution Kit | Tbdscience | Cat#TBD0018SOP | Contains animal spleen tissue lymphocyte separation liquid, tissue sample diluent (cat no. 2010C1119), sample cleaning solution (cat no. 2010X1118), sample washing solution (cat no. TBTDM-W), tissue homogenate flushing liquid (F2013TBD) |
| ChamQ SYBR qPCR Master Mix (High ROX Premixed) | Vazyme | Cat#Q341-02 | https://www.vazymebiotech.com/product-center/chamq-sybr-qpcr-master-mix-high-rox-premixed-q341.html. |
| Fixation/permeabilization Concentrate | invitrogen | Cat#00-5123-43 | |
| Fixation / Permeabilization Diluent | invitrogen | Cat#00-5223-56 | |
| Fixable Viability Dye EF506 | invitrogen | Cat#65-0866 | |
| HiScript II Q RT SuperMix for qPCR (+gDNA wiper) | Vazyme | Cat#R223-01 | https://www.vazymebiotech.com/product-center/hiscript-ii-q-rt-supermix-for-qpcr-gdna-wiper-r223.html. |
| Leukocyte Activation Cocktail | BD | Cat#550583 | |
| Mouse IL-17A (Interleukin 17A) ELISA Kit | Elabscience® | Cat#E-EL-M0047 | |
| Naïve CD4+ T cells isolation kit, mouse | STEMCELL | Cat#19765 | EasySep kit contains mouse CD4+ T cell isolation cocktail [cat no. 19852C.1], mouse memory T cell depletion cocktail [cat no. 18766C], streptavidin RapidSphered 50001 [cat no. 50001], normal rat serum [cat no. 13551]); only store rat serum at -20 °C; other components to be stored at 2-8 °C. |
| Permeabilization Buffer | invitrogen | Cat#00-8333-56 | |
| SPARKeasy Cell RNA Kit | Sparkjade | Cat#AC0205-B | https://www.sparkjade.com/product/detail?id=85 |
| Experimental models: Organisms/strains | |||
| Mouse: C57BL/6 | Gempharmatech | Cat#000013 | |
| Oligonucleotides | |||
| Mouse Il17a TaqMan primers with probe | ribobio | NA | |
| Mouse Il17f TaqMan primers with probe | ribobio | NA | |
| Mouse Il23r TaqMan primers with probe | ribobio | NA | |
| Mouse Rora TaqMan primers with probe | ribobio | NA | |
| β-actin TaqMan primers with probe | ribobio | NA | |
| Software and algorithms | |||
| Cytek Aurora | Cytek | https://spectrum.cytekbio.com/ | |
| FlowJo v10.8.1 | Tree Star | https://www.flowjo.com | |
| GraphPad prism 9 | GraphPad Software | https://www.graphpad-prism.cn | |
| Other | |||
| 1 mL syringe | Kindly | NA | |
| 1.5 mL Centrifuge tubes | Eppendorf | Cat#MCT-150-C | |
| 5 mL Round-bottom tubes | Corning | Cat#352235 | |
| 15 mL Centrifuge tubes | NEST | Cat#601052 | |
| 48-Well tissue culture plate (flatten bottom) | Corning | Cat#3548 | |
| 50 mL Centrifuge tubes | NEST | Cat#602052 | |
| 70 µm Cell strainer | Biosharp | Cat#BS-70-XBS | |
| 96-well Unskirted qPCR Plates | VIOX scientific | Cat#V4801-M | |
| 100 mm Petri dish | Corning | Cat#430167 | |
| Centrifuge | Eppendorf | 5425R | |
| Cell culture CO2 incubator | Thermo Fisher | HEPA Class100 | |
| Cytek Aurora | Cytek | NA | |
| dissecting scissors | RWD | S12003-09 | |
| Hemocytometer | AlphaCell | Cat#J633201 | |
| NanoDrop 2000 Spectrophotometer | Thermo Fisher | ND-2000 | |
| Real-time PCR System | Roche | LightCycler96 | |
| Surgical tweezers | RWD | F12005-10 | |
| Thermal cycler | Bio-Rad | C1000 Touch |
Références
- Korn, T., Bettelli, E., Oukka, M., Kuchroo, V. K. IL-17 and Th17 cells. Annu Rev Immunol. 27, 485-517 (2009).
- Bhaumik, S., Basu, R. Cellular and molecular dynamics of Th17 differentiation and its developmental plasticity in the intestinal immune response. Front Immunol. 8, 254 (2017).
- Bettelli, E., Korn, T., Oukka, M., Kuchroo, V. K. Induction and effector functions of T(H)17 cells. Nature. 453 (7198), 1051-1057 (2008).
- Korn, T., et al. IL-21 initiates an alternative pathway to induce proinflammatory T(H)17 cells. Nature. 448 (7152), 484-487 (2007).
- Bettelli, E., et al. Reciprocal developmental pathways for the generation of pathogenic effector TH17 and regulatory T cells. Nature. 441 (7090), 235-238 (2006).
- Yang, X. O., et al. T helper 17 lineage differentiation is programmed by orphan nuclear receptors ROR alpha and ROR gamma. Immunity. 28 (1), 29-39 (2008).
- Ghoreschi, K., et al. Generation of pathogenic T(H)17 cells in the absence of TGF-β signalling. Nature. 467 (7318), 967-971 (2010).
- Lee, Y., et al. Induction and molecular signature of pathogenic TH17 cells. Nat Immunol. 13 (10), 991-999 (2012).
- McGeachy, M. J., et al. The interleukin 23 receptor is essential for the terminal differentiation of interleukin 17-producing effector T helper cells in vivo. Nat Immunol. 10 (3), 314-324 (2009).
- Wu, B., et al. The TGF-β superfamily cytokine Activin-A is induced during autoimmune neuroinflammation and drives pathogenic Th17 cell differentiation. Immunity. 54 (2), 308-323 (2021).
- Ramesh, R., et al. Pro-inflammatory human Th17 cells selectively express P-glycoprotein and are refractory to glucocorticoids. J Exp Med. 211 (1), 89-104 (2014).
- Basdeo, S. A., et al. Ex-Th17 (nonclassical Th1) cells are functionally distinct from classical Th1 and Th17 cells and are not constrained by regulatory T cells. J Immunol. 198 (6), 2249-2259 (2017).
- Cua, D. J., et al. Interleukin-23 rather than interleukin-12 is the critical cytokine for autoimmune inflammation of the brain. Nature. 421 (6924), 744-748 (2003).
- Awasthi, A., et al. Cutting edge: IL-23 receptor gfp reporter mice reveal distinct populations of IL-17-producing cells. J Immunol. 182 (10), 5904-5908 (2009).
- Jäger, A., Dardalhon, V., Sobel, R. A., Bettelli, E., Kuchroo, V. K. Th1, Th17, and Th9 effector cells induce experimental autoimmune encephalomyelitis with different pathological phenotypes. J Immunol. 183 (11), 7169-7177 (2009).
- Lee, Y., Collins, M., Kuchroo, V. K. Unexpected targets and triggers of autoimmunity. J Clin Immunol. 34, S56-S60 (2014).
- Chang, D., et al. The conserved non-coding aequences CNS6 and CNS9 control cytokine-induced Rorc transcription during T helper 17 cell differentiation. Immunity. 53 (3), 614-626 (2020).
- Bunte, K., Beikler, T. Th17 cells and the IL-23/IL-17 axis in the pathogenesis of periodontitis and immune-mediated inflammatory diseases. Int J Mol Sci. 20 (14), 3394 (2019).
- Zhao, Y., Liu, Z., Qin, L., Wang, T., Bai, O. Insights into the mechanisms of Th17 differentiation and the Yin-Yang of Th17 cells in human diseases. Mol Immunol. 134, 109-117 (2021).
- Berghmans, N., et al. Interferon-γ orchestrates the number and function of Th17 cells in experimental autoimmune encephalomyelitis. J Interferon Cytokine Res. 31 (7), 575-587 (2011).
- Wu, B., Wan, Y. Molecular control of pathogenic Th17 cells in autoimmune diseases. Int Immunopharmacol. 80, 106187 (2020).
- Du, L., et al. Growth hormone releasing hormone signaling promotes Th17 cell differentiation and autoimmune inflammation. Nat Commun. 14 (1), 3298 (2023).
- Ernst, D. N., Weigle, W. O., Noonan, D. J., McQuitty, D. N., Hobbs, M. V. The age-associated increase in IFN-gamma synthesis by mouse CD8+ T cells correlates with shifts in the frequencies of cell subsets defined by membrane CD44, CD45RB, 3G11, and MEL-14 expression. J Immunol. 151 (2), 575-587 (1993).
- Radtke, A. J., et al. IBEX: an iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- El-Hindi, K., et al. T-cell-specific CerS4 depletion prolonged inflammation and enhanced tumor burden in the AOM/DSS-induced CAC model. Int J Mol Sci. 23 (3), 1866 (2022).
- Cooney, L. A., Towery, K., Endres, J., Fox, D. A. Sensitivity and resistance to regulation by IL-4 during Th17 maturation. J Immunol. 187 (9), 4440-4450 (2011).
- Zhu, X., et al. A novel interleukin-2-based fusion molecule, HCW9302, differentially promotes regulatory T cell expansion to treat atherosclerosis in mice. Front Immunol. 14, 1114802 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon