Method Article
Differenziazione in vitro di cellule T CD4+ naive in cellule Th17 patogene nel topo
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive la differenziazione di cellule T CD4+ naive in cellule Th17 patogene in vitro. In particolare, se combinato con un approccio basato sulla citometria a flusso multiparametrica, è possibile ottenere una purezza del 90% delle cellule Th17 patogene da cellule T CD4+ naive utilizzando questo metodo di differenziazione.
Abstract
In vitro Le tecniche di differenziazione delle cellule T sono essenziali per le indagini sia funzionali che meccanicistiche delle cellule T CD4+ . Le cellule Th17 patogene sono state collegate a un'ampia gamma di malattie negli ultimi tempi, tra cui la sclerosi multipla (SM), l'artrite reumatoide, la sindrome da distress respiratorio acuto (ARDS), la sepsi e altri disturbi autoimmuni. Tuttavia, i protocolli di differenziazione in vitro attualmente noti hanno difficoltà a raggiungere un'elevata purezza delle cellule Th17 patogene, con un'efficienza di induzione spesso inferiore al 50%, che rappresenta una sfida chiave negli esperimenti in vitro . In questo protocollo, proponiamo un protocollo avanzato di coltura e differenziazione in vitro per cellule Th17 patogene, che viene utilizzato per differenziare direttamente le cellule T CD4+ naive isolate dalla milza di topo in cellule Th17 patogene. Questo protocollo fornisce istruzioni dettagliate sull'isolamento degli splenociti, la purificazione delle cellule T CD4+ naive e la differenziazione delle cellule Th17 patogene. Attraverso questo protocollo, possiamo raggiungere una purezza di differenziazione di circa il 90% per le cellule Th17 patogene, che soddisfa le esigenze di base di molti esperimenti cellulari.
Introduzione
Dopo aver lasciato il timo, i linfociti T CD4+ naive passano attraverso gli organi linfoidi secondari. Le cellule presentanti l'antigene che trasmettono antigeni omologhi alle cellule T CD4+ naive li attivano, avviando una serie di programmi di differenziazione che alla fine portano alla produzione di linee cellulari T helper (Th) altamente specializzate1. La produzione di interleuchina 17 (IL-17) caratterizza le cellule Th17, una sottopopolazione di cellule Th pro-infiammatorie2. Le cellule Th17 svolgono un ruolo nella difesa dell'ospite contro i patogeni extracellulari e nella patogenesi di molte malattie autoimmuni, come l'uveite autoimmune e la sclerosi multipla. I segnali provenienti dai recettori delle cellule T e dalle citochine IL-6 e dal fattore di crescita trasformante β (TGF-β) inducono la differenziazione delle cellule T naive in cellule Th17 attraverso la fosforilazione del trasduttore di segnale e dell'attivatore della trascrizione (STAT)33. STAT3 è ulteriormente amplificato in un ciclo di feedback positivo attraverso la segnalazione mediata da IL-23 e IL-21 4,5. La fosforilazione di STAT3 può indurre l'espressione dei fattori di trascrizione RORγt e RORα, che agiscono come interruttori master che regolano il profilo delle citochine di IL-17A, IL-17F, IL-21 e IL-22 nelle cellule Th176. Tuttavia, è stato riportato che le cellule Th17 indotte da IL-6 e TGF-β sono insufficienti per scatenare malattie autoimmuni, che richiedono la co-stimolazione da parte di IL-23 o una co-stimolazione separata di IL-6, IL-1β e IL-23 in assenza di TGF-β 7,8.
I sottogruppi Th17 che non sono in grado di indurre efficacemente l'encefalomielite autoimmune sperimentale (EAE) sono talvolta indicati come Th17 non patogeni, mentre i sottogruppi Th17 che possono indurre EAE sono noti come Th17 9 patogeni. Gli studi attuali hanno dimostrato che, sebbene il Th17 patogeno e il Th17 non patogeno esprimano il fattore di trascrizione principale RORγt, ci sono grandi differenze nella capacità di produrre IL-17A e nelle proprietà pro-infiammatorie e antinfiammatorie10. Oltre all'elevata espressione di RORγt, CCR6, STAT4 e RUNX4, che sono fattori di trascrizione caratteristici comuni di Th17, le cellule Th17 patogene mostrano anche ulteriori caratteristiche di espressione del segnale genico correlate alla malattia, come TBX21, IFN-γ e CXCR3, che hanno le caratteristiche dei sottogruppi di cellule Th1. Le cellule Th17 patogene possono secernere alti livelli di fattore stimolante le colonie di granulociti-macrofagi (GM-CSF), IFN-γ, TNF-α e altre citochine11,12. Il fenotipo delle cellule Th17 non patogene è instabile e solo sotto la stimolazione di CD3 e citochina IL-2 alcune di queste cellule possono differenziarsi in cellule Th17 patogene. Pertanto, nei modelli clinici di malattia comuni come l'artrite reumatoide, la sclerosi multipla e la sindrome da distress respiratorio acuto, le cellule Th17 patogene esercitano principalmente effetti patogeni.
Le cellule Th17 patogene e non patogene possono differenziarsi in vitro sotto l'influenza di diverse citochine. Negli ultimi anni, diversi studi hanno proposto metodi per indurre la differenziazione delle cellule Th17 utilizzando diversi tipi e concentrazioni di citochine. Le cellule Th17 sono stimolate da una combinazione di IL-6, IL-1β e IL-23 13,14,15,16. E' stato dimostrato che IL-6 e TGF-β, due citochine necessarie per il differenziamento delle cellule Th17, regolano sinergicamente l'espressione del differenziamento delle cellule RORγt e Th17 interagendo con due diverse sequenze di DNA conservate non codificanti presso il locus17 del gene Rorc. La fase stabile delle cellule Th17 patogene è mantenuta principalmente da IL-2318,19. IL-23 si lega al suo recettore e attiva la via di segnalazione JAK-STAT20, causando così la fosforilazione di Jak2 e Tyk2 e promuovendo la fosforilazione di STAT1, STAT3, STAT4 e STAT5. IL-4 e IFN-γ sono regolatori negativi di questa via. Tuttavia, gli studi hanno dimostrato che IL-1β può regolare positivamente la trascrizione di Rorα e Rorγt attraverso la via mTOR per mantenere la stabilità del fenotipo21 delle cellule Th17.
A causa dell'eterogeneità di numerosi studi, abbiamo scelto i protocolli di induzione per cellule Th17 patogene e non patogene dalle ultime ricerche come controlli22. I risultati indicano che, supponendo che tutto venga eseguito secondo questo protocollo, dopo 5 giorni di coltura a condizione di generare Th17 patogeno, oltre il 90% delle cellule sopravvissute può essere costituito da cellule Th17 patogene.
Protocollo
Il Comitato di Revisione Istituzionale per gli Studi Animali della Southeast University ha approvato tutti gli studi sugli animali che sono dettagliati in questo studio, che sono stati condotti in conformità con gli standard degli uffici locali e istituzionali. I campioni di milza sono stati prelevati da topi C57BL6/J. In questo studio sono stati inclusi sia topi femmine che maschi, di età compresa tra 5 e 8 settimane. Il terreno di coltura e il tampone sono stati conservati a 4 °C per un massimo di 1 mese. Gli strumenti chirurgici sono stati sterilizzati in autoclave prima dell'uso. Indossare guanti e maschere in lattice per evitare la contaminazione della pelle, degli occhi e degli indumenti con i reagenti; Usa molta acqua o risciacquo salino per la pelle e gli occhi.
1. Prerivestimento della piastra di coltura tissutale a 24 pozzetti con anti-CD3
- Diluire l'anti-CD3 a una concentrazione finale di 1 μg/mL in soluzione salina sterile tamponata con fosfato (PBS) o in terreno RPMI 1640.
- Riempire un pozzetto per piastra di coltura tissutale a 24 pozzetti con 1 mL di soluzione anti-CD3 (1 μg/mL); Quindi, coprire il pozzetto con parafilm.
- Mettere la piastra rivestita anti-CD3 in frigorifero (2 °C-8 °C) per 16 ore o conservarla a 37 °C in un incubatore cellulare per 2-3 ore.
2. Isolamento della milza di topo e preparazione della sospensione unicellulare della milza
- Indurre l'anestesia completa in C57BL/6J con isoflurano al 3% e sopprimere inalando il 100% di anidride carbonica per 2 minuti. Immediatamente dopo la morte dei topi, immergili in etanolo al 75% per la disinfezione per prevenire la contaminazione.
- Posiziona il topo in posizione supina sulla panca pulita, aprilo lungo la linea mediana dell'addome e separa le strutture addominali strato per strato. Aprire delicatamente il grande omento e lo stomaco con una pinzetta, estrarre delicatamente la milza con il legamento gastrosplenico e separare bruscamente la milza dai tessuti e dai legamenti circostanti per ottenere una milza intatta (fare attenzione a evitare lo schiacciamento o la rottura della milza).
NOTA: Per mantenere uno stato sterile, la seguente operazione viene eseguita in un banco sterile super pulito. - Posizionare un filtro cellulare da 70 μm in una piastra di coltura sterile da 100 mm, aggiungere la milza al filtro cellulare e frantumarla con uno stantuffo della siringa. Allo stesso tempo, aggiungere 5-8 mL di liquido di lavaggio omogeneizzato per spingere tutte le cellule attraverso il filtro nella piastra di coltura.
NOTA: La composizione del kit del liquido per la separazione dei linfociti può essere visualizzata nella Tabella dei Materiali. - Centrifugare il filtrato a 450 × g per 5 minuti a temperatura ambiente. Scartare il surnatante.
- Risospendere il pellet cellulare con il diluente campione fornito con il kit di separazione dei linfociti o con il terreno PBS o RPMI 1640. Regolare la concentrazione della sospensione cellulare su 2 × 108-1 × 109 cellule/mL.
NOTA: In genere, sono necessari 4-6 ml di diluente per campioni per una milza. - In una provetta da centrifuga, prelevare la stessa quantità di soluzione di separazione dei linfociti della sospensione tissutale a singola cellula. Pipettare con cautela la sospensione monocellulare sulla superficie della soluzione di separazione dei linfociti e centrifugare a 800 × g, 25 °C per 30 minuti. Impostare l'accelerazione e la decelerazione della centrifuga su un intervallo appropriato (ad esempio, impostato su 3 se ci sono le solite marce da 1 a 9).
NOTA: La soluzione di separazione dei linfociti non deve essere inferiore a 3 ml. - Dopo la centrifugazione, osservare i quattro strati nella provetta da centrifuga dall'alto verso il basso. Il primo strato corrisponde al diluente del campione, seguito dallo strato anulare di linfociti bianchi lattiginosi. Il terzo strato è il liquido di separazione e il quarto strato è costituito da globuli rossi.
- Utilizzare una pipetta per aspirare con cura il secondo strato di linfociti bianchi lattiginosi anulari in un'altra provetta da centrifuga e aggiungere 10 ml di soluzione detergente alla provetta da centrifuga per miscelare le cellule.
- Centrifugare a 250 × g per 5 min a temperatura ambiente. Scartare il surnatante. Utilizzare 10 mL di terreno RPMI 1640 per risospendere il pellet cellulare per il conteggio delle cellule.
3. Purificazione di cellule T CD4+ naive basata sulla selezione negativa di biglie magnetiche
NOTA: Isolare le cellule T CD4+ naive intatte e altamente purificate (CD4+CD44lowCD62Lhigh) da splenociti di topo mediante selezione immunomagnetica negativa.
- Preparare il campione in modo che abbia 1 × 108 cellule/mL nell'intervallo di volume di 0,1-2 mL.
- Aggiungere 50 μL/mL di siero di ratto e trasferire il campione in una provetta di polistirene a fondo tondo da 5 mL.
NOTA: La composizione del kit di smistamento delle cellule T CD4 naive può essere vista nella Tabella dei Materiali. - Aggiungere 50 μL/mL di Isolation Cocktail al campione, mescolare bene e incubare per 7,5 minuti a temperatura ambiente.
- Aggiungere 50 μL/mL di Depletion Cocktail al campione, mescolare bene e incubare per 2,5 minuti a temperatura ambiente.
- Agitare le perline magnetiche per garantire una dispersione uniforme. Aggiungere 75 μL/mL di microsfere magnetiche al campione, mescolare bene e incubare per 2,5 minuti a temperatura ambiente.
NOTA: Le particelle dovrebbero apparire uniformemente disperse; Il tempo di vortice non deve essere inferiore a 30 s. - Rabboccare il campione con il terreno RPMI 1640 fino a un volume finale di 2,5 mL. Mescolare più volte pipettando delicatamente su e giù.
- Posizionare la provetta (senza coperchio) nel magnete e incubare per 2,5 minuti a temperatura ambiente.
- Prelevando il magnete, capovolgere il magnete e il tubo con un movimento continuo e versare la sospensione cellulare arricchita in un nuovo tubo.
NOTA: Tenere il tubo e il magnete capovolti per 2-3 secondi prima di girarli in posizione verticale. Non disturbare eventuali gocce che potrebbero rimanere sulla bocca del tubo. - Determinare il numero di cellule utilizzando un emocitometro.
- Eseguire l'analisi citofluorimetrica per le cellule T CD4+ naive prima e dopo l'isolamento (Figura 1).
- Trasferire le cellule in una nuova provetta da centrifuga, centrifugare a 400 × g per 5 minuti a temperatura ambiente ed eliminare il surnatante.
- Risospendere le cellule in 200-500 μL di terreno RPMI 1640 (integrato con il 10% di siero fetale bovino). Per ogni 1 mL di coltura cellulare (ad esempio, ~106 cellule/mL), aggiungere 2 μL di cocktail di attivazione leucocitaria e mescolare. Coltivare le cellule in un incubatore a CO2 a 37 °C e umidità satura per 4-6 ore.
NOTA: Le celle sono state vortex una volta ogni 1-2 ore. - Centrifugare le celle a 400 × g, 4 °C per 5 min. Scartare con cautela il surnatante e risospendere le cellule in 200 μL di PBS. Aggiungere 1 μL di bloccante Fc alle cellule (ad esempio, ~106 cellule/mL). Incubare le cellule per 10 minuti a 4 °C.
- Lavare le cellule una volta con 1 mL di PBS centrifugando a 400 × g per 5 minuti a 4 °C. Scartare il surnatante e risospendere le cellule in 200 μL di PBS.
- Aggiungere un cocktail di anticorpi (Fixable Viability Dye, 1:1.000; anti-CD4, 1:200; anti-CD44, 1:200; anti-CD62L, 1:200) e incubare le cellule per 15-20 minuti al buio a 4 °C.
- Lavare le celle con 2 x 1 mL di PBS e centrifugare a 400 × g per 5 minuti a 4 °C.
- Scartare il surnatante, risospendere le cellule in 250 μL di tampone fisso e incubarle per 40-60 minuti al buio a 4 °C.
NOTA: Il tampone di fissaggio viene fornito con il tampone di colorazione Foxp3/fattore di trascrizione, che è una soluzione madre 4x che deve essere diluita con diluente di permeabilizzazione. - Aggiungere 1 mL di tampone di permeabilizzazione 1x per lavare le cellule 2x centrifugando le cellule a 300 × g, 4 °C per 5 minuti.
- Scartare il surnatante, risospendere le cellule in 200 μL di tampone di permeabilizzazione e aggiungere l'anticorpo (Foxp3 [nucleare], 1:200) alle cellule. Dopo la fissazione e la rottura della membrana, incubare le miscele cellula-anticorpo al buio per 40-60 minuti agitando di tanto in tanto per brevi periodi a 4 °C.
NOTA: La soluzione di permeabilizzazione/lavaggio è una soluzione madre 10x che deve essere diluita prima dell'uso con PBS.
4. Induzione di cellule Th17 patogene in vitro
- Rimuovere il PBS sterile dalle piastre a 24 pozzetti prerivestite prima dell'uso.
- Utilizzare il terreno di coltura cellulare Th17 (Tabella 1) e il mezzo di contrasto per la coltura, compreso il terreno di coltura cellulare Th0 (Tabella 1), il terreno di coltura Th17 classico non patogeno (Tabella 1) e il terreno di coltura Th17 patogeno classico (Tabella 1). Risospendere le cellule T CD4 + naive arricchite e dispensarle in pozzetti diversi nella stessa piastra da 24 pozzetti (prerivestita con anti-CD3), regolando la concentrazione a 4 × 105 cellule/mL, con 1 mL di terreno in ciascun pozzetto.
- Aggiungere 1 μL/mL di soluzione anti-CD28 (concentrazione finale: diluita con terreno a 2 μg/mL) in ciascun pozzetto. Coltivare le cellule in un incubatore al 5% di CO2 a 37 °C per 5 giorni.
- Sostituire il terreno surnatante di coltura cellulare con un terreno di coltura (Th0, Th17 non patogeno, Th17 patogeno classico e Th17 obiettivo) pipettando attentamente per distribuire uniformemente le cellule, scartare metà del terreno contenente le cellule e quindi aggiungere un nuovo terreno pari al volume di scarto il giorno 2. Ripeti il giorno 4.
- Osservare le cellule al microscopio ottico il giorno 5 (Figura 2). Raccogliere i surnatanti cellulari da ciascun gruppo e crioconservarli a -80 °C.
5. Analisi citofluorimetrica per il differenziamento patogeno di cellule Th17 e Th0
- Raccogliere tutte le cellule (per pozzetto) dopo 5 giorni dall'inizio della coltura. Eseguire i passaggi 3.12-3.15.
- Aggiungere un cocktail di anticorpi (Fixable Viability Dye, 1:1.000; anti-CD4, 1:200) e incubare le cellule per 15-20 minuti al buio a 4 °C.
- Eseguire i passaggi 3.17-3.19.
- Scartare il surnatante, risospendere le cellule in 200 μL di tampone di permeabilizzazione e aggiungere il cocktail di anticorpi (IL-17A [intracellulare], 1:200; RORγT [nucleare], 1:200) alle cellule. Dopo la fissazione e la rottura della membrana, incubare le miscele cellula-anticorpo al buio per 40-60 minuti agitando di tanto in tanto per brevi periodi a 4 °C.
- Aggiungere 1 mL di 1x soluzione di permeabilizzazione/lavaggio per lavare le cellule 2x, centrifugando le cellule a 400 × g per 5 minuti a 4 °C; Quindi, scartare il surnatante.
- Esaminare le cellule su un citometro a flusso e analizzare i dati utilizzando il software (Figura 3).
- Accendere il citometro a flusso e attivare il processo di pulizia e calibrazione ad avvio automatico.
- Seleziona il canale laser utilizzato in questo esperimento e imposta un tubo vuoto, tubi colorati singolarmente e tubi campione.
- Fare clic sull'opzione del tubo grezzo e testare sulla macchina; Regolare la tensione in modo che l'evento cella sia, per quanto possibile, al centro della scatola di raccolta.
- Raccogliere le cellule provette colorate singolarmente ed eseguire il rilevamento spettrale dei canali corrispondenti per confermare che l'anticorpo fluorescente sia stato aggiunto correttamente.
- Fare clic sul pulsante di dismix e lasciare che la macchina regoli automaticamente la compensazione spettrale. Con altri strumenti di citometria a flusso, regolare normalmente la compensazione della fluorescenza.
- Raccogliere ogni cella della provetta del campione, salvare il formato dei dati come file FCS ed esportare. Analizza e traccia nel software di analisi dei dati di citometria a flusso pertinente.
- Circondare la popolazione cellulare principale e rimuovere le possibili aderenze cellulari attraverso i cancelli circolari diagonali di FSC-H e FSC-A.
- Cerchio di cellule vive secondo l'area del cerchio negativo FVS; quindi, cerchia la popolazione cellulare CD4+.
- Costruire il cancello a croce con l'asse orizzontale come IL-17 A e l'asse verticale come RORγT; la doppia selezione positiva nell'area Q2 è la popolazione di cellule Th17.
6. Saggio di immunoassorbimento enzimatico (ELISA) per la rilevazione della secrezione di IL-17A indotta da diversi mezzi
- Scongelare i surnatanti delle cellule congelate raccolti nella Sezione 4.5.
- Aggiungere 100 μl dello standard diluito, del bianco e del campione nei pozzetti designati. Utilizzare il sigillante per coprire la piastra e incubare per 90 minuti a 37 °C.
NOTA: Aggiungere bene le soluzioni sul fondo della piastra micro-ELISA senza toccare la parete interna o causare formazione di schiuma. - Decantare il liquido da ciascun pozzetto e aggiungere immediatamente 100 μl di soluzione di lavoro per anticorpi di rilevamento biotinilati. Utilizzare un nuovo sigillante per coprire la piastra e incubare per 1 ora a 37 °C.
- Decantare la soluzione e aggiungere 350 μl di tampone di lavaggio a ciascun pozzetto. Dopo 1 minuto, aspirare o decantare la soluzione da ogni pozzetto e asciugarla tamponando su carta assorbente pulita. Ripetere questo passaggio di lavaggio 3 volte.
- Aggiungere 100 μl della soluzione di lavoro coniugato perossidasi di rafano in ciascun pozzetto. Utilizzare un nuovo sigillante per coprire la piastra e incubare per 30 minuti a 37 °C.
- Decantare la soluzione da ogni pozzetto e ripetere il processo di lavaggio 5 volte come descritto al punto 6.4.
- Aggiungere 90 μl di reagente substrato a ciascun pozzetto e incubare per circa 15 minuti a 37 °C con un nuovo sigillante, proteggendo la piastra dalla luce.
NOTA: Il tempo di reazione può essere modificato in base all'effettivo cambiamento di colore, ma non deve superare i 30 minuti. Lasciare che il lettore di micropiastre si riscaldi per ~15 minuti prima della misurazione OD. - Aggiungere 50 μL/pozzetto di soluzione di arresto nello stesso ordine della soluzione di substrato.
- Determinare la densità ottica (valore OD) di ciascun pozzetto contemporaneamente con un lettore di micropiastre impostato su 450 nm.
- Ottenere i valori OD per i campioni standard e replicare i pozzetti dei campioni, quindi sottrarre i valori OD dei pozzetti bianchi per ottenere i valori corretti. Quindi, eseguite l'adattamento lineare con la concentrazione come ascissa e il valore OD come ordinata. Sulla base dell'equazione adattata, calcolare i valori di concentrazione di IL-17A per i pozzetti del campione.
NOTA: Dopo 4 giorni di differenziazione, il contenuto di IL-17A nel surnatante di ciascun gruppo è mostrato nella Figura 4. Il metodo di adattamento può fare riferimento al software Orgin21. La curva standard ottenuta da questo esperimento ha un valore R2 di 0,9946.
7. Differenziazione delle cellule T mediante saggi di espressione genica mediante PCR quantitativa (qPCR)
NOTA: Per escludere la possibilità di instabilità della citometria a flusso dovuta agli effetti dei coloranti e della fissazione/rottura, abbiamo rilevato l'espressione di geni caratteristici mediante qPCR per chiarire l'effetto di differenziazione del Th17 patogeno a livello trascrizionale.
- Misurare il numero di cellule al termine della coltura di cellule T. Raccogliere le cellule in provette da centrifuga da 1,5 mL e centrifugare a 400 × g per 5 minuti. Scartare con cura tutti i surnatanti.
- Estrazione dell'RNA
NOTA: Il processo di estrazione deve essere effettuato in una cappa aspirante o in un banco ultra-pulito per evitare la contaminazione dell'enzima RNA.- Aggiungere 500 μl di tampone di lisi (ad es. a 1-2 × 106 cellule) alla provetta del campione (passaggio 7.1), agitare e mescolare immediatamente fino a quando non c'è massa cellulare e lasciarlo riposare per 1 minuto.
- Aggiungere la miscela alla colonna di adsorbimento posta in una provetta di raccolta, centrifugare a 13.400 × g per 30 s ed eliminare il filtrato.
- Aggiungere la quantità specificata di etanolo assoluto al flacone del tampone di lavaggio prima di utilizzarlo per la prima volta. Aggiungere 500 μl di tampone di lavaggio alla colonna di adsorbimento, centrifugare a 13.400 × g per 30 s ed eliminare il filtrato.
- Ripetere il lavaggio al punto 7.2.3.
- Posizionare la colonna di adsorbimento RA nella provetta di raccolta vuota e centrifugare a 13.400 × g per 2 minuti per rimuovere la soluzione di risciacquo.
NOTA: La soluzione di risciacquo deve essere rimossa per evitare che l'etanolo residuo nella soluzione di risciacquo inibisca la reazione a valle. - Rimuovere la colonna di adsorbimento RA e metterla in una provetta da centrifuga pulita e priva di RNasi. Aggiungere 50 μL di H2O privo di RNasi al centro della membrana di adsorbimento, lasciarlo riposare per 1 minuto a temperatura ambiente e centrifugare a 9.600 × g per 1 minuto.
- Misurare la purezza e la concentrazione dell'RNA.
- Sintetizzare il primo filamento di cDNA seguendo le istruzioni del produttore del kit.
- Rimuovi il DNA genomico. Prelevare 500 ng dell'RNA stampo estratto in una provetta da centrifuga priva di RNasi; aggiungere ddH2O senza RNasi e 4x miscela di gDNA per formare una miscela. Frullare delicatamente con una pipetta e porre a bagnomaria a 42 °C per 2 minuti.
NOTA: La quantità della miscela di reazione e la quantità aggiunta sono correlate alla concentrazione di RNA; Fare riferimento alle istruzioni in dettaglio. In questo esperimento è stato utilizzato il sistema di reazione da 20 μl. - Aggiungere direttamente un sistema di reazione di trascrizione inversa 5x nella provetta di reazione del passaggio precedente. Prelevare 4 μl del sistema di reazione di trascrizione inversa e 16 μl della miscela del passaggio precedente e mescolare delicatamente con una pipetta.
NOTA: Il sistema di trascrizione inversa include tutta la trascrittasi inversa richiesta, che è un sistema di trascrizione inversa configurato direttamente. - Impostare la reazione di trascrizione inversa: 37 °C 15 min, 85 °C, 5 s e infine raffreddare a 4 °C.
- Rimuovi il DNA genomico. Prelevare 500 ng dell'RNA stampo estratto in una provetta da centrifuga priva di RNasi; aggiungere ddH2O senza RNasi e 4x miscela di gDNA per formare una miscela. Frullare delicatamente con una pipetta e porre a bagnomaria a 42 °C per 2 minuti.
- Impostare la reazione PCR seguendo le istruzioni del produttore.
- Aggiungere 10 μl di miscela enzimatica di reazione PCR 2x nella provetta qPCR, 0,4 μl di primer 1, 0,4 μl di primer 2, 0,4 μl di 50x di colorante di riferimento ROX 1, 2 μl di cDNA stampo e 6,8 μl di ddH2O per preparare una miscela da 20 μl.
- Eseguire la PCR nello strumento di PCR quantitativa a fluorescenza in tempo reale utilizzando le seguenti impostazioni: fase 1, 95 °C, 30 s, Rep x 1; stadio 2, primi 95 °C, 10 s, poi 60 °C, 30 s, Rep x 40; stadio 3, primi 95 °C, 15 s, successivi 60 °C, 60 s, infine 95 °C, 15 s, Rep x 1.
NOTA: Dopo 4 giorni di differenziazione, i livelli di espressione relativi di Il17a, Il17f, Rra e Il23r nelle cellule T CD4+ naive sono mostrati nella Figura 5.
Risultati
Il nostro protocollo è stato sviluppato sulla base di precedenti ricerche sulla differenziazione patogena delle cellule Th17. Il primo passo dell'esperimento consiste nel rilevare la purezza delle cellule T CD4+ naive isolate dalla milza mediante smistamento di biglie magnetiche, che è la base per il successo della nostra successiva differenziazione patogena delle cellule Th17. La purezza delle cellule T CD4+ naive è stata rilevata utilizzando i marcatori di superficie CD62L23 e CD4424 , mentre FOXP325 è stato utilizzato come marcatore delle cellule Treg. Abbiamo scoperto che il contenuto di cellule Treg era significativamente ridotto dopo lo smistamento e la purezza delle cellule T CD4+ naive poteva raggiungere almeno il 95% (Figura 1). Per confrontare la differenziazione delle cellule Th17 patogene, le cellule T CD4+ naive sono state coltivate con terreno Th0 (Tabella 1) e terreno di differenziazione Th17 (Tabella 1) per un totale di 5 giorni. È stato riscontrato che le cellule T mostravano una crescita a grappolo sia nei terreni Th0 che in quelli Th17 (Figura 2).
Successivamente, le cellule sono state fissate, permeabilizzate e marcate con anticorpi contro diverse citochine in cellule T CD4+ differenziate in base alla citometria a flusso. Abbiamo esaminato IL17A26 e RORγT27 come citochine distintive della differenziazione delle cellule Th17 e abbiamo scoperto che il 90% delle cellule T CD4+ naive si è differenziato con successo in cellule Th17 patogene sotto la stimolazione di un nuovo terreno di coltura cellulare Th17 (Figura 3). La Figura 5 mostra i geni caratteristici delle cellule Th17 rilevate mediante PCR, che hanno dimostrato che le cellule Th17 patogene che abbiamo ottenuto per differenziazione erano stabilmente esprimesse.
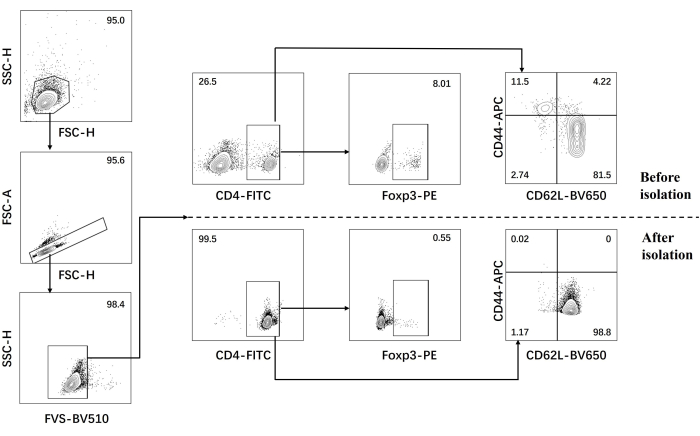
Figura 1: Strategia di gating per l'analisi delle citochine di firma nel topo C57BL/6J prima e dopo l'isolamento naive delle cellule T CD4+ . Abbreviazioni: FSC-H = altezza del picco di dispersione in avanti; SSC-H = altezza scatter-peah laterale; FSC-A = area di picco di dispersione in avanti; FITC = isotiocianato di fluoresceina; PE = ficoeritrina. Clicca qui per visualizzare una versione più grande di questa figura.
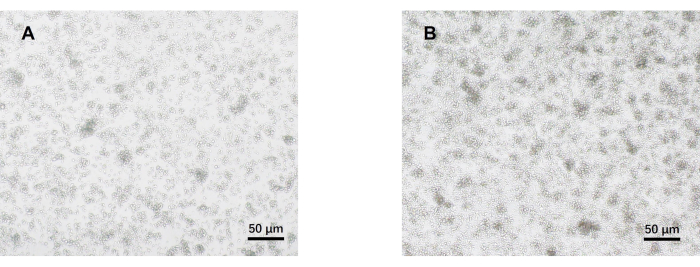
Figura 2: Immagini rappresentative di cellule T CD4+ naïve al topo coltivate in condizioni patogene Th17 e Th0 per 5 giorni. (A) cellule Th0; (B) cellule Th17 patogene. Barre di scala = 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
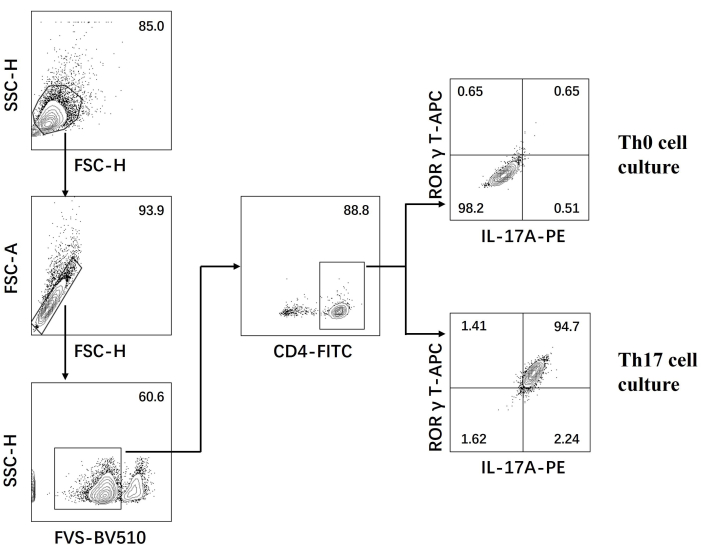
Figura 3: Analisi della citometria a flusso dopo il differenziamento indotto dal terreno di coltura cellulare Th0 e dal terreno di coltura cellulare Th17. Abbreviazioni: FSC-H = altezza del picco di dispersione in avanti; SSC-H = altezza scatter-peah laterale; FSC-A = area di picco di dispersione in avanti; FITC = isotiocianato di fluoresceina; PE = ficoeritrina. Clicca qui per visualizzare una versione più grande di questa figura.
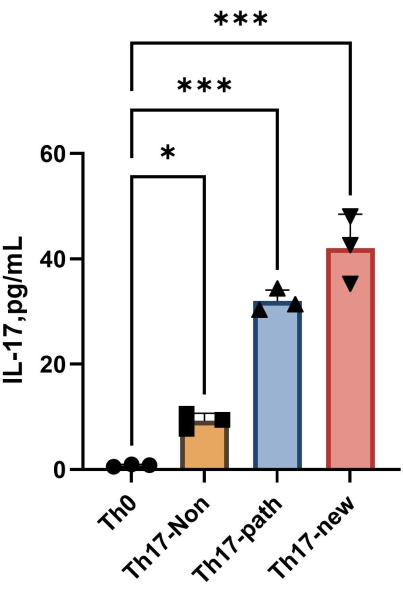
Figura 4: Il contenuto di IL-17A nel surnatante del terreno di coltura cellulare Th0 e del terreno di coltura cellulare Th17 dopo l'induzione del differenziamento. Fare clic qui per visualizzare una versione più grande di questa figura.
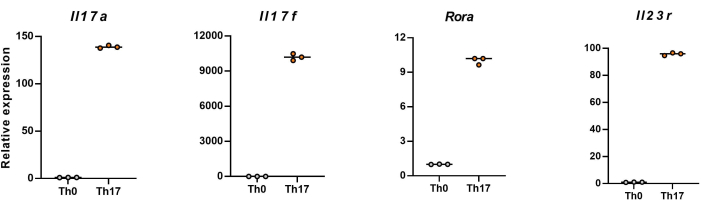
Figura 5: Risultati rappresentativi dei livelli di espressione delle citochine di firma nelle cellule T CD4+ differenziate di topo C57BL/6J. Clicca qui per visualizzare una versione più grande di questa figura.
| Terreno di coltura cellulare Th17 patogeno target | Terreno di coltura cellulare Th0 | Terreno di coltura cellulare Th17 classico non patogeno | Terreno di coltura cellulare Th17 patogeno classico. | |||||||||||
| Reagente | Concentrazione finale | Importo | Concentrazione finale | Importo | Concentrazione finale | Importo | Concentrazione finale | Importo | ||||||
| Penicillina-Streptomicina (100x) | 1x | 500 μl | 1x | 500 μl | 1x | 500 μl | 1x | 500 μl | ||||||
| Siero fetale bovino | 10% | 5 ml | 10% | 5 ml | 10% | 5 ml | 10% | 5 ml | ||||||
| β-mercaptoetanolo | 50 μM | 50 μl | 50 μM | 50 μl | 50 μM | 50 μl | 50 μM | 50 μl | ||||||
| Integratore GlutaMAXTM (100x) | 1x | 500 μl | 1x | 500 μl | 1x | 500 μl | 1x | 500 μl | ||||||
| Soluzione di piruvato di sodio (100x) | 1 mM | 500 μl | 1 mM | 500 μl | 1 mM | 500 μl | 1 mM | 500 μl | ||||||
| Anti-topo IFN gamma (1 mg/mL) | 5 μg/mL | 250 μl | 10 μg/mL | 500 μl | 10 μg/mL | 500 μl | ||||||||
| Anti-Mouse IL-4 (2 mg/mL) | 5 μg/mL | 125 μl | 5 μg/mL | 250 μl | 10 μg/mL | 250 μl | ||||||||
| Mouse rIL-1 beta (20 μg) | 20 ng/mL | NA | ||||||||||||
| Mouse rIL-6 (20 μg) | 20 ng/mL | NA | 50 ng/mL | NA | 50 ng/mL | NA | ||||||||
| Mouse rIL-23 (50 μg) | 50 ng/mL | NA | 10 ng/mL | NA | ||||||||||
| TGF beta del topo (100 μg) | 3 ng/mL | NA | 1 ng/mL | NA | 1 ng/mL | NA | ||||||||
| IL-2 murina (5 μg) | 20 ng/mL | NA | ||||||||||||
| RPMI 1640 | NA | Fino a 50 mL | NA | Fino a 50 mL | NA | Fino a 50 mL | ||||||||
| Totale | NA | 50 ml | NA | 50 ml | NA | 50 ml | ||||||||
Tabella 1: Colture cellulari Th17 patogene target, colture cellulari Th0, terreni di coltura cellulare Th17 classici non patogeni e patogeni.
Discussione
Questa procedura ha offerto un modo produttivo per aumentare il numero di cellule T CD4+ naïve allo splenic di topo per la produzione in vitro di cellule Th17 patogene. Sebbene utilizziamo più citochine rispetto ad altri terreni di coltura cellulare Th17 riportati, ci impegniamo a ottimizzare le condizioni di crescita delle cellule Th17 patogene. Stiamo valutando un'ulteriore ottimizzazione del nostro protocollo di differenziazione indotta.
In questo caso, abbiamo semplicemente utilizzato la citometria a flusso e la qPCR per esaminare la produzione di citochine distintive. Con alcune piccole modifiche, questo approccio può essere utilizzato anche per altri test funzionali, come la proliferazione cellulare.
Abbiamo utilizzato un kit di isolamento dei linfociti prodotto in Cina per isolare i linfociti della milza di topo perché è efficace e fa risparmiare tempo. Le soluzioni di separazione dei linfociti basate su altre marche possono anche raggiungere lo scopo di separare i linfociti della milza di topo attraverso diversi passaggi. Un altro metodo consiste nel lisare direttamente i globuli rossi della sospensione di cellule della milza ottenuta; Tuttavia, abbiamo scoperto che i globuli rossi della milza spesso non possono essere lisati in una sola volta.
Durante l'esecuzione di questo protocollo possono sorgere alcuni problemi. In primo luogo, il numero di cellule T CD4+ naive ottenute mediante smistamento con biglie magnetiche può essere molto basso (fase 3 del protocollo). Ciò potrebbe essere attribuito al fatto che il processo di frantumazione degli organi è insufficiente. È importante assicurarsi che l'organo sia adeguatamente omogeneizzato. L'aumento della frequenza di risciacquo durante il processo di omogeneizzazione migliorerà il tasso di recupero. Per ottenere un numero maggiore di cellule T CD4+ naïve alla milza, suggeriamo di utilizzare topi più giovani (6-10 settimane di età). Sono disponibili vari metodi per separare i linfociti della milza e la resa può variare a seconda del liquido di separazione utilizzato. Si consiglia di utilizzare un liquido di separazione universalmente certificato e cercare di estrarre il più possibile lo strato linfocitario.
In secondo luogo, la proporzione di cellule T CD4+ nella citometria a flusso può essere del <80% (fase 5 del protocollo). Una possibile causa di questo problema potrebbe essere una conta degli splenociti imprecisa, con conseguente conta cellulare maggiore di quella del cocktail di anticorpi aggiuntivi e delle microsfere magnetiche. Per aumentare l'efficacia della purificazione naive delle cellule T CD4+ , il conteggio delle cellule deve essere preciso. Inoltre, è possibile utilizzare il 10% in più di cocktail di anticorpi e microsfere magnetiche rispetto a quanto raccomandato da questo protocollo. Infine, la citometria a flusso può essere eseguita immediatamente dopo lo smistamento delle cellule T CD4+ naive.
In terzo luogo, potrebbero non esserci molti cluster di cellule T formati durante la coltura e la maggior parte delle cellule potrebbe essere morta durante la differenziazione delle cellule T (fase 4 del protocollo). La potenziale ragione di questo problema potrebbe essere la determinazione imprecisa del numero di cellule per le cellule T CD4+ naive prima della semina, con conseguente bassa densità cellulare. Si raccomanda di adottare un metodo di conteggio più accurato per ottenere la densità cellulare desiderata di circa 4 × 105 cellule/mL per ciascun pozzetto in una piastra a 48 pozzetti. Un'altra possibile causa potrebbe essere costituita da problemi tecnici con l'incubatore di CO2 , come una temperatura o una concentrazione di CO2 errate. Infine, una forza eccessiva durante la modifica del terreno di coltura cellulare potrebbe potenzialmente causare la morte cellulare.
In quarto luogo, i livelli di espressione relativa dei geni delle citochine possono essere bassi (fase 7 del protocollo). Per garantire l'autenticità dell'RNA estratto, si consiglia di utilizzare una concentrazione di rilevamento di nanogoccioline superiore a 100 ng/mL. La potenziale ragione della diminuzione della concentrazione può essere la natura malsana delle cellule in coltura, ad esempio che gran parte delle cellule raccolte sono morte o in fase di morte. Per ottenere la vera concentrazione di RNA, è necessario risolvere la situazione che può portare a una scarsa crescita durante la coltura cellulare. Una ragione alternativa alla base di questa preoccupazione potrebbe essere la conta cellulare finale eccessivamente bassa raggiunta durante l'estrazione dell'RNA, probabilmente a causa della perdita involontaria di cellule durante lo smaltimento del surnatante. L'impiego di tecniche avanzate di estrazione dell'RNA, come i kit di estrazione dell'RNA in un solo passaggio, potrebbe rivelarsi vantaggioso. Il rapporto OD260/OD280 ideale, misurato da Nanodrop, dovrebbe rientrare nell'intervallo 1,9-2,1. In caso di un rapporto eccessivamente basso, la contaminazione proteica diventa una possibilità. Aumentare la frequenza del lavaggio con tampone privo di RNasi può aiutare a mitigare questo problema. Al contrario, un rapporto insolitamente basso implica una potenziale degradazione dell'RNA. Per contrastare questo problema, si consiglia di utilizzare acqua priva di RNasi e provette da 1,5 ml per la decontaminazione con RNasi.
In conclusione, l'attuale protocollo descrive l'uso di nuovi terreni di coltura cellulare per indurre direttamente le cellule T CD4+ naive a differenziarsi in cellule Th17 patogene in vitro. Rispetto alla separazione diretta, non c'è dubbio che questo metodo sia più diretto, economico e più efficiente. Anche la configurazione del terreno è molto semplice in modo che le cellule Th17 costruite possano essere utilizzate in modo più intuitivo per esperimenti successivi, fornendo un ottimo modello cellulare per lo studio di molte malattie.
Divulgazioni
Gli autori dichiarano che non vi sono conflitti di interesse in merito alla pubblicazione di questo articolo.
Riconoscimenti
Il lavoro è stato sostenuto dal National Key R&D Program of China (No.2022YFC2304604), dalla National Natural Science Foundation of China (No.81971812), dalla National Natural Science Foundation of China (No.82272235), dalla Science Foundation della Commissione della Salute della Provincia di Jiangsu (No.8272235), dalla Science Foundation of the Health della provincia di Jiangsu (No.822YFC2304604). ZDB2020009), Progetto speciale del programma chiave di ricerca, sviluppo (sviluppo sociale) della provincia di Jiangsu (n. BE2021734), Programma di ricerca e sviluppo chiave nazionale del Ministero della Scienza e della Tecnologia (n. 2020YFC083700), Laboratorio chiave della provincia di Jiangsu di medicina di terapia intensiva (BM2020004), Progetto chiave della Fondazione nazionale di scienze naturali della Cina (81930058), Fondazione nazionale di scienze naturali della Cina (82171717), Fondi di ricerca di base delle università centrali (2242022K4007), e il Progetto Generale della Fondazione Provinciale di Scienze Naturali del Jiangsu (BK20211170).
Materiali
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| Rat anti-mouse CD62L, BV650, clone MEL-14, 1:200 dilution | BD | Cat# 564108; RRID: AB_2738597 | |
| Rat monoclonal anti-CD4, FITC, clone RM4-5, 1:200 dilution | BioLegend | Cat#100509; RRID: AB_312712 | |
| Rat monoclonal anti-IL-17A, PE, clone TC11-18H10.1, 1:200 dilution | BioLegend | Cat#506903; RRID: AB_ 315463 | |
| Rat monoclonal anti mouse/human CD44, APC, clone IM&, 1:200 dilution | BioLegend | Cat#103012; RRID: AB_312963 | |
| Rat monoclonal anti-RORγT, APC, clone B2D, 1:200 dilution | Invitrogen | Cat#17-6981-80; RRID: AB_2573253 | |
| Rat monoclonal FOXP3 antibody, PE, clone FJK-16s, 1:200 dilution | Invitrogen | Cat#12-5773-82; RRID: AB_465936 | |
| Chemicals, peptides, and recombinant proteins | |||
| Anti-Mouse CD3 SAFIRE purified | biogems | Cat#05112-25 | |
| Anti-Mouse CD28 SAFIRE purified | biogems | Cat#10312-25 | |
| Anti-Mouse IFN gamma | biogems | Cat#80822-25 | |
| Anti-Mouse IL-4 | biogems | Cat#81112-25 | |
| Ethanol | Xilong scientific | Cat#64-17-5 | |
| Fetal bovine serum | Gibco | Cat#10437-028 | |
| FcR Blocking reagent | Miltenyi Biotec | Cat#130-092-575 | |
| GlutaMAX supplement | gibco | Cat#35050079 | |
| Mouse rIL-1 beta | Sino Biological | Cat#50101-MNAE | |
| Mouse rIL-6 | Sino Biological | Cat#50136-MNAE | |
| Mouse rIL-23 | Sino Biological | Cat#CT028-M08H | |
| Mouse TGF beta 1 | Sino Biological | Cat#50698-M08H | |
| PBS | Procell | Cat#PB180327 | |
| Recombinant Murine IL-2 | peprotech | Cat#212-12 | |
| RPMI 1640 with L-glutamine | Gibco | Cat#11875-119 | |
| Penicillin-streptomycin solution | Gibco | Cat#15070063 | |
| β-mercaptoethanol | Sigma-Aldrich | Cat#M6250-100ML | |
| Critical commercial assays | |||
| Animal Organ Lymphocyte Separation Solution Kit | Tbdscience | Cat#TBD0018SOP | Contains animal spleen tissue lymphocyte separation liquid, tissue sample diluent (cat no. 2010C1119), sample cleaning solution (cat no. 2010X1118), sample washing solution (cat no. TBTDM-W), tissue homogenate flushing liquid (F2013TBD) |
| ChamQ SYBR qPCR Master Mix (High ROX Premixed) | Vazyme | Cat#Q341-02 | https://www.vazymebiotech.com/product-center/chamq-sybr-qpcr-master-mix-high-rox-premixed-q341.html. |
| Fixation/permeabilization Concentrate | invitrogen | Cat#00-5123-43 | |
| Fixation / Permeabilization Diluent | invitrogen | Cat#00-5223-56 | |
| Fixable Viability Dye EF506 | invitrogen | Cat#65-0866 | |
| HiScript II Q RT SuperMix for qPCR (+gDNA wiper) | Vazyme | Cat#R223-01 | https://www.vazymebiotech.com/product-center/hiscript-ii-q-rt-supermix-for-qpcr-gdna-wiper-r223.html. |
| Leukocyte Activation Cocktail | BD | Cat#550583 | |
| Mouse IL-17A (Interleukin 17A) ELISA Kit | Elabscience® | Cat#E-EL-M0047 | |
| Naïve CD4+ T cells isolation kit, mouse | STEMCELL | Cat#19765 | EasySep kit contains mouse CD4+ T cell isolation cocktail [cat no. 19852C.1], mouse memory T cell depletion cocktail [cat no. 18766C], streptavidin RapidSphered 50001 [cat no. 50001], normal rat serum [cat no. 13551]); only store rat serum at -20 °C; other components to be stored at 2-8 °C. |
| Permeabilization Buffer | invitrogen | Cat#00-8333-56 | |
| SPARKeasy Cell RNA Kit | Sparkjade | Cat#AC0205-B | https://www.sparkjade.com/product/detail?id=85 |
| Experimental models: Organisms/strains | |||
| Mouse: C57BL/6 | Gempharmatech | Cat#000013 | |
| Oligonucleotides | |||
| Mouse Il17a TaqMan primers with probe | ribobio | NA | |
| Mouse Il17f TaqMan primers with probe | ribobio | NA | |
| Mouse Il23r TaqMan primers with probe | ribobio | NA | |
| Mouse Rora TaqMan primers with probe | ribobio | NA | |
| β-actin TaqMan primers with probe | ribobio | NA | |
| Software and algorithms | |||
| Cytek Aurora | Cytek | https://spectrum.cytekbio.com/ | |
| FlowJo v10.8.1 | Tree Star | https://www.flowjo.com | |
| GraphPad prism 9 | GraphPad Software | https://www.graphpad-prism.cn | |
| Other | |||
| 1 mL syringe | Kindly | NA | |
| 1.5 mL Centrifuge tubes | Eppendorf | Cat#MCT-150-C | |
| 5 mL Round-bottom tubes | Corning | Cat#352235 | |
| 15 mL Centrifuge tubes | NEST | Cat#601052 | |
| 48-Well tissue culture plate (flatten bottom) | Corning | Cat#3548 | |
| 50 mL Centrifuge tubes | NEST | Cat#602052 | |
| 70 µm Cell strainer | Biosharp | Cat#BS-70-XBS | |
| 96-well Unskirted qPCR Plates | VIOX scientific | Cat#V4801-M | |
| 100 mm Petri dish | Corning | Cat#430167 | |
| Centrifuge | Eppendorf | 5425R | |
| Cell culture CO2 incubator | Thermo Fisher | HEPA Class100 | |
| Cytek Aurora | Cytek | NA | |
| dissecting scissors | RWD | S12003-09 | |
| Hemocytometer | AlphaCell | Cat#J633201 | |
| NanoDrop 2000 Spectrophotometer | Thermo Fisher | ND-2000 | |
| Real-time PCR System | Roche | LightCycler96 | |
| Surgical tweezers | RWD | F12005-10 | |
| Thermal cycler | Bio-Rad | C1000 Touch |
Riferimenti
- Korn, T., Bettelli, E., Oukka, M., Kuchroo, V. K. IL-17 and Th17 cells. Annu Rev Immunol. 27, 485-517 (2009).
- Bhaumik, S., Basu, R. Cellular and molecular dynamics of Th17 differentiation and its developmental plasticity in the intestinal immune response. Front Immunol. 8, 254 (2017).
- Bettelli, E., Korn, T., Oukka, M., Kuchroo, V. K. Induction and effector functions of T(H)17 cells. Nature. 453 (7198), 1051-1057 (2008).
- Korn, T., et al. IL-21 initiates an alternative pathway to induce proinflammatory T(H)17 cells. Nature. 448 (7152), 484-487 (2007).
- Bettelli, E., et al. Reciprocal developmental pathways for the generation of pathogenic effector TH17 and regulatory T cells. Nature. 441 (7090), 235-238 (2006).
- Yang, X. O., et al. T helper 17 lineage differentiation is programmed by orphan nuclear receptors ROR alpha and ROR gamma. Immunity. 28 (1), 29-39 (2008).
- Ghoreschi, K., et al. Generation of pathogenic T(H)17 cells in the absence of TGF-β signalling. Nature. 467 (7318), 967-971 (2010).
- Lee, Y., et al. Induction and molecular signature of pathogenic TH17 cells. Nat Immunol. 13 (10), 991-999 (2012).
- McGeachy, M. J., et al. The interleukin 23 receptor is essential for the terminal differentiation of interleukin 17-producing effector T helper cells in vivo. Nat Immunol. 10 (3), 314-324 (2009).
- Wu, B., et al. The TGF-β superfamily cytokine Activin-A is induced during autoimmune neuroinflammation and drives pathogenic Th17 cell differentiation. Immunity. 54 (2), 308-323 (2021).
- Ramesh, R., et al. Pro-inflammatory human Th17 cells selectively express P-glycoprotein and are refractory to glucocorticoids. J Exp Med. 211 (1), 89-104 (2014).
- Basdeo, S. A., et al. Ex-Th17 (nonclassical Th1) cells are functionally distinct from classical Th1 and Th17 cells and are not constrained by regulatory T cells. J Immunol. 198 (6), 2249-2259 (2017).
- Cua, D. J., et al. Interleukin-23 rather than interleukin-12 is the critical cytokine for autoimmune inflammation of the brain. Nature. 421 (6924), 744-748 (2003).
- Awasthi, A., et al. Cutting edge: IL-23 receptor gfp reporter mice reveal distinct populations of IL-17-producing cells. J Immunol. 182 (10), 5904-5908 (2009).
- Jäger, A., Dardalhon, V., Sobel, R. A., Bettelli, E., Kuchroo, V. K. Th1, Th17, and Th9 effector cells induce experimental autoimmune encephalomyelitis with different pathological phenotypes. J Immunol. 183 (11), 7169-7177 (2009).
- Lee, Y., Collins, M., Kuchroo, V. K. Unexpected targets and triggers of autoimmunity. J Clin Immunol. 34, S56-S60 (2014).
- Chang, D., et al. The conserved non-coding aequences CNS6 and CNS9 control cytokine-induced Rorc transcription during T helper 17 cell differentiation. Immunity. 53 (3), 614-626 (2020).
- Bunte, K., Beikler, T. Th17 cells and the IL-23/IL-17 axis in the pathogenesis of periodontitis and immune-mediated inflammatory diseases. Int J Mol Sci. 20 (14), 3394 (2019).
- Zhao, Y., Liu, Z., Qin, L., Wang, T., Bai, O. Insights into the mechanisms of Th17 differentiation and the Yin-Yang of Th17 cells in human diseases. Mol Immunol. 134, 109-117 (2021).
- Berghmans, N., et al. Interferon-γ orchestrates the number and function of Th17 cells in experimental autoimmune encephalomyelitis. J Interferon Cytokine Res. 31 (7), 575-587 (2011).
- Wu, B., Wan, Y. Molecular control of pathogenic Th17 cells in autoimmune diseases. Int Immunopharmacol. 80, 106187 (2020).
- Du, L., et al. Growth hormone releasing hormone signaling promotes Th17 cell differentiation and autoimmune inflammation. Nat Commun. 14 (1), 3298 (2023).
- Ernst, D. N., Weigle, W. O., Noonan, D. J., McQuitty, D. N., Hobbs, M. V. The age-associated increase in IFN-gamma synthesis by mouse CD8+ T cells correlates with shifts in the frequencies of cell subsets defined by membrane CD44, CD45RB, 3G11, and MEL-14 expression. J Immunol. 151 (2), 575-587 (1993).
- Radtke, A. J., et al. IBEX: an iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- El-Hindi, K., et al. T-cell-specific CerS4 depletion prolonged inflammation and enhanced tumor burden in the AOM/DSS-induced CAC model. Int J Mol Sci. 23 (3), 1866 (2022).
- Cooney, L. A., Towery, K., Endres, J., Fox, D. A. Sensitivity and resistance to regulation by IL-4 during Th17 maturation. J Immunol. 187 (9), 4440-4450 (2011).
- Zhu, X., et al. A novel interleukin-2-based fusion molecule, HCW9302, differentially promotes regulatory T cell expansion to treat atherosclerosis in mice. Front Immunol. 14, 1114802 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon