Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Cell-free dot blot en tant que plateforme d’immunodosage pratique et adaptable pour la détection de la réponse anticorps dans les sérums humains et animaux
Dans cet article
Résumé
Nous décrivons une plateforme d’immunoessais récemment développée basée sur les principes de la biologie synthétique acellulaire et la technique dot-blot pour la détection personnalisable de la réponse anticorps dans les sérums humains et animaux.
Résumé
La série d’épidémies pathogènes mondiales au cours des deux dernières décennies a mis en évidence l’importance des stratégies de sérosurveillance. Les plateformes d’immunodosage qui servent à détecter les anticorps spécifiques de la maladie dans le sérum des patients sont au cœur de la sérosurveillance. Les exemples courants incluent les tests immuno-enzymatiques et les tests à flux latéral ; Cependant, bien qu’il s’agisse de méthodes de référence, elles nécessitent des consommables spécifiques aux agents pathogènes et des équipements spécialisés, ce qui limite leur utilisation en dehors des laboratoires disposant de ressources suffisantes.
Nous avons récemment mis au point une nouvelle plateforme d’immunodosage appelée Cell-Free Dot-Blot (CFDB) et l’avons validée à l’aide de sérums humains et animaux contre le SRAS-CoV-2. Contrairement aux immunoessais conventionnels, les échantillons de sérum de patients CFDB sont immobilisés dans une phase solide (membrane de nitrocellulose), tandis que l’antigène cible est en suspension dans la phase mobile du test. Pour améliorer l’accès aux capacités de sérosurveillance, les antigènes CFDB sont produits à la demande et avec une infrastructure à faible charge en utilisant l’expression in vitro des protéines. Ici, l’antigène est fusionné avec une étiquette peptidique qui peut être détectée à l’aide d’une seule protéine rapporteuse universelle pour tout test CFDB. Par conséquent, la CFDB n’a pas besoin d’avoir accès à un lecteur de plaques multipuits ou à des composants d’essai moléculaire commerciaux purifiés. Grâce à ces considérations de conception, CFDB comble les lacunes des plateformes d’immunoessais existantes en offrant l’accessibilité aux laboratoires non centralisés, l’adaptabilité aux agents pathogènes émergents et l’abordabilité aux communautés à faible revenu.
Dans le présent article, nous fournirons un protocole étape par étape pour préparer et effectuer un dosage immunologique CFDB. En utilisant nos récents travaux sur le SARS-CoV-2 CFDB à titre d’exemple, nous couvrirons la conception de l’ADN antigénique pour la production acellulaire à la demande, suivie de la préparation de la protéine rapporteure CFDB, de l’immobilisation d’échantillons de sérum sur la phase solide, et enfin, des étapes de liaison et de détection de l’antigène du test. Nous prévoyons qu’en suivant ces instructions, les chercheurs seront en mesure d’adapter le test CFDB pour détecter les réponses immunitaires dans les sérums humains et animaux à n’importe quel agent pathogène donné.
Introduction
La pandémie de COVID-19 a révélé le besoin critique d’outils de diagnostic abordables et évolutifs, en particulier pour les milieux à faibles ressources1. Les immunotests conventionnels tels que les tests immuno-enzymatiques (ELISA) se sont avérés essentiels pour détecter les réponses immunitaires 2,3. Cependant, leur coût élevé, leur dépendance à l’égard de réactifs complexes et leur dépendance à l’égard d’équipements spécialisés limitent leur accessibilité, en particulier pendant les crises sanitaires mondiales. En réponse à ces défis, nous avons développé le Cell-Free Dot Blot (CFDB), une plateforme d’immunodosage adaptable et peu coûteuse conçue pour la détection d’anticorps anti-SRAS-CoV-2 dans les sérums humains et animaux.
La CFDB tire parti de la biologie synthétique acellulaire pour la production rapide et à la demande d’antigènes viraux à l’aide de modèles d’ADN linéaires 4,5. Cela élimine le besoin de processus traditionnels de clonage, d’expression et de purification basés sur les cellules, accélérant considérablement la production d’antigènes tout en réduisant les coûts. La méthode CFDB simplifie la détection des anticorps en utilisant un format de transfert de points, où les sérums sont directement repérés sur les membranes de nitrocellulose. Ce système évite d’avoir besoin de plaques multipuits coûteuses et d’équipements de laboratoire spécialisés, ce qui permet un flux de travail simple pour les étapes d’incubation et de lavage. La plate-forme utilise également un système SpyCatcher-SpyTag, où une chimère de peroxydase SpyCatcher2-Apex2 agit comme un réactif de détection secondaire universel 5,6. Celui-ci est produit à l’aide de l’expression standard basée sur Escherichia coli, ce qui élimine la dépendance aux conjugués d’anticorps commerciaux coûteux. En conséquence, le système CFDB peut effectuer des tests sérologiques avec des performances comparables à celles des ELISA à un coût nettement inférieur - environ 3 USD par test de 96 échantillons contre plus de 300 USD pour un kit ELISAcommercial 5.
Pour démontrer l’efficacité de la CFDB, nous avons testé sa capacité à détecter les anticorps dans des sérums humains et animaux précaractérisés. Nos résultats sont étroitement corrélés à l’ELISA pour l’identification des échantillons positifs et négatifs au COVID-19. En plus du diagnostic humain, nous avons évalué l’utilité de la CFDB dans des modèles animaux, en testant des sérums de hamsters infectés par le SRAS-CoV-2 et de ceux vaccinés avec la protéine recombinante de la nucléocapside. Ces tests ont confirmé le potentiel d’utilisation de CFDB dans les diagnostics humains et vétérinaires, ce qui en fait un outil polyvalent pour surveiller les réponses immunitaires de toutes les espèces. L’un des principaux avantages de la CFDB est sa flexibilité. En modifiant simplement la matrice d’ADN codant pour l’antigène d’intérêt, la plateforme peut être rapidement adaptée pour détecter les anticorps contre différents agents pathogènes, ce qui la rend précieuse pour la préparation aux futures pandémies. Son faible coût, son flux de travail simple et ses exigences minimales en matière d’infrastructure le rendent particulièrement adapté aux laboratoires décentralisés et aux environnements à faibles ressources, où l’accès aux diagnostics commerciaux est limité.
Dans ce travail, nous fournirons des instructions étape par étape pour la préparation et la réalisation d’un test CFDB. Tout d’abord, nous couvrons la conception et la synthèse de matrices d’ADN linéaires pour la production acellulaire d’antigènes, qui sont les principaux réactifs de détection du test. Nous décrivons ensuite les étapes de préparation du réactif de détection secondaire du test SpyCatcher2-Apex2. Ensuite, nous fournissons des instructions pour la production acellulaire et le contrôle de la qualité des antigènes eux-mêmes. Enfin, nous décrivons en détail le processus de réalisation d’un test CFDB sur des échantillons de sérum humain ou animal.
Protocole
Toutes les expériences sur les hamsters ont été réalisées au Laboratoire national de microbiologie (LNM) de l’Agence de la santé publique du Canada, avec l’approbation du Centre scientifique canadien de santé humaine et animale et conformément aux lignes directrices du Conseil canadien de protection des animaux. Tous les échantillons de sérum et de plasma humains ont été obtenus commercialement pour des analyses à l’interne ou fournis par des collaborateurs cliniques au LNM pour des analyses indépendantes au LNM.
1. Conception et préparation de modèles d’expression linéaire d’antigènes (LETs)
- Concevoir les LET d’expression acellulaire de l’antigène cible en se basant sur les instructions de Norouzi et al.7 et contenant un SpyTag N/C-terminal comme décrit dans Norouzi et al.5.
REMARQUE : Nous fournissons ici des détails pour la préparation de la protéine de nucléocapside du SRAS-CoV-2 (NP, acides aminés 2-419).- Assurez-vous que le LET pour le NP du SARS-CoV-2 (figure 1 et fichier supplémentaire 1) contient des sites Ter 5' et 3' avec une séquence tampon de 50 paires de bases avant le promoteur T7 et après le codon stop.
- Marquez la protéine NP sur l’extrémité N-terminale avec His 6-SpyTag-TEV.
- Obtenez la séquence protéique de la base de données UniProt (code d’accès P0DTC9) et optimisez-la pour l’expression basée sur E. coli à l’aide de l’outil d’optimisation des codons IDT8. Commandez cette séquence pour la synthèse commerciale sous la forme d’un fragment d’ADN simple brin remis en suspension dans l’eau à 10 ng/μL.
REMARQUE : Le site de clivage de la protéase TEV et la balise His6 ne sont pas des caractéristiques essentielles dans la conception du LET.
- La PCR amplifie le fragment d’ADN avec le Ter FW universel (GGCTCCGAATAAGTATGTTGTAACTAAAGTGCGGCC
ACGATGCGTCCGGCGTAGAGGATCG) et Ter RV (CCGAGGCAATAAGTATGTTGTAACTAAAGTGCTCAG
CTTCCTTTCGGGCTTTGTTAGCAGCC) à l’aide du kit d’ADN polymérase haute fidélité (voir le tableau des matériaux)- Mettre en place une réaction de 100 μL comme suit : 75 μL d’eau exempte de nucléases, 20 μL de tampon d’ADN polymérase 5x, 2 μL de 10 mM de dNTPs (concentration finale de 200 μM), 0,5 μL d’amorce Ter-FW de 100 μM, 0,5 μL d’amorce Ter-RV de 100 μM, 1 μL de LET simple brin de 10 ng/μL, 1 μL(2U) d’ADN polymérase.
- Utiliser les paramètres PCR suivants : dénaturation initiale à 98 °C pendant 30 s ; 35 cycles : 98 °C pendant 6 s, 60 °C pendant 15 s, 72 °C pendant 90 s ; maintenir à 4 °C.
- Purifiez le produit PCR à l’aide d’un kit de purification PCR commercial, vérifiez sa qualité en analysant un échantillon sur un gel d’agarose à 1 % et mesurez sa concentration sur un spectrophotomètre UV-Vis.
REMARQUE : Les produits PCR bruts peuvent également être utilisés directement pour l’expression acellulaire. Cependant, la purification permet une procédure plus standardisée.

Figure 1 : Modèle d’expression linéaire pour le SARS-CoV-2-NP. Un schéma représentant les caractéristiques de la matrice d’ADN linéaire His-SpyTag-SARS-CoV-2 NP. Les éléments clés du modèle d’ADN sont étiquetés. La séquence codante de la protéine d’intérêt, ici la NP, est placée sous le contrôle transcriptionnel d’un promoteur T7 pour une expression efficace. À l’extrémité N-terminale, la protéine NP est ajoutée à un SpyTag pour une détection spécifique à l’aide du réactif de détection SpyCatcher2-Apex2. Les sites de protéase x6His-tag et TEV, bien qu’inclus dans la conception générale du LET, sont superflus aux fins de la CFDB. Aux extrémités de la matrice d’ADN linéaire, les sites Ter « en amont » et « en aval », chacun précédé de séquences tampons respectives de 50 paires de bases, sont inclus pour la protection médiée par Tus contre la dégradation de l’ADN exonucléolytique dans le lysat acellulaire. Abréviations : NP = protéine de nucléocapside ; LET = modèle d’expression linéaire ; TEV = virus de la gravure du tabac ; CFDB = tache de point acellulaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Purification de la protéine rapporteure SpyCatcher2-Apex2
- Utilisez le plasmide pET24b-SpyCatcher2-Apex2, construit à l’origine dans Norouzi et al.5 (séquence complète dans le fichier supplémentaire 2 et carte du plasmide dans la figure supplémentaire S1) pour préparer la protéine rapporteure SpyCatcher2-Apex-2.
- Préparez des plaques de gélose et un bouillon de lysogénie (LB) contenant 50 μg/mL de kanamycine. Transformez les cellules E . coli BL21 (DE3) avec le plasmide pET24b-SpyCatcher2-Apex2. Inoculer une seule colonie dans une culture de démarrage LB de 15 mL et cultiver pendant la nuit à 37 °C en agitant à 250 tr/min.
- Le lendemain, ajouter 10 mL de la culture de démarrage dans 500 mL de milieu frais contenant 50 μg/mL de kanamycine. Incuber à 37 °C avec agitation à 250 tr/min jusqu’à ce que la culture atteigne une densité optique à 600 nm de 0,6-0,8 (~3 h).
- Induire l’expression de SpyCatcher2-Apex2 en complétant la culture avec 0,5 mM d’isopropyl-β-D-1-thiogalactopyranoside et 1 mM de chlorhydrate d’acide 5-aminolévulinique. Réduire la température de croissance à 30 °C et laisser la culture incuber pendant encore 4 h.
- Récoltez les bactéries par centrifugation à 8 000 × g pendant 15 min. Procédez à la lyse cellulaire ou stockez la pastille à -80 °C jusqu’à ce qu’elle soit utilisée.
- Remettre la pastille en suspension dans 20 mL de tampon de lyse contenant 50 mM de Tris-HCl (pH 7,8), 300 mM de NaCl, 1 mg/mL de lysozyme, un comprimé d’inhibiteur de protéase sans EDTA et 1 mM de dithiothréitol (DTT). Lyse les cellules par sonication à 50 % d’amplitude avec des intervalles de 5 s ON et 10 s OFF pour un temps ON total de 3 min.
- Clarifier le lysat par centrifugation à 20 000 × g pendant 1 h à 4 °C. Passez le surnageant à travers un filtre à seringue de 0,2 μm.
- Ajouter du chlorure d’hémine à une concentration finale de 250 μM dans le lysat clarifié et incuber pendant une nuit à 4 °C. Procéder à la purification des protéines.
REMARQUE : L’incubation avec du chlorure d’hémine maximise l’incorporation de l’hème dans la peroxydase Apex-2 pour une activité enzymatique optimale. - Ajouter 2,5 mL de résine de Ni dans le lysat clarifié et incuber à 4 °C en agitant doucement pendant 45 min. Appliquez le mélange sur une colonne d’écoulement par gravité et lavez la résine avec 50 mL de tampon Tris (50 mM de Tris-HCl (pH 7,8), 300 mM de NaCl et 1 mM de DTT).
- Éluer la protéine SpyCatcher2-Apex2 dans 25 mL de tampon Tris contenant 400 mM d’imidazole. Le concentré et le tampon échangent l’éluat dans le tampon Tris à l’aide d’une unité de filtration centrifuge, en visant un volume final de 0,5 à 1,0 mL.
- Utilisez le coefficient d’extinction molaire de SpyCatcher2-Apex2 (27 390 M-1 cm-1) pour déterminer la concentration en protéines sur un spectrophotomètre UV-Vis. Ajouter le glycérol à une concentration finale de 40 % et stocker les aliquotes à -20 °C.
REMARQUE : Ce protocole devrait produire environ 40 mg de SpyCatcher2-Apex2 hautement pur et actif, ce qui est suffisant pour 400 analyses CFDB sur une taille de transfert standard de 96 échantillons. - Pour la détection des protéines marquées SpyTagged à l’aide du western blot9 conventionnel et pour les expériences CFDB, toujours bloquer les membranes dans du lait écrémé en poudre à 5 % dans 1 solution saline tamponnée au Tris contenant 0,05 % de Tween-20 (TBST). Utilisez la protéine SpyCatcher2-Apex2 à une concentration finale de 10 μg/mL dans une solution bloquante.
REMARQUE : Le rapporteur SpyCatcher2-Apex2 nécessite une solution de chimiluminescence améliorée (ECL) pour le développement du signal. La solution ECL peut être obtenue dans le commerce ou préparée en interne selon les instructions de Mruk et al.10. La solution finale d’ECL se compose ici de 0,4 mM d’acide p-coumarique, de 2,5 mM de luminol et de 0,015 % de H2O2 dans 100 mM de Tris-HCl (pH 8,6).
3. Production acellulaire et contrôle de la qualité des antigènes
- Préparez et assemblez un lysat d’expression acellulaire d’E . coli BL21 et des composants de réaction en suivant les instructions de Levine et al.11 et Norouzi et al.5. Compléter le mélange réactionnel final avec 5 μM de protéine Tus et 1,2 μM d’ARN polymérase T7 comme suit pour une réaction représentative de 100 μL : 14,6 μL de solution A, 14 μL de solution B, 33,3 μL de lysat d’E. coli BL21, 2,5 μL de protéine Tus 200 mM, 1,2 μL d’ARN polymérase 100 mM T7, 1 μL de matrice d’ADN linéaire de 1,5 μM, 33,4 μL d’eau sans nucléases.
REMARQUE : Voir le fichier supplémentaire 3 pour des instructions clés sur la préparation d’un lysat acellulaire d’E . coli BL21 et le tableau supplémentaire S1 pour une recette détaillée des solutions A et B. Le système d’expression acellulaire d’E . coli peut également être préparé à l’aide d’autres protocoles ou obtenu commercialement, à condition que la réaction soit complétée par de l’ARN polymérase Tus et T7. - Effectuez un test d’expression initial à l’échelle de 5 μL en ajoutant 10 % (v/v) de produit PCR brut à la réaction acellulaire dans un tube de PCR. Incuber sans agiter à 30 °C pendant 15 h.
- Vérifiez la qualité d’expression de l’antigène NP en chargeant 1 μL de la réaction acellulaire sur un gel de polyacrylamide de dodécylsulfate de sodium à 12 % (SDS-PAGE), puis en le transférant sur une membrane de nitrocellulose pour le transfert Western, comme décrit à l’étape 2.12. Utilisez la protéine rapporteure SpyCatcher2-Apex2 pour le marquage par transfert via le SpyTag, et éventuellement, un anticorps commercial spécifique (ici un anti-SARS-CoV-2-NP) contre l’antigène cible.
- Assembler une réaction acellulaire à l’échelle de 1 mL avec un produit LET purifié de 15 nM à utiliser pour le test CFDB. Incuber le mélange d’expression dans un tube conique de 15 mL en agitant à 80 tr/min pendant 15 h à 30 °C. Vérifier la qualité de l’expression à l’aide du transfert Western et stocker les aliquotes de 50 μL à -20 °C.
4. Échantillons de sérum
- Obtenir des échantillons de sérum (ou de plasma) humain/animal auprès de sources commerciales, de collaborateurs cliniques ou à l’interne en utilisant les procédures standard et en suivant les lignes directrices du comité d’éthique de la recherche humaine de l’établissement ou du comité sur le soin et l’utilisation des animaux. S’assurer que des mesures appropriées de prétraitement des échantillons ont été prises pour atténuer les risques de contamination.
REMARQUE : Les mesures de prétraitement peuvent inclure un traitement au solvant-détergent, l’inactivation de la chaleur et l’analyse des marqueurs de virus transmissibles par le sang. Il existe un protocole utile pour l’isolement du sérum et du plasma à partir du sang total12. Comme pour d’autres plateformes d’immunoessais, les échantillons de sérum prélevés tôt (<3 semaines) après l’infection initiale ou le début de la maladie ne devraient pas contenir des niveaux adéquats de réponse anticorps et leurs résultats doivent être traités avec prudence. - Pour la vérification de la qualité des réactifs SARS-CoV-2 NP CFDB, utilisez le panel de référence international de l’Organisation mondiale de la santé (OMS) du National Institute for Biological Standards and Control (NIBSC) pour l’immunoglobuline anti-SARS-CoV-2, qui contient x1 échantillon pré-COVID-19 et x4 échantillons positifs au SARS-CoV-2 avec des niveaux variables d’immunoglobulines anti-NP13. Alternativement, utilisez des échantillons de sérum sains et positifs précaractérisés provenant d’autres sources.
- Pour les expériences CFDB, obtenir ou préparer un échantillon de contrôle négatif en regroupant plusieurs (>3) échantillons de sérum sain afin d’augmenter la précision et de faciliter l’analyse et l’interprétation des résultats.
5. Procédure de transfert de points acellulaire (CFDB)
- Téléchargez et imprimez le fichier image de la grille maîtresse (figure supplémentaire S2). La grille maîtresse est un motif de 6 x 6 cm contenant 12 x 12 cercles de 2 mm de diamètre chacun et offre une capacité de repérage similaire à celle d’une plaque à 96 puits.
- Placez la grille en sandwich entre deux couches de film d’étanchéité adhésif pour plaque PCR et coupez-la sur mesure le long des bords extérieurs de la grille. À l’aide d’un poinçon de biopsie de 2 mm, vous pouvez creuser chaque cercle marqué.
REMARQUE : Cette grille maîtresse peut être réutilisée plusieurs fois après avoir été essuyée avec de l’éthanol à 70 %. - Coupez un morceau de membrane nitrocellulosique de 6,5 x 6,5 cm et placez-le sous la grille maîtresse sur une surface propre, en fixant l’installation à l’aide de ruban adhésif, comme illustré à la figure 2 et à la figure supplémentaire S3. À l’aide d’un marqueur, marquez les positions des cercles les plus externes sur la membrane de nitrocellulose afin de les guider pour couper la membrane après le repérage de l’échantillon.
- Diluer les échantillons de sérum 1/10 dans 1x solution saline tamponnée au phosphate (pH 7,4). À l’aide d’une micropipette, versez des volumes triples de 0,4 μL de chaque échantillon sur la membrane de nitrocellulose à des positions de grille prédéterminées. Il faudra environ 15 s par point pour distribuer.
REMARQUE : Assurez-vous d’inclure des échantillons de contrôle négatifs et positifs avec chaque essai, car cela sera nécessaire pour l’analyse des résultats. - Attendez 10 min à température ambiante pour que les échantillons repérés se lient et sèchent. À l’aide d’une pince à épiler, récupérez soigneusement la membrane de nitrocellulose et coupez le long des cercles extérieurs marqués.
- Bloquer la membrane dans 10 mL de solution bloquante (5 % de lait écrémé en poudre dans du TBST) dans une boîte de Pétri de 10 cm pendant 30 min à température ambiante, en agitant doucement à 100 tr/min.
- Décongeler et ajouter une aliquote de 50 μL du mélange d’expression d’antigène acellulaire à 5 mL de solution bloquante dans une boîte de Pétri de 10 cm. Transférez la membrane directement dans cette solution contenant de l’antigène et incubez à température ambiante pendant 1 h, en agitant à 100 tr/min.
REMARQUE : Il s’agit de l’étape de détection primaire au cours de laquelle l’antigène SpyTagged se liera aux positions ponctuelles contenant des anticorps, le cas échéant. - Rincez la membrane, lavez-la pendant 5 minutes et rincez-la à nouveau au TBST avant de passer à l’étape de détection secondaire.
- Incuber la membrane dans 10 mL de tampon de blocage contenant 10 μg/mL de protéine SpyCatcher2-Apex2 purifiée, pendant 1 h tout en agitant à 100 tr/min.
- Rincez la membrane et lavez 2 x 5 min en TBST, avec un rinçage final en TBS.
- Collez et fixez un morceau de parafilm sur une zone de travail propre près de l’instrument d’imagerie par transfert. Retirez l’excès de liquide en tapotant la membrane à l’aide d’une pince à épiler et placez la membrane sur le parafilm.
- Ajouter immédiatement 3 ml (100 μL/cm2) de solution ECL sur la membrane et incuber à température ambiante pendant exactement 90 s.
- Séchez immédiatement la membrane par tapotement et transférez-la dans un instrument d’imagerie compatible avec la chimiluminescence pour la visualisation des résultats.
REMARQUE : Le temps d’imagerie optimal peut varier selon les instruments. Il est recommandé d’utiliser les paramètres de temps d’acquisition automatique par défaut de l’instrument et, en cas d’absence, d’utiliser des échantillons de sérum précaractérisés pour optimiser le temps d’acquisition de l’image. - Utilisez la fonction d’analyse d’images de l’instrument pour obtenir des intensités ponctuelles, y compris pour trois positions blanches (arrière-plan) sur la membrane de nitrocellulose ; Maintenir un volume de mesure constant par point. Exportez les données vers une feuille de calcul, en n’oubliant pas d’étiqueter correctement chaque position de spot.
- Calculez la moyenne et l’écart-type (ET) des intensités ponctuelles triples. Ensuite, soustrayez le fond de la membrane de nitrocellulose de tous les échantillons.
- Utilisez l’équation suivante pour obtenir une valeur seuil pour l’interprétation des résultats -/+ :
(Moyenne des témoins négatifs) + (3 x écart-type des témoins négatifs). Considérons les échantillons positifs si leur intensité moyenne est supérieure à la valeur seuil et négatifs si leur intensité moyenne est inférieure à la valeur seuil.
REMARQUE : Un schéma de la procédure CFDB est fourni à la figure 3.
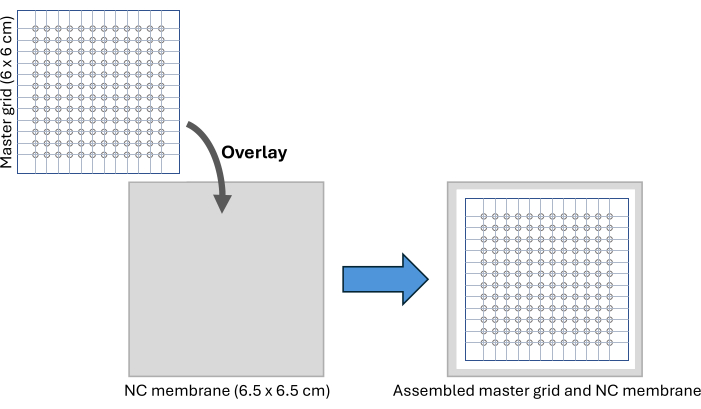
Figure 2 : Assemblage de la CFDB. Schéma de la configuration de la grille maîtresse CFDB et de l’assemblage de la membrane CN. La grille maîtresse est superposée sur la membrane NC pour fournir un motif régulier et adressable pour le repérage et l’immobilisation des échantillons de sérum. Abréviations : CFDB = tache de point acellulaire ; NC = nitrocellulose. Veuillez cliquer ici pour voir une version agrandie de cette figure.
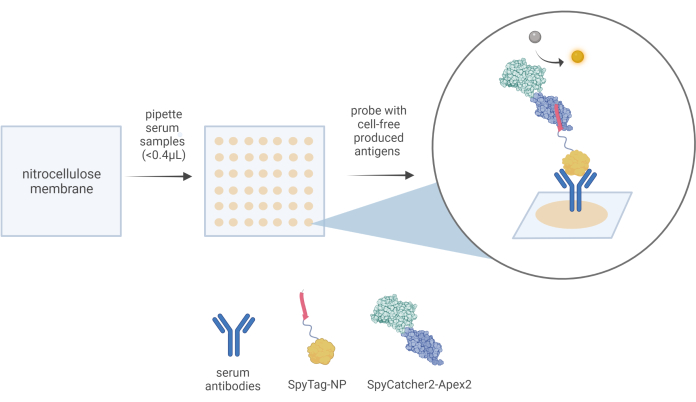
Figure 3 : Représentation schématique du flux de travail CFDB. Dans un test CFDB, une petite quantité (<0,4 μL) d’échantillons de sérum 10x dilués est distribuée manuellement sur une membrane de nitrocellulose prédécoupée (panneau de gauche) à des endroits discrets et adressables (panneau du milieu). Déposer un échantillon de sérum par point en trois taches et immobiliser le contenu protéique, y compris le réservoir total d’anticorps du sérum, sur le substrat NC solide (taches beiges dans le panneau central). Dans cet exemple, les anticorps anti-NP contenus dans les échantillons de sérum peuvent être d’abord liés par le réactif de détection primaire CFDB SpyTag-NP et enfin détectés par le réactif de détection secondaire CFDB SpyCatcher2-Apex2 (bulle agrandie dans le panneau de droite). Cette figure est tirée de Norouzi et al.5. Abréviations : CFDB = tache de point acellulaire ; NP = protéine de nucléocapside ; LET = modèle d’expression linéaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Amplification par PCR du modèle d’expression linéaire de l’antigène cible
Pour amplifier par PCR le SARS-CoV-2 NP LET, des amorces Ter directes et inverses universelles ont été utilisées comme décrit dans la section 1.2 du protocole et 1 μL du produit a été vérifié sur un gel d’agarose (Figure 4) avant de procéder à la purification du produit PCR.
<...
Discussion
La COVID-19 a mis en évidence l’importance de diagnostics accessibles et robustes pour contrôler les épidémies d’infection et optimiser les stratégies de santé mondiale. Les tests sérologiques qui détectent les anticorps protecteurs se sont avérés essentiels pour suivre les profils de transmissibilité des nouveaux variants, identifier les points chauds, guider la mise au point d’un vaccin, trier les cas suspects et protéger les populations vulnérables...
Déclarations de divulgation
M.N. et K.P. sont les co-inventeurs de la méthode du transfert de points acellulaires. Une demande de brevet provisoire liée à ces travaux a été déposée (PCT/CA2024/050097, déposée en janvier 2024).
Remerciements
S.S. et R.Z. sont soutenus par un financement de la Defense Advanced Research Projects Agency (DARPA), contrat n° N66001-23-2-4042. Les points de vue, opinions et/ou conclusions exprimés sont ceux des auteurs et ne doivent pas être interprétés comme représentant les points de vue ou les politiques officiels du ministère de la Défense ou du gouvernement américain. Ces travaux ont été financés par des fonds versés à K.P. dans le cadre du programme de subventions de la Fondation des IRSC (201610FDN-375469), du Programme des chaires de recherche du Canada des IRSC (950-231075 et 950-233107), de l’initiative Medicine by Design de l’Université de Toronto, qui reçoit des fonds du Fonds d’excellence en recherche Apogée Canada, et des fonds versés à K.P., du Programme canadien pour la sûreté et la sécurité de Recherche et développement pour la défense Canada (contrat 39903-200137). La figure 1 et la figure supplémentaire S1 ont été créées à l’aide de SnapGene Viewer.
matériels
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
Références
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722 (2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- . IDT codon optimization tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2025)
- . General protocol for western blotting Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025)
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- . Plasma and serum preparation Available from: https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025)
- Mattiuzzo, G., et al. . WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405 (2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon