È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cell-Free Dot Blot come piattaforma di immunodosaggio pratica e adattabile per il rilevamento della risposta anticorpale in sieri umani e animali
In questo articolo
Riepilogo
Descriviamo una piattaforma di immunodosaggio di recente sviluppo basata sui principi della biologia sintetica cell-free e sulla tecnica dot-blot per il rilevamento personalizzabile della risposta anticorpale in sieri umani e animali.
Abstract
La serie di focolai patogeni globali negli ultimi due decenni ha evidenziato l'importanza delle strategie di sierosorveglianza. Le piattaforme di immunodosaggio che servono a rilevare gli anticorpi specifici della malattia nei sieri dei pazienti sono al centro della sierosorveglianza. Esempi comuni includono i saggi di immunoassorbimento enzimatico e i saggi a flusso laterale; Tuttavia, sebbene si tratti di metodi gold standard, richiedono materiali di consumo specifici per agenti patogeni e attrezzature specializzate, il che ne limita l'uso al di fuori di laboratori dotati di risorse adeguate.
Di recente abbiamo sviluppato una nuova piattaforma di immunodosaggio chiamata Cell-Free Dot-Blot (CFDB) e l'abbiamo convalidata utilizzando sieri umani e animali contro SARS-CoV-2. A differenza dei dosaggi immunologici convenzionali, i campioni di siero dei pazienti CFDB vengono immobilizzati in una fase solida (membrana di nitrocellulosa), mentre l'antigene bersaglio viene sospeso nella fase mobile del test. Per migliorare l'accesso alle capacità di sierosorveglianza, gli antigeni CFDB sono prodotti su richiesta e con un'infrastruttura a basso carico utilizzando l'espressione proteica in vitro . Qui, l'antigene viene fuso con un tag peptidico che può essere rilevato utilizzando una singola proteina reporter universale per qualsiasi test CFDB. Il risultato è che il CFDB non richiede l'accesso a un lettore di piastre multipozzetto o a componenti di saggi molecolari commerciali purificati. Con queste considerazioni di progettazione, CFDB affronta le carenze delle piattaforme di immunodosaggio esistenti fornendo accessibilità a laboratori non centralizzati, adattabilità per i patogeni emergenti e convenienza per le comunità a basso reddito.
In questo articolo, forniremo un protocollo passo dopo passo per preparare ed eseguire un test immunologico CFDB. Utilizzando il nostro recente lavoro su SARS-CoV-2 CFDB come esempio, tratteremo la progettazione del DNA dell'antigene per la produzione senza cellule su richiesta, seguita dalla preparazione della proteina reporter CFDB, dall'immobilizzazione dei campioni di siero sulla fase solida e, infine, dalle fasi di legame e rilevamento dell'antigene del test. Prevediamo che, seguendo queste istruzioni, i ricercatori saranno in grado di adattare il test CFDB per rilevare le risposte immunitarie nei sieri umani e animali a qualsiasi agente patogeno.
Introduzione
La pandemia di COVID-19 ha rivelato la necessità critica di strumenti diagnostici convenienti e scalabili, in particolare per contesti con risorse limitate1. I test immunologici convenzionali, come i test di immunoassorbimento enzimatico (ELISA), si sono dimostrati essenziali per rilevare le risposte immunitarie 2,3. Tuttavia, il loro costo elevato, la dipendenza da reagenti complessi e la dipendenza da attrezzature specializzate ne limitano l'accessibilità, soprattutto durante le crisi sanitarie globali. In risposta a queste sfide, abbiamo sviluppato il Cell-Free Dot Blot (CFDB), una piattaforma di immunodosaggio a basso costo e adattabile, progettata per la rilevazione di anticorpi anti-SARS-CoV-2 in sieri umani e animali.
CFDB sfrutta la biologia sintetica senza cellule per la produzione rapida e on-demand di antigeni virali utilizzando modelli di DNA lineari 4,5. Ciò elimina la necessità dei tradizionali processi di clonazione, espressione e purificazione basati su cellule, accelerando significativamente la produzione di antigeni e riducendo i costi. Il metodo CFDB semplifica il rilevamento degli anticorpi utilizzando un formato dot blot, in cui i sieri vengono individuati direttamente sulle membrane di nitrocellulosa. Questo sistema elimina la necessità di costose piastre multipozzetto e attrezzature di laboratorio specializzate, consentendo un semplice flusso di lavoro di "immersione" per le fasi di incubazione e lavaggio. La piattaforma utilizza anche un sistema SpyCatcher-SpyTag, in cui una chimera di perossidasi SpyCatcher2-Apex2 funge da reagente di rilevamento secondario universale 5,6. Questo è prodotto utilizzando l'espressione standard a base di Escherichia coli, che elimina la dipendenza dai costosi coniugati anticorpali commerciali. Di conseguenza, il sistema CFDB è in grado di eseguire saggi sierologici con prestazioni paragonabili a quelle degli ELISA a un costo significativamente inferiore, circa 3 dollari per 96 campioni rispetto agli oltre 300 dollari diun kit ELISA 5 commerciale.
Per dimostrare l'efficacia di CFDB, abbiamo testato la sua capacità di rilevare gli anticorpi in sieri umani e animali precaratterizzati. I nostri risultati sono strettamente correlati all'ELISA nell'identificazione di campioni positivi e negativi al COVID-19. Oltre alla diagnostica umana, abbiamo valutato l'utilità di CFDB in modelli animali, testando sieri di criceti infetti da SARS-CoV-2 e di quelli vaccinati con la proteina Nucleocapside ricombinante. Questi test hanno confermato il potenziale di CFDB per l'uso sia nella diagnostica umana che veterinaria, rendendolo uno strumento versatile per il monitoraggio delle risposte immunitarie tra le specie. Uno dei principali vantaggi di CFDB è la sua flessibilità. Semplicemente modificando il modello di DNA che codifica l'antigene di interesse, la piattaforma può essere rapidamente adattata per rilevare gli anticorpi contro diversi agenti patogeni, rendendola preziosa per la preparazione alle future pandemie. Il suo basso costo, il flusso di lavoro semplice e i requisiti minimi dell'infrastruttura lo rendono particolarmente adatto per laboratori decentralizzati e ambienti con poche risorse, dove l'accesso alla diagnostica commerciale è limitato.
In questo lavoro forniremo istruzioni dettagliate per la preparazione e l'esecuzione di un test CFDB. In primo luogo, ci occupiamo della progettazione e della sintesi di modelli lineari di DNA per la produzione di antigeni privi di cellule, che sono i reagenti di rilevamento primari del test. Descriviamo quindi le fasi per la preparazione del reagente di rilevamento secondario del test SpyCatcher2-Apex2. Successivamente, forniamo istruzioni per la produzione senza cellule e il controllo di qualità degli antigeni stessi. Infine, descriviamo in dettaglio il processo per condurre un test CFDB su campioni di siero umano o animale.
Protocollo
Tutti gli esperimenti sui criceti sono stati eseguiti presso il National Microbiology Laboratory (NML) presso l'Agenzia per la salute pubblica del Canada, approvati dal Canadian Science Centre for Human and Animal Health e seguendo le linee guida del Canadian Council on Animal Care. Tutti i campioni di siero/plasma umano sono stati ottenuti commercialmente per test interni o forniti da collaboratori clinici all'NML per test indipendenti presso l'NML.
1. Progettazione e preparazione di pattern di espressione lineare (LET) dell'antigene
- Progettare i LET di espressione cell-free per l'antigene bersaglio sulla base delle istruzioni di Norouzi et al.7 e contenenti uno SpyTag N/C-terminale come descritto in Norouzi et al.5.
NOTA: Qui forniamo i dettagli per la preparazione della proteina nucleocapside SARS-CoV-2 (NP, aminoacidi 2-419).- Assicurarsi che il LET per il SARS-CoV-2 NP (Figura 1 e File supplementare 1) contenga siti Ter 5' e 3' con una sequenza di tamponi di 50 coppie di basi prima del promotore T7 e dopo il codone di stop.
- Etichettare la proteina NP sull'N-terminale con il suo 6-SpyTag-TEV.
- Ottenere la sequenza proteica dal database UniProt (codice di accesso P0DTC9) e ottimizzarla per l'espressione basata su E. coli utilizzando lo strumento di ottimizzazione del codone IDT8. Ordina questa sequenza per la sintesi commerciale come un frammento di DNA a filamento singolo risospeso in acqua a 10 ng/μL.
NOTA: Il sito di scissione della proteasi TEV e il tag His6 non sono caratteristiche essenziali nel progetto LET.
- La PCR amplifica il frammento di DNA con il Ter FW universale (GGCTCCGAATAAGTATGTTGTAACTAAAGTGCGGCC
ACGATGCGTCCGGCGTAGAGGATCG) e Ter RV (CCGAGGCAATAAGTATGTTGTAACTAAAGTGCTCAG
CTTCCTTTCGGGCTTTGTTAGCAGCC) utilizzando il kit DNA polimerasi ad alta fedeltà (vedere la Tabella dei materiali)- Impostare una reazione da 100 μL come segue: 75 μL di acqua priva di nucleasi, 20 μL di tampone DNA polimerasi 5x, 2 μL di dNTP da 10 mM (concentrazione finale 200 μM), 0,5 μL di primer Ter-FW da 100 μM, 0,5 μL di primer Ter-RV da 100 μM, 1 μL di LET a filamento singolo da 10 ng/μL, 1 μL(2U) di DNA polimerasi.
- Utilizzare le seguenti impostazioni PCR: denaturazione iniziale a 98 °C per 30 s; 35 cicli: 98 °C per 6 s, 60 °C per 15 s, 72 °C per 90 s; mantenere a 4 °C.
- Purificare il prodotto PCR utilizzando un kit di purificazione PCR commerciale, verificarne la qualità eseguendo un campione su un gel di agarosio all'1% e misurarne la concentrazione su uno spettrofotometro UV-Vis.
NOTA: I prodotti grezzi della PCR possono anche essere utilizzati direttamente per l'espressione libera da cellule. Tuttavia, la purificazione consente una procedura più standardizzata.

Figura 1: Modello di espressione lineare per SARS-CoV-2-NP. Uno schema che rappresenta le caratteristiche del modello di DNA lineare His-SpyTag-SARS-CoV-2 NP. Gli elementi chiave del modello DNA sono etichettati. La sequenza codificante per la proteina di interesse, in questo caso la NP, è posta sotto il controllo trascrizionale di un promotore T7 per un'espressione efficiente. All'N-terminale, alla proteina NP viene aggiunto uno SpyTag per la rilevazione specifica utilizzando il reagente di rilevamento SpyCatcher2-Apex2. I siti x6His-tag e TEV proteasici, sebbene inclusi come parte del progetto generale LET, sono superflui ai fini del CFDB. Ai termini del modello lineare di DNA, i siti Ter "a monte" e "a valle", ciascuno preceduto da rispettive 50 sequenze tampone di 50 coppie di basi, sono inclusi per la protezione mediata da Tu contro la degradazione del DNA esonucleolitico nel lisato libero da cellule. Abbreviazioni: NP = proteina nucleocapside; LET = modello di espressione lineare; TEV = virus dell'incisione del tabacco; CFDB = macchia a punti senza cellule. Clicca qui per visualizzare una versione più grande di questa figura.
2. Purificazione della proteina reporter SpyCatcher2-Apex2
- Utilizzare il plasmide pET24b-SpyCatcher2-Apex2, originariamente costruito in Norouzi et al.5 (sequenza completa nel file supplementare 2 e mappa plasmidica nella figura supplementare S1) per preparare la proteina reporter SpyCatcher2-Apex-2.
- Preparare piastre di agar e brodo di lisogenia (LB) contenente 50 μg/mL di kanamicina. Trasformare le cellule BL21 (DE3) di E. coli con il plasmide pET24b-SpyCatcher2-Apex2. Inoculare una singola colonia in una coltura LB starter da 15 ml e far crescere durante la notte a 37 °C con agitazione a 250 giri/min.
- Il giorno successivo, aggiungere 10 mL di coltura starter in 500 mL di terreno LB fresco contenente 50 μg/mL di kanamicina. Incubare a 37 °C agitando a 250 giri/min fino a quando la coltura raggiunge una densità ottica a 600 nm di 0,6-0,8 (~3 h).
- Indurre l'espressione di SpyCatcher2-Apex2 integrando la coltura con 0,5 mM di isopropil-β-D-1-tiogalattopiranoside e 1 mM di acido 5-aminolevulinico cloridrato. Ridurre la temperatura di crescita a 30 °C e lasciare incubare la coltura per altre 4 ore.
- Raccogliere i batteri mediante centrifugazione a 8.000 × g per 15 minuti. Procedere alla lisi cellulare o in alternativa conservare il pellet a -80 °C fino al momento dell'utilizzo.
- Risospendere il pellet in 20 mL di tampone di lisi contenente 50 mM di Tris-HCl (pH 7,8), 300 mM di NaCl, 1 mg/mL di lisozima, una compressa di inibitore della proteasi priva di EDTA e 1 mM di ditiotreitolo (DTT). Lisare le cellule mediante sonicazione al 50 % di ampiezza con intervalli di 5 s ON e 10 s OFF per un tempo totale di ON di 3 minuti.
- Chiarificare il lisato mediante centrifugazione a 20.000 × g per 1 ora a 4 °C. Passare il surnatante attraverso un filtro per siringa da 0,2 μm.
- Aggiungere cloruro di emina a una concentrazione finale di 250 μM al lisato chiarificato e incubare per una notte a 4 °C. Procedere alla purificazione delle proteine.
NOTA: L'incubazione con cloruro di emina massimizza l'incorporazione dell'eme nella perossidasi Apex-2 per un'attività enzimatica ottimale. - Aggiungere 2,5 mL di resina di Ni al lisato chiarificato e incubare a 4 °C agitando delicatamente per 45 minuti. Applicare la miscela su una colonna di flusso per gravità e lavare la resina con 50 mL di tampone Tris (50 mM Tris-HCl (pH 7,8), 300 mM NaCl e 1 mM DTT).
- Eluire la proteina SpyCatcher2-Apex2 in 25 mL di tampone Tris contenente 400 mM di imidazolo. Il concentrato e il tampone scambiano l'eluato in tampone Tris utilizzando un'unità di filtro centrifuga, con l'obiettivo di ottenere un volume finale di 0,5-1,0 mL.
- Utilizzare il coefficiente di estinzione molare di SpyCatcher2-Apex2 (27.390 M-1 cm-1) per determinare la concentrazione proteica su uno spettrofotometro UV-Vis. Aggiungere glicerolo ad una concentrazione finale del 40% e conservare le aliquote a -20 °C.
NOTA: Si prevede che questo protocollo produca circa 40 mg di SpyCatcher2-Apex2 altamente puro e attivo, sufficienti per 400 esecuzioni CFDB su una dimensione standard di 96 campioni di blot. - Per il rilevamento di proteine SpyTagged utilizzando il western blot9 convenzionale e per gli esperimenti CFDB, bloccare sempre le membrane nel latte in polvere scremato al 5% in 1x soluzione salina tamponata con Tris contenente lo 0,05% di Tween-20 (TBST). Utilizzare la proteina SpyCatcher2-Apex2 a una concentrazione finale di 10 μg/mL in soluzione bloccante.
NOTA: Il reporter SpyCatcher2-Apex2 richiede una soluzione di chemiluminescenza avanzata (ECL) per lo sviluppo del segnale. La soluzione ECL può essere ottenuta in commercio o preparata internamente secondo le istruzioni di Mruk et al.10. La soluzione finale di ECL è composta da 0,4 mM di acido p-cumarico, 2,5 mM di luminolo e 0,015% di H2O2 in 100 mM di Tris-HCl (pH 8,6).
3. Produzione cell-free e controllo di qualità degli antigeni
- Preparare e assemblare un lisato di espressione cellulare libero BL21 di E. coli BL21 e i componenti di reazione seguendo le istruzioni in Levine et al.11 e Norouzi et al.5. Integrare la miscela di reazione finale con 5 μM di proteina Tus e 1,2 μM di RNA polimerasi T7 come segue per una reazione rappresentativa di 100 μL: 14,6 μL di soluzione A, 14 μl di soluzione B, 33,3 μl di lisato BL21 di E. coli , 2,5 μl di proteina Tus 200 mM, 1,2 μl di RNA polimerasi T7 da 100 mM, 1 μL di stampo di DNA lineare da 1,5 μM, 33,4 μL di acqua priva di nucleasi.
NOTA: Vedere il file supplementare 3 per le istruzioni chiave sulla preparazione di un lisato libero da cellule BL21 di E. coli e la tabella supplementare S1 per una ricetta dettagliata delle soluzioni A e B. Il sistema di espressione libera da cellule di E. coli può anche essere preparato utilizzando protocolli alternativi o ottenuto commercialmente, purché la reazione sia integrata con Tus e RNA polimerasi T7. - Eseguire un test iniziale di espressione su scala di 5 μL aggiungendo il 10% (v/v) di prodotto PCR grezzo alla reazione priva di cellule in una provetta per PCR. Incubare senza agitare a 30 °C per 15 ore.
- Verificare la qualità dell'espressione dell'antigene NP caricando 1 μL della reazione libera da cellule su un gel di poliacrilammide dodecil solfato di sodio al 12% (SDS-PAGE) e successivamente trasferirlo su una membrana di nitrocellulosa per il western blot come descritto al punto 2.12. Utilizzare la proteina reporter SpyCatcher2-Apex2 per la marcatura dei blot tramite SpyTag e, facoltativamente, un anticorpo commerciale specifico (in questo caso un anti-SARS-CoV-2-NP) contro l'antigene bersaglio.
- Assemblare una reazione priva di cellule su scala da 1 mL con prodotto LET purificato da 15 nM da utilizzare per il test CFDB. Incubare la miscela di espressione in una provetta conica da 15 mL agitando a 80 giri/min per 15 ore a 30 °C. Controllare la qualità dell'espressione utilizzando il western blot e conservare aliquote da 50 μl a -20 °C.
4. Campioni di siero
- Ottenere campioni di siero (o plasma) umano/animale da fonti commerciali o collaboratori clinici o internamente utilizzando procedure standard e seguendo le linee guida del comitato etico istituzionale per la ricerca umana o del comitato per la cura e l'uso degli animali. Assicurarsi che siano state adottate adeguate misure di pretrattamento dei campioni per attenuare i rischi di contaminazione.
NOTA: Le misure di pretrattamento possono includere il trattamento solvente-detergente, l'inattivazione del calore e l'analisi dei marcatori virali trasmissibili per via ematica. È disponibile un protocollo utile per l'isolamento del siero e del plasma dal sangue intero12. Come con altre piattaforme di immunodosaggio, i campioni di siero raccolti precocemente (<3 settimane) dopo l'infezione iniziale o l'insorgenza della malattia non dovrebbero contenere livelli adeguati di risposta anticorpale e i loro risultati devono essere trattati con cautela. - Per la verifica della qualità dei reagenti SARS-CoV-2 NP CFDB, utilizzare il pannello di riferimento internazionale dell'Organizzazione mondiale della sanità (OMS) del National Institute for Biological Standards and Control (NIBSC) per l'immunoglobulina anti-SARS-CoV-2, che contiene campioni x1 pre-COVID-19 e x4 positivi al SARS-CoV-2 con livelli variabili di immunoglobuline anti-NP13. In alternativa, utilizzare campioni di siero sani e positivi precaratterizzati provenienti da altre fonti.
- Per gli esperimenti CFDB, ottenere o preparare un campione di controllo negativo raggruppando più (>3) campioni di siero sano per aumentare l'accuratezza e facilitare l'analisi e l'interpretazione dei risultati.
5. Procedura CFDB (Cell-Free Dot Blot)
- Scaricare e stampare il file di immagine della griglia master (Figura S2 supplementare). La griglia principale è un modello di 6 x 6 cm contenente 12 x 12 cerchi di 2 mm di diametro ciascuno e fornisce una capacità di spotting simile a una piastra a 96 pozzetti.
- Inserire saldamente la griglia tra due strati di pellicola sigillante adesiva per piastre PCR e tagliarla a misura lungo i bordi esterni della griglia. Utilizzare un punzone per biopsia da 2 mm per incavare ogni cerchio contrassegnato.
NOTA: Questa griglia principale può essere riutilizzata più volte dopo la pulizia con etanolo al 70%. - Tagliare un pezzo di membrana in nitrocellulosa di 6,5 x 6,5 cm e posizionarlo sotto la griglia principale su una superficie pulita, fissando l'installazione con nastro adesivo come mostrato nella Figura 2 e nella Figura S3 supplementare. Utilizzare un pennarello per contrassegnare le posizioni del cerchio più esterno sulla membrana di nitrocellulosa da utilizzare come guida per tagliare la membrana dopo l'individuazione del campione.
- Diluire i campioni di siero 1/10 in 1x soluzione salina tamponata con fosfato (pH 7,4). Utilizzare una micropipetta per erogare volumi triplicati di 0,4 μl di ciascun campione sulla membrana in nitrocellulosa in posizioni predeterminate della griglia. Ci vorranno circa 15 secondi per punto per l'erogazione.
NOTA: Assicurarsi di includere sia i campioni di controllo negativi che quelli positivi con ogni test, poiché ciò sarà necessario per l'analisi dei risultati. - Attendere 10 minuti a temperatura ambiente affinché i campioni macchiati si leghino e si asciughino. Usando una pinzetta, recupera con cura la membrana di nitrocellulosa e taglia lungo i cerchi esterni contrassegnati.
- Bloccare la membrana in 10 mL di soluzione bloccante (5% di latte scremato in polvere in TBST) in una piastra di Petri da 10 cm per 30 minuti a temperatura ambiente, agitando delicatamente a 100 giri/min.
- Scongelare e aggiungere 50 μl di aliquota della miscela di espressione dell'antigene libero da cellule a 5 mL di soluzione bloccante in una piastra di Petri da 10 cm. Trasferire la membrana direttamente in questa soluzione contenente antigene e incubare a temperatura ambiente per 1 ora, agitando a 100 giri/min.
NOTA: Questa sarà la fase di rilevamento principale in cui l'antigene SpyTagged si legherà alle posizioni dei punti contenenti anticorpi, se presenti. - Sciacquare la membrana, lavare per 5 minuti e risciacquare nuovamente in TBST prima di procedere alla fase di rilevamento secondaria.
- Incubare la membrana in 10 mL di tampone bloccante contenente 10 μg/mL di proteina purificata SpyCatcher2-Apex2, per 1 ora agitando a 100 giri/min.
- Risciacquare la membrana e lavare 2 x 5 min in TBST, con un risciacquo finale in TBS.
- Incollare e fissare un pezzo di parafilm su un'area di lavoro pulita vicino allo strumento di imaging blot. Rimuovere il liquido in eccesso picchiettando la membrana con una pinzetta e posizionare la membrana sopra il parafilm.
- Aggiungere immediatamente 3 mL (100 μL/cm2) di soluzione ECL sulla parte superiore della membrana e incubare a temperatura ambiente per esattamente 90 s.
- Asciugare immediatamente la membrana e trasferirla a uno strumento di imaging compatibile con la chemiluminescenza per la visualizzazione dei risultati.
NOTA: Il tempo di imaging ottimale può variare a seconda degli strumenti. Si consiglia di utilizzare le impostazioni predefinite del tempo di acquisizione automatica dello strumento e, in caso di assenza, di utilizzare campioni di siero precaratterizzati per ottimizzare il tempo di acquisizione dell'immagine. - Utilizzare la funzione di analisi delle immagini dello strumento per ottenere intensità spot, anche per tre posizioni vuote (sfondo) sulla membrana di nitrocellulosa; Mantenere un volume di misurazione costante per punto. Esporta i dati in un foglio di calcolo, ricordando di etichettare correttamente ogni posizione del punto.
- Calcolare la media e la deviazione standard (SD) delle intensità spot triplicate. Quindi, sottrarre il fondo della membrana di nitrocellulosa da tutti i campioni.
- Utilizzare la seguente equazione per ottenere un valore di cut-off per l'interpretazione dei risultati -/+:
(Media dei controlli negativi) + (3 x SD dei controlli negativi). Considerare i campioni positivi se la loro intensità media scende al di sopra del valore di cut-off e negativi se la loro intensità media scende al di sotto del valore di cut-off.
NOTA: Uno schema della procedura CFDB è fornito nella Figura 3.
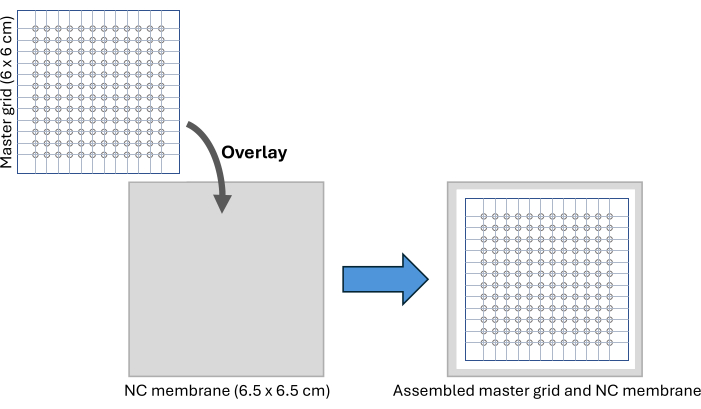
Figura 2: Assemblaggio CFDB. Uno schema della griglia master CFDB e della configurazione dell'assemblaggio della membrana NC. La griglia master è sovrapposta alla membrana NC per fornire un modello regolare e indirizzabile per l'individuazione e l'immobilizzazione dei campioni di siero. Abbreviazioni: CFDB = cell-free dot blot; NC = nitrocellulosa. Clicca qui per visualizzare una versione più grande di questa figura.
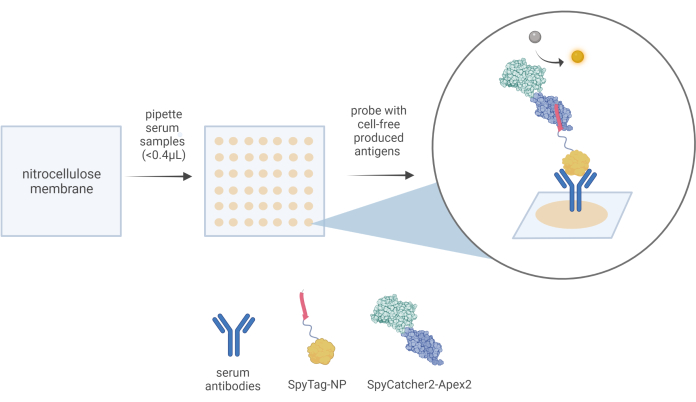
Figura 3: Una rappresentazione schematica del flusso di lavoro CFDB. In un test CFDB, una piccola quantità (<0,4 μl) di campioni di siero diluiti 10x viene dispensata manualmente su una membrana di nitrocellulosa pretagliata (pannello sinistro) in posizioni discrete e indirizzabili (pannello centrale). Depositare un campione di siero per punto in punti triplicati e immobilizzare il contenuto proteico, compreso il serbatoio totale di anticorpi del siero, sul substrato solido NC (macchie beige nel pannello centrale). In questo esempio, gli anticorpi anti-NP contenuti nei campioni di siero possono essere prima legati dal reagente di rilevamento primario CFDB SpyTag-NP e infine rilevati dal reagente di rilevamento secondario CFDB SpyCatcher2-Apex2 (bolla ingrandita nel pannello di destra). Questa cifra è stata presa da Norouzi et al.5. Abbreviazioni: CFDB = cell-free dot blot; NP = proteina nucleocapside; LET = modello di espressione lineare. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Amplificazione PCR del modello di espressione lineare per l'antigene target
Per amplificare con PCR il SARS-CoV-2 NP LET, sono stati utilizzati primer universali Ter in avanti e indietro come descritto nella sezione 1.2 del protocollo e 1 μL del prodotto è stato controllato su un gel di agarosio (Figura 4) prima di procedere alla purificazione del prodotto per PCR.
<...
Discussione
Il COVID-19 ha evidenziato l'importanza di una diagnostica accessibile e solida per il controllo dei focolai di infezione e l'ottimizzazione delle strategie sanitarie globali. I test sierologici che rilevano gli anticorpi protettivi si sono rivelati essenziali per tracciare i modelli di trasmissibilità di nuove varianti, identificare i punti caldi, guidare lo sviluppo del vaccino, valutare i casi sospetti e proteggere le popolazioni vulnerabili14. La pandemia ha ...
Divulgazioni
M.N. e K.P. sono co-inventori del metodo del dot blot cell-free. È stata depositata una domanda di brevetto provvisoria relativa a questo lavoro (PCT/CA2024/050097, depositata nel gennaio 2024).
Riconoscimenti
S.S. e R.Z. sono supportati da finanziamenti della Defense Advanced Research Projects Agency (DARPA), contratto n. N66001-23-2-4042. I punti di vista, le opinioni e/o i risultati espressi sono quelli degli autori e non devono essere interpretati come rappresentanti i punti di vista o le politiche ufficiali del Dipartimento della Difesa o del Governo degli Stati Uniti. Questo lavoro è stato sostenuto da fondi a K.P. dal programma di sovvenzioni della Fondazione CIHR (201610FDN-375469), dal programma CIHR Canada Research Chair (950-231075 e 950-233107), dall'iniziativa Medicine by Design dell'Università di Toronto, che riceve finanziamenti dal Canada First Research Excellence Fund e fondi a K.P., dal Defense Research and Development Canada's, Canadian Safety and Security Program (contratto 39903-200137). La Figura 1 e la Figura S1 supplementare sono state create utilizzando SnapGene Viewer.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
Riferimenti
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722 (2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- . IDT codon optimization tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2025)
- . General protocol for western blotting Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025)
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- . Plasma and serum preparation Available from: https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025)
- Mattiuzzo, G., et al. . WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405 (2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon