A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קביעה מבוססת צמיחה ואישור ביוכימית של דרישות גנטיות לפירוק חלבונים ב
In This Article
Summary
This article describes a yeast growth-based assay for the determination of genetic requirements for protein degradation. It also demonstrates a method for rapid extraction of yeast proteins, suitable for western blotting to biochemically confirm degradation requirements. These techniques can be adapted to monitor degradation of a variety of proteins.
Abstract
פירוק חלבונים מוסדר הוא חיוני עבור כמעט כל פונקציה סלולרית. הרבה ממה שידוע על המנגנונים המולקולריים וגנטיות לדרישות פירוק חלבונים האיקריוטים בתחילה הוקם בSaccharomyces cerevisiae. ניתוחים קלאסיים של פירוק חלבונים יש לסמוך על מתודולוגיות דופק מרדף וcycloheximide-מרדף ביוכימיים. בעוד טכניקות אלה מספקים אמצעים רגישים להתבוננות פירוק חלבונים, שהם, זמן רב מייגע, ונמוכה-תפוקה. גישות אלה אינן ניתנות לסינון מהיר או בקנה מידה גדולה למוטציות המונעות פירוק חלבונים. כאן, assay המבוסס על צמיחת שמרים לזיהוי הקליל של דרישות גנטיות לפירוק חלבונים מתואר. ב assay זה, אנזים כתב הנדרש לצמיחה בתנאים סלקטיבית מסוימים התמזג חלבון יציב. תאים חסרי אנזים כתב אנדוגני אך המבטא את חלבון ההיתוך יכול לגדול תחת Sele גודלתנאי ctive רק כאשר חלבון ההיתוך הוא התייצב (כלומר, כאשר פירוק חלבונים נפגעת). בassay הצמיחה המתואר כאן, דילולים סידוריים של wild-type ותאי שמרי מוטציה מחסה פלסמיד קידוד חלבון היתוך הם הבחינו על מדיום סלקטיבית ולא סלקטיבי. צמיחה בתנאים סלקטיבית עולה בקנה אחד עם ליקוי השפלה ידי מוטציה נתון. שפע חלבון מוגבר צריך להיות מאושר ביוכימית. שיטה להפקת המהירה של שמרי חלבונים בצורה מתאימה לאלקטרופורזה ומערבית סופג הוא הוכיח גם. קריאה מבוססת צמיחה ליציבות חלבון, בשילוב עם פרוטוקול פשוט להפקת חלבון לאנליזה ביוכימית, מאפשרת זיהוי מהיר של דרישות גנטיות לפירוק חלבונים. ניתן להתאים את הטכניקות הללו כדי לפקח השפלה של מגוון רחב של חלבונים קצרים ימים. בדוגמא המוצגת, אנזים HIS3, אשר נדרש לסינתזת היסטידין, התמזגלDeg1 -Sec62. Deg1 -Sec62 מיועד לפירוק לאחר שaberrantly עוסק translocon reticulum endoplasmic. תאי מחסה Deg1 -Sec62-HIS3 היו מסוגלים לגדול בתנאים סלקטיבית כאשר החלבון התייצב.
Introduction
פירוק חלבוני סלקטיבי הוא חיוני לחיים אוקריוטים, ופירוק חלבונים שינה תורם למספר מצבים רפואיים, כוללים מספר סוגים של הסרטן, מחלה ניוונית של מערכת עצבים, מחלות לב, וסיסטיק פיברוזיס 1-5. מערכת היוביקוויטין-פרוטאזום (UPS), המזרז פירוק חלבונים סלקטיבית, היא יעד טיפולי המתעוררים לתנאים אלה 6-10. ligases יוביקוויטין קוולנטית לצרף פולימרים של היוביקוויטין חומצת אמינו 76-לחלבוני 11. חלבונים שסומנו עם רשתות polyubiquitin מוכרים וproteolyzed על ידי ~ 2.5 megadalton 26s פרוטאזום 12. מחקרים שיזמו באורגניזם אוקריוטים מודל Saccharomyces cerevisiae (שמרי ניצנים) היו מכוננים בהבהרת מנגנוני פירוק חלבונים בתאים אוקריוטים. המצע הפיזיולוגי הפגין הראשון של UPS היה מדכא תעתיק שמרי 13 MATα2, 14 ורכיבים שמורים ביותר, רבים של UPS זוהו לראשונה או מתאפיינים בשמרים (למשל 15-26). התגליות שנעשו באורגניזם מודל רב-תכליתי וצייתן גנטי זה, צפויות להמשיך ולספק תובנות חשובות מנגנוני שימור של השפלה בתיווך היוביקוויטין.
הכרה והשפלה של רוב מצעי UPS דורשים פעולה מתואמת של חלבונים מרובים. לכן, מטרה חשובה באפיון השפלה המוסדרת של חלבון יציב נתון היא לקבוע את הדרישות הגנטיות לproteolysis. גישות קלאסיות (למשל דופק מרדף וניסויים cycloheximide-מרדף 27) לניטור פירוק חלבונים בתאי יונקים או שמרים הן מייגע וגוזל זמן. בעוד אלו סוגים של המתודולוגיה לספק אמצעי רגיש מאוד לאיתור פירוק חלבונים, שהם אינם מתאימים לניתוח מהיר של פירוק חלבונים או screeni בקנה מידה גדולהng למוטציות המונעות פירוק חלבונים. כאן, assay המבוסס על צמיחת שמרים לזיהוי המהיר של דרישות גנטיות לפירוק של חלבונים לא יציבים מוצג.
בשיטה המבוססת על צמיחת שמרים לניתוח פירוק חלבונים, חלבון יציב של עניין (או אות השפלה) מותך, במסגרת, לחלבון שנדרש לשמרי צמיחה בנסיבות מסוימות. התוצאה היא מצע מלאכותי שעשויות לשמש ככלי רב עוצמה כדי לקבוע את הדרישות הגנטיות של פירוק חלבונים של החלבון יציב של עניין. נוח, זני שמרי מעבדה הנפוצים ביותר בנמל פנל של מוטציות בגני מקודדי אנזימים מעורבים בחילוף חומרי הסינתזה של חומצות אמינו מסוימות או בסיסים חנקניים (למשל 20,28-30). אנזימים אלה חיוניים לשגשוג תאי בהעדר מטבוליטים סיפקו אקסוגני בסינתזה שהאנזימים להשתתף. כזהאנזימים מטבוליים עשויים כך לתפקד ככתבים המבוסס על צמיחה לפירוק של חלבונים לא יציבים שאליו הם התמזגו. הדרישות הגנטיות לפירוק חלבונים אפשר לבאר בקלות, שכן מוטציות המונעות proteolysis תאפשר תאי מחסה כתב השפלה לגדול בתנאים סלקטיבית.
יתרון צמיחה הוא אינדיקציה עקיפה שמוטציה מסוימת מגדילה את השפע של החלבון של עניין. עם זאת, נדרשת אנליזה ביוכימית ישירה כדי לאשר שמוטציה מאפשרת צמיחה דרך רמות חלבון מוגברות ולא באמצעות גורמים עקיפים או artifactual. ההשפעה של מוטציה בחלבון שפע עשויה להיות מאושרת על ידי ניתוח כתם המערבי של רמות חלבון במצב יציב בתאים שעושים ולא נמל מוטציה מסוימת. שיטה למיצוי המהיר ויעיל של חלבוני שמרים (דגירה רציפה של תאי שמרים עם נתרן הידרוקסידי וחיץ מדגם) בצורה מתאימהלניתוח על ידי מערבי סופג מוצג גם 31. יחד, ניסויים אלה להקל על זיהוי המהיר של רגולטורי מועמד של פירוק חלבונים.
Protocol
1. Assay צמיחת שמרים לזיהוי מוטציות מועמד פגומים בפירוק חלבונים
- להפוך wild-type ותאי שמרים שעברו מוטציה עם קידוד פלסמיד חלבון יציב התמזגו, במסגרת, לאנזים מטבולי כתב.
- לחסן transformants ב 5 מיליליטר של סינטטי מוגדר בינוני מינימאלי (SD) שהוא סלקטיבי לתאי חסת מולקולות פלסמיד. דגירה הלילה בשעה 30 ° C, מסתובבת.
- מדוד את הצפיפות האופטית ב 600 ננומטר (OD 600) של כל תרבות לילה.
הערה: לאחר דגירה הלילה, תאים בתרבית יכולה להיות בכל שלב צמיחת לוגריתמים או נייח אבל צריך להגיע OD מינימאלי 600 של 0.2. שמרי זנים גדלים לאט מאוד עשויים לדרוש פעמים דגירה יותר מלילה אחד, או חיסון של מספר גדול יותר של תאים, כפי שנקבעו באופן אמפירי. - הכן פי שישה דילולים סדרתי של תאי שמרים הפכו בצלחת 96-היטב סטרילי, החל בתאים מדוללים לn OD 600 של 0.2. מניחים כל transformant שמרים להיות מאושרים בשורה שונה בצלחת 96-היטב.
- עבור כל transformant, לחשב את הנפח של תרבות הלילה נדרשת לדלל תאים לOD 600 של 0.2 בנפח סופי של 200 μl. הוספת נפח זה של תרבות הלילה למתאים היטב בטור 1. הוסף את הכמות המתאימה של מים סטריליים כדי להביא את הנפח 200 μl.
- עבור כל שורה של שמרים, להוסיף 125 μl של מים סטריליים לבארות בעמודות 2, 3, ו -4.
הערה: 96-גם צלחות סטרילי עטופות בנפרד עשויות להיות ארוזות עם מכסי סטרילי. המכסים עשויים לשמש כמאגרים למים סטריליים שמופץ בשלב זה. זה מאפשר העברה סימולטנית של מים סטריליים לכל הבארות בעמודת נתונה עם pipettor רב-ערוצי. - מערבבים את התוכן של העמודה הראשונה (השמרים מדוללים לOD 600 של 0.2) על ידי pipetting מעלה ומטה עם pipettor רב-ערוצי.
- Transfאה 25 μl של שמרים מהטור 1 לעמודה 2, באמצעות pipettor רב-ערוצי. מערבבים על ידי pipetting מעלה ומטה. העברה 25 μl מטור 2 לטור 3, ו -25 μl מהעמודה 3 לעמודה 4 (ערבב היטב בכל שלב).
- מערבבים כל דגימה עם pipettor רב-ערוצי. ימשיך מרוב לדלל לדלל לפחות עמודות של שמרים, פיפטה 4 μl של כל דגימה על שתי צלחות המכילות מצע סלקטיבי המתאים. השתמש בצלחת אחת עם מדיום ששומרת על בחירת פלסמיד (צלחת זה משמשת כתצפית שמרים ושליטה צמיחה). השתמש בצלחת שנייה עם מדיום שבוחר עבור תחזוקת פלסמיד וביטוי של החלבון יציב התמזג אנזים הכתב. משום שמרים להתיישב במהירות, לערבב תאים על ידי pipetting מעלה ומטה במרווחי זמן קבועים.
הערה: צלחות Drier יותר בקלות תספוג נוזלים מאשר צלחות מוכנות טרי ולכן מומלצים לניסויים אלה. צלחות לחות עשויות להיות מיובשות על ידי דגירה בטמפרטורת חדר בloלחות w עבור 1-2 ימים או incubations קצר יותר בזרימה למינרית. צלחות עלולות להתייבש בצורה לא אחידה, אם את זרימת האוויר למינרית היא מקבילה לספסל. שימוש בתבנית עושה את זה קל יותר לזהות תאי שמרים במרחקים קבועים. שתי תבניות מדגם מסופקות באיור 1. אלה עשויים להיות מודפסים, לגזור, ומודבקים לחלק הפנימי של מכסה צלחת פטרי. - לאפשר צלחות לייבוש על גבי הספסל.
- דגירה צלחות על C ° 30 ל2-6 ימים.
- לצלם כל צלחת לאחר דגירה.
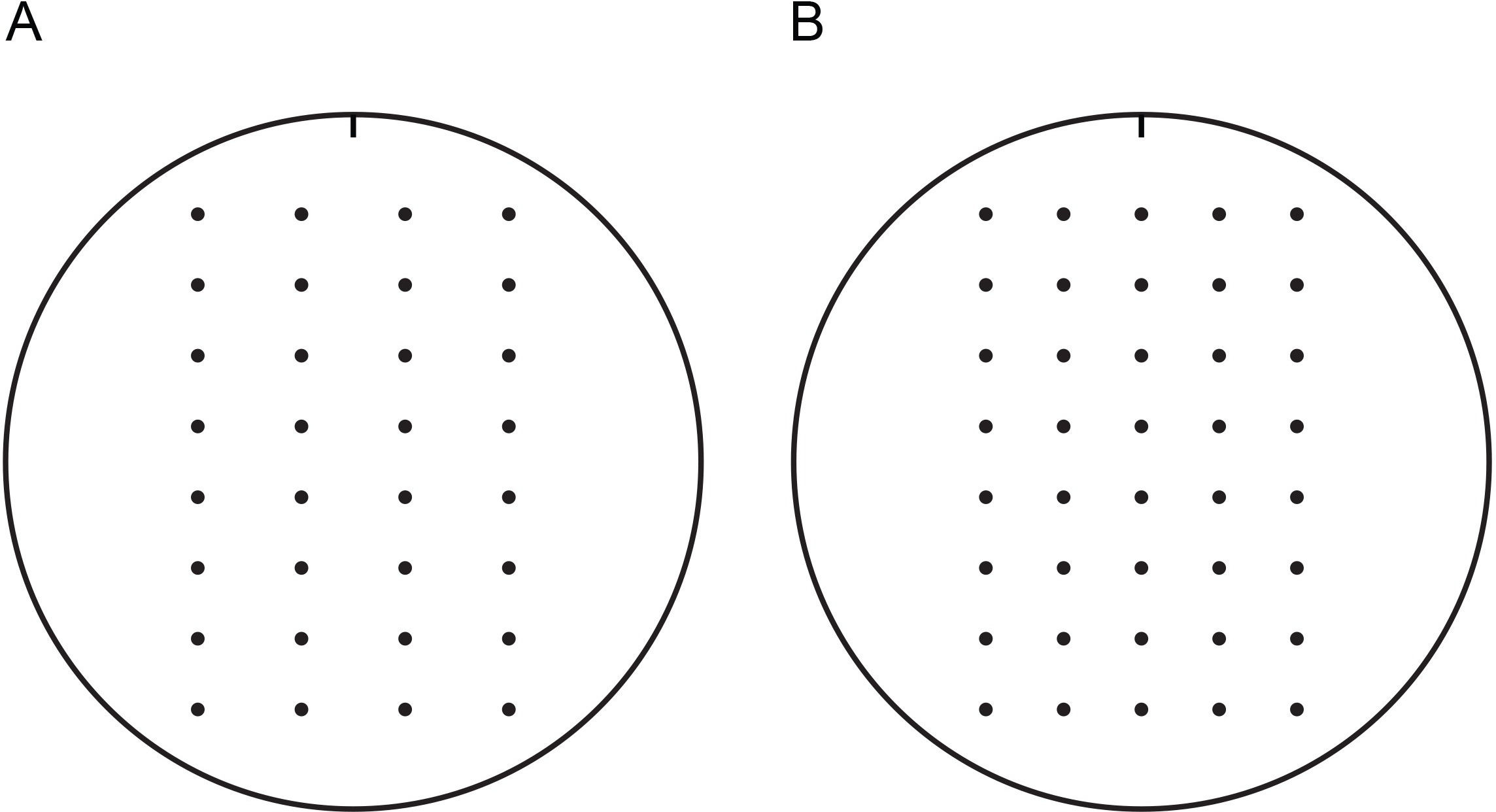
איור 1. תבניות לאיתור תאי שמרים על צלחות אגר 100 מ"מ. תבניות אלו עשויות לשמש כדי להקל על איתור שמרים במרחקים קבועים עם pipettor רב-ערוצי. תבניות ניתן להדפיס, לגזור, ומודבקים לחלק הפנימי של מכסה צלחת פטרי. מניחים צלחת פטרי עם צמיחהמכסה בתוך בינוני עם תבנית מודבקת. תבניות מסומנות בחריץ כדי לעקוב אחר כיוון. מומלץ צלחות המשמשות במבחני צמיחה תסומן באופן דומה עם חריץ כדי לעקוב אחר כיוון. תבניות לאיתור ארבעה (א) או חמישה (B) דילולים סדרתי של תאי שמרים מסופקים. אנא לחץ כאן כדי לצפות בגרסה להדפסה של נתון זה עם תבניות 100 מ"מ.
2. אישור ביוכימית של Assay צמיחת שמרים
- צמיחה של תאי שמרים והפקת חלבון Post-אלקליין (שונה מ -31)
- להפוך wild-type ותאי שמרים שעברו מוטציה עם פלסמיד המקודד את החלבון יציב.
- לחסן transformants ב 5 מיליליטר של מדיום SD שהוא סלקטיבי לתאי חסת מולקולות פלסמיד. דגירה הלילה בשעה 30 ° C, מסתובבת.
- מדוד את OD 600 של כל מ"ק לילהlture.
הערה: לאחר דגירה הלילה, תאים יכולים להיות בכל שלב צמיחת לוגריתמים או נייח אבל צריך להגיע OD 600 שיאפשר דילול לOD 600 של 0.2 ב 10 מיליליטר בינוני טרי סלקטיבית (צעד 2.1.4). שמרי זנים גדלים לאט מאוד עשויים לדרוש פעמים דגירה יותר מלילה אחד, או חיסון של מספר גדול יותר של תאים, כפי שנקבעו באופן אמפירי. - לדלל תאי שמרים לOD 600 של 0.2 ב 10 מיליליטר בינוני סלקטיבית טרי.
- תמשיך לדגור תאים על 30 מעלות צלזיוס, מסתובב או רועד, עד תרבויות להגיע OD 600 בין 0.8 ו -1.2 (כלומר נמצאות בצמיחת אמצע לוגריתמים).
הערה: אם החלבון לא יציב של עניין הוא תחת שליטתו של אמרגן ויסות, התזמון האופטימלי של אינדוקציה של ביטוי חלבון וקציר תא עשוי להשתנות בהתאם למחקרים קודמים או תצפיות אמפיריות. - לאסוף 2.5 OD 600 יחידות של התרבות ב 15 מ"ל חרוטיאל צינור על ידי צנטריפוגה בXG 5000 במשך 5 דקות בטמפרטורת חדר. הסר supernatant ידי pipetting או שאיפה.
הערה: OD אחת 600 יחידה מוגדרת כסכום של שמרים הנוכחיים ב 1 מיליליטר של התרבות בOD 600 של 1.0. הנפח של התרבות (במ"ל) נדרש למסוק 2.5 OD 600 יחידות (V) ניתן לקבוע באמצעות המשוואה הבאה: V = 2.5 OD 600 יחידות OD נמדד / 600 - תאי Resuspend ב 1 מיליליטר מים מזוקקים. העבר את התאים מושעים לצינור microcentrifuge.
- גלולה תאים על ידי צנטריפוגה ב6,500 XG במשך 30 שניות בטמפרטורת חדר. הסר supernatant ידי pipetting או שאיפה.
- תאי Resuspend ב 100 μl מים מזוקקים על ידי pipetting מעלה ומטה או vortexing, ולהוסיף 100 μl 0.2 M NaOH. מערבבים על ידי pipetting מעלה ומטה. דגירה דגימות במשך 5 דקות בטמפרטורת חדר.
- תאים גלולה (שרובם עדיין לא שוחררו חלבונים ועדיין קיימא) על ידי צנטריפוגה ב18,000 xגרם במשך 5 דקות. הסר supernatant ידי pipetting או שאיפה.
- גלולה גלולה ב50-100 μl חיץ מדגם Laemmli 1x, אשר lyse תאים, על ידי pipetting מעלה ומטה או vortexing.
הערה: הסרת supernatant אלקליין הבא צנטריפוגה וresuspension הבא של תאים במאגר מדגם Laemmli תמציות חלבונים בpH תואם עם ג'ל אלקטרופורזה סולפט-polyacrylamide (SDS-PAGE) dodecyl נתרן באמצעות מערכת חיץ ריצת טריס-גליצין ומערבי סופג. - לפגל באופן מלא חלבונים, דגירה lysates על 95 מעלות צלזיוס למשך 5 דקות.
הערה: חלבוני צבירה מועדים (למשל חלבונים עם כמה קטעים הטרנסממברני) עשויים להיות בלתי ניתנים לפתרון, כאשר טופחו על 95 מעלות צלזיוס. לכן, צריך להיות מודגרות lysates בטמפרטורות נמוכות יותר (למשל 37 ° C - 70 ° C) במשך 10 - 30 דקות, כאמפירי שנקבע, לניתוח של חלבונים מסוג זה. - lysates מגניב על ידי הנחת על קרח למשך 5 דקות.
- lysates צנטריפוגות ב 18,000 XG 1 דקות בטמפרטורת חדר כדי גלולה חומר מסיס. הפרד את supernatant (solubilized חלבון חילוץ) על ידי SDS-PAGE לפני ניתוח שלאחר מכן מערבי כתם (סעיף 2.2). לחלופין, lysates החנות ב -20 ° C.
- פרוטוקול מערבי סופג נציג
- לטעון נפח אמפירי נחוש של lysates בג'ל SDS-PAGE.
- הפעל ג'ל ב 200 V עד לפני לצבוע הגיע לתחתית הג'ל.
- העבר את החלבונים מן ג'ל לפלואוריד polyvinylidene קרום (PVDF) על ידי העברה רטובה ב 20 V ל60-90 דקות ב 4 מעלות צלזיוס.
- קרום בלוק על ידי דוגרים ב 5% חלב דל שומן בטריס שנאגרו מלוח (TBS), נדנדה, עבור שעה 1 בטמפרטורת חדר או הלילה ב 4 מעלות צלזיוס.
- למזוג פתרון חסימה.
- דגירה הממברנה עם נוגדן ראשוני ספציפי לחלבון של עניין (או תג epitope ממנו) ב 1% חלב דל שומן בTBS עם 0.1% Tween-20 (TBS / T) לשעה 1 בחדר temperature, נדנדה.
- למזוג פתרון נוגדן, ולשטוף קרום דק '3 x 5 עם TBS / T בטמפרטורת חדר, נדנדה.
- דגירה קרום עם נוגדנים משני fluorophore מצומדות מתאימים ב 1% חלב דל שומן בכפות / T עבור שעה 1 בטמפרטורת חדר, נדנדה.
הערה: מאחר fluorophores הן רגישה לאור, דילולים של נוגדני fluorophore מצומדות צריכים להיות מוכנים בחושך. בנוסף, דגירה של ממברנות בנוכחות נוגדני fluorophore מצומדות צריכה להתרחש במיכלים lightproof. זה יכול להיות מושלם על ידי עטיפת מגשי דגירה ברדיד אלומיניום. - למזוג פתרון נוגדן, ולשטוף קרום דק '3 x 5 עם TBS / T בטמפרטורת חדר, נדנדה.
- לרכוש תמונה של קרום באמצעות Li-Cor אודיסיאה CLX ותוכנת Studio התמונה (או ציוד הדמיה דומה ותוכנה), על פי המלצות יצרן.
- לאחר ההדמיה קרום, דגירה הממברנה עם נוגדן ראשוני ספציפי לעומסing חלבון שליטה ב 1% חלב דל שומן בכפות / T עבור שעה 1 בטמפרטורת חדר, נדנדה.
- למזוג פתרון נוגדן, ולשטוף קרום דק '3 x 5 עם TBS / T בטמפרטורת חדר, נדנדה.
- דגירה קרום עם נוגדנים משני fluorophore מצומדות מתאימים ב 1% חלב דל שומן בכפות / T עבור שעה 1 בטמפרטורת חדר, נדנדה.
- למזוג פתרון נוגדן, ולשטוף קרום דק '3 x 5 עם TBS / T בטמפרטורת חדר, נדנדה.
- לרכוש תמונה של קרום באמצעות Li-Cor אודיסיאה CLX ותוכנת Studio התמונה (או ציוד הדמיה דומה ותוכנה), על פי המלצות יצרן.
תוצאות
כדי להמחיש מתודולוגיה זו, אנזים HIS3 כבר התמזג carboxy-הסופי של reticulum endoplasmic מודל השפלה -associated (ER) (ERAD) מצע, Deg1 -Sec62 (איור 2 א) כדי ליצור Deg1 -Sec62-HIS3 (איור 3) . Deg1 -Sec62 מייצג חבר מייסד של כיתת רומן של מצעי ERAD ממוקדות הבאים עמותה מתמשכת, סוטה עם translocon, הער?...
Discussion
המתודולוגיה המוצגת כאן מאפשרת קביעה המהירה ואישור ביוכימיים של דרישות גנטיות לפירוק חלבונים בתאי שמרים. ניסויים אלה מדגישים את השירות ואת כוחו של שמרי כאורגניזם מודל אוקריוטים (כמה ביקורות מצוינות של שמרי ביולוגיה וליקוטים של פרוטוקולים לטיפול, אחסון, ומניפולצית ת...
Disclosures
יש לי המחברים אין לחשוף.
Acknowledgements
אנו מודים לחברי הווה ובעבר של המעבדה רובינשטיין למתן סביבת מחקר תומכת ונלהבת. אנו מודים ריאן ט גיבסון לסיוע בפרוטוקול אופטימיזציה. אנו מודים למארק Hochstrasser (אוניברסיטת ייל) ודיטר וולף (Universität שטוטגרט) לזני שמרים ופלסמידים. אנו מודים למבקרים האנונימיים שלנו לעזרתם בשיפור הבהירות והשירות של כתב היד הזה. עבודה זו נתמכה על ידי הפרס מחקר מהפרק סטייט הכדור של Sigma Xi לSGW, המכונים לאומי לבריאות מענק (R15 GM111713) לEMR, הפרס מחקר כדור מדינה לשאוף לאוניברסיטת EMR, וקרנות מאוניברסיטת מדינת הכדור משרד המכללה והמחלקה לביולוגיה.
Materials
| Name | Company | Catalog Number | Comments |
| Desired yeast strains, plasmids, standard medium and buffer components | Yeast strains with desired mutations may be generated in the investigator's laboratory. Wild-type yeast and a variety of mutants are also commercially available (e.g. from GE Healthcare). Plasmids encoding fusion proteins may be generated in the investigator's laboratory. | ||
| 3-amino-1H-1,2,4-triazole | Fisher Scientific | AC264571000 | Competitive inhibitor of His3 enzyme. May be included in medium to increase stringency of growth assay using His3 reporter constructs. |
| Endoglycosidase H (recombinant form from Streptomyces plicatus) | Roche | 11088726001 | May be used to assess N-glycosylation of proteins; compatible with SDS and beta-mercaptoethanol concentrations found in 1x Laemmli sample buffer. |
| Disposable borosilicate glass tubes | Fisher Scientific | 14-961-32 | Available from a variety of manufacturers |
| Temperature-regulated incubator (e.g. Heratherm Incubator Model IMH180) | Dot Scientific | 51028068 | Available from a variety of manufacturers |
| New Brunswick Interchangeable Drum for 18 mm tubes (tube roller) | New Brunswick | M1053-0450 | Tube roller is recommended to maintain overnight yeast starter cultures of yeast cells in suspension. A platform shaker or tube roller may be used to maintain larger cultures in suspension. |
| New Brunswick TC-7 Roller Drum 120V 50/60 H | New Brunswick | M1053-4004 | For use with tube roller |
| SmartSpec Plus Spectrophotometer | Bio-Rad | 170-2525 | Available from a variety of manufacturers |
| Sterile 96-well flat bottom microtest plates with lid individually wrapped | Sarstedt | 82.1581.001 | Available from a variety of manufacturers |
| Pipetman Neo P8x20N, 2-20 μl | Gilson | F14401 | Available from a variety of manufacturers |
| [header] | |||
| Pipetman Neo P8x200N, 20-200 μl | Gilson | F14403 | Single-channel and multichannel pipettors are used at various stages of the protocol. While multichannel pipettors reduce the pipetting burden at several steps, single-channel pipettors may be used throughout the entire protocol. Available from a variety of manufacturers. |
| Centrifuge 5430 | Eppendorf | 5427 000.216 | Rotor that is sold with unit holds 1.5 and 2.0 ml microcentrifuge tubes. Rotor may be swapped for one that holds 15 ml and 50 ml conical tubes. |
| Plate imaging system (e.g. Gel Doc XR+ System) | Bio-Rad | 170-8195 | A variety of systems may be used to image plates, including sophisticated imaging systems, computer scanners, and camera phones. |
| Fixed-Angle Rotor F-35-6-30 with Lid and Adapters for Centrifuge Model 5430/R, 15/50 ml Conical Tubes, 6-Place | Eppendorf | F-35-6-30 | |
| 15 ml screen printed screw cap tube 17 x 20 mm conical, polypropylene | Sarstedt | 62.554.205 | Available from a variety of manufacturers |
| 1.5 ml flex-tube, PCR clean, Natural microcentrifuge tubes | Eppendorf | 22364120 | Available from a variety of manufacturers |
| Analog Dri-Bath Heater | Fisher Scientific | 1172011AQ | Boiling water bath with hot plate may also be used to denature proteins |
| SDS-PAGE running and transfer apparatuses, power supplies, and imaging equipment or darkrooms for SDS-PAGE and transfer to membrane | Will vary by lab and application | ||
| Western blot imaging system (e.g. Li-Cor Odyssey CLx scanner and Image Studio Software) | Li-Cor | 9140-01 | Will vary by lab and application |
| EMD Millipore Immobilon PVDF Transfer Membranes | Fisher Scientific | IPFL00010 | Will vary by lab and application |
| Primary antibodies (e.g. Phosphoglycerate Kinase (Pgk1) Monoclonal antibody, mouse (clone 22C5D8)) | Life Technologies | 459250 | Will vary by lab and application |
| Secondary antibodies (e.g. Alexa-Fluor 680 Rabbit Anti-Mouse IgG (H+L)) | Life Technologies | A-21065 | Will vary by lab and application |
References
- Goldberg, A. L. Protein degradation and protection against misfolded or damaged proteins. Nature. 426, 895-899 (2003).
- Guerriero, C. J., Brodsky, J. L. The delicate balance between secreted protein folding and endoplasmic reticulum-associated degradation in human physiology. Physiological Reviews. 92, 537-576 (2012).
- Pagan, J., Seto, T., Pagano, M., Cittadini, A. Role of the ubiquitin proteasome system in the heart. Circulation Research. 112, 1046-1058 (2013).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443, 780-786 (2006).
- Turnbull, E. L., Rosser, M. F., Cyr, D. M. The role of the UPS in cystic fibrosis. BMC Biochemistry. 8, S11 (2007).
- Bedford, L., Lowe, J., Dick, L. R., Mayer, R. J., Brownell, J. E. Ubiquitin-like protein conjugation and the ubiquitin-proteasome system as drug targets. Nature Reviews Drug Discovery. 10, 29-46 (2011).
- Kar, G., Keskin, O., Fraternali, F., Gursoy, A. Emerging role of the ubiquitin-proteasome system as drug targets. Current Pharmaceutical Design. 19, 3175-3189 (2013).
- Paul, S. Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches. BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology. 30, 1172-1184 (2008).
- Shen, M., Schmitt, S., Buac, D., Dou, Q. P. Targeting the ubiquitin-proteasome system for cancer therapy. Expert Opinion on Therapeutic Targets. 17 (9), 1091-1108 (2013).
- Finley, D., Ulrich, H. D., Sommer, T., Kaiser, P. The ubiquitin-proteasome system of Saccharomyces cerevisiae. Genetics. 192, 319-360 (2012).
- Scheffner, M., Nuber, U., Huibregtse, J. M. Protein ubiquitination involving an E1-E2-E3 enzyme ubiquitin thioester cascade. Nature. 373, 81-83 (1995).
- Ravid, T., Hochstrasser, M. Diversity of degradation signals in the ubiquitin-proteasome system. Nature Reviews Molecular Cell Biology. 9, 679-690 (2008).
- Hochstrasser, M., Ellison, M. J., Chau, V., Varshavsky, A. The short-lived MAT alpha 2 transcriptional regulator is ubiquitinated in vivo. Proceedings of the National Academy of Sciences of the United States of America. 88, 4606-4610 (1991).
- Hochstrasser, M., Varshavsky, A. In vivo degradation of a transcriptional regulator: the yeast alpha 2 repressor. Cell. 61, 697-708 (1990).
- Bays, N. W., Gardner, R. G., Seelig, L. P., Joazeiro, C. A., Hampton, R. Y. Hrd1p/Der3p is a membrane-anchored ubiquitin ligase required for ER-associated degradation. Nature Cell Biology. 3, 24-29 (2001).
- Hampton, R. Y., Gardner, R. G., Rine, J. Role of 26S proteasome and HRD genes in the degradation of 3-hydroxy-3-methylglutaryl-CoA reductase, an integral endoplasmic reticulum membrane protein. Molecular Biology of the Cell. 7, 2029-2044 (1996).
- Swanson, R., Locher, M., Hochstrasser, M. A conserved ubiquitin ligase of the nuclear envelope/endoplasmic reticulum that functions in both ER-associated and Matalpha2 repressor degradation. Genes & Development. 15, 2660-2674 (2001).
- Goebl, M. G., et al. The yeast cell cycle gene CDC34 encodes a ubiquitin-conjugating enzyme. Science. 241, 1331-1335 (1988).
- Bachmair, A., Finley, D., Varshavsky, A. In vivo half-life of a protein is a function of its amino-terminal residue. Science. 234, 179-186 (1986).
- Chen, P., Johnson, P., Sommer, T., Jentsch, S., Hochstrasser, M. Multiple ubiquitin-conjugating enzymes participate in the in vivo degradation of the yeast MAT alpha 2 repressor. Cell. 74, 357-369 (1993).
- Varshavsky, A. Discovery of the biology of the ubiquitin system. JAMA: The Journal of the American Medical Association. 311, 1969-1970 (2014).
- Heinemeyer, W., Kleinschmidt, J. A., Saidowsky, J., Escher, C., Wolf, D. H. Proteinase yscE, the yeast proteasome/multicatalytic-multifunctional proteinase: mutants unravel its function in stress induced proteolysis and uncover its necessity for cell survival. The EMBO Journal. 10, 555-562 (1991).
- Sommer, T., Jentsch, S. A protein translocation defect linked to ubiquitin conjugation at the endoplasmic reticulum. Nature. 365, 176-179 (1993).
- Hiller, M. M., Finger, A., Schweiger, M., Wolf, D. H. ER degradation of a misfolded luminal protein by the cytosolic ubiquitin-proteasome pathway. Science. 273, 1725-1728 (1996).
- Seufert, W., Jentsch, S. In vivo function of the proteasome in the ubiquitin pathway. The EMBO Journal. 11, 3077-3080 (1992).
- Knop, M., Finger, A., Braun, T., Hellmuth, K., Wolf, D. H. Der1, a novel protein specifically required for endoplasmic reticulum degradation in yeast. The EMBO Journal. 15, 753-763 (1996).
- Zattas, D., Adle, D. J., Rubenstein, E. M., Hochstrasser, M. N-terminal acetylation of the yeast Derlin Der1 is essential for Hrd1 ubiquitin-ligase activity toward luminal ER substrates. Molecular Biology of the Cell. 24, 890-900 (2013).
- Brachmann, C. B., et al. Designer deletion strains derived from Saccharomyces cerevisiae S288C: a useful set of strains and plasmids for PCR-mediated gene disruption and other applications. Yeast. 14, 115-132 (1998).
- Sikorski, R. S., Hieter, P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae. Genetics. 122, 19-27 (1989).
- Ralser, M., et al. The Saccharomyces cerevisiae W303-K6001 cross-platform genome sequence: insights into ancestry and physiology of a laboratory mutt. Open Biology. 2, 120093 (2012).
- Kushnirov, V. V. Rapid and reliable protein extraction from yeast. Yeast. 16, 857-860 (2000).
- Rubenstein, E. M., Kreft, S. G., Greenblatt, W., Swanson, R., Hochstrasser, M. Aberrant substrate engagement of the ER translocon triggers degradation by the Hrd1 ubiquitin ligase. The Journal of Cell Biology. 197, 761-773 (2012).
- Mayer, T. U., Braun, T., Jentsch, S. Role of the proteasome in membrane extraction of a short-lived ER-transmembrane protein. The EMBO Journal. 17, 3251-3257 (1998).
- Scott, D. C., Schekman, R. Role of Sec61p in the ER-associated degradation of short-lived transmembrane proteins. The Journal of Cell Biology. 181, 1095-1105 (2008).
- Kim, I., et al. The Png1-Rad23 complex regulates glycoprotein turnover. The Journal of Cell Biology. 172, 211-219 (2006).
- Fisher, E. A., et al. The degradation of apolipoprotein B100 is mediated by the ubiquitin-proteasome pathway and involves heat shock protein 70. The Journal of Biological Chemistry. 272, 20427-20434 (1997).
- Pariyarath, R., et al. Co-translational interactions of apoprotein B with the ribosome and translocon during lipoprotein assembly or targeting to the proteasome. The Journal of Biological Chemistry. 276, 541-550 (2001).
- Yeung, S. J., Chen, S. H., Chan, L. Ubiquitin-proteasome pathway mediates intracellular degradation of apolipoprotein. B. Biochemistry. 35, 13843-13848 (1996).
- Mumberg, D., Muller, R., Funk, M. Regulatable promoters of Saccharomyces cerevisiae: comparison of transcriptional activity and their use for heterologous expression. Nucleic Acids Research. 22, 5767-5768 (1994).
- Bruckner, A., Polge, C., Lentze, N., Auerbach, D., Schlattner, U. Yeast two-hybrid, a powerful tool for systems biology. International Journal of Molecular Sciences. 10, 2763-2788 (2009).
- Amberg, D. C., Burke, D., Strathern, J. N., Burke, D. . Methods in Yeast Genetics: A Cold Spring Harbor Laboratory Course Manual. , (2005).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: a primer on the Saccharomyces cerevisiae model system. Genetics. 197, 33-48 (2014).
- Guthrie, C., Fink, G. R. . Guide to Yeast Genetics and Molecular and Cell Biology. , (2004).
- Sherman, F. Getting started with yeast. Methods in Enzymology. 350, 3-41 (2002).
- Xie, Y., Rubenstein, E. M., Matt, T., Hochstrasser, M. SUMO-independent in vivo activity of a SUMO-targeted ubiquitin ligase toward a short-lived transcription factor. Genes & Development. 24, 893-903 (2010).
- Ravid, T., Kreft, S. G., Hochstrasser, M. Membrane and soluble substrates of the Doa10 ubiquitin ligase are degraded by distinct pathways. The EMBO Journal. 25, 533-543 (2006).
- Metzger, M. B., Maurer, M. J., Dancy, B. M., Michaelis, S. Degradation of a cytosolic protein requires endoplasmic reticulum-associated degradation machinery. The Journal of Biological Chemistry. 283, 32302-32316 (2008).
- Medicherla, B., Kostova, Z., Schaefer, A., Wolf, D. H. A genomic screen identifies Dsk2p and Rad23p as essential components of ER-associated degradation. EMBO Reports. 5, 692-697 (2004).
- Kohlmann, S., Schafer, A., Wolf, D. H. Ubiquitin ligase Hul5 is required for fragment-specific substrate degradation in endoplasmic reticulum-associated degradation. The Journal of Biological Chemistry. 283, 16374-16383 (2008).
- Crouse, G. F. Mutagenesis assays in yeast. Methods. 22, 116-119 (2000).
- Le Douarin, B., Pierrat, B., vom Baur, E., Chambon, F., Losson, R. A new version of the two-hybrid assay for detection of protein-protein interactions. Nucleic Acids Research. 23, 876-878 (1995).
- Boeke, J. D., Trueheart, J., Natsoulis, G., Fink, G. R. 5-Fluoroorotic acid as a selective agent in yeast molecular genetics. Methods in Enzymology. 154, 164-175 (1987).
- Toyn, J. H., Gunyuzlu, P. L., White, W. H., Thompson, L. A., Hollis, G. F. A counterselection for the tryptophan pathway in yeast: 5-fluoroanthranilic acid resistance. Yeast. 16, 553-560 (2000).
- Schafer, A., Wolf, D. H. Endoplasmic reticulum-associated protein quality control and degradation: genome-wide screen for ERAD components. Methods in Molecular Biology. 301, 289-292 (2005).
- Griggs, D. W., Johnston, M. Regulated expression of the GAL4 activator gene in yeast provides a sensitive genetic switch for glucose repression. Proceedings of the National Academy of Sciences of the United States of America. 88, 8597-8601 (1991).
- Duennwald, M. L. Growth assays to assess polyglutamine toxicity in yeast. The Journal of Visualized Experiments. (61), e3791 (2012).
- Baryshnikova, A., et al. Synthetic genetic array (SGA) analysis in Saccharomyces cerevisiae and Schizosaccharomyces pombe. Methods in Enzymology. 470, 145-179 (2010).
- Szymanski, E. P., Kerscher, O. Budding yeast protein extraction and purification for the study of function, interactions, and post-translational modifications. The Journal of Visualized Experiments. (80), e50921 (2013).
- Hjelm, H., Sjodahl, J., Sjoquist, J. Immunologically active and structurally similar fragments of protein A from Staphylococcus aureus. European Journal of Biochemistry. 57, 395-403 (1975).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved