Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détermination basée sur la croissance et la confirmation biochimique des exigences génétiques pour la dégradation des protéines dans
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
This article describes a yeast growth-based assay for the determination of genetic requirements for protein degradation. It also demonstrates a method for rapid extraction of yeast proteins, suitable for western blotting to biochemically confirm degradation requirements. These techniques can be adapted to monitor degradation of a variety of proteins.
Résumé
La dégradation des protéines réglementé est crucial pour pratiquement toutes les fonctions cellulaires. Une grande partie de ce qui est connu sur les mécanismes moléculaires et génétiques exigences pour la dégradation des protéines eucaryotes a été initialement créé en Saccharomyces cerevisiae. Analyses classiques de la dégradation des protéines se sont appuyés sur pulse-chase et cycloheximide-chase méthodes biochimiques. Bien que ces techniques fournissent des moyens sensibles pour observer la dégradation des protéines, ils sont laborieux, de temps, et à faible débit. Ces approches ne sont pas aptes à la sélection rapide ou à grande échelle pour des mutations qui empêchent la dégradation des protéines. Ici, un test basé sur une croissance de la levure pour l'identification facile des exigences génétiques pour la dégradation des protéines est décrite. Dans ce dosage, une enzyme rapporteur nécessaire à la croissance dans des conditions sélectives spécifiques est fusionnée à une protéine instable. Les cellules dépourvues de l'enzyme rapporteur endogène mais qui expriment la protéine de fusion peut se développer sous sélective conditions que lorsque la protéine de fusion est stabilisé (lorsque la dégradation des protéines est compromise). Dans l'essai de croissance décrit ici, des dilutions en série de type sauvage et des cellules de levure mutantes qui hébergent un plasmide codant pour une protéine de fusion sont déposés sur un milieu sélectif et non-sélectif. Croissance dans des conditions sélectives est compatible avec la dégradation dépréciation par une mutation donnée. L'abondance de la protéine accrue devrait être confirmé biochimiquement. Procédé pour l'extraction rapide de protéines de levure sous une forme appropriée pour l'électrophorèse et transfert de Western est également démontrée. Une lecture basée sur la croissance pour la stabilité de la protéine, combinée avec un simple protocole d'extraction de protéines pour l'analyse biochimique, facilite l'identification rapide des exigences génétiques pour la dégradation des protéines. Ces techniques peuvent être adaptées pour surveiller la dégradation d'une variété de protéines de courte durée. Dans l'exemple présenté, l'enzyme His3, qui est nécessaire pour la biosynthèse de l'histidine, a été fusionnéà DEG1 -Sec62. DEG1 -Sec62 est prévue pour dégradation après ce qu'il se engage aberrante le réticulum endoplasmique translocon. Cellules hébergeant DEG1 -Sec62-His3 sont capables de croître dans des conditions sélectives lorsque la protéine a été stabilisé.
Introduction
La dégradation sélective des protéines est essentielle à la vie eucaryote, et la dégradation de la protéine altérée contribue à un certain nombre de maladies, y compris plusieurs types de cancer, les maladies neurodégénératives, les maladies cardiovasculaires, et la fibrose kystique 1-5. Le système ubiquitine-protéasome (UPS), qui catalyse la dégradation sélective des protéines, est une cible thérapeutique émergeant de ces conditions 6-10. ubiquitine ligases attachent de manière covalente des polymères de l'acide 76-ubiquitine aminés aux protéines 11. Les protéines qui ont été marqués avec des chaînes de polyubiquitine sont reconnus et protéolysée par les ~ 2,5 mégadaltons protéasome 26S 12. Études initiées dans l'organisme eucaryote modèle Saccharomyces cerevisiae (levure en herbe) ont été fondamental dans l'élucidation des mécanismes de dégradation des protéines dans les cellules eucaryotes. Le premier substrat physiologique démontré de l'onduleur était la levure répresseur transcriptionnel MATα2 13, 14, et de nombreux composants hautement conservées de la première UPS ont été identifiés ou caractérisés dans la levure (par exemple 15-26). Les découvertes faites dans cet organisme modèle polyvalent et génétiquement traitable sont susceptibles de continuer à fournir des informations importantes sur les mécanismes conservés de la dégradation des ubiquitine.
Reconnaissance et de la dégradation de la plupart des substrats UPS requièrent une action concertée de plusieurs protéines. Par conséquent, un objectif important dans la caractérisation de la dégradation régulée d'une protéine donnée est instable pour déterminer les exigences génétiques pour la protéolyse. Les approches classiques (par exemple pulse-chase et expériences cycloheximide-chase 27) pour surveiller la dégradation des protéines dans des cellules de mammifère ou de levure sont laborieuses et prennent du temps. Bien que ces types de méthodologie fournissent des moyens très sensibles pour détecter la dégradation des protéines, ils ne conviennent pas pour l'analyse rapide de la dégradation des protéines ou à grande échelle screening pour les mutations qui empêchent la dégradation des protéines. Ici, une analyse basée sur la croissance de la levure pour l'identification rapide des exigences génétiques pour la dégradation des protéines instables est présentée.
Dans la méthode de la croissance de la levure pour l'analyse de la dégradation des protéines, une protéine instable d'intérêt (ou un signal de dégradation) est fusionnée, en cadre, à une protéine qui est nécessaire pour la croissance de la levure dans des circonstances particulières. Le résultat est un substrat artificiel qui peut servir comme un outil puissant pour déterminer les exigences génétiques de la dégradation des protéines de la protéine d'intérêt instable. Idéalement, les souches de levure de laboratoire les plus couramment utilisés abritent un panel de mutations dans les gènes codant pour des enzymes métaboliques impliquées dans la biosynthèse de certains acides aminés ou de bases azotées (par exemple, 20,28-30). Ces enzymes sont essentiels pour la prolifération cellulaire en l'absence de métabolites dans exogène prévues dont la synthèse des enzymes participent. Telenzymes métaboliques peuvent ainsi fonctionner comme rapporteurs basées sur la croissance de la dégradation des protéines instables à laquelle ils sont fusionnés. Les exigences génétiques pour la dégradation des protéines peuvent être aisément élucidés, puisque des mutations qui empêchent la protéolyse permettront cellules hébergeant le reporter de dégradation de se développer dans des conditions sélectives.
Un avantage de croissance est une indication indirecte qu'une mutation particulier augmente l'abondance de la protéine d'intérêt. Cependant, l'analyse biochimique directe est nécessaire pour confirmer qu'une mutation permet la croissance à travers des niveaux accrus en protéines plutôt que par des causes indirectes ou artefacts. L'effet d'une mutation sur la protéine abondance peut être confirmée par analyse western blot des niveaux de protéines à l'état stable dans les cellules qui font et ne abritent la mutation particulière. Procédé pour l'extraction rapide et efficace de protéines de levure (incubation séquentielle de cellules de levure avec de l'hydroxyde de sodium et un tampon échantillon) sous une forme adaptéepour analyse par western blot est également présenté 31. Ensemble, ces expériences faciliter l'identification rapide des régulateurs de candidats de la dégradation des protéines.
Protocole
1. levure croissance essai pour identifier des mutants candidats défectueux dans la dégradation des protéines
- Transform de type sauvage et des cellules de levure mutantes avec un plasmide codant pour une protéine fusionnée instable, en cadre, à une enzyme reporter métabolique.
- Ensemencer transformants dans 5 ml de synthèse défini (SD) de milieu minimal qui est sélectif pour les cellules portant des molécules plasmidiques. Incuber pendant une nuit à 30 ° C, en rotation.
- Mesurer la densité optique à 600 nm (DO600) de chaque culture d'une nuit.
REMARQUE: Après incubation pendant une nuit, les cellules en culture peut être soit en phase de croissance logarithmique ou stationnaire, mais devrait avoir atteint une DO 600 de 0,2 minimal. Souches de levure à croissance très lente peuvent nécessiter des temps d'incubation plus d'une nuit, ou l'inoculation d'un plus grand nombre de cellules, tel que déterminé de manière empirique. - Préparer six dilutions en série de cellules de levure transformées dans une plaque à 96 puits stériles, à partir de cellules diluées à unen DO600 de 0,2. Placez chaque transformant de levure à doser dans un enregistrement différent de la plaque de 96 puits.
- Pour chaque transformant, on calcule le volume de culture d'une nuit nécessaire pour diluer les cellules à une DO 600 de 0,2 dans un volume final de 200 ul. Ajouter ce volume de culture de la nuit à l'puits correspondant dans la colonne 1. Ajouter la quantité appropriée de l'eau stérile pour porter le volume à 200 pi.
- Pour chaque ligne de la levure, ajouter 125 pi d'eau stérile pour les puits dans les colonnes 2, 3 et 4.
REMARQUE: des plaques à 96 puits stériles emballés individuellement peuvent être emballés avec des couvercles stériles. Les couvercles peuvent être utilisés comme réservoirs pour l'eau stérile qui est répartie dans cette étape. Ceci permet un transfert simultané de l'eau stérile à tous les puits dans une colonne donnée avec une pipette à canaux multiples. - Mélanger le contenu de la première colonne (levure diluée jusqu'à une DO 600 de 0,2) par pipetage de haut en bas avec une pipette à canaux multiples.
- Transfer 25 pi de levure de la colonne 1 à la colonne 2, en utilisant une pipette multicanaux. Mélanger en pipetant. Transfert 25 ul de la colonne 2 à la colonne 3, et 25 pi de la colonne 3 à la colonne 4 (en mélangeant bien à chaque étape).
- Mélanger chaque échantillon avec une pipette multicanaux. Partant de la plus diluée pour diluer moins colonnes de levure, une pipette 4 pi de chaque échantillon sur deux plaques contenant le milieu sélectif approprié. Utilisez une plaque avec un milieu qui maintient sélection de plasmide (cette plaque sert de taches de levure et le contrôle de la croissance). Utiliser une seconde plaque avec un milieu qui sélectionne pour le maintien du plasmide et l'expression de la protéine instable fusionné à l'enzyme rapporteur. Parce que la levure se installer rapidement, mélanger les cellules par pipetage de haut en bas à intervalles réguliers.
NOTE: Séchoir plaques seront plus facilement absorber le liquide que les plaques fraîchement préparés et sont donc recommandés pour ces expériences. Plaques humides peuvent être séchés par incubation à température ambiante dans lol'humidité w pour 1-2 jours ou incubations courtes dans une hotte à flux laminaire. Plaques peuvent sécher inégalement si le flux d'air laminaire est parallèle à la magistrature. Utilisation d'un gabarit, il est plus facile de repérer les cellules de levure à des distances régulières. Deux exemples de modèles sont fournis à la figure 1. Ceux-ci peuvent être imprimés, découpés et fixés à l'intérieur d'une boîte de Petri couvercle. - Permettre aux plaques à sécher sur la paillasse.
- Incuber les plaques à 30 ° C pendant 2-6 jours.
- Photographier chaque plaque après incubation.
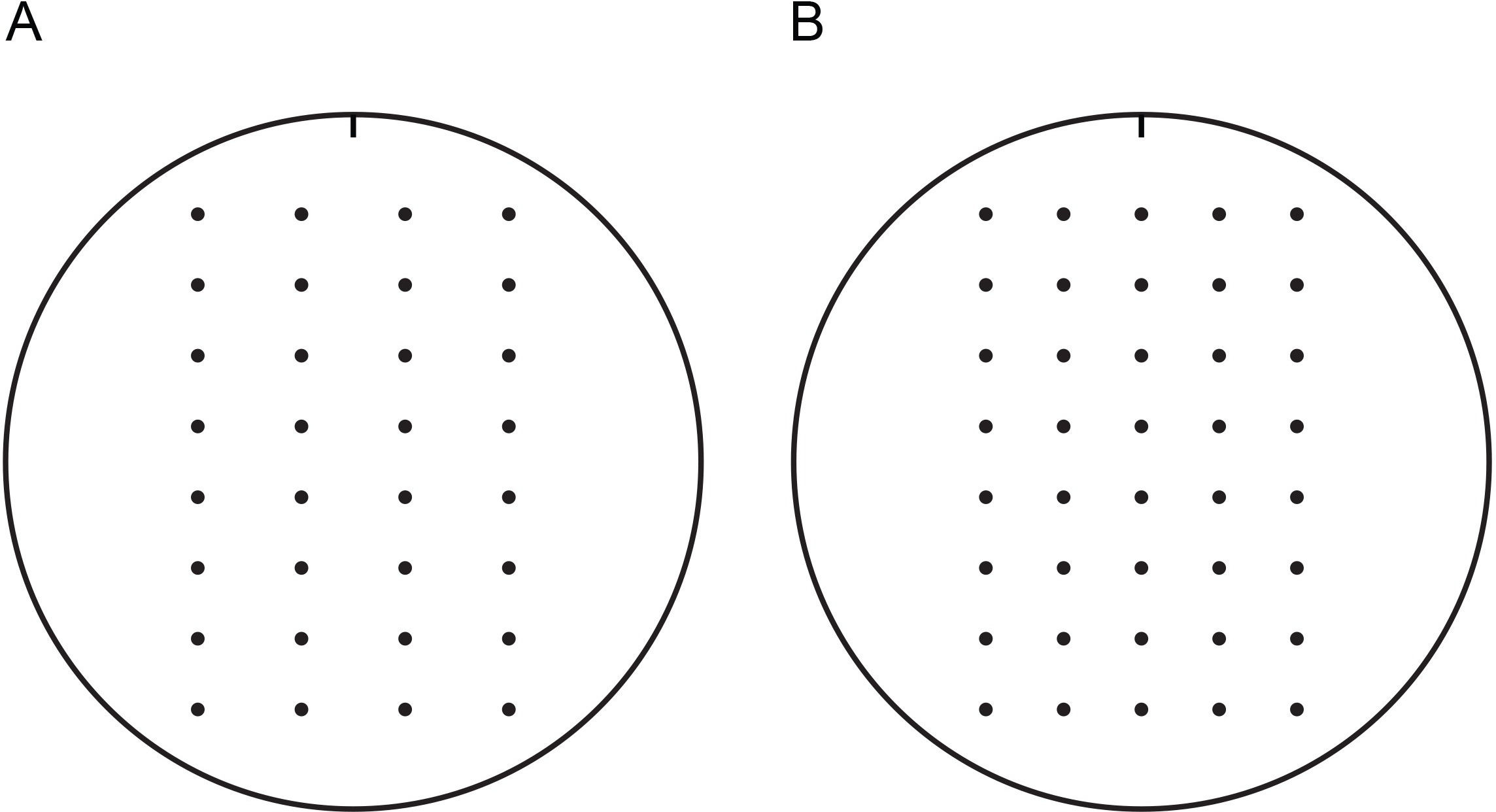
Figure 1. Modèles pour repérer des cellules de levure sur des plaques d'agar de 100 mm. Ces modèles peuvent être utilisés pour faciliter repérer la levure à des distances régulières avec une pipette multicanaux. Les modèles peuvent être imprimés, découpés et fixés à l'intérieur d'une boîte de Petri couvercle. Placez boîte de Pétri avec la croissanceintérieur du couvercle moyenne avec modèle apposé. Les modèles sont marqués d'une encoche pour suivre l'orientation. Il est recommandé que les plaques utilisées dans des dosages de croissance être marqués de manière similaire avec une encoche pour suivre l'orientation. Modèles pour repérer quatre (A) ou cinq (B) des dilutions en série de cellules de levure sont fournis. Se il vous plaît cliquez ici pour afficher une version imprimable de ce chiffre avec 100 mm modèles.
2. Confirmation biochimique de croissance de la levure Assay
- La croissance des cellules de levure et d'extraction de protéines post-alcaline (modifiée du 31)
- Transform de type sauvage et des cellules de levure mutantes avec un plasmide codant pour la protéine instable.
- Ensemencer les transformants dans 5 ml de milieu SD qui est sélectif pour les cellules hébergeant molécules de plasmide. Incuber pendant une nuit à 30 ° C, en rotation.
- Mesurer la DO 600 de chaque cu nuitlture.
REMARQUE: Après incubation pendant une nuit, les cellules peuvent être soit en phase de croissance logarithmique ou stationnaire, mais devrait avoir atteint une DO 600 qui permettra dilution jusqu'à une DO 600 de 0,2 à 10 ml de milieu sélectif frais (étape 2.1.4). Souches de levure à croissance très lente peuvent nécessiter des temps d'incubation plus d'une nuit, ou l'inoculation d'un plus grand nombre de cellules, tel que déterminé de manière empirique. - Diluer les cellules de levure à une DO 600 de 0,2 à 10 ml de milieu sélectif frais.
- Continuer à incuber les cellules à 30 ° C, la rotation ou l'agitant, jusqu'à cultures atteignent une DO 600 entre 0,8 et 1,2 (ce est à dire sont en croissance mi-logarithmique).
NOTE: Si la protéine d'intérêt est instable sous le contrôle d'un promoteur régulable, le moment optimal pour l'induction de l'expression de la protéine et la récolte des cellules peut varier selon les études antérieures ou des observations empiriques. - Recueillir 2,5 OD 600 unités de la culture dans un 15 ml coniqueal tuyau par centrifugation à 5000 xg pendant 5 min à température ambiante. Retirer le surnageant par la pipette ou l'aspiration.
REMARQUE: Un DO600 unité est définie comme la quantité de levure présente dans 1 ml de culture à DO 600 de 1,0. Le volume de la culture (en ml) nécessaire pour récolter 2,5 OD 600 unités (V) peut être déterminée en utilisant l'équation suivante: V = 2,5 unités DO 600 / DO mesurée 600 - Resuspendre les cellules dans 1 ml d'eau distillée. Transfert cellules en suspension dans un tube à centrifuger.
- Pellet les cellules par centrifugation à 6500 xg pendant 30 sec à température ambiante. Retirer le surnageant par la pipette ou l'aspiration.
- Resuspendre les cellules dans 100 pi d'eau distillée à la pipette de haut en bas ou de vortex, et ajouter 100 ul NaOH 0,2 M. Mélanger en pipetant. Incuber les échantillons pendant 5 min à température ambiante.
- cellules Pellet (dont la plupart ne ont pas encore libérés des protéines et sont encore viables) par centrifugation à 18 000 xg pendant 5 min. Retirer le surnageant par la pipette ou l'aspiration.
- Remettre en suspension le culot dans 50 - 100 pi de tampon d'échantillon Laemmli 1x, qui lyser les cellules, par pipetage de haut en bas ou vortex.
NOTE: L'élimination du surnageant alcalin après la centrifugation et remise en suspension ultérieure des cellules dans du tampon d'échantillon de Laemmli extrait de protéines à un pH compatible avec dodécylsulfate de sodium électrophorèse sur gel de sulfate-polyacrylamide (SDS-PAGE) en utilisant un système tampon de roulement Tris-glycine et western blotting. - Pour dénaturer complètement protéines, incuber les lysats à 95 ° C pendant 5 min.
NOTE: Agrégation protéines-protéines exposées (par exemple avec plusieurs segments transmembranaires) peuvent devenir insolubles lorsqu'ils sont incubés à 95 ° C. Par conséquent, les lysats doivent être incubées à des températures plus basses (par exemple 37 ° C - 70 ° C) pendant 10 à 30 min, comme déterminé empiriquement, pour l'analyse de ces protéines. - Lysats frais en le plaçant sur la glace pendant 5 min.
- Lysats centrifuger à 18 000 g pendant 1 min à température ambiante pour obtenir un culot insoluble. Séparer le surnageant (extrait de protéine solubilisée) par SDS-PAGE avant l'analyse western blot ultérieure (section 2.2). Alternativement, lysats de conserver à -20 ° C.
- Représentant Protocole Western Blot
- Charger le volume déterminé de manière empirique des lysats dans un gel de SDS-PAGE.
- Exécuter gel à 200 V jusqu'à ce que le colorant avant a atteint le fond du gel.
- Transfert de protéines à partir de gel de fluorure de polyvinylidène (PVDF) par transfert humide à 20 V pendant 60 à 90 min à 4 ° C.
- Bloquer membrane par incubation dans 5% de lait écrémé dans du Tris-solution saline tamponnée (TBS), bascule, pendant 1 heure à température ambiante ou une nuit à 4 ° C.
- Décanter la solution de blocage.
- Incuber la membrane avec l'anticorps primaire spécifique pour la protéine d'intérêt (ou épitope tag de celui-ci) dans 1% de lait écrémé dans du TBS avec 0,1% de Tween-20 (TBS / T) pendant 1 h à la salle température, bascule.
- Décanter solution d'anticorps, et laver membrane 3 x 5 min avec le SCT / T à la température ambiante, à bascule.
- Incuber la membrane avec un anticorps secondaire conjugué à un fluorophore approprié dans 1% de lait écrémé dans du TBS / T pendant 1 heure à la température ambiante, à bascule.
NOTE: En raison fluorophores sont sensibles à la lumière, des dilutions d'anticorps fluorophores conjugué doivent être préparés dans l'obscurité. En outre, l'incubation de membranes en présence d'anticorps conjugué à un fluorophore doit se produire dans des récipients étanches à la lumière. Ceci peut être réalisé en enroulant plateaux d'incubation dans de l'aluminium. - Décanter solution d'anticorps, et laver membrane 3 x 5 min avec le SCT / T à la température ambiante, à bascule.
- Acquérir image de membrane en utilisant Li-Cor Odyssey CLx et logiciel Image Studio (ou de l'équipement d'imagerie comparables et logiciels), selon les recommandations du fabricant.
- Après l'imagerie membrane, incuber la membrane avec un anticorps primaire spécifique d'une chargeing protéine de contrôle dans 1% de lait écrémé dans du TBS / T pendant 1 heure à température ambiante, à bascule.
- Décanter solution d'anticorps, et laver membrane 3 x 5 min avec le SCT / T à la température ambiante, à bascule.
- Incuber la membrane avec un anticorps secondaire conjugué à un fluorophore approprié dans 1% de lait écrémé dans du TBS / T pendant 1 heure à la température ambiante, à bascule.
- Décanter solution d'anticorps, et laver membrane 3 x 5 min avec le SCT / T à la température ambiante, à bascule.
- Acquérir image de membrane en utilisant Li-Cor Odyssey CLx et logiciel Image Studio (ou de l'équipement d'imagerie comparables et logiciels), selon les recommandations du fabricant.
Résultats
Pour illustrer cette méthode, l'enzyme His3 a été fusionné à l'extrémité carboxy-terminale du modèle réticulum endoplasmique (RE) associée au gène de dégradation (ERAD) substrat, DEG1 -Sec62 (figure 2A) pour créer DEG1 -Sec62-His3 (Figure 3) . DEG1 -Sec62 représente un membre fondateur d'une nouvelle classe de substrats ERAD qui sont ciblées suivant, les association persistante aberrante avec le translocon, le canal principal respons...
Discussion
La méthodologie présentée ici permet la détermination rapide et confirmation biochimique des exigences génétiques pour la dégradation des protéines dans des cellules de levure. Ces expériences mettent en évidence l'utilité et la puissance de la levure comme modèle organisme eucaryote (plusieurs excellentes critiques de levure biologie et compilations de protocoles pour la manipulation, le stockage et la manipulation de cellules de levure (par exemple 41-44) sont disponibles pour les e...
Déclarations de divulgation
Les auteurs ne ont rien à divulguer.
Remerciements
Nous remercions les membres actuels et anciens du laboratoire Rubenstein pour fournir un environnement de recherche et enthousiaste. Nous remercions Ryan T. Gibson pour l'assistance dans l'optimisation de protocole. Nous remercions Mark Hochstrasser (Université de Yale) et Dieter Wolf (Universität Stuttgart) pour les souches de levures et plasmides. Nous remercions nos lecteurs anonymes pour leur aide dans l'amélioration de la clarté et l'utilité de ce manuscrit. Ce travail a été soutenu par une bourse de recherche de la section de l'Université Ball State de Sigma Xi à SGW, un National Institutes of Health subvention (R15 GM111713) au DME, une bourse de recherche de Ball State University aspirent à DME, et les fonds de l'Université Ball State Bureau du prévôt et Département de biologie.
matériels
| Name | Company | Catalog Number | Comments |
| Desired yeast strains, plasmids, standard medium and buffer components | Yeast strains with desired mutations may be generated in the investigator's laboratory. Wild-type yeast and a variety of mutants are also commercially available (e.g. from GE Healthcare). Plasmids encoding fusion proteins may be generated in the investigator's laboratory. | ||
| 3-amino-1H-1,2,4-triazole | Fisher Scientific | AC264571000 | Competitive inhibitor of His3 enzyme. May be included in medium to increase stringency of growth assay using His3 reporter constructs. |
| Endoglycosidase H (recombinant form from Streptomyces plicatus) | Roche | 11088726001 | May be used to assess N-glycosylation of proteins; compatible with SDS and beta-mercaptoethanol concentrations found in 1x Laemmli sample buffer. |
| Disposable borosilicate glass tubes | Fisher Scientific | 14-961-32 | Available from a variety of manufacturers |
| Temperature-regulated incubator (e.g. Heratherm Incubator Model IMH180) | Dot Scientific | 51028068 | Available from a variety of manufacturers |
| New Brunswick Interchangeable Drum for 18 mm tubes (tube roller) | New Brunswick | M1053-0450 | Tube roller is recommended to maintain overnight yeast starter cultures of yeast cells in suspension. A platform shaker or tube roller may be used to maintain larger cultures in suspension. |
| New Brunswick TC-7 Roller Drum 120V 50/60 H | New Brunswick | M1053-4004 | For use with tube roller |
| SmartSpec Plus Spectrophotometer | Bio-Rad | 170-2525 | Available from a variety of manufacturers |
| Sterile 96-well flat bottom microtest plates with lid individually wrapped | Sarstedt | 82.1581.001 | Available from a variety of manufacturers |
| Pipetman Neo P8x20N, 2-20 μl | Gilson | F14401 | Available from a variety of manufacturers |
| [header] | |||
| Pipetman Neo P8x200N, 20-200 μl | Gilson | F14403 | Single-channel and multichannel pipettors are used at various stages of the protocol. While multichannel pipettors reduce the pipetting burden at several steps, single-channel pipettors may be used throughout the entire protocol. Available from a variety of manufacturers. |
| Centrifuge 5430 | Eppendorf | 5427 000.216 | Rotor that is sold with unit holds 1.5 and 2.0 ml microcentrifuge tubes. Rotor may be swapped for one that holds 15 ml and 50 ml conical tubes. |
| Plate imaging system (e.g. Gel Doc XR+ System) | Bio-Rad | 170-8195 | A variety of systems may be used to image plates, including sophisticated imaging systems, computer scanners, and camera phones. |
| Fixed-Angle Rotor F-35-6-30 with Lid and Adapters for Centrifuge Model 5430/R, 15/50 ml Conical Tubes, 6-Place | Eppendorf | F-35-6-30 | |
| 15 ml screen printed screw cap tube 17 x 20 mm conical, polypropylene | Sarstedt | 62.554.205 | Available from a variety of manufacturers |
| 1.5 ml flex-tube, PCR clean, Natural microcentrifuge tubes | Eppendorf | 22364120 | Available from a variety of manufacturers |
| Analog Dri-Bath Heater | Fisher Scientific | 1172011AQ | Boiling water bath with hot plate may also be used to denature proteins |
| SDS-PAGE running and transfer apparatuses, power supplies, and imaging equipment or darkrooms for SDS-PAGE and transfer to membrane | Will vary by lab and application | ||
| Western blot imaging system (e.g. Li-Cor Odyssey CLx scanner and Image Studio Software) | Li-Cor | 9140-01 | Will vary by lab and application |
| EMD Millipore Immobilon PVDF Transfer Membranes | Fisher Scientific | IPFL00010 | Will vary by lab and application |
| Primary antibodies (e.g. Phosphoglycerate Kinase (Pgk1) Monoclonal antibody, mouse (clone 22C5D8)) | Life Technologies | 459250 | Will vary by lab and application |
| Secondary antibodies (e.g. Alexa-Fluor 680 Rabbit Anti-Mouse IgG (H+L)) | Life Technologies | A-21065 | Will vary by lab and application |
Références
- Goldberg, A. L. Protein degradation and protection against misfolded or damaged proteins. Nature. 426, 895-899 (2003).
- Guerriero, C. J., Brodsky, J. L. The delicate balance between secreted protein folding and endoplasmic reticulum-associated degradation in human physiology. Physiological Reviews. 92, 537-576 (2012).
- Pagan, J., Seto, T., Pagano, M., Cittadini, A. Role of the ubiquitin proteasome system in the heart. Circulation Research. 112, 1046-1058 (2013).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443, 780-786 (2006).
- Turnbull, E. L., Rosser, M. F., Cyr, D. M. The role of the UPS in cystic fibrosis. BMC Biochemistry. 8, S11 (2007).
- Bedford, L., Lowe, J., Dick, L. R., Mayer, R. J., Brownell, J. E. Ubiquitin-like protein conjugation and the ubiquitin-proteasome system as drug targets. Nature Reviews Drug Discovery. 10, 29-46 (2011).
- Kar, G., Keskin, O., Fraternali, F., Gursoy, A. Emerging role of the ubiquitin-proteasome system as drug targets. Current Pharmaceutical Design. 19, 3175-3189 (2013).
- Paul, S. Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches. BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology. 30, 1172-1184 (2008).
- Shen, M., Schmitt, S., Buac, D., Dou, Q. P. Targeting the ubiquitin-proteasome system for cancer therapy. Expert Opinion on Therapeutic Targets. 17 (9), 1091-1108 (2013).
- Finley, D., Ulrich, H. D., Sommer, T., Kaiser, P. The ubiquitin-proteasome system of Saccharomyces cerevisiae. Genetics. 192, 319-360 (2012).
- Scheffner, M., Nuber, U., Huibregtse, J. M. Protein ubiquitination involving an E1-E2-E3 enzyme ubiquitin thioester cascade. Nature. 373, 81-83 (1995).
- Ravid, T., Hochstrasser, M. Diversity of degradation signals in the ubiquitin-proteasome system. Nature Reviews Molecular Cell Biology. 9, 679-690 (2008).
- Hochstrasser, M., Ellison, M. J., Chau, V., Varshavsky, A. The short-lived MAT alpha 2 transcriptional regulator is ubiquitinated in vivo. Proceedings of the National Academy of Sciences of the United States of America. 88, 4606-4610 (1991).
- Hochstrasser, M., Varshavsky, A. In vivo degradation of a transcriptional regulator: the yeast alpha 2 repressor. Cell. 61, 697-708 (1990).
- Bays, N. W., Gardner, R. G., Seelig, L. P., Joazeiro, C. A., Hampton, R. Y. Hrd1p/Der3p is a membrane-anchored ubiquitin ligase required for ER-associated degradation. Nature Cell Biology. 3, 24-29 (2001).
- Hampton, R. Y., Gardner, R. G., Rine, J. Role of 26S proteasome and HRD genes in the degradation of 3-hydroxy-3-methylglutaryl-CoA reductase, an integral endoplasmic reticulum membrane protein. Molecular Biology of the Cell. 7, 2029-2044 (1996).
- Swanson, R., Locher, M., Hochstrasser, M. A conserved ubiquitin ligase of the nuclear envelope/endoplasmic reticulum that functions in both ER-associated and Matalpha2 repressor degradation. Genes & Development. 15, 2660-2674 (2001).
- Goebl, M. G., et al. The yeast cell cycle gene CDC34 encodes a ubiquitin-conjugating enzyme. Science. 241, 1331-1335 (1988).
- Bachmair, A., Finley, D., Varshavsky, A. In vivo half-life of a protein is a function of its amino-terminal residue. Science. 234, 179-186 (1986).
- Chen, P., Johnson, P., Sommer, T., Jentsch, S., Hochstrasser, M. Multiple ubiquitin-conjugating enzymes participate in the in vivo degradation of the yeast MAT alpha 2 repressor. Cell. 74, 357-369 (1993).
- Varshavsky, A. Discovery of the biology of the ubiquitin system. JAMA: The Journal of the American Medical Association. 311, 1969-1970 (2014).
- Heinemeyer, W., Kleinschmidt, J. A., Saidowsky, J., Escher, C., Wolf, D. H. Proteinase yscE, the yeast proteasome/multicatalytic-multifunctional proteinase: mutants unravel its function in stress induced proteolysis and uncover its necessity for cell survival. The EMBO Journal. 10, 555-562 (1991).
- Sommer, T., Jentsch, S. A protein translocation defect linked to ubiquitin conjugation at the endoplasmic reticulum. Nature. 365, 176-179 (1993).
- Hiller, M. M., Finger, A., Schweiger, M., Wolf, D. H. ER degradation of a misfolded luminal protein by the cytosolic ubiquitin-proteasome pathway. Science. 273, 1725-1728 (1996).
- Seufert, W., Jentsch, S. In vivo function of the proteasome in the ubiquitin pathway. The EMBO Journal. 11, 3077-3080 (1992).
- Knop, M., Finger, A., Braun, T., Hellmuth, K., Wolf, D. H. Der1, a novel protein specifically required for endoplasmic reticulum degradation in yeast. The EMBO Journal. 15, 753-763 (1996).
- Zattas, D., Adle, D. J., Rubenstein, E. M., Hochstrasser, M. N-terminal acetylation of the yeast Derlin Der1 is essential for Hrd1 ubiquitin-ligase activity toward luminal ER substrates. Molecular Biology of the Cell. 24, 890-900 (2013).
- Brachmann, C. B., et al. Designer deletion strains derived from Saccharomyces cerevisiae S288C: a useful set of strains and plasmids for PCR-mediated gene disruption and other applications. Yeast. 14, 115-132 (1998).
- Sikorski, R. S., Hieter, P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae. Genetics. 122, 19-27 (1989).
- Ralser, M., et al. The Saccharomyces cerevisiae W303-K6001 cross-platform genome sequence: insights into ancestry and physiology of a laboratory mutt. Open Biology. 2, 120093 (2012).
- Kushnirov, V. V. Rapid and reliable protein extraction from yeast. Yeast. 16, 857-860 (2000).
- Rubenstein, E. M., Kreft, S. G., Greenblatt, W., Swanson, R., Hochstrasser, M. Aberrant substrate engagement of the ER translocon triggers degradation by the Hrd1 ubiquitin ligase. The Journal of Cell Biology. 197, 761-773 (2012).
- Mayer, T. U., Braun, T., Jentsch, S. Role of the proteasome in membrane extraction of a short-lived ER-transmembrane protein. The EMBO Journal. 17, 3251-3257 (1998).
- Scott, D. C., Schekman, R. Role of Sec61p in the ER-associated degradation of short-lived transmembrane proteins. The Journal of Cell Biology. 181, 1095-1105 (2008).
- Kim, I., et al. The Png1-Rad23 complex regulates glycoprotein turnover. The Journal of Cell Biology. 172, 211-219 (2006).
- Fisher, E. A., et al. The degradation of apolipoprotein B100 is mediated by the ubiquitin-proteasome pathway and involves heat shock protein 70. The Journal of Biological Chemistry. 272, 20427-20434 (1997).
- Pariyarath, R., et al. Co-translational interactions of apoprotein B with the ribosome and translocon during lipoprotein assembly or targeting to the proteasome. The Journal of Biological Chemistry. 276, 541-550 (2001).
- Yeung, S. J., Chen, S. H., Chan, L. Ubiquitin-proteasome pathway mediates intracellular degradation of apolipoprotein. B. Biochemistry. 35, 13843-13848 (1996).
- Mumberg, D., Muller, R., Funk, M. Regulatable promoters of Saccharomyces cerevisiae: comparison of transcriptional activity and their use for heterologous expression. Nucleic Acids Research. 22, 5767-5768 (1994).
- Bruckner, A., Polge, C., Lentze, N., Auerbach, D., Schlattner, U. Yeast two-hybrid, a powerful tool for systems biology. International Journal of Molecular Sciences. 10, 2763-2788 (2009).
- Amberg, D. C., Burke, D., Strathern, J. N., Burke, D. . Methods in Yeast Genetics: A Cold Spring Harbor Laboratory Course Manual. , (2005).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: a primer on the Saccharomyces cerevisiae model system. Genetics. 197, 33-48 (2014).
- Guthrie, C., Fink, G. R. . Guide to Yeast Genetics and Molecular and Cell Biology. , (2004).
- Sherman, F. Getting started with yeast. Methods in Enzymology. 350, 3-41 (2002).
- Xie, Y., Rubenstein, E. M., Matt, T., Hochstrasser, M. SUMO-independent in vivo activity of a SUMO-targeted ubiquitin ligase toward a short-lived transcription factor. Genes & Development. 24, 893-903 (2010).
- Ravid, T., Kreft, S. G., Hochstrasser, M. Membrane and soluble substrates of the Doa10 ubiquitin ligase are degraded by distinct pathways. The EMBO Journal. 25, 533-543 (2006).
- Metzger, M. B., Maurer, M. J., Dancy, B. M., Michaelis, S. Degradation of a cytosolic protein requires endoplasmic reticulum-associated degradation machinery. The Journal of Biological Chemistry. 283, 32302-32316 (2008).
- Medicherla, B., Kostova, Z., Schaefer, A., Wolf, D. H. A genomic screen identifies Dsk2p and Rad23p as essential components of ER-associated degradation. EMBO Reports. 5, 692-697 (2004).
- Kohlmann, S., Schafer, A., Wolf, D. H. Ubiquitin ligase Hul5 is required for fragment-specific substrate degradation in endoplasmic reticulum-associated degradation. The Journal of Biological Chemistry. 283, 16374-16383 (2008).
- Crouse, G. F. Mutagenesis assays in yeast. Methods. 22, 116-119 (2000).
- Le Douarin, B., Pierrat, B., vom Baur, E., Chambon, F., Losson, R. A new version of the two-hybrid assay for detection of protein-protein interactions. Nucleic Acids Research. 23, 876-878 (1995).
- Boeke, J. D., Trueheart, J., Natsoulis, G., Fink, G. R. 5-Fluoroorotic acid as a selective agent in yeast molecular genetics. Methods in Enzymology. 154, 164-175 (1987).
- Toyn, J. H., Gunyuzlu, P. L., White, W. H., Thompson, L. A., Hollis, G. F. A counterselection for the tryptophan pathway in yeast: 5-fluoroanthranilic acid resistance. Yeast. 16, 553-560 (2000).
- Schafer, A., Wolf, D. H. Endoplasmic reticulum-associated protein quality control and degradation: genome-wide screen for ERAD components. Methods in Molecular Biology. 301, 289-292 (2005).
- Griggs, D. W., Johnston, M. Regulated expression of the GAL4 activator gene in yeast provides a sensitive genetic switch for glucose repression. Proceedings of the National Academy of Sciences of the United States of America. 88, 8597-8601 (1991).
- Duennwald, M. L. Growth assays to assess polyglutamine toxicity in yeast. The Journal of Visualized Experiments. (61), e3791 (2012).
- Baryshnikova, A., et al. Synthetic genetic array (SGA) analysis in Saccharomyces cerevisiae and Schizosaccharomyces pombe. Methods in Enzymology. 470, 145-179 (2010).
- Szymanski, E. P., Kerscher, O. Budding yeast protein extraction and purification for the study of function, interactions, and post-translational modifications. The Journal of Visualized Experiments. (80), e50921 (2013).
- Hjelm, H., Sjodahl, J., Sjoquist, J. Immunologically active and structurally similar fragments of protein A from Staphylococcus aureus. European Journal of Biochemistry. 57, 395-403 (1975).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon