Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Growth-basierte Bestimmung und biochemische Bestätigung der genetischen Voraussetzungen für Proteinabbau in
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
This article describes a yeast growth-based assay for the determination of genetic requirements for protein degradation. It also demonstrates a method for rapid extraction of yeast proteins, suitable for western blotting to biochemically confirm degradation requirements. These techniques can be adapted to monitor degradation of a variety of proteins.
Zusammenfassung
Geregelten Proteinabbau ist entscheidend für praktisch jede Zellfunktion. Vieles von dem, was über die molekularen Mechanismen und genetischen Erfordernisse für eukaryotische Proteinabbau bekannt wurde zunächst in Saccharomyces cerevisiae hergestellt. Klassische Analysen der Proteinabbau haben biochemische Pulse-Chase und Cycloheximid-Chase-Methoden verlassen. Während diese Techniken empfindliche Mittel zur Beobachtung des Proteinabbaus sind mühsam, zeitaufwendig und niedrigem Durchsatz. Diese Ansätze sind nicht geeignet, um eine schnelle und großflächige Screening auf Mutationen, die den Proteinabbau zu verhindern. Hier wird ein Wachstum der Hefe-basierenden Assay für die leichte Identifizierung von genetischen Erfordernisse für den Proteinabbau beschrieben. In diesem Test wird ein Reporterenzym für das Wachstum unter bestimmten Selektionsbedingungen erforderlich, um ein instabiles Protein fusioniert ist. Zellen, denen das endogene Reporter-Enzym exprimieren, aber das Fusionsprotein unter sele wachsenctive Bedingungen nur, wenn das Fusionsprotein stabilisiert ist (dh, wenn der Proteinabbau gefährdet ist). Bei dem hier beschriebenen Wachstumstest werden Verdünnungsreihen von Wildtyp und mutierten Hefezellen ein Plasmid ein Fusionsprotein kodiert, auf selektiven und nicht-selektiven Medium entdeckt. Wachstum unter selektiven Bedingungen entspricht Abbau Beeinträchtigung von einer bestimmten Mutation. Erhöhte Proteinmenge sollte biochemisch bestätigt werden. Ein Verfahren zur schnellen Extraktion von Hefe-Proteinen in einer für die Elektrophorese und Western-Blotting Form wird auch gezeigt. Ein Wachstum basierende Auslesen für die Proteinstabilität, kombiniert mit einer einfachen Protokoll für die Proteinextraktion zur biochemischen Analyse, erleichtert die schnelle Identifizierung von genetischen Erfordernisse für den Proteinabbau. Diese Techniken können angepaßt, um den Abbau einer Vielzahl von kurzlebigen Proteinen zu überwachen. In dem dargestellten Beispiel des HIS3-Enzym, das für die Biosynthese von Histidin erforderlich ist, fusioniertum deg1 -Sec62. deg1 -Sec62 ist für den Abbau gezielt nach der aberrant die Endoplasmatischen Retikulum Translokon einrastet. Zellen, die deg1 -Sec62-His3 konnten unter selektiven Bedingungen wachsen, wenn das Protein stabilisiert wurde.
Einleitung
Selektive Proteinabbau ist für eukaryontische Leben und veränderten Proteinabbau trägt zu einer Reihe von Erkrankungen, einschließlich verschiedene Arten von Krebs, neurodegenerativen Erkrankungen, Herz-Kreislaufkrankheiten und Mukoviszidose 1-5. Das Ubiquitin-Proteasom-System (UPS), die selektive Proteinabbau katalysiert, ist eine aufstrebende therapeutisches Ziel für diesen Bedingungen 10.6. Ubiquitin-Ligase kovalent an Polymere der 76-Aminosäure-Ubiquitin an Proteine 11. Proteine, die mit Polyubiquitinketten markiert wurden, werden erkannt und durch die ~ 2,5 Megadalton 26S Proteasom proteolysiert 12. Studien im Modell eukaryotischen Organismus Saccharomyces cerevisiae (Bäckerhefe) initiiert wurden grund bei der Aufklärung von Proteinabbaumechanismen in eukaryotischen Zellen. Der erste Beweis physiologischen Substrat der USV war die Hefe Transkriptionsrepressor MATα2 13, 14, und viele hoch konservierte Komponenten des UPS wurden zuerst identifiziert oder gekennzeichnet Hefe (zB 15-26). Entdeckungen in diesem vielseitigen und genetisch manipulierbaren Modellorganismus hergestellt werden wahrscheinlich weiterhin wichtige Einblicke in konservierten Mechanismen des Ubiquitin-vermittelten Abbau liefern.
Anerkennung und Abbau der meisten UPS Substrate erfordern die konzertierte Aktion von mehreren Proteinen. Daher ist ein wichtiges Ziel bei der Charakterisierung des geregelten Abbau einer bestimmten instabilen Protein die genetischen Erfordernisse für die Proteolyse zu bestimmen. Klassische Ansätze (zB Pulse-Chase und Cycloheximid-Chase-Experimente 27) zur Überwachung der Proteinabbau in Säugetier- oder Hefezellen sind mühsam und zeitaufwendig. Während diese Art von Methode bereitzustellen hochempfindliche Einrichtung zum Erfassen des Proteinabbaus für eine schnelle Analyse des Proteinabbaus oder Groß screeni sind sie nichtng für Mutationen, die den Proteinabbau zu verhindern. Hier wird ein Wachstum der Hefe-basierenden Assay zur schnellen Identifizierung von genetischen Erfordernisse für den Abbau von instabilen Proteinen präsentiert.
Im Hefewachstum basiertes Verfahren zur Analyse von Protein-Abbau, ein instabiles Protein von Interesse (oder Abbausignals) fusioniert, im Rahmen, ein Protein, das für das Wachstum der Hefe unter bestimmten Umständen erforderlich ist. Das Ergebnis ist ein künstliches Substrat, das als ein mächtiges Werkzeug, um die genetischen Voraussetzungen des Proteinabbaus der instabilen Protein von Interesse festzustellen, dienen können. Zweckmßigerweise beherbergen am häufigsten verwendeten Labor Hefestämme eine Gruppe von Mutationen in Genen, die Stoffwechselenzyme in der Biosynthese von bestimmten Aminosäuren oder stickstoffhaltigen Basen (zB 20,28-30) beteiligt kodiert. Diese Enzyme sind für die zelluläre Proliferation in Abwesenheit von exogen bereitgestellt Metaboliten in deren Synthese der Enzyme beteiligt. Sometabolische Enzyme können so als Wachstumsbasis Reporter für den Abbau von instabilen Proteinen, denen sie fusioniert sind. Die genetischen Voraussetzungen für den Proteinabbau kann leicht erklärt werden, da Mutationen, die Proteolyse zu verhindern, können Zellen, die das Abbau Reporter, unter selektiven Bedingungen wachsen.
Einen Wachstumsvorteil ist ein indirekter Hinweis auf eine bestimmte Mutation erhöht die Häufigkeit des Proteins von Interesse. Allerdings ist direkte biochemische Analyse erforderlich, um zu bestätigen, dass eine Mutation ermöglicht Wachstum durch erhöhte Protein-Ebene und nicht durch indirekte oder Artefakt Ursachen. Die Wirkung einer Mutation auf Proteinmenge kann durch Western-Blot-Analyse der Steady-State-Protein-Spiegel in den Zellen, und tun nicht die bestimmte Mutation beherbergen bestätigt werden. Ein Verfahren für die schnelle und effiziente Gewinnung von Hefeproteinen (sequentielle Inkubation der Hefezellen mit Natriumhydroxid und Probenpuffer) in einer geeigneten Formfür die Analyse durch Western-Blot wird ebenfalls vorgestellt 31. Zusammen stellen diese Experimente ermöglichen die schnelle Identifizierung von Kandidatenregulatoren der Proteinabbau.
Protokoll
1. Hefe-Wachstumstest zur Kandidaten Mutanten Defekte in Proteinabbau identifizieren
- Transformieren Wildtyp- und mutierten Hefe-Zellen mit einem Plasmid einen instabilen Protein fusioniert codiert, im Rahmen, mit einem Reporter-Stoffwechselenzym.
- Beimpfen von Transformanten in 5 ml synthetischem definiert (SD) Minimalmedium, das selektiv für Zellen, die Plasmid-Moleküle sind. Inkubieren über Nacht bei 30 ° C, rotiert.
- Anschließend wird die optische Dichte bei 600 nm (OD 600) von jeder Übernachtkultur.
HINWEIS: Nach Inkubation über Nacht, können die Zellen in Kultur in entweder logarithmisch oder stationären Wachstumsphase sein, sollte aber eine minimale OD 600 von 0,2 erreicht. Sehr langsam wachsenden Hefestämme können Inkubationszeiten von einer größeren Anzahl von Zellen benötigen länger als eine Nacht oder Inokulation empirisch bestimmt. - Bereiten sechsfache serielle Verdünnungen von transformierten Hefezellen in einem sterilen 96-Well-Platte, beginnend mit den Zellen, um eine verdünnten OD 600 von 0,2. Platzieren jedes Hefetransformante in einer anderen Reihe in der Platte mit 96 Vertiefungen untersucht werden.
- Für jede Transformante, die Berechnung des Volumens der Übernacht-Kultur erforderlich ist, um Zellen zu einer OD 600 von 0,2 in einem Endvolumen von 200 ul verdünnt. Fügen Sie diese Volumen von Übernacht-Kultur, um die entsprechende Vertiefung in Spalte 1. Fügen Sie die entsprechende Menge an sterilem Wasser, um das Volumen auf 200 ul zu bringen.
- Für jede Reihe von Hefe, fügen 125 ul sterilem Wasser, um die Vertiefungen in den Spalten 2, 3 und 4.
HINWEIS: Einzeln verpackte sterile 96-Well-Platten mit einer sterilen Deckeln verpackt werden. Der Deckel kann als Reservoir für das sterile Wasser, das in dieser Stufe verteilt werden. Dies ermöglicht die gleichzeitige Übertragung von sterilem Wasser zu allen Vertiefungen in einer bestimmten Spalte mit einer Mehrkanalpipette. - Mischen Sie den Inhalt der ersten Spalte (Hefe zu einer OD 600 von 0,2 verdünnt) durch Auf- und Abpipettieren mit einer Mehrkanalpipette.
- Transfer 25 ul Hefe aus Spalte 1 bis Spalte 2, mit einer Mehrkanalpipette. Mischen Sie durch Auf- und Abpipettieren. Transfer 25 ul aus Spalte 2 bis Spalte 3, und 25 ul aus Spalte 3 bis Spalte 4 (gut mischen bei jedem Schritt).
- Mischen Sie jede Probe mit einer Mehrkanalpipette. Ausgehend von den meisten verdünnten in wenigstens verdünnte Spalten von Hefe, einer Pipette 4 ul jeder Probe auf beiden Platten, die den entsprechenden Selektionsmedium. Verwenden Sie eine Platte mit Medium, das Plasmid-Selektion (diese Platte dient als Hefe-Spotting und Wachstumskontrolle) unterhält. Verwenden einer zweiten Platte mit Medium, das für die Erhaltung des Plasmids und Expression des instabilen Protein an das Reporterenzym fusioniert auswählt. Da Hefe begleichen schnell, mischen Zellen durch Auf- und Abpipettieren in regelmäßigen Abständen.
HINWEIS: Trockenplatten werden leichter absorbiert Flüssigkeit als frisch zubereiteten Platten und sind daher für diese Versuche zu empfehlen. Feuchten Platten können durch Inkubation bei Raumtemperatur in lo trocknendenw Luftfeuchtigkeit für 1 - 2 Tage oder kürzer Inkubationen in einer Haube mit laminarer Strömung. Platten können ungleichmäßig trocknen, wenn sich die laminare Luftströmung parallel zu der Bank. Verwendung einer Schablone erleichtert die Hefezellen in regelmäßigen Abständen zu erkennen. Zwei Mustervorlagen sind in Figur 1 zur Verfügung gestellt. Diese können gedruckt werden, ausgeschnitten und in das Innere der Petrischale mit Deckel versehen. - Lassen Platten auf der Bank oben trocknen.
- Inkubation erfolgt bei 30 ° C für 2 - 6 Tagen haben.
- Fotografieren Sie jede Platte nach Inkubation.
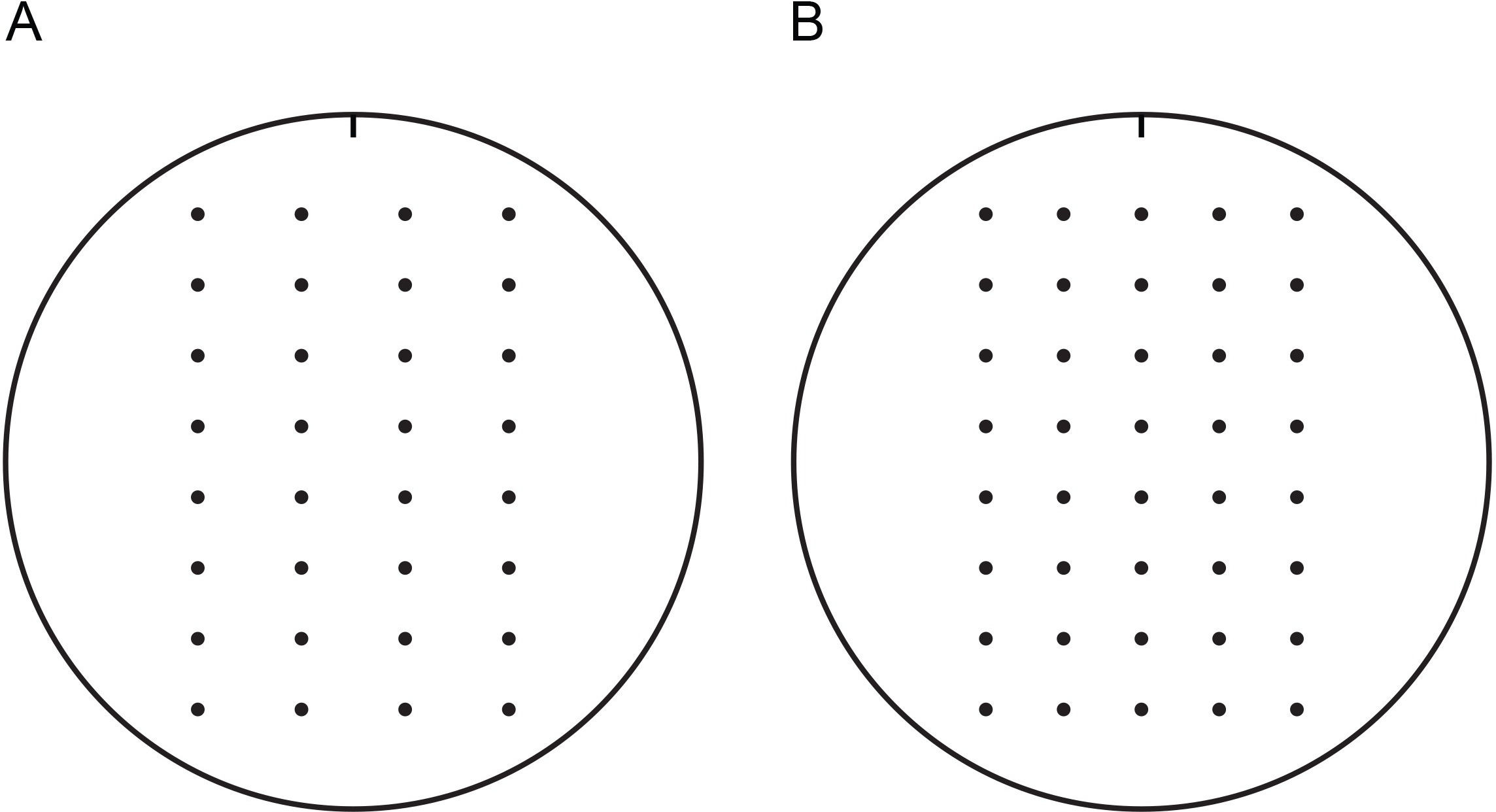
Abbildung 1. Vorlagen für das Auffinden Hefezellen auf 100-mm-Agar-Platten. Diese Vorlagen können verwendet werden, um zu ermöglichen Spotting Hefe in regelmäßigen Abständen mit einem Mehrkanalpipette werden. Templates können ausgedruckt, ausgeschnitten und in das Innere der Petrischale mit Deckel versehen. Zeigen Petrischale mit WachstumMedium im Deckel mit Schablone angebracht. Vorlagen werden mit einer Einkerbung markiert, um Orientierung zu verfolgen. Es wird empfohlen, dass die Platten in Wachstumsassays verwendet in ähnlicher Weise mit einer Kerbe markiert werden, um die Orientierung zu verfolgen. Vorlagen für die Erkennung von vier (A) oder fünf (B) Reihenverdünnungen von Hefezellen zur Verfügung gestellt werden. Bitte klicken Sie hier um eine druckfähige Version dieser Figur mit 100-mm-Vorlagen zu sehen.
2. Biochemische Bestätigung des Hefe-Wachstumstest
- Das Wachstum der Hefezellen und Post-Alkaline Proteinextraktion (von 31 modifiziert)
- Transform-Wildtyp und mutierten Hefe-Zellen mit einem Plasmid der instabilen Protein kodiert.
- Beimpfen von Transformanten in 5 ml SD-Medium, das selektiv für Zellen, die Plasmid-Moleküle sind. Inkubieren über Nacht bei 30 ° C, rotiert.
- Messen Sie die OD600 von jeder Nacht culture.
HINWEIS: Nach Inkubation über Nacht, können die Zellen entweder in logarithmische oder stationäre Wachstumsphase sein, sollte aber eine OD 600 die Verdünnung bis zu einer OD 600 von 0,2 in 10 ml frisches Selektivmedium (Schritt 2.1.4) erlauben wird erreicht. Sehr langsam wachsenden Hefestämme können Inkubationszeiten von einer größeren Anzahl von Zellen benötigen länger als eine Nacht oder Inokulation empirisch bestimmt. - Verdünne Hefezellen bis zu einer OD 600 von 0,2 in 10 ml frischem selektivem Medium.
- Weiter zu den Zellen bei 30 ° C inkubieren, Drehen oder Schütteln, bis Kulturen erreichen einer OD 600 zwischen 0,8 und 1,2 (dh sind Mitte logarithmischen Wachstums).
Hinweis: Wenn der instabile Protein von Interesse unter der Kontrolle eines regulierbaren Promotors, kann die optimale Zeitsteuerung der Induktion der Proteinexpression und der Zellgewinnung nach den vorherigen Studien oder empirischen Beobachtungen variieren. - Sammeln 2,5 OD 600 Einheiten der Kultur in 15 ml konischenal Rohr durch Zentrifugation bei 5.000 × g für 5 min bei Raumtemperatur. Überstand entfernen durch Pipettieren oder Absaugen.
Hinweis: eine OD600-Einheit wird als die Menge der vorhandenen Hefe von 1 ml der Kultur bei einer OD 600 von 1,0 definiert. V = 2,5 OD 600 Einheiten / Mess OD 600: Das Volumen der Kultur (in ml) erforderlich, um 2,5 OD 600 Einheiten (V) zu ernten kann mit der folgenden Gleichung bestimmt werden - Zellen in 1 ml destilliertem Wasser. Übertragen suspendierten Zellen in ein Mikrozentrifugenrohr.
- Pelle Zellen durch Zentrifugation bei 6.500 xg für 30 sec bei Raumtemperatur. Überstand entfernen durch Pipettieren oder Absaugen.
- Zellen in 100 ul destilliertem Wasser durch Auf- und Abpipettieren oder Verwirbelung, und fügen Sie 100 ul 0,2 M NaOH. Mischen Sie durch Auf- und Abpipettieren. Proben für 5 min bei Raumtemperatur.
- Pellet die Zellen durch Zentrifugation bei 18.000 x (von denen die meisten noch nicht veröffentlicht Proteine und sind noch lebensfähig)g für 5 min. Überstand entfernen durch Pipettieren oder Absaugen.
- Pellet in 50 bis 100 ul 1x Laemmli-Probenpuffer, die Zellen zu lysieren wird, durch Auf- und Abpipettieren oder Verwirbeln.
HINWEIS: Die Entfernung des Alkaliüberstand nach Zentrifugation und anschließende Resuspension der Zellen in Laemmli-Probenpuffer extrahiert Proteine bei einem pH-Wert mit Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) kompatibel mit einem Tris-Glycin-Laufpuffersystem und Western Blotting. - Um vollständig zu denaturieren, Inkubation Lysate bei 95 ° C für 5 min.
HINWEIS: Aggregation neigende Proteine (zB Proteine mit mehreren Transmembransegmente) können unlöslich werden, wenn sie bei 95 ° C inkubiert. Daher sollte Lysate bei niedrigeren Temperaturen inkubiert werden (zB 37 ° C - 70 ° C) für 10 bis 30 min, wie empirisch ermittelt, für die Analyse von solchen Proteinen. - Kühle Lysaten durch Anordnen auf Eis für 5 min.
- Zentrifuge Lysate bei 18000 g für 1 min bei Raumtemperatur, um unlösliches Material zu pelletisieren. Trennen Sie den Überstand (in Lösung extrahierte Protein) durch SDS-PAGE vor der anschließenden Western Blot-Analyse (Abschnitt 2.2). Alternativ Shop Lysate bei -20 ° C.
- Vertreter Western Blot Protokoll
- Laden empirisch ermittelten Volumen Lysate in einem SDS-PAGE-Gel.
- Führen Gel bei 200 V, bis Farbstofffront den Boden des Gels erreichte.
- Transferproteine aus Gel auf Polyvinylidenfluorid (PVDF) -Membran durch Nasstransfer bei 20 V für 60 bis 90 min bei 4 ° C.
- Block Membran durch Inkubation in 5% Magermilch in Tris-gepufferter Saline (TBS), schaukeln, für 1 h bei Raumtemperatur oder über Nacht bei 4 ° C.
- Dekantieren Blockierungslösung.
- Dazu werden die Membranen mit primärem Antikörper, der für das Protein von Interesse (oder Epitoptag davon) in 1% Magermilch in TBS mit 0,1% Tween-20 (TBS / T) für 1 h bei Raum temperatur, schaukeln.
- Dekantier-Antikörperlösung und Waschen der Membran 3 x 5 min mit TBS / T bei Raumtemperatur schaukeln.
- Dazu werden die Membranen mit geeigneten Fluorophor-konjugierten sekundären Antikörpers in 1% Magermilch in TBS / T für 1 Stunde bei Raumtemperatur schaukeln.
Hinweis: Da Fluorophore sind lichtempfindlich, sollte Verdünnungen fluorophorkonjugierten Antikörper im Dunkeln hergestellt werden. Zusätzlich sollte die Inkubation von Membranen in Gegenwart von fluorophorkonjugierten Antikörpern in lichtundurchlässigen Behältern auftreten. Dies kann durch Umwickeln Inkubationsschalen in Aluminiumfolie durchgeführt werden. - Dekantier-Antikörperlösung und Waschen der Membran 3 x 5 min mit TBS / T bei Raumtemperatur schaukeln.
- Erwerben Bild der Membran unter Verwendung von Li-Cor Odyssey CLx und Bild Studio-Software (oder einem vergleichbaren bildgebenden Geräten und Software), entsprechend den Empfehlungen des Herstellers.
- Nach Abbilden Membran inkubieren der Membran mit einem primären Antikörper, der spezifisch für einen Lasting Kontrollprotein in 1% Magermilch in TBS / T für 1 Stunde bei Raumtemperatur schaukeln.
- Dekantier-Antikörperlösung und Waschen der Membran 3 x 5 min mit TBS / T bei Raumtemperatur schaukeln.
- Dazu werden die Membranen mit geeigneten Fluorophor-konjugierten sekundären Antikörpers in 1% Magermilch in TBS / T für 1 Stunde bei Raumtemperatur schaukeln.
- Dekantier-Antikörperlösung und Waschen der Membran 3 x 5 min mit TBS / T bei Raumtemperatur schaukeln.
- Erwerben Bild der Membran unter Verwendung von Li-Cor Odyssey CLx und Bild Studio-Software (oder einem vergleichbaren bildgebenden Geräten und Software), entsprechend den Empfehlungen des Herstellers.
Ergebnisse
Diese Methodik zu veranschaulichen, hat das HIS3-Enzyms zu dem Carboxy-Terminus des Modells endoplasmatischen Retikulum (ER) assoziierte Abbau fusioniert (ERAD) -Substrat deg1 -Sec62 (2A) zu deg1 -Sec62-His3 schaffen (Figur 3) . deg1 -Sec62 eine Gründungsmitglied einer neuen Klasse von ERAD Substrate, die folgenden persistenten, anomale Verbindung mit dem Translokon ausgerichtet sind, wird der Kanal in erster Linie für die Bewegung Proteine durch die E...
Diskussion
Die hier vorgestellte Methode erlaubt die schnelle Bestimmung und biochemischen Bestätigung genetischen Erfordernisse für den Proteinabbau in Hefezellen. Diese Experimente unterstreichen den Nutzen und die Kraft der Hefe als Modell eukaryotischen Organismus (mehrere ausgezeichnete Bewertungen von Hefe Biologie und Zusammenstellungen von Protokollen für die Handhabung, Speicherung und Manipulation von Hefezellen (zB 41 bis 44) sind für die Ermittler neue für den Organismus verfügbar). Die Techni...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir bedanken uns bei aktuellen und ehemaligen Mitgliedern des Rubenstein Labor zur Bereitstellung eines unterstützenden und begeisterten Forschungsumfeld. Wir danken Ryan T. Gibson um Hilfe bei der Protokolloptimierung. Wir danken Markus Hochstrasser (Yale University) und Dieter Wolf (Universität Stuttgart) für Hefestämme und Plasmide. Wir danken unseren anonymen Gutachtern für ihre Hilfe bei der Verbesserung der Klarheit und Nützlichkeit dieses Manuskript. Diese Arbeit wurde von einem Forschungspreis der Ball State University Kapitel der Sigma Xi zu SGW unterstützt, ein National Institutes of Health Zuschuss (R15 GM111713) zu EMR, ein Ball State University ASPiRE Forschungspreis zur EMR und Mittel aus der Ball State University Provost Büro und Fachbereich Biologie.
Materialien
| Name | Company | Catalog Number | Comments |
| Desired yeast strains, plasmids, standard medium and buffer components | Yeast strains with desired mutations may be generated in the investigator's laboratory. Wild-type yeast and a variety of mutants are also commercially available (e.g. from GE Healthcare). Plasmids encoding fusion proteins may be generated in the investigator's laboratory. | ||
| 3-amino-1H-1,2,4-triazole | Fisher Scientific | AC264571000 | Competitive inhibitor of His3 enzyme. May be included in medium to increase stringency of growth assay using His3 reporter constructs. |
| Endoglycosidase H (recombinant form from Streptomyces plicatus) | Roche | 11088726001 | May be used to assess N-glycosylation of proteins; compatible with SDS and beta-mercaptoethanol concentrations found in 1x Laemmli sample buffer. |
| Disposable borosilicate glass tubes | Fisher Scientific | 14-961-32 | Available from a variety of manufacturers |
| Temperature-regulated incubator (e.g. Heratherm Incubator Model IMH180) | Dot Scientific | 51028068 | Available from a variety of manufacturers |
| New Brunswick Interchangeable Drum for 18 mm tubes (tube roller) | New Brunswick | M1053-0450 | Tube roller is recommended to maintain overnight yeast starter cultures of yeast cells in suspension. A platform shaker or tube roller may be used to maintain larger cultures in suspension. |
| New Brunswick TC-7 Roller Drum 120V 50/60 H | New Brunswick | M1053-4004 | For use with tube roller |
| SmartSpec Plus Spectrophotometer | Bio-Rad | 170-2525 | Available from a variety of manufacturers |
| Sterile 96-well flat bottom microtest plates with lid individually wrapped | Sarstedt | 82.1581.001 | Available from a variety of manufacturers |
| Pipetman Neo P8x20N, 2-20 μl | Gilson | F14401 | Available from a variety of manufacturers |
| [header] | |||
| Pipetman Neo P8x200N, 20-200 μl | Gilson | F14403 | Single-channel and multichannel pipettors are used at various stages of the protocol. While multichannel pipettors reduce the pipetting burden at several steps, single-channel pipettors may be used throughout the entire protocol. Available from a variety of manufacturers. |
| Centrifuge 5430 | Eppendorf | 5427 000.216 | Rotor that is sold with unit holds 1.5 and 2.0 ml microcentrifuge tubes. Rotor may be swapped for one that holds 15 ml and 50 ml conical tubes. |
| Plate imaging system (e.g. Gel Doc XR+ System) | Bio-Rad | 170-8195 | A variety of systems may be used to image plates, including sophisticated imaging systems, computer scanners, and camera phones. |
| Fixed-Angle Rotor F-35-6-30 with Lid and Adapters for Centrifuge Model 5430/R, 15/50 ml Conical Tubes, 6-Place | Eppendorf | F-35-6-30 | |
| 15 ml screen printed screw cap tube 17 x 20 mm conical, polypropylene | Sarstedt | 62.554.205 | Available from a variety of manufacturers |
| 1.5 ml flex-tube, PCR clean, Natural microcentrifuge tubes | Eppendorf | 22364120 | Available from a variety of manufacturers |
| Analog Dri-Bath Heater | Fisher Scientific | 1172011AQ | Boiling water bath with hot plate may also be used to denature proteins |
| SDS-PAGE running and transfer apparatuses, power supplies, and imaging equipment or darkrooms for SDS-PAGE and transfer to membrane | Will vary by lab and application | ||
| Western blot imaging system (e.g. Li-Cor Odyssey CLx scanner and Image Studio Software) | Li-Cor | 9140-01 | Will vary by lab and application |
| EMD Millipore Immobilon PVDF Transfer Membranes | Fisher Scientific | IPFL00010 | Will vary by lab and application |
| Primary antibodies (e.g. Phosphoglycerate Kinase (Pgk1) Monoclonal antibody, mouse (clone 22C5D8)) | Life Technologies | 459250 | Will vary by lab and application |
| Secondary antibodies (e.g. Alexa-Fluor 680 Rabbit Anti-Mouse IgG (H+L)) | Life Technologies | A-21065 | Will vary by lab and application |
Referenzen
- Goldberg, A. L. Protein degradation and protection against misfolded or damaged proteins. Nature. 426, 895-899 (2003).
- Guerriero, C. J., Brodsky, J. L. The delicate balance between secreted protein folding and endoplasmic reticulum-associated degradation in human physiology. Physiological Reviews. 92, 537-576 (2012).
- Pagan, J., Seto, T., Pagano, M., Cittadini, A. Role of the ubiquitin proteasome system in the heart. Circulation Research. 112, 1046-1058 (2013).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443, 780-786 (2006).
- Turnbull, E. L., Rosser, M. F., Cyr, D. M. The role of the UPS in cystic fibrosis. BMC Biochemistry. 8, S11 (2007).
- Bedford, L., Lowe, J., Dick, L. R., Mayer, R. J., Brownell, J. E. Ubiquitin-like protein conjugation and the ubiquitin-proteasome system as drug targets. Nature Reviews Drug Discovery. 10, 29-46 (2011).
- Kar, G., Keskin, O., Fraternali, F., Gursoy, A. Emerging role of the ubiquitin-proteasome system as drug targets. Current Pharmaceutical Design. 19, 3175-3189 (2013).
- Paul, S. Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches. BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology. 30, 1172-1184 (2008).
- Shen, M., Schmitt, S., Buac, D., Dou, Q. P. Targeting the ubiquitin-proteasome system for cancer therapy. Expert Opinion on Therapeutic Targets. 17 (9), 1091-1108 (2013).
- Finley, D., Ulrich, H. D., Sommer, T., Kaiser, P. The ubiquitin-proteasome system of Saccharomyces cerevisiae. Genetics. 192, 319-360 (2012).
- Scheffner, M., Nuber, U., Huibregtse, J. M. Protein ubiquitination involving an E1-E2-E3 enzyme ubiquitin thioester cascade. Nature. 373, 81-83 (1995).
- Ravid, T., Hochstrasser, M. Diversity of degradation signals in the ubiquitin-proteasome system. Nature Reviews Molecular Cell Biology. 9, 679-690 (2008).
- Hochstrasser, M., Ellison, M. J., Chau, V., Varshavsky, A. The short-lived MAT alpha 2 transcriptional regulator is ubiquitinated in vivo. Proceedings of the National Academy of Sciences of the United States of America. 88, 4606-4610 (1991).
- Hochstrasser, M., Varshavsky, A. In vivo degradation of a transcriptional regulator: the yeast alpha 2 repressor. Cell. 61, 697-708 (1990).
- Bays, N. W., Gardner, R. G., Seelig, L. P., Joazeiro, C. A., Hampton, R. Y. Hrd1p/Der3p is a membrane-anchored ubiquitin ligase required for ER-associated degradation. Nature Cell Biology. 3, 24-29 (2001).
- Hampton, R. Y., Gardner, R. G., Rine, J. Role of 26S proteasome and HRD genes in the degradation of 3-hydroxy-3-methylglutaryl-CoA reductase, an integral endoplasmic reticulum membrane protein. Molecular Biology of the Cell. 7, 2029-2044 (1996).
- Swanson, R., Locher, M., Hochstrasser, M. A conserved ubiquitin ligase of the nuclear envelope/endoplasmic reticulum that functions in both ER-associated and Matalpha2 repressor degradation. Genes & Development. 15, 2660-2674 (2001).
- Goebl, M. G., et al. The yeast cell cycle gene CDC34 encodes a ubiquitin-conjugating enzyme. Science. 241, 1331-1335 (1988).
- Bachmair, A., Finley, D., Varshavsky, A. In vivo half-life of a protein is a function of its amino-terminal residue. Science. 234, 179-186 (1986).
- Chen, P., Johnson, P., Sommer, T., Jentsch, S., Hochstrasser, M. Multiple ubiquitin-conjugating enzymes participate in the in vivo degradation of the yeast MAT alpha 2 repressor. Cell. 74, 357-369 (1993).
- Varshavsky, A. Discovery of the biology of the ubiquitin system. JAMA: The Journal of the American Medical Association. 311, 1969-1970 (2014).
- Heinemeyer, W., Kleinschmidt, J. A., Saidowsky, J., Escher, C., Wolf, D. H. Proteinase yscE, the yeast proteasome/multicatalytic-multifunctional proteinase: mutants unravel its function in stress induced proteolysis and uncover its necessity for cell survival. The EMBO Journal. 10, 555-562 (1991).
- Sommer, T., Jentsch, S. A protein translocation defect linked to ubiquitin conjugation at the endoplasmic reticulum. Nature. 365, 176-179 (1993).
- Hiller, M. M., Finger, A., Schweiger, M., Wolf, D. H. ER degradation of a misfolded luminal protein by the cytosolic ubiquitin-proteasome pathway. Science. 273, 1725-1728 (1996).
- Seufert, W., Jentsch, S. In vivo function of the proteasome in the ubiquitin pathway. The EMBO Journal. 11, 3077-3080 (1992).
- Knop, M., Finger, A., Braun, T., Hellmuth, K., Wolf, D. H. Der1, a novel protein specifically required for endoplasmic reticulum degradation in yeast. The EMBO Journal. 15, 753-763 (1996).
- Zattas, D., Adle, D. J., Rubenstein, E. M., Hochstrasser, M. N-terminal acetylation of the yeast Derlin Der1 is essential for Hrd1 ubiquitin-ligase activity toward luminal ER substrates. Molecular Biology of the Cell. 24, 890-900 (2013).
- Brachmann, C. B., et al. Designer deletion strains derived from Saccharomyces cerevisiae S288C: a useful set of strains and plasmids for PCR-mediated gene disruption and other applications. Yeast. 14, 115-132 (1998).
- Sikorski, R. S., Hieter, P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae. Genetics. 122, 19-27 (1989).
- Ralser, M., et al. The Saccharomyces cerevisiae W303-K6001 cross-platform genome sequence: insights into ancestry and physiology of a laboratory mutt. Open Biology. 2, 120093 (2012).
- Kushnirov, V. V. Rapid and reliable protein extraction from yeast. Yeast. 16, 857-860 (2000).
- Rubenstein, E. M., Kreft, S. G., Greenblatt, W., Swanson, R., Hochstrasser, M. Aberrant substrate engagement of the ER translocon triggers degradation by the Hrd1 ubiquitin ligase. The Journal of Cell Biology. 197, 761-773 (2012).
- Mayer, T. U., Braun, T., Jentsch, S. Role of the proteasome in membrane extraction of a short-lived ER-transmembrane protein. The EMBO Journal. 17, 3251-3257 (1998).
- Scott, D. C., Schekman, R. Role of Sec61p in the ER-associated degradation of short-lived transmembrane proteins. The Journal of Cell Biology. 181, 1095-1105 (2008).
- Kim, I., et al. The Png1-Rad23 complex regulates glycoprotein turnover. The Journal of Cell Biology. 172, 211-219 (2006).
- Fisher, E. A., et al. The degradation of apolipoprotein B100 is mediated by the ubiquitin-proteasome pathway and involves heat shock protein 70. The Journal of Biological Chemistry. 272, 20427-20434 (1997).
- Pariyarath, R., et al. Co-translational interactions of apoprotein B with the ribosome and translocon during lipoprotein assembly or targeting to the proteasome. The Journal of Biological Chemistry. 276, 541-550 (2001).
- Yeung, S. J., Chen, S. H., Chan, L. Ubiquitin-proteasome pathway mediates intracellular degradation of apolipoprotein. B. Biochemistry. 35, 13843-13848 (1996).
- Mumberg, D., Muller, R., Funk, M. Regulatable promoters of Saccharomyces cerevisiae: comparison of transcriptional activity and their use for heterologous expression. Nucleic Acids Research. 22, 5767-5768 (1994).
- Bruckner, A., Polge, C., Lentze, N., Auerbach, D., Schlattner, U. Yeast two-hybrid, a powerful tool for systems biology. International Journal of Molecular Sciences. 10, 2763-2788 (2009).
- Amberg, D. C., Burke, D., Strathern, J. N., Burke, D. . Methods in Yeast Genetics: A Cold Spring Harbor Laboratory Course Manual. , (2005).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: a primer on the Saccharomyces cerevisiae model system. Genetics. 197, 33-48 (2014).
- Guthrie, C., Fink, G. R. . Guide to Yeast Genetics and Molecular and Cell Biology. , (2004).
- Sherman, F. Getting started with yeast. Methods in Enzymology. 350, 3-41 (2002).
- Xie, Y., Rubenstein, E. M., Matt, T., Hochstrasser, M. SUMO-independent in vivo activity of a SUMO-targeted ubiquitin ligase toward a short-lived transcription factor. Genes & Development. 24, 893-903 (2010).
- Ravid, T., Kreft, S. G., Hochstrasser, M. Membrane and soluble substrates of the Doa10 ubiquitin ligase are degraded by distinct pathways. The EMBO Journal. 25, 533-543 (2006).
- Metzger, M. B., Maurer, M. J., Dancy, B. M., Michaelis, S. Degradation of a cytosolic protein requires endoplasmic reticulum-associated degradation machinery. The Journal of Biological Chemistry. 283, 32302-32316 (2008).
- Medicherla, B., Kostova, Z., Schaefer, A., Wolf, D. H. A genomic screen identifies Dsk2p and Rad23p as essential components of ER-associated degradation. EMBO Reports. 5, 692-697 (2004).
- Kohlmann, S., Schafer, A., Wolf, D. H. Ubiquitin ligase Hul5 is required for fragment-specific substrate degradation in endoplasmic reticulum-associated degradation. The Journal of Biological Chemistry. 283, 16374-16383 (2008).
- Crouse, G. F. Mutagenesis assays in yeast. Methods. 22, 116-119 (2000).
- Le Douarin, B., Pierrat, B., vom Baur, E., Chambon, F., Losson, R. A new version of the two-hybrid assay for detection of protein-protein interactions. Nucleic Acids Research. 23, 876-878 (1995).
- Boeke, J. D., Trueheart, J., Natsoulis, G., Fink, G. R. 5-Fluoroorotic acid as a selective agent in yeast molecular genetics. Methods in Enzymology. 154, 164-175 (1987).
- Toyn, J. H., Gunyuzlu, P. L., White, W. H., Thompson, L. A., Hollis, G. F. A counterselection for the tryptophan pathway in yeast: 5-fluoroanthranilic acid resistance. Yeast. 16, 553-560 (2000).
- Schafer, A., Wolf, D. H. Endoplasmic reticulum-associated protein quality control and degradation: genome-wide screen for ERAD components. Methods in Molecular Biology. 301, 289-292 (2005).
- Griggs, D. W., Johnston, M. Regulated expression of the GAL4 activator gene in yeast provides a sensitive genetic switch for glucose repression. Proceedings of the National Academy of Sciences of the United States of America. 88, 8597-8601 (1991).
- Duennwald, M. L. Growth assays to assess polyglutamine toxicity in yeast. The Journal of Visualized Experiments. (61), e3791 (2012).
- Baryshnikova, A., et al. Synthetic genetic array (SGA) analysis in Saccharomyces cerevisiae and Schizosaccharomyces pombe. Methods in Enzymology. 470, 145-179 (2010).
- Szymanski, E. P., Kerscher, O. Budding yeast protein extraction and purification for the study of function, interactions, and post-translational modifications. The Journal of Visualized Experiments. (80), e50921 (2013).
- Hjelm, H., Sjodahl, J., Sjoquist, J. Immunologically active and structurally similar fragments of protein A from Staphylococcus aureus. European Journal of Biochemistry. 57, 395-403 (1975).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten