このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
増殖に基づく決定とタンパク質分解に対する遺伝的要件の生化学的確認で
要約
This article describes a yeast growth-based assay for the determination of genetic requirements for protein degradation. It also demonstrates a method for rapid extraction of yeast proteins, suitable for western blotting to biochemically confirm degradation requirements. These techniques can be adapted to monitor degradation of a variety of proteins.
要約
安定化されたタンパク質分解は、事実上すべての細胞機能のために重要である。真核生物のタンパク質分解のための分子機構と遺伝的要件について知られていることの多くは、最初は出芽酵母に設立されました。タンパク質分解の古典の分析は、生化学的なパルスチェイスとシクロヘキシミドチェイスの方法論に依存してきた。これらの技術は、タンパク質の分解を観察するための高感度の方法を提供するが、それらは、面倒で時間がかかり、低スループットである。これらのアプローチは、タンパク質の分解を防ぐ変異についての迅速なまたは大規模なスクリーニングに適していない。ここでは、タンパク質分解のための遺伝的要件の容易な同定のための酵母の増殖に基づくアッセイが記載されている。このアッセイでは、特定の選択条件下で増殖に必要なレポーター酵素は不安定なタンパク質に融合される。細胞の内因性レポーター酵素を欠いているが、融合タンパク質を発現するセレ下で成長することができ融合タンパク質が安定化されるだけctive条件( すなわちタンパク質分解が損なわれている場合)。ここに記載の増殖アッセイにおいて、野生型および融合タンパク質をコードするプラスミドを有する変異体酵母細胞の段階希釈物を選択的および非選択培地上にスポットされる。選択条件下で成長が所定の変異による分解障害と一致する。増加したタンパク質存在量は、生化学的に確認すべきである。電気泳動及びウエスタンブロッティングに適した形態の酵母タンパク質の迅速な抽出のための方法も示されている。生化学分析のためのタンパク質抽出のための単純なプロトコルと組み合わせたタンパク質の安定性のための成長ベースの読み出しは、タンパク質分解のための遺伝的要件の迅速な同定を容易にします。これらの技術は、短命の種々のタンパク質の分解を監視するように適合させることができる。示した例では、ヒスチジン生合成に必要とされるのHis3酵素は、融合したそれが異常に小胞体トランスロコンに係合後にDeg1に-Sec62。Deg1 -Sec62は分解の標的とされている。 Deg1 -Sec62-のHis3を保有する細胞は、タンパク質が安定した時に選択的条件下で増殖することができた。
概要
選択的なタンパク質分解は、真核生物の生活のために不可欠であり、変化したタンパク質分解は、数種類の癌、神経変性疾患、心血管疾患、および嚢胞性線維症1-5を含む医学的状態の数に寄与する。選択的なタンパク質分解を触媒するユビキチン-プロテアソーム系(UPS)は、これらの条件6-10のための新たな治療標的である。ユビキチンリガーゼは共有結合したタンパク質11に76アミノ酸ユビキチンのポリマーを添付してください。ポリユビキチン鎖でマークされたタンパク質は、12プロテアソーム 〜2.5メガダルトン26Sによって認識され、タンパク質分解されている。モデル真核生物サッカロミセス·セレビシエ (出芽酵母)で開始研究は、真核細胞におけるタンパク質分解メカニズムの解明における基礎となっている。 UPSの最初の実証生理的基質は、酵母転写抑制因子MATα213だった、UPSの14、および多くの高度に保存されたコンポーネントは、最初に同定されたか( 例えば 15-26)酵母特徴。この汎用性と遺伝的に扱いやすいモデル生物で行われた発見は、ユビキチン媒介分解の保存されたメカニズムに重要な洞察を提供し続ける可能性が高い。
認識とほとんどのUPSの基質の分解は、複数のタンパク質の協調行動が必要です。したがって、与えられた不安定なタンパク質の調節された劣化を特徴付ける重要な目標は、タンパク質分解のための遺伝的要件を決定することです。哺乳動物または酵母細胞におけるタンパク質分解を監視するための古典的なアプローチ( 例えば 、パルスチェイスとシクロヘキシミドチェイス実験27)は、面倒で時間がかかる。方法のこれらのタイプのタンパク質分解を検出するための高感度の手段を提供するが、それらは、タンパク質分解または大規模screeniの迅速な分析には適していないタンパク質分解を防ぐ突然変異についてngの。ここでは、不安定なタンパク質の分解のための遺伝的要件の迅速な同定のための酵母増殖に基づくアッセイが提示されている。
タンパク質分解を分析するための酵母の増殖に基づく方法では、対象(または劣化信号)の不安定なタンパク質は、特定の状況下での酵母の増殖に必要とされるタンパク質を、インフレームで融合されている。結果は、関心のある不安定なタンパク質のタンパク質分解の遺伝的要件を決定するための強力なツールとして機能することができる人工的な基質である。好都合なことに、最も一般的に使用される実験室酵母株は、特定のアミノ酸または窒素含有塩基( 例えば、20,28-30)の生合成に関与する代謝酵素をコードする遺伝子における変異のパネルを保有する。これらの酵素は、合成酵素が参加する中で、外因的に提供代謝物の非存在下での細胞増殖のために必須である。このような代謝酵素は、従って、それらが融合されるため、不安定なタンパク質の分解のための増殖に基づくレポーターとして機能することができる。タンパク質分解を防ぐ変異が劣化レポーターを有する細胞が選択的条件下で成長することができますので、タンパク質分解のための遺伝的要件は容易に解明することができます。
増殖の利点は、特定の変異が、目的のタンパク質の存在量を増加させる間接的な指標である。しかし、直接生化学分析は、変異が増加したタンパク質のレベルを介してではなく、間接的または人為的な原因を経由して成長を可能することを確認する必要がある。タンパク質存在量における変異の効果を行い、特定の変異を保有しない細胞における定常状態タンパク質レベルのウェスタンブロット分析によって確認することができる。適した形態の酵母タンパク質(水酸化ナトリウムおよびサンプル緩衝液を用いて酵母細胞の逐次インキュベーション)の迅速かつ効率的に抽出するための方法ウェスタンブロッティングによる分析のためにも31を提示する。一緒に、これらの実験は、タンパク質分解の候補調節因子の迅速な同定を容易にする。
プロトコル
タンパク質分解に欠陥候補突然変異体を同定する1.酵母増殖アッセイ
- レポーター代謝酵素と、フレームで融合された不安定なタンパク質をコードするプラスミドを、野生型および突然変異体酵母細胞を形質転換する。
- プラスミド分子を保有する細胞に対して選択的である合成に定義(SD)最小培地5ml中の形質転換体を接種する。回転、30℃で一晩インキュベートする。
- 各一晩培養物を600nm(OD 600)での光学密度を測定します。
注:一晩のインキュベーション後、培養中の細胞のいずれか、対数または定常増殖期にあってもよいが、0.2の最小限のOD 600に達している必要があります。経験的に決定されるような非常に成長の遅い酵母株は、1晩、または細胞のより多くの接種より長いインキュベーション時間が必要な場合があります。 - に希釈した細胞をはじめとする無菌の96ウェルプレート中で形質転換された酵母細胞の六倍連続希釈液を調製するN OD 0.2の600。 96ウェルプレート内の異なる行でアッセイされる各酵母形質転換体を配置します。
- 各形質転換のために、200μlの最終容量で0.2のOD 600に細胞を希釈するために必要な一晩培養の体積を計算。コラム1に対応するウェルに一晩培養のこのボリュームを追加200μlにボリュームをもたらすために滅菌水の適切な量を追加します。
- 酵母の各行について、列2、3、および4のウェルに滅菌水125μlを添加する。
注:個別に包装された無菌の96ウェルプレートは、滅菌蓋でパッケージされてもよい。蓋は、このステップで配布され、滅菌水のリザーバとして使用することができる。これはマルチチャンネルピペッターに与えられた列のすべてのウェルに滅菌水の同時転送を可能にする。 - ピによってマルチチャンネルピ ペッターを下の最初の列(OD 0.2の600に希釈した酵母)の内容を混ぜる。
- TRANSFマルチチャンネルピペッターを使用して列2に列1から酵母25μlの、えー。ピペッティングにより混和する。転送列3の列2から25μL、列3から列4に25μL(各ステップでよく混合)。
- マルチチャンネルピペッターで各サンプルを混ぜる。最も希薄から出発すると、少なくとも適切な選択培地を含む2プレートに酵母、ピペット各サンプルの4μlの列を希釈する。プラスミド選択を(このプレートは酵母スポッティングと成長制御となります)維持培地で一つのプレートを使用してください。プラスミド維持とレポーター酵素に融合された不安定なタンパク質の発現のために選択した媒体と第二のプレートを使用してください。酵母は、急速に沈降し、一定の間隔で上下にピペッティングして細胞を混ぜているため。
注:乾燥機のプレートをより容易に、新たに調製したプレートよりも液体吸収し、したがって、これらの実験のために推奨されている。湿ったプレートを見よ中、室温でインキュベートすることによって乾燥させることができる層流フード中で2日またはより短いインキュベーション - 1ワット湿度。層流ベンチに対して平行である場合、プレートを不均一に乾燥することができる。テンプレートの使用は、簡単に正規の距離で酵母細胞を発見することができる。 2つのサンプルテンプレートが図1に提供される。これらは、印刷された切り出し、ペトリ皿の蓋の内側に取り付けることができる。 - プレートが作業台の上に乾燥することができます。
- 6日 - 2、30℃でプレートをインキュベートする。
- インキュベーション後に各プレートを撮影。
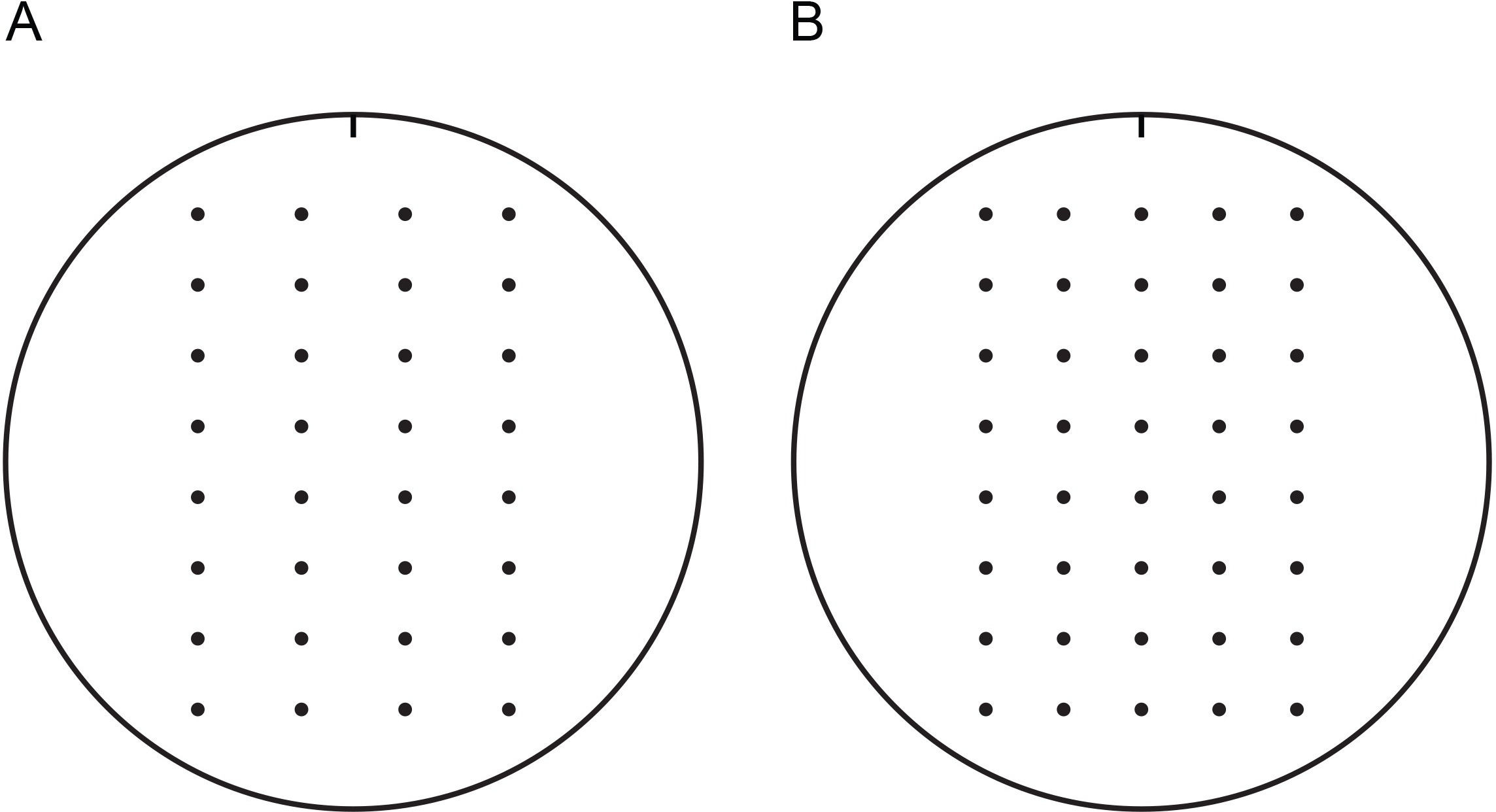
100mmの寒天プレート上の酵母細胞をスポッティングするための図1のテンプレート。これらのテンプレートは、マルチチャンネルピ ペッターを持つ通常の距離で酵母をスポッティング容易にすることができる。テンプレートは、印刷された切り出し、ペトリ皿の蓋の内側に取り付けることができる。成長にシャーレを置きテンプレートとメディア内部の蓋が固定された。テンプレートは、向きを追跡するためにノッチが付いています。これは、成長アッセイで使用したプレートは、同様に方向性を追跡するためにノッチでマークすることをお勧めします。 4(A)または5(B)酵母細胞の連続希釈が提供されてスポッティングためのテンプレート。 100 mmのテンプレートと、この図の印刷可能バージョンを表示するには、こちらをクリックしてください。
酵母増殖アッセイの2生化学確認
- 酵母細胞およびポストアルカリ性タンパク質抽出の成長(31から変更)
- 不安定なタンパク質をコードするプラスミドを、野生型および突然変異体酵母細胞を形質転換する。
- プラスミド分子を保有する細胞に選択的であるSD培地5ml中で形質転換体を接種する。回転、30℃で一晩インキュベートする。
- 各一晩、CuのOD 600を測定しますlture。
注:一晩のインキュベーション後に、細胞を、対数または定常増殖期にあってもよいが、0.2 10ミリリットルで新鮮な選択培地(ステップ2.1.4)のOD 600に希釈を可能にするOD 600に達している必要があります。経験的に決定されるような非常に成長の遅い酵母株は、1晩、または細胞のより多くの接種より長いインキュベーション時間が必要な場合があります。 - 0.2mlの10の新鮮な選択培地のOD 600に酵母細胞を希釈する。
- (中期対数増殖しているIE)培養物は0.8と1.2の間のOD 600に到達するまで、回転または振盪、30℃で細胞をインキュベートし続けます。
注:関心のある不安定なタンパク質は、調節可能なプロモーターの制御下にある場合には、タンパク質の発現および細胞収穫の誘導の最適なタイミングは、以前の研究又は経験的観察に応じて変化し得る。 - 15 mlの円錐での培養の2.5 OD 600単位を収集室温で5分間、5000×gでの遠心分離によってらチューブ。ペッティングまたは吸引により上清を除去します。
注:One OD 600単位を1.0のOD 600での培養液1ml中に存在する酵母の量として定義される。 V = 2.5 OD 600単位/測定OD 600:2.5のOD 600単位(V)を収穫するために必要な(ミリリットル)の培養物の容量は、以下の式を用いて決定することができる - 1ミリリットル中に細胞を再懸濁し、水を蒸留した。マイクロ遠心チューブに懸濁した細胞を転送します。
- 室温で30秒間、6500×gでの遠心分離によって細胞をペレット。ペッティングまたは吸引により上清を除去します。
- 100μl中に細胞を再懸濁がダウンしているかボルテックスピペッティングして蒸留水を、そして100μl、0.2MのNaOHを追加します。ピペッティングにより混和する。室温で5分間、サンプルをインキュベートする。
- 18000 xでの遠心分離により細胞をペレット化(そのほとんどはまだタンパク質をリリースし、まだ生存しているしていない)5分間のグラム。ペッティングまたは吸引により上清を除去します。
- 上下にピペッティングまたはボルテックスによって、細胞を溶解します100μlの1×Laemmliサンプルバッファー、 - 50でペレットを再懸濁。
NOTE:Laemmliサンプル緩衝液中で遠心分離し、細胞のその後の再懸濁以下アルカリ上清の除去はトリス - グリシンランニング緩衝系およびウェスタンブロッティングを使用して、ドデシル硫酸ナトリウム - ポリアクリルアミドゲル電気泳動(SDS-PAGE)と互換性のpHでタンパク質を抽出する。 - 完全にタンパク質を変性させ、5分間95℃で溶解物をインキュベートする。
注:95℃でインキュベートしたときに凝集しやすいタンパク質(いくつかの膜貫通セグメントを持つ例えば、タンパク質)が不溶性になることがあります。経験的にこのようなタンパク質の分析のために、決定されるように、30分- 10について-このため、溶解物は、より低い温度(70℃、 例えば 37℃)でインキュベートされるべきである。 - 氷上で5分間置くことによって溶解物を冷却する。
- 室温で1分間、18000×gで遠心分離溶解物の不溶性物質をペレット化する。事前のその後のウエスタンブロット分析(セクション2.2)に、SDS-PAGEにより上清(可溶化抽出したタンパク質)を分離。また、-20℃で保存溶解物。
- 代表ウェスタンブロッティングプロトコル
- SDS-PAGEゲルで溶解物の実験的に決定されたボリュームをロードします。
- 色素フロントがゲルの底に到達するまで200Vでゲルを実行します。
- 4℃で90分間 - 60 20 Vでウェット移送によりポリフッ化ビニリデン(PVDF)膜にゲルからタンパク質を移す。
- 4℃で一晩、室温で1時間、揺動または、トリス緩衝生理食塩水(TBS)中の5%スキムミルク中でインキュベートすることによりブロック膜。
- ブロッキング溶液デカントします。
- 室温テで1時間、0.1%のTween-20(TBS / T)を含むTBS中の1%スキムミルク中の目的のタンパク質(またはそのエピトープタグ)に特異的な一次抗体で膜をインキュベートするmperature、ロッキング。
- 抗体溶液をデカントし、そして室温でTBS / Tで膜3×5分間洗浄し、ロッキング。
- ロッキング、室温で1時間、TBS / T中の1%スキムミルク中の適切なフルオロフォア結合二次抗体で膜をインキュベートする。
注:蛍光団は光に敏感であるため、フォア標識抗体の希釈は暗闇の中で準備する必要があります。さらに、フォア標識抗体の存在下での膜のインキュベーションは、遮光容器内に発生する必要があります。これはアルミ箔でインキュベーショントレイをラップすることによって達成することができる。 - 抗体溶液をデカントし、そして室温でTBS / Tで膜3×5分間洗浄し、ロッキング。
- 製造業者の推奨に従って、LI-CORオデッセイCLxのを用いた膜と画像Studioソフトウェア(または同等の画像機器およびソフトウェア)の画像を取得する。
- 膜を画像化した後、ロード用の具体的な一次抗体で膜をインキュベートロッキング、室温で1時間、TBS / T中の1%スキムミルクで制御タンパク質る。
- 抗体溶液をデカントし、そして室温でTBS / Tで膜3×5分間洗浄し、ロッキング。
- ロッキング、室温で1時間、TBS / T中の1%スキムミルク中の適切なフルオロフォア結合二次抗体で膜をインキュベートする。
- 抗体溶液をデカントし、そして室温でTBS / Tで膜3×5分間洗浄し、ロッキング。
- 製造業者の推奨に従って、LI-CORオデッセイCLxのを用いた膜と画像Studioソフトウェア(または同等の画像機器およびソフトウェア)の画像を取得する。
結果
この方法論を説明するために、のHis3酵素はモデル小胞体(ER)関連分解のカルボキシ末端に融合されている(ERAD)基板、Deg1 -Sec62( 図2A)Deg1 -Sec62-のHis3を作成する( 図3) 。Deg1 -Sec62はロコンとの永続的な、異常な関連付けを次のターゲットとしているERAD基質の新規クラスの創立メンバーを表し、ER膜32-34間でタンパク質を移動するため?...
ディスカッション
ここで紹介する方法は、迅速な決定と酵母細胞内のタンパク質分解のための遺伝的要件の生化学的に確認することができます。これらの実験は、モデル真核生物として酵母の有用性とパワーを強調(処理するための酵母生物学およびプロトコルのコンピレーションのいくつかの優れたレビューは、保管、および酵母細胞を操作する( 例えば 41-44)生物に新たな研究者のために?...
開示事項
著者らは、開示することは何もない。
謝辞
私たちは、協力的かつ熱狂的な研究環境を提供するためのルーベンラボの現在および過去のメンバーに感謝。私たちは、プロトコルの最適化の支援のためにライアンT.ギブソンに感謝。我々は、酵母株およびプラスミドのためにマークHochstrasser(エール大学)とディーター·ウルフ(シュトゥットガルト大学)を感謝。我々は、この原稿の明快さと実用性の改善に彼らの助けのために私たちの匿名の査読者に感謝。この作品は、ボール州立大学からEMR、EMRにボール州立大学ASPIRE研究賞、および資金にSGWにシグマXIのボール州立大学の章から研究賞、国立衛生研究所の助成金(R15 GM111713)によってサポートされていました学長のOfficeと生物学科。
資料
| Name | Company | Catalog Number | Comments |
| Desired yeast strains, plasmids, standard medium and buffer components | Yeast strains with desired mutations may be generated in the investigator's laboratory. Wild-type yeast and a variety of mutants are also commercially available (e.g. from GE Healthcare). Plasmids encoding fusion proteins may be generated in the investigator's laboratory. | ||
| 3-amino-1H-1,2,4-triazole | Fisher Scientific | AC264571000 | Competitive inhibitor of His3 enzyme. May be included in medium to increase stringency of growth assay using His3 reporter constructs. |
| Endoglycosidase H (recombinant form from Streptomyces plicatus) | Roche | 11088726001 | May be used to assess N-glycosylation of proteins; compatible with SDS and beta-mercaptoethanol concentrations found in 1x Laemmli sample buffer. |
| Disposable borosilicate glass tubes | Fisher Scientific | 14-961-32 | Available from a variety of manufacturers |
| Temperature-regulated incubator (e.g. Heratherm Incubator Model IMH180) | Dot Scientific | 51028068 | Available from a variety of manufacturers |
| New Brunswick Interchangeable Drum for 18 mm tubes (tube roller) | New Brunswick | M1053-0450 | Tube roller is recommended to maintain overnight yeast starter cultures of yeast cells in suspension. A platform shaker or tube roller may be used to maintain larger cultures in suspension. |
| New Brunswick TC-7 Roller Drum 120V 50/60 H | New Brunswick | M1053-4004 | For use with tube roller |
| SmartSpec Plus Spectrophotometer | Bio-Rad | 170-2525 | Available from a variety of manufacturers |
| Sterile 96-well flat bottom microtest plates with lid individually wrapped | Sarstedt | 82.1581.001 | Available from a variety of manufacturers |
| Pipetman Neo P8x20N, 2-20 μl | Gilson | F14401 | Available from a variety of manufacturers |
| [header] | |||
| Pipetman Neo P8x200N, 20-200 μl | Gilson | F14403 | Single-channel and multichannel pipettors are used at various stages of the protocol. While multichannel pipettors reduce the pipetting burden at several steps, single-channel pipettors may be used throughout the entire protocol. Available from a variety of manufacturers. |
| Centrifuge 5430 | Eppendorf | 5427 000.216 | Rotor that is sold with unit holds 1.5 and 2.0 ml microcentrifuge tubes. Rotor may be swapped for one that holds 15 ml and 50 ml conical tubes. |
| Plate imaging system (e.g. Gel Doc XR+ System) | Bio-Rad | 170-8195 | A variety of systems may be used to image plates, including sophisticated imaging systems, computer scanners, and camera phones. |
| Fixed-Angle Rotor F-35-6-30 with Lid and Adapters for Centrifuge Model 5430/R, 15/50 ml Conical Tubes, 6-Place | Eppendorf | F-35-6-30 | |
| 15 ml screen printed screw cap tube 17 x 20 mm conical, polypropylene | Sarstedt | 62.554.205 | Available from a variety of manufacturers |
| 1.5 ml flex-tube, PCR clean, Natural microcentrifuge tubes | Eppendorf | 22364120 | Available from a variety of manufacturers |
| Analog Dri-Bath Heater | Fisher Scientific | 1172011AQ | Boiling water bath with hot plate may also be used to denature proteins |
| SDS-PAGE running and transfer apparatuses, power supplies, and imaging equipment or darkrooms for SDS-PAGE and transfer to membrane | Will vary by lab and application | ||
| Western blot imaging system (e.g. Li-Cor Odyssey CLx scanner and Image Studio Software) | Li-Cor | 9140-01 | Will vary by lab and application |
| EMD Millipore Immobilon PVDF Transfer Membranes | Fisher Scientific | IPFL00010 | Will vary by lab and application |
| Primary antibodies (e.g. Phosphoglycerate Kinase (Pgk1) Monoclonal antibody, mouse (clone 22C5D8)) | Life Technologies | 459250 | Will vary by lab and application |
| Secondary antibodies (e.g. Alexa-Fluor 680 Rabbit Anti-Mouse IgG (H+L)) | Life Technologies | A-21065 | Will vary by lab and application |
参考文献
- Goldberg, A. L. Protein degradation and protection against misfolded or damaged proteins. Nature. 426, 895-899 (2003).
- Guerriero, C. J., Brodsky, J. L. The delicate balance between secreted protein folding and endoplasmic reticulum-associated degradation in human physiology. Physiological Reviews. 92, 537-576 (2012).
- Pagan, J., Seto, T., Pagano, M., Cittadini, A. Role of the ubiquitin proteasome system in the heart. Circulation Research. 112, 1046-1058 (2013).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443, 780-786 (2006).
- Turnbull, E. L., Rosser, M. F., Cyr, D. M. The role of the UPS in cystic fibrosis. BMC Biochemistry. 8, S11 (2007).
- Bedford, L., Lowe, J., Dick, L. R., Mayer, R. J., Brownell, J. E. Ubiquitin-like protein conjugation and the ubiquitin-proteasome system as drug targets. Nature Reviews Drug Discovery. 10, 29-46 (2011).
- Kar, G., Keskin, O., Fraternali, F., Gursoy, A. Emerging role of the ubiquitin-proteasome system as drug targets. Current Pharmaceutical Design. 19, 3175-3189 (2013).
- Paul, S. Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches. BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology. 30, 1172-1184 (2008).
- Shen, M., Schmitt, S., Buac, D., Dou, Q. P. Targeting the ubiquitin-proteasome system for cancer therapy. Expert Opinion on Therapeutic Targets. 17 (9), 1091-1108 (2013).
- Finley, D., Ulrich, H. D., Sommer, T., Kaiser, P. The ubiquitin-proteasome system of Saccharomyces cerevisiae. Genetics. 192, 319-360 (2012).
- Scheffner, M., Nuber, U., Huibregtse, J. M. Protein ubiquitination involving an E1-E2-E3 enzyme ubiquitin thioester cascade. Nature. 373, 81-83 (1995).
- Ravid, T., Hochstrasser, M. Diversity of degradation signals in the ubiquitin-proteasome system. Nature Reviews Molecular Cell Biology. 9, 679-690 (2008).
- Hochstrasser, M., Ellison, M. J., Chau, V., Varshavsky, A. The short-lived MAT alpha 2 transcriptional regulator is ubiquitinated in vivo. Proceedings of the National Academy of Sciences of the United States of America. 88, 4606-4610 (1991).
- Hochstrasser, M., Varshavsky, A. In vivo degradation of a transcriptional regulator: the yeast alpha 2 repressor. Cell. 61, 697-708 (1990).
- Bays, N. W., Gardner, R. G., Seelig, L. P., Joazeiro, C. A., Hampton, R. Y. Hrd1p/Der3p is a membrane-anchored ubiquitin ligase required for ER-associated degradation. Nature Cell Biology. 3, 24-29 (2001).
- Hampton, R. Y., Gardner, R. G., Rine, J. Role of 26S proteasome and HRD genes in the degradation of 3-hydroxy-3-methylglutaryl-CoA reductase, an integral endoplasmic reticulum membrane protein. Molecular Biology of the Cell. 7, 2029-2044 (1996).
- Swanson, R., Locher, M., Hochstrasser, M. A conserved ubiquitin ligase of the nuclear envelope/endoplasmic reticulum that functions in both ER-associated and Matalpha2 repressor degradation. Genes & Development. 15, 2660-2674 (2001).
- Goebl, M. G., et al. The yeast cell cycle gene CDC34 encodes a ubiquitin-conjugating enzyme. Science. 241, 1331-1335 (1988).
- Bachmair, A., Finley, D., Varshavsky, A. In vivo half-life of a protein is a function of its amino-terminal residue. Science. 234, 179-186 (1986).
- Chen, P., Johnson, P., Sommer, T., Jentsch, S., Hochstrasser, M. Multiple ubiquitin-conjugating enzymes participate in the in vivo degradation of the yeast MAT alpha 2 repressor. Cell. 74, 357-369 (1993).
- Varshavsky, A. Discovery of the biology of the ubiquitin system. JAMA: The Journal of the American Medical Association. 311, 1969-1970 (2014).
- Heinemeyer, W., Kleinschmidt, J. A., Saidowsky, J., Escher, C., Wolf, D. H. Proteinase yscE, the yeast proteasome/multicatalytic-multifunctional proteinase: mutants unravel its function in stress induced proteolysis and uncover its necessity for cell survival. The EMBO Journal. 10, 555-562 (1991).
- Sommer, T., Jentsch, S. A protein translocation defect linked to ubiquitin conjugation at the endoplasmic reticulum. Nature. 365, 176-179 (1993).
- Hiller, M. M., Finger, A., Schweiger, M., Wolf, D. H. ER degradation of a misfolded luminal protein by the cytosolic ubiquitin-proteasome pathway. Science. 273, 1725-1728 (1996).
- Seufert, W., Jentsch, S. In vivo function of the proteasome in the ubiquitin pathway. The EMBO Journal. 11, 3077-3080 (1992).
- Knop, M., Finger, A., Braun, T., Hellmuth, K., Wolf, D. H. Der1, a novel protein specifically required for endoplasmic reticulum degradation in yeast. The EMBO Journal. 15, 753-763 (1996).
- Zattas, D., Adle, D. J., Rubenstein, E. M., Hochstrasser, M. N-terminal acetylation of the yeast Derlin Der1 is essential for Hrd1 ubiquitin-ligase activity toward luminal ER substrates. Molecular Biology of the Cell. 24, 890-900 (2013).
- Brachmann, C. B., et al. Designer deletion strains derived from Saccharomyces cerevisiae S288C: a useful set of strains and plasmids for PCR-mediated gene disruption and other applications. Yeast. 14, 115-132 (1998).
- Sikorski, R. S., Hieter, P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae. Genetics. 122, 19-27 (1989).
- Ralser, M., et al. The Saccharomyces cerevisiae W303-K6001 cross-platform genome sequence: insights into ancestry and physiology of a laboratory mutt. Open Biology. 2, 120093 (2012).
- Kushnirov, V. V. Rapid and reliable protein extraction from yeast. Yeast. 16, 857-860 (2000).
- Rubenstein, E. M., Kreft, S. G., Greenblatt, W., Swanson, R., Hochstrasser, M. Aberrant substrate engagement of the ER translocon triggers degradation by the Hrd1 ubiquitin ligase. The Journal of Cell Biology. 197, 761-773 (2012).
- Mayer, T. U., Braun, T., Jentsch, S. Role of the proteasome in membrane extraction of a short-lived ER-transmembrane protein. The EMBO Journal. 17, 3251-3257 (1998).
- Scott, D. C., Schekman, R. Role of Sec61p in the ER-associated degradation of short-lived transmembrane proteins. The Journal of Cell Biology. 181, 1095-1105 (2008).
- Kim, I., et al. The Png1-Rad23 complex regulates glycoprotein turnover. The Journal of Cell Biology. 172, 211-219 (2006).
- Fisher, E. A., et al. The degradation of apolipoprotein B100 is mediated by the ubiquitin-proteasome pathway and involves heat shock protein 70. The Journal of Biological Chemistry. 272, 20427-20434 (1997).
- Pariyarath, R., et al. Co-translational interactions of apoprotein B with the ribosome and translocon during lipoprotein assembly or targeting to the proteasome. The Journal of Biological Chemistry. 276, 541-550 (2001).
- Yeung, S. J., Chen, S. H., Chan, L. Ubiquitin-proteasome pathway mediates intracellular degradation of apolipoprotein. B. Biochemistry. 35, 13843-13848 (1996).
- Mumberg, D., Muller, R., Funk, M. Regulatable promoters of Saccharomyces cerevisiae: comparison of transcriptional activity and their use for heterologous expression. Nucleic Acids Research. 22, 5767-5768 (1994).
- Bruckner, A., Polge, C., Lentze, N., Auerbach, D., Schlattner, U. Yeast two-hybrid, a powerful tool for systems biology. International Journal of Molecular Sciences. 10, 2763-2788 (2009).
- Amberg, D. C., Burke, D., Strathern, J. N., Burke, D. . Methods in Yeast Genetics: A Cold Spring Harbor Laboratory Course Manual. , (2005).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: a primer on the Saccharomyces cerevisiae model system. Genetics. 197, 33-48 (2014).
- Guthrie, C., Fink, G. R. . Guide to Yeast Genetics and Molecular and Cell Biology. , (2004).
- Sherman, F. Getting started with yeast. Methods in Enzymology. 350, 3-41 (2002).
- Xie, Y., Rubenstein, E. M., Matt, T., Hochstrasser, M. SUMO-independent in vivo activity of a SUMO-targeted ubiquitin ligase toward a short-lived transcription factor. Genes & Development. 24, 893-903 (2010).
- Ravid, T., Kreft, S. G., Hochstrasser, M. Membrane and soluble substrates of the Doa10 ubiquitin ligase are degraded by distinct pathways. The EMBO Journal. 25, 533-543 (2006).
- Metzger, M. B., Maurer, M. J., Dancy, B. M., Michaelis, S. Degradation of a cytosolic protein requires endoplasmic reticulum-associated degradation machinery. The Journal of Biological Chemistry. 283, 32302-32316 (2008).
- Medicherla, B., Kostova, Z., Schaefer, A., Wolf, D. H. A genomic screen identifies Dsk2p and Rad23p as essential components of ER-associated degradation. EMBO Reports. 5, 692-697 (2004).
- Kohlmann, S., Schafer, A., Wolf, D. H. Ubiquitin ligase Hul5 is required for fragment-specific substrate degradation in endoplasmic reticulum-associated degradation. The Journal of Biological Chemistry. 283, 16374-16383 (2008).
- Crouse, G. F. Mutagenesis assays in yeast. Methods. 22, 116-119 (2000).
- Le Douarin, B., Pierrat, B., vom Baur, E., Chambon, F., Losson, R. A new version of the two-hybrid assay for detection of protein-protein interactions. Nucleic Acids Research. 23, 876-878 (1995).
- Boeke, J. D., Trueheart, J., Natsoulis, G., Fink, G. R. 5-Fluoroorotic acid as a selective agent in yeast molecular genetics. Methods in Enzymology. 154, 164-175 (1987).
- Toyn, J. H., Gunyuzlu, P. L., White, W. H., Thompson, L. A., Hollis, G. F. A counterselection for the tryptophan pathway in yeast: 5-fluoroanthranilic acid resistance. Yeast. 16, 553-560 (2000).
- Schafer, A., Wolf, D. H. Endoplasmic reticulum-associated protein quality control and degradation: genome-wide screen for ERAD components. Methods in Molecular Biology. 301, 289-292 (2005).
- Griggs, D. W., Johnston, M. Regulated expression of the GAL4 activator gene in yeast provides a sensitive genetic switch for glucose repression. Proceedings of the National Academy of Sciences of the United States of America. 88, 8597-8601 (1991).
- Duennwald, M. L. Growth assays to assess polyglutamine toxicity in yeast. The Journal of Visualized Experiments. (61), e3791 (2012).
- Baryshnikova, A., et al. Synthetic genetic array (SGA) analysis in Saccharomyces cerevisiae and Schizosaccharomyces pombe. Methods in Enzymology. 470, 145-179 (2010).
- Szymanski, E. P., Kerscher, O. Budding yeast protein extraction and purification for the study of function, interactions, and post-translational modifications. The Journal of Visualized Experiments. (80), e50921 (2013).
- Hjelm, H., Sjodahl, J., Sjoquist, J. Immunologically active and structurally similar fragments of protein A from Staphylococcus aureus. European Journal of Biochemistry. 57, 395-403 (1975).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved