Method Article
הפקת DNA-RNA סימולטני החוף משקעים וכימות של 16S rRNA גנים תעתיקי ידי בזמן אמת PCR
In This Article
Summary
A protocol to quantify bacterial 16S rRNA genes and transcripts from coastal sediments via real-time PCR is provided. The methodology includes the co-extraction of DNA and RNA; preparation of DNA-free RNA; and 16S rRNA gene and transcript quantification via RT-q-PCR, including standard curve construction.
Abstract
תגובת שרשרת פולימראז Real Time הידוע גם בשם PCR כמותי (q-PCR) הוא כלי בשימוש נרחב אקולוגיה מיקרוביאלית לכמת שכיחותם הגן של קבוצות טקסונומיות ופונקציונלי דגימות סביבתיות. להשתמש בשילוב עם תגובת transcriptase הפוכה (RT-q-PCR), זה יכול גם להיות מועסק לכמת תמלילי גן. Q-PCR עושה שימוש כימי איתור פלורסנט רגישות גבוהה המאפשרות כימותים של amplicons PCR בשלב המעריכים של התגובה. לכן, ההטיות הקשורות 'הקצה' PCR זוהו בשלב הרמה של תגובת PCR נמנעות. פרוטוקול לכמת גני rRNA 16S חיידקי תמלילים ממשקעי החוף דרך בזמן אמת PCR מסופק. ראשית, שיטה שיתוף החילוץ של DNA ו- RNA ממשקעים החוף, כולל הצעדים הנוספים הדרושים לעריכת RNA DNA ללא, מפורט. שנית, מדריך צעד-אחר-צעד עבור כימות של גני 16S rRNA ו TRanscripts מן חומצות גרעין שחולצו באמצעות q-PCR ו RT-q-PCR המתואר. מידע זה כולל פרטים לבניית עקומות סטנדרטיות DNA ו- RNA. שיקולים עיקריים לשימוש מבחני RT-q-PCR באקולוגיה מיקרוביאלית כלולים.
Introduction
מיקרואורגניזמים הם אבן הפינה של הפונקציה האקולוגית נהיגה הביוספרה. רוב מיקרואורגניזמים להישאר חסרי תרבות 1. לכן גישות מולקולריות מבוססות הם יסוד לקדם את הבנתנו את המגוון והתפקוד של מיקרואורגניזמים בסביבה. מרכז גישות אלה הוא המיצוי של חומצות גרעין מ דגימות סביבתיות לבין ההגברה הבאה של גני המטרה באמצעות תגובת שרשרת פולימראז (PCR).
הצעד הראשון של מיצוי DNA / RNA שואפת lyse את דופן התא של ההווה קהילת חיידקים, להסיר מולקולות גרעין-חומצה הלא רצויים (למשל, חומרים אורגניים ואי-אורגניים) ולשמר DNA / RNA בתמיסה לניתוח נוסף במורד הזרם. בין מספר אפשרויות זמינות בספרות 2,3,4,5, כוללים מגוון רחב של ערכות הפקה מסחריות, שיטת גריפיתס 6 מועסק נרחב 7,8,9. זהו חסכוני particularly גם מתאים משקע כפי שהיא משתמשת צעד להכות חרוז כדי lyse תאים משלב צעדים כדי למזער את שאיבת שיתוף של מעכבי ה- PCR, כגון חומצות humic, תוך מחל DNA ו- RNA זמנית.
הצעד השני משתמש תגובת שרשרת פולימראז (PCR) כדי להגביר גני המטרה, כגון סמן 16S rRNA טקסונומיות, מן חומצות גרעין שחולצו. יש גישה זו וממשיכה להקל על החיפושים של 10,11 קופסה שחורה מיקרוביאלי uncharacterized. עם זאת, בשיטות מבוססות PCR קצה לסבול ממגבלות שונות שיכולות להטות את האפיון של קהילות מיקרוביאלי 12. כדי לכמת במדויק שכיחותם הגן / תמליל, בזמן אמת PCR, הידוע גם בשם PCR כמותי (qPCR) חייב לשמש. qPCR מנצל מערכות צבע כתב ניאון העוקבות אחר הצטברות amplicon לאחר כל מחזור של PCR. זה משמעותי כי זה אומר כי כימות יכול להתרחש במהלך שלב המעריכים מוקדם, ולאמתום הנקודות, השלב של תגובת PCR, כאשר תשואת amplicon עדיין ביחס ישר השפע הראשוני של גן המטרה.
שתי מערכות הכתב משמשות: כתם חומצות גרעין intercalating 13 ו 5 '3' פעילות exonuclease של פולימראז DNA 14. מאחר שרשת הכתב לשעבר נקשרה indistinctively לכל הדנ"א הדו-גדילית, זה עלול להוביל יתר של רצף היעד אם amplicons שאינו ספציפי הרצוי או פריימר דיימר ידי מוצרים להתרחש. כדי לעקוף זאת, אופטימיזציה הנרחבת של הגברה ייתכן שתידרש. במערכת האחרונה, הגברת תבנית הוא אתר באמצעות שילוב של פעילות nuclease 5 'של פולימראז תקי מסלקת fluorophore מן התחקיר פנימי. תכונה זו מגבירה את הספציפיות של assay בשל הניצול של חללית fluorogenic שקושר רק sequenc היעד ספציפי משליםדואר בין זוג פריימר. בשתי כימי כימות מושגת על ידי קביעת הנקודה-המעבר (CP) שם ההצטברות של amplicons PCR, כפי שהיא נמדדת על ידי גידול קרינה, היא משמעותי מעל קרינת רקע.
qPCR כבר נעשה שימוש נרחב באקולוגיה מיקרוביאלית לקבוע שכיחותם גן בסביבות שונות 15. יתר על כן, שעתוק לאחור של RNA ל cDNA בשילוב עם qPCR ו RT-qPCR לכמת ביטוי גנים. לפיכך, qPCR ו RT-qPCR מייצגים מהירים, שיטות יעילות עבור כימות של מספרי גן ו / או תמליל בתוך דגימות סביבתיות.
מיקרואורגניזמים במשקעי החוף לנהוג תהליכים אקולוגיים שונים, כוללים מינרליזציה של חומר אורגני, השפלתה של מזהמים ואת אופני biogeochemical של macronutrients כגון 16,17,18 חנקן. ההבנה הממצה של השינויים הללו דורשת accoun הקיףt של אוכלוסיות חיידקים התורמות, כוללים נתונים כמותיים על שכיחותם גני תמליל. כאן אנחנו מציגים סדרה של בדיקות מקיפות, נהלים פשוטים הסטנדרטיים עבור כימות של חיידקי 16S rRNA שכיחותם גני תמליל במשקעי החוף. הפרוטוקול מתאר אוסף מדגם, DNA סימולטני מיצוי RNA, הכנת RNA חופשי-DNA, בדיקת איכות של חומצות גרעין שחולצו, דור של 16S rRNA-DNA וסטנדרטי -RNA וכימות של דגימות סביבתיות. נתונים כמותיים נגזרו השיטות שתוארו כאן יש צורך לשפוך אור על קהילות חיידקי נהיגת מערכות אקולוגיות החוף.
Protocol
1. DNA & RNA הפקה ממשקעי החוף הימי
- הכנת ribonuclease (RNAse) חומר -חינם וסביבת עבודה
- הכן מים מזוקקים RNAse ללא (DH 2 O) על ידי טיפול עם 0.1% diethylpyrocarbonate (DEPC) ו לדגור על 37 מעלות CO / N. הסר DEPC שיורי על ידי מעוקר DH 2 O ב 121 מעלות צלזיוס במשך 15 דקות. השתמש DEPC שטופלו DH 2 O כדי להפוך את כל פתרונות.
- אופים את כל כלי זכוכית ב 180 ° CO / N. הסר מכסי פלסטיק חישוקים, להשרות אותן בתמיסה של 2 M NaOH O / N, ולשטוף אותם עם DEPC שטופלו DH 2 O לפני השימוש.
- כן פתרון חיץ CTAB-פוספט לפי טבלת 1.
- הכן את הפתרון הממטר PEG-NaCl לפי טבלה 2.
- נקו את כל משטחי עבודה (כלומר, ספסל, microcentrifuge, קטר-ברדס) ו micropipettes באמצעות RNAse-decontaminating פתרון לפני שמתחילים. יש להשתמש בכפפות במהלך ההליך כולו.
- השתמש plasticware RNAse ללא יחד עם טיפים מסננים micropipette כדי למנוע זיהום צולבות מדגם.
- איסוף ואחסון מדגם סביבה
- אסוף משקעים החוף במהלך שפל באמצעות צינור בז סטרילית 50 מ"ל מן 2.5 ס"מ העליון. אסוף כ 15-20 גרם של משקעים. בזהירות ולסגור את המכסה לאחר איסוף דגימה. התחבורה מיד למעבדה בצידנית ב 4 ° C לעיבוד מיידי.
- Homogenize המדגם על ידי ערבוב עם מרית aliquot סטרילי 0.5 גרם של משקעים משקל רטוב לתוך צינור מכות חרוז 2 מ"ל. משכפל נוסף Aliquot, פלאש להקפיא ידי צבת בחנקן נוזלי. חנות aliquots ב -80 מעלות צלזיוס. זהירות: יש להשתמש בכפפות מגן ומשקפי בטיחות בעת טיפול בחנקן נוזלי.
הערה: אם אתר השדה אינו ממוקם קרוב למעבדה, aliquot 0.5 גרם של משקעים לתוך 2 מיליליטר צינורות microcentrifuge סטרילית באתר השדה. פלאש להקפיא את הצינורות על ידי לשים אותם מיד לתוך liחנקן תחבורה פאונד למעבדה. חנות דגימות ב -80 ° C עד עיבוד.
- Co-הפקת DNA ו- RNA משקעים
הערה: הפרוטוקול הבא הוא גרסה שונה של שיטת גריפיתס 6.- הוספת 500 חיץ μl CTAB-פוספט 500 פנול μl: כלורופורם: isoamyl אלכוהול (25: 24: 1) אל הצינור 2 מ"ל חרוז-lysing המכיל 0.5 גרם של משקעים להפוך את הצינור 5-10 פעמים כדי homogenize המדגם.
זהירות: בצע פעולה זו בתוך קטר-ברדס לובש ציוד מגן מתאים (כלומר, מעיל מעבדה, כפפות ומשקפי בטיחות) בכל עת. - וורטקס במלוא המהירות עבור 2.5 דקות. צנטריפוגה ב XG 16,000 במשך 10 דקות ב 4 °.
- במנדף-קטר לחלץ את השכבה המימית העליון ולהעביר צינור microcentrifuge סטרילי 1.5 מ"ל חדש. שמירה על דגימות קרח, להוסיף 500 μl של כלורופורם קר כקרח: אלכוהול isoamyl (24: 1).
- היפוך מספר פעמים עד אמולסיה היא visible. צנטריפוגה במשך 10 דקות ב XG 16,000 ב 4 ° C.
- במנדף-קטר לחלץ את השכבה המימית העליון ומניחים אותו בתוך שפופרת סטרילית 1.5 מ"ל חדש. לזרז את חומצות גרעין על ידי הוספת שני כרכים של 30% פתרון PEG- NaCl ומערבבים היטב.
- לדגור על הקרח במשך שעה 2 או לעזוב דגימות ב 4 ° CO / N.
- בסוף דגימות צנטריפוגות הדגרה ב XG 16,000 במשך 20 דקות ב 4 ° C.
הערה: גלולה עשויה להיות גלויה בתחתית הצינור. - הוצא בעדינות את supernatant באמצעות micropipette, לוודא שלא להפריע את הכדור. השאירו סביב 10 μl של פתרון PEG בצינור ולהוסיף 1 מ"ל של אתנול 70% קר כקרח.
- צנטריפוגה במשך 20 דקות ב XG 16,000 ב 4 ° C. לאט להסיר אתנול עם micropipette נזהרת שלא לגעת גלולה.
- צנטריפוגות במשך 5 שניות ולהסיר אתנול שיורית באמצעות micropipette. השאר גלולה לייבוש באוויר למשך כ 5 דקות.
- Re- להשעות גלולה ב 50 DEPC שטופלו μlH 2 O. בדוק את האיכות ואת התשואה של ה- DNA / RNA על ידי ג'ל אלקטרופורזה agarose לרוץ 5 μl על ג'ל 1% agarose 19. מחלקים את חומצות גרעין לשתי aliquots, 15 μl עבור דנ"א 30 μl עבור RNA.
- אחסן את ה- DNA ב -80 ° C עד הצורך. לבודד RNA כפי שמתואר בסעיף הבא.
- הוספת 500 חיץ μl CTAB-פוספט 500 פנול μl: כלורופורם: isoamyl אלכוהול (25: 24: 1) אל הצינור 2 מ"ל חרוז-lysing המכיל 0.5 גרם של משקעים להפוך את הצינור 5-10 פעמים כדי homogenize המדגם.
2. הכנת בדוק RNA ואיכות RNA DNA ללא
- עיכול של ה- DNA
- הוסף 3 μl של DNAse אני הצפת 1.5 μl של DNAse אני 30 נפח μl של חומצות גרעין, לדגור על 37 מעלות צלזיוס למשך 30 דקות.
- מערבבים את מגיב איון DNAse ידי vortexing למשך 30 שניות. להוסיף 4.8 μl של הפתרון המדגם. לדגור על RT במשך 5 דקות, הקש מעת לעת בקרקעית של הצינורות כדי לערבב את הפתרון.
- צנטריפוגה ב XG 10,000 עבור 90 שניות ב RT, להעביר את supernatant (RNA) לתוך צינור חדש, נזהר שלא להעביר משקע אשר יכול לעכב תגובות במורד זרם.
- בדיקת איכות RNA
- הוסף 2 μl של רנ"א 18 μl של DH RNAse ללא סטרילי 2 O לדלל 1:10.
- בתגובות PCR נפרד, להוסיף 1 μl של דילול מסודר או 1:10 של RNA לצינור 0.5 מ"ל PCR סטרילי. הוספת רכיבי תגובת PCR כדי להגביר את גן 16S rRNA לפי טבלה 3. כלול "חיובית" (למשל., DNA שחולץ מן תרבות חיידקים טהורה), ה- DNA "PCR מעכב" שליטה (כלומר, 1 μl של ההתרבות הטהורה חיובי 1 μl של RNA חילוץ מסודר) ושלט "שלילית" של סטרילית RNAse ללא dH 2 O.
- הגדר את thermocycler עם הפרמטרים הגברה הבאים: 95 ° C 5 דקות, (95 מעלות צלזיוס 30 שניות, 57 ° C 30 שניות, 72 ° C 1 דקות) x 35 מחזורים, 72 ° C 10 דקות.
- הפעל 10% מהתוצר PCR על ג'ל agarose 1% ב 85 V עבור 40 דקות. יש לחזור על טיפול DNAse אני אם מוצר PCR נוצר ב RN הסביבתידגימה.
- הוסף 2 μl של רנ"א 18 μl של DH RNAse ללא סטרילי 2 O לדלל 1:10.
דור 3. סטרנד ראשון cDNA מ- RNA
- מוסיפים את ריאגנטים לפי טבלה 4 על RNAse / DNAse חינם צינור 0.2 מ"ל PCR.
- דגירה המדגם על 65 מעלות צלזיוס 5 דקות. מניחים על קרח למשך דקות לפחות 1 ולאחר מכן לדגור על 25 מעלות צלזיוס במשך 10 דקות.
- כדי הצינור אותו ולהוסיף ריאגנטים המפורטים בטבלה 5.
- דגירה המדגם על 55 מעלות צלזיוס למשך 50 דקות. להשבית את התגובה על 72 מעלות צלזיוס למשך 10 דקות.
- cDNA חנות ב -20 ° C עד השימוש.
4. PCR כמותי
- הדור של תקני ה- DNA עבור qPCR
- להגביר חיידקי 16S rRNA רצף 20 (או כל רצף אחרים בעלי עניין) מ- DNA הגנומי חילוץ מתרבות טהור עם פריימרים q-PCR (1369F & 1492R 21).
- לטהר את מוצר ה- PCR באמצעות ערכה מסחרית בהתאם להוראות היצרן.
- לשכפל את ה- PCR מטוהריםמוצר אל סככת 3'-T שיבוט מערכת וקטור פי הוראות היצרן.
- Transform וקטור המכיל את PCR להכניס לתוך coli Escherichia תאים המוסמכות JM109 פי הוראות היצרן.
- פלייט 50 μl של טרנספורמציה על אמפיצילין (100 מיקרוגרם / מ"ל), X-gal (20 מיקרוגרם / מ"ל) ו IPTG (0.5 מ"מ) לוריא Bertani אגר צלחות (Tryptone 10 גר '/ ל, תמצית שמרים 5 גר' / ל, NaCl 10 גרם / L, אגר 15 גר '/ ל). דגירה O / N ב 37 מעלות צלזיוס.
- בחר transformant לבן חיובי אחת מהצלחת. לחסן את transformant חיובי 50 מ"ל מרק LB המכיל אמפיצילין 100 מיקרוגרם / מ"ל ו דגירה O / N רועד ב 250 סל"ד ב 37 מעלות צלזיוס.
- מתרבה O / N, לבודד ולטהר פלסמיד באמצעות ערכה מסחרית בהתאם להוראות היצרן.
- Linearize הפלסמיד באמצעות אנזים הגבלה מתאים כי הוא מסוגל לשסף את הפלסמיד פעם (למשל, EcoRI). > לנתח את הטוהר של פלסמיד על ידי מדידת היחס הקליט 260/280 19. אם היחס הוא מתחת 1.8, לחלץ שנית את הפלסמיד עם פנול, כלורופורם 19. הערה: דנ"א טהור יש יחס 1.8.
- קבע את הריכוז של ה- DNA פלסמיד על ידי ספיגה ב 260 ננומטר באמצעות ספקטרופוטומטר 19.
הערה: לחילופין, את הדיוק של כימות ניתן לשפר על ידי שימוש לצבוע DNA מחייבת פלורסנט הפועל בשיתוף עם fluorometer. - רצף הפלסמיד להכניס לאשר את גודל היעד בבסיסים. לחשב את הסכום (כלומר, אחוז) של DNA להכניס פלסמיד (למשל, ב וקטור 3,000 נ"ב, קטע DNA 123 נ"ב הוא 3.9% של ה- DNA הכולל) הכפל אותו ב הריכוז שהושג 4.1.10, כדי לקבוע את ריכוז ה- DNA של היעד (להוסיף).
- השתמש המשוואה הבאה כדי לחשב את העותקים של יעד לכל μl:
עומס / 54067 / 54067eq1.jpg "/> - חישוב המשקל המולקולרי היעד (TMW) על ידי הכפלת מספר זוגות בסיסים של DNA היעד על ידי המשקל המולקולרי הממוצע של גדיל DNA הכפול (dsDNA, 660 Daltons לכל זוג בסיס).
- לדלל את ה- DNA היעד בטווח בין 10 10 כדי 10 2 עותקים באמצעות 2 μl של דנ"א 18 μl של סטרילי DH 2 O. מערבבים היטב כל דילול ומסובב בקצרה לפני ביצוע דילול הבא.
- הדור של עקומת סטנדרט RNA עבור qPCR
- משלב 4.1.5, מסך מספר מושבות לבנות (כ 5) על ידי מושבת PCR 19 באמצעות פריימר הפוך היעד (כלומר, R1492) ואת פריימר פלסמיד הווקטור קדימה M13F.
הערה: זה שילוב של פריימרים יגביר הוסיף משובט בכיוון התחושה הנכון עם אתר אמרגן T7 במעלה זרם. - לטהר את מוצר ה- PCR באמצעות ערכת מסחרי ממלאים את ההוראות של היצרן. לכמת את p PCR מטוהריםroduct ב 260 ננומטר באמצעות ספקטרופוטומטר.
- הוספת 200 ננוגרם של המוצר PCR בכרך האחרון של 20 μl עם 7.5 מ"מ של כל ribonucleotide, 1 חיץ התגובה T7 ואנזים פולימראז 1 T7 עבור התגובה שעתוק במבחנה.
- דגירת התגובה במשך 4 שעות ב 37 מעלות צלזיוס. הוסף 1 יחידה של RNase ללא DNase אני על התגובה, דגירה במשך 15 דקות ב 37 ° C כדי להסיר תבנית ה- DNA.
- שחזר את הרנ"א עבד במבחנה על ידי אתנול ממטרים 19 ולכמת תשואות RNA באמצעות fluorimeter או על ידי ספיגה ב 260 ננומטר.
- חישוב מספר תמליל לכל μl של רנ"א. מוסיף את מספר זוגות בסיסים ב RNA היעד (למשל, 123 בסיסים) האורך של אמרגן T7 (153 בסיסים) ולהתרבות על ידי המשקל המולקולרי הממוצע של RNA, 340 Daltons לכל בסיס.
- להוסיף 500 ננוגרם של רנ"א היעד כמותית על transcriptase הפוכה תגובה (RT) כפי שמתואר בסעיף 3. סדר לדלל את cDNA כפי שמתואר 4.1.13 לשימושכמו העקומה הסטנדרטית.
- משלב 4.1.5, מסך מספר מושבות לבנות (כ 5) על ידי מושבת PCR 19 באמצעות פריימר הפוך היעד (כלומר, R1492) ואת פריימר פלסמיד הווקטור קדימה M13F.
- PCR בזמן אמת
- DNA הסביבתי הפשרה / דגימות cDNA, תקנים ריאגנטים qPCR על קרח. הגן על החללית fluorogenic מן האור.
- לדלל את הסטנדרטים של עקומת סטנדרט כפי שמתואר 4.1.14. הפוך דילולים 1:10 של ה- DNA סביבתיים / cDNA מן מסודר 10 -3 על ידי הוספת 2 μl של המדגם ל -18 μl של סטרילי DH 2 O.
- הפוך תגובת תערובת אב המספר הכולל של תגובות בתוספת 10% על פי לוח 6 (פריימרים ואת החללית) וטבלת 7 (תערובת תגובה). מערבבים היטב בקצרה צנטריפוגות.
- להוסיף 19 μl של תמהיל מאסטר ו 1 μl של תבנית (סטנדרטי, דיגום סביבתי או מים במשך בקרה שלילית) לכל היטב צלחת 96-היטב אופטי qPCR. בצע כל תגובה (תקנים, דגימות סביבתיות ובקרות שליליות) בשלושת עותקים.
- מכסים את צלחת 96-היטב עם כיסוי אופטי q-PCR. צנטריפוגה הצלחת בקצרה. טענת אתהצלחתי לתוך בלוק החימום של מכונת qPCR ולסגור את המכסה.
- פתח את תוכנת מנהל qPCR. לחץ על הכרטיסייה "פרוטוקול", לחץ על "צור חדש", להוסיף את תנאי ההגברה הבאות: 3 דקות 95 ° C, (10 שניות 95 ° C, 30 שניות 60 ° C) x 40 מחזורים. נפח דגימת גדר עד 20 μl, לחץ על "אישור" ולשמור את הפרוטוקול.
- לחץ על הכרטיסייה "פלייט". תחת "ערוך נבחר" לחץ "בחר fluorophore", להוסיף צבע דוח הבדיקה מתאימה, למשל, והעמסה. לחץ על "אישור".
- דגש בארות המכילות את הסטנדרטים, בחר "רגיל" מהתפריט "סוג המדגם". לחץ "לשכפל סדרה" לשנות "לשכפל גודל" 3, לחץ על "החל". לחץ על "סדרת דילול" להוסיף ריכוז התחלה שחושב 1 סטנדרטי, לציין את הגורם לדילול (10), בחר "ירידה" או "הגדלה" תלוי כדי עקום סטנדרט נוסףלצלחת. לחץ על "חל".
- דגש היטב עמדות המכילות דגימות הידועות, בחר "לא ידוע" מן "סוג המדגם" לתפריט נשלף. לחץ "לשכפל סדרה", ולשנות "לשכפל גודל" עד 3. לחץ על "החל". עריכת שמות מדגם תחת "שם מדגם". הליך חזור על אין בקרות תבנית בארות (NTC).
- לחץ על "אישור" על קופסא עורך הצלחת ולשמור קובץ. לחץ על "להתחיל לרוץ".
- עם השלמה בטווח, חלון ניתוח נתונים יפתח. לחץ על הכרטיסייה "כימות" כדי להציג את עקומת הסטנדרט, מתארי עקומת סטנדרט (לוח 8) ומגרשי הגברה.
- לחץ על כרטיסיית "נתוני כימותים" עבור ערכי Cp ומקביל החל מכמות הגנים (SQ) של דגימות ידועות. ייצא טבלה לתוכנה גליונות אלקטרוניים, אם נדרש.
- שכיחותם גן כפל ממדגמים לא ידועים על ידי גרם לדילול, כמות המשקעים extracטד מ ואת חומצות גרעין הנפח הכולל היו eluted כדי להשיג עותקי גן לגרם של משקעי משקל רטובים.
הערה: Elute דנ"א 0.5 גרם של משקעים 50 μl, לדלל 1/10, להוסיף 1 μl כתבנית לתגובה q-PCR; עותקים גן לחשב g -1 משקעים ידי הכפלה: עותקי גן μl של נפח elution x גורם לדילול x DNA x 2.
תוצאות
הפקת DNA באיכות טובה ו- RNA ממשקעים היא הצעד הראשון בכימות שכיחותם גני תמליל. תשואות חילוץ מוצלחות להקות ה- DNA ו- RNA ברורות כמצוין באיור 1 עבור המדגם AC, שבו 23S ו 16S חדו להקות rRNA גלויות בנוסף להקת הדנ"א הגנומי המשקל המולקולרית הגבוהה.
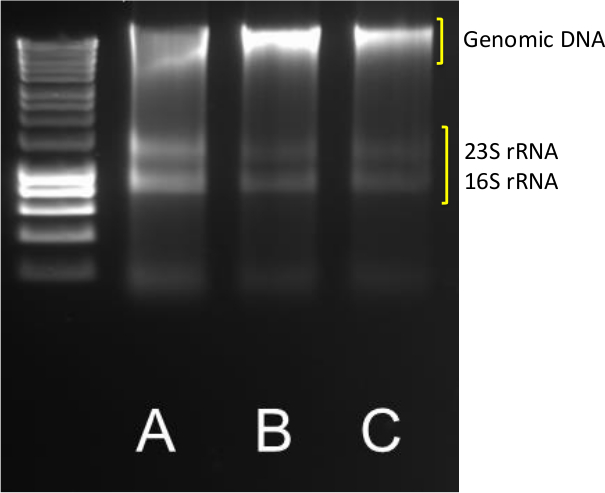
באיור 1. DNA / RNA החילוץ. תוצאות אופייניות מ- DNA / מיצוי RNA מן בשלושה עותקים (ABC) 0.5 גרם משקעים החוף. 5 μl של רנ"א DNA / נדרס על agarose 1.4% ב 85 V עבור 40 דקות. סמן מולקולרי בטווח 10,037-200 נ"ב היה בשימוש. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
-together.within-page = "1"> כדי להכין RNA, עיכול של ה- DNA שיתוף חילוץ הוא חובה. זה חייב להיות מלווה 16S rRNA נקודת הסיום PCR של רנ"א על מנת להבטיח כי ה- DNA הוסר בהצלחה. אם ה- DNA הוסר לחלוטין רק להקה בבקרה החיובית הוא ציין. חשוב להשתמש בשני מסודר 1:10 דילול של RNA כדי להבטיח מעכבים אינם מונעים ההיווצרות של מוצר ה- PCR. RNA יכול עכשיו לעבור את התגובה transcriptase הפוכה להמירו cDNA. זה יכול להתבצע עם פריימרים משני או hexamers אקראי גן ספציפי. בדרך כלל, התגובה מתבצעת במכונת PCR, כדי להבטיח שפרופיל טמפרטורה אופטימלי. cDNA זה משמש כתבנית תגובת qPCR הבאה. DNA ו- RNA הוא לכמת לקביעת ריכוז התבנית בשימוש כל תגובה.
פרוטוקול התגובה qPCR המתואר מיקוד 16S rRNA DNA. עבור 16S rRNA תמלילים להחליף את מ"ק התקןRVE ותבנית עם cDNA. כמו דגימות ידועות הן לכמת נגד עקומת הסטנדרט, זה הכרחי כדי להבטיח את העקומה הסטנדרטית היא באיכות טובה. תרשים 2 מציג את ההכנה (א), עקומות סטנדרטיות דילולי DNA הסביבתי, (B), ההגברה של עקומת הסטנדרט דגימות סביבתיות (C) מרה של עקומת סטנדרט כדי רגרסיה ליניארית וחישוב של מספרי עותק הגן. כאשר מגוון דילול של פי 10 מוכן כראוי ומוגברי הבדל 3.3 מחזור בין כל דילול סטנדרטי נתפסת (זה לוקח 3.3 מחזורי גידול של פי עשר תבנית ב 100% יעילים הגברה) (איור 2Bi). מתארי עקומות סטנדרטיים, כוללים ערכי 2 R של 0.99 ויעילויות PCR בתוך בין 90 ל- 110% (איור 2 ג) הם רצויים. חשוב לדווח על נתוני Cp מן אין שליטת תבנית (NTC) אם הוא קיים. במקרה כזה הפסקת Cp עבור תקני דגימות ידועות 3.3 מחזורים ( כלומר, קפל יומן) גבוה משווי Cp של NTC מוטל 20 (איור 2Cii). תבנית לא ידועה מופקת DNA המשקע הייתה לכמת מ מסודר, 10 -1, 10 -2 ו 10 -3 דילולים (B). המדגם המסודר לא להגביר, ערכי Cp של 10 -1 עד 10 -3 דילולים היו 24.12, 26.02 ו 28.40 בהתאמה. NTC Cp היה 30.5, גידול הפסקת NCT של 27.2 הוטל. המרת שכיחותם גן g -1 משקעי משקל רטובים הביאו 2.5 x 10 7 ו 7.1 x 10 7 ל -10 -1 ו -10 -2 בהתאמה עבור מגוון הדילול. 10 -3 Cp דילול היה מתחת לציון NTC, והיה ולכן לא נעשה שימוש. במקרה זה 10 -2 נבחרת דילול התבנית האופטימלית.

איור 2. 16S rRNA גן QPCהגברת R של תקני DNA הסביבה מופק משקעי החוף. הכנה (א) i) עקום DNA סטנדרטי ii) דילולי DNA הסביבתי הגברת qPCR, (B) qPCR הגברה של i) עקום סטנדרט DNA ו- II) דגימות סביבתיות, Cp עבור כל דגימה מוצגות. (C) i) רגרסיה לינארית של עקומת סטנדרט עם מתארי עקומת סטנדרט; ii) חישוב שכיחותם גנים בין דגימות סביבתיות. NTC:. בקרת תבנית שלילית אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| מֵגִיב | כמות |
| CTAB | 5 גרם |
| K 2 HPO 4 x3H 2 O | 2.58 גרם |
| KH 2 PO 4 | 0.1 גרם |
| NaCl | 2.05 גרם |
| DH 2 O | עד 100 מ"ל |
הכנת טבלה 1. חיץ CTAB-פוספט.
| מֵגִיב | כמות |
| PEG 6000 | 30 גרם |
| NaCl | 9.35 גרם |
| DH 2 O | עד 100 מ"ל |
| הערה: להוסיף PEG 6000 לאט מתחת לחימום ערבוב מתון של 50 מ"ל של DH 2 O. | |
בתוך-page = "1"> הכנת טבלה 2. פתרון ממטרים PEG-NaCl.
| מֵגִיב | כמות |
| RNA חי RNA / 01:10 מדולל | 1.0 מיקרוליטר |
| 10x PCR הצפה | 5.0 μl |
| dNTPs 10 מ"מ | 1.0 מיקרוליטר |
| פריימר 63F קדימה | 1.0 מיקרוליטר |
| CAG GCC TAA GTC CAA CAC ATG | |
| פריימר 518R ההפוך | 1.0 מיקרוליטר |
| ATT ACC מהפקולטה לרוקחות אוכלים GCT GCT GG | |
| פולימראז תקי | 0.4 μl |
| DH RNAse ללא 2 O | 40.6 μl |
e = "1"> לוח 3. שילוב הורים PCR בדיקת איכות RNA ללא DNA.
| מֵגִיב | כמות |
| RNA | 0.5-5 מיקרוגרם |
| dNTPs 10 מ"מ | 1 μl |
| פריימר הפוך 10 מיקרומטר / Random hexamers 50 מיקרומטר | 1 μl |
| DEPC-H 2 O | עד 13 μl |
תמהיל לוח 4. cDNA תגובת סינתזת א
| מֵגִיב | כמות |
| מאגר 5x | 4 μl |
| DTT | 1 μl |
| RNAse מונע | 1 μl |
| transcriptase הפוכה | 1 μl |
תמהיל לוח 5. cDNA תגובת סינתזת B.
| יַעַד | שם פריימר | סדר פעולות | T חישול (° C) | התייחסות |
| 16S rRNA חיידקים | Bact1369F | CGG TGA ATA CGT TCY CGG | 56 | סוזוקי et al 2000. 19 |
| Prok1492R | GGW TAC CTT GTT ACG ACT T | |||
| TM1389F (בדיקה) | CTT GTA CAC ACC GCC CGT C |
| מֵגִיב | כמות |
| 2x מיקס מאסטר | 10 μl |
| פריימר קדימה (10 מיקרומטר) | 0.8 μl |
| פריימר הפוך (10 מיקרומטר) | 0.8 μl |
| Probe (10 מיקרומטר) | 0.4 μl |
| מַיִם | 7.0 μl |
| נפח סופי | 19 μl |
טבלה 7. בזמן אמת תגובת PCR לערבב.
| מתאר | מַשְׁמָעוּת |
| מקדם המתאם (R 2) | מדד של הליניאריות של עקומת סטנדרט. באופן אידיאלי זה צריך להיות R 2 = 1. ערך של R 2 = 0.98-0.99 כשרים. |
| סלופ (α) | מדד של יעילות התגובה. באופן אידיאלי זה צריך להיות שווה ערך ל -3.32. ערך בטווח שבין -3.58 ו -3.1. |
| יעילות (= 10 (-1 / שיפוע) -1) | אם זה 100%, את התבניות מכפילות לאחר כל מחזור תרמי במהלך הגברת מעריכים. מגוון יעיל טוב הוא בין 90 ו -110%. |
| Y יירוט (β) | לגבול התיאורטי של זיהוי של התגובה. אמנם לא משמש לכמת רגישות התגובה, חשוב כאשר משווים עקומות סטנדרטיות שונות של אותה מטרה. |
לוח 8: QPCמתארי תגובת R.
Discussion
השילוב של מיצוי DNA / RNA עם qPCR מספק שיטה מהירה, מדויקת, יחסית חסכונית עבור הכימות הרגיש של שכיחותם גני תמליל מתוך מגוון רחב של דגימות סביבתיות, כגון משקעי החוף.
מיצוי חומצות הגרעין הראשוני הוא השלב הקריטי כדי להבטיח תצוגת נציג של ההווה קהילת חיידקים מושגים 22. מספר מגבלות צריכים להילקח בחשבון עבור פרוטוקול החילוץ: א) השגת תמוגה התא הכולל, ב) שיתוף החילוץ של תרכובות מעכבות (למשל, חומצות humic או polyphenolics), ג) זיהום DNA בשבריר RNA, ד) שפלה מהירה של חומצות גרעין שחולצו כאשר הם מאוחסנים. אמצעי זהירות יש לנקוט כדי לעקוף מגבלות אלו. לדוגמא, תשומת לב מיוחדת יש לנקוט על מנת להבטיח כי החילוץ של חומצות גרעין מותאם במיוחד עבור סוג המדגם (למשל, משקעים, אדמה, שפכים, וכו '). שיפורים משמעותיים תשואת חומצות גרעין ואיכות הוא DNA ו- RNA יכולים להיות מושגת על ידי ביצוע ניסויים ראשוניים 23. כדי למזער את ההשפעה של תרכובות מעכבות על יישומים מבוססי PCR 24, לבדוק מגוון של דילולים מ- DNA ו- RNA חילוץ. כדי לעקוף את השפלה המהירה של DNA / RNA מופק להקפיא להפשיר מחזורים מרובים למנוע אובדן אפשרי של מידע גנטי, aliquots נפח קטן מרובים ולאחסן ב -80 מעלות צלזיוס.
כאשר תוכנן בקפידה, qPCR היא שיטה חזקה, מאוד לשחזור ורגישה. יש לציין, כי שיטות הגברה ובנייה עקומת בתקן המפורט בפרוטוקול זה יכול להיות מותאם לכל יעד הגן של עניין, כולל סמנים פילוגנטי אחרים (כלומר, rRNA 16S archaeal, rRNA 18S פטרייתי) או גנים המעורבים פונקציות חשובות בסביבה. מגבלות ידועות בשימוש qPCR הן: א) הדור של באיכות גבוהה לשחזורעקומת tandard כימות מוחלטת, ב) הבחירה של פריימר / בדיקות ואופטימיזציה של תנאי assay q-PCR, ג) השימוש באיכות נמוכה / חומצות גרעין טעון, ד) הבחירה של דילול עובד של RNA DNA / להימנע עיכוב . יתר על כן, זה חייב להיחשב כי טכניקת qPCR מספקת גן / שכיחותם תמליל אשר לא יכול להשוות לתא ספירות: זה נכון במיוחד במקרה כאשר 16S ו 18S גני rRNA ממוקדים, כמו שיש מיקרואורגניזמים מספרים עותק שונה של גן ריבוזומלי בגנום שלהם 25.
עקומות סטנדרטיות באיכות ירודה תגרומנה כימות מדויק של הגן של עניין. זה תרגול טוב כדי לאחסן מלאי של סטנדרטי ריכוז גבוהים aliquots הקטן שממנו עקומות סטנדרטיות טריות יכולות להתבצע. אין לאחסן עקומות סטנדרטיות, תמיד לעשות דילולים טריים מלאה של הריכוז הגבוה ביותר בכל פעם. עבור כימות מדויק של דגימות סביבתיות להבטיח את טווח הריכוזים של סטןעקומת dard משתרעת על פני ערכי Cp הצפויים של הדגימות הידועות. כאשר לכימותי תמלילים, לבנות את עקומת סטנדרט מ- RNA לא להכפיל גדילי הדנ"א. במידת האפשר, כימות של דגימות כדי להשוות ישירות צריך להסתיים בתוך assay אחת להימנע וריאציה-assay היתר 20. זה לא תמיד אפשרי. לכן, כדי להשוות שכיחותם גן שנוצרה בין מבחנים רצוי יש סט-משנה אקראי של דגימות המשוכפלות בין מבחנים. מבחר רחב של ערכות פריימר בדיקה זמין כרגע עבור qPCR מיקוד מיני מיקרוביאליים קבוצות פונקציונליות 15. בחינה קפדנית יש צורך בבחירה אלה כדי להבטיח הן כיסוי מרבי וספציפי קבוצת היעד. אם יעילות התגובה של assay qPCR אינה משביעת רצון, פתרון בעיות מתחילות עם בדיקת תנאי רכיבת תרמית שונה (ככל שזמן חישול ו / או טמפרטורה) ו / או תנאי תגובה, למשל, ריכוזים שונים פריימר-בדיקה. ONCE Q-PCR assay ותנאי התגובה ממוטבים, תמיד לערוך בדיקה ראשונית של מגוון רחב של דילולים DNA / cDNA כדי לקבוע את הריכוז תבנית מתאימה. בחר את טווח הדילול וכתוצאה מכך מספר העותק הגבוה ביותר כמו דילול התבנית האופטימלי עבור מבחנים נוספים.
נכון לעכשיו, טכנולוגיות רצף הדור הבא לשפוך אור ביעילות על מבנה הקהילה חיידקים ופונקציות בתוך שפע של סביבות 26,27,28. עם זאת, מערכי נתונים אלה מבוססים לעתים קרובות על ספריות amplicon נקודת סיום PCR ולכן מספקים רק ערכות וכמותיות של השפע של מינים מסוימים. לפיכך, את היכולת של טכניקת PCR מבוססת בזמן האמת למקד סמנים טקסונומיות ספציפיים (מדומיין גבוה עד לרמת מתח) מאפשר אימות יעילה של התוצאות שהושגו על ידי רצף של הדור הבא. יתר על כן, qPCR שמש בהצלחה בשילוב עם שיטות מולקולריות אקולוגיה מיקרוביאלית אחרות כגון ISO היציבהלהשתכר חיטוט (SIP) או microarrays פילוגנטי / תפקודי. בשילוב עם הכלי לשעבר, qPCR יכול לשמש כדי לכמת את הקהילה פעילה מבחינה מטבולית 29,30. בשילוב עם ניתוח microarray, qPCR מספק פרשנות כמותית המפתח של מבוססי סמן פילוגנטי גן פונקציונלי סקרים של סביבות 31,32.
לכן, אם להשתמש לבד או בשילוב עם אחרים (לעתים קרובות תהליך מבוסס) ערכות של תפקוד מערכת אקולוגי, PCR כמוני הוא כלי חיוני עבור אקולוגים מיקרוביאלי בחיפוש של הקישור החמקמק בין קהילות חיידקי פונקציות מערכת אקולוגיות.
Disclosures
The authors have nothing to disclose.
Acknowledgements
פרסום זה נובע מן המחקר נערך בתמיכת הכספיים של המועצה למחקר הסביבה הטבעית (NERC) תחת מספר מענק NERC NE / JO11959 / 1 ו קרן מדע אירלנד & the COFUND פעולת מארי קירי תחת גרנט מספר 11 / SIRG / B2159awarded כדי CJs ואת קונסורציום המחקר האקדמי המזרח (מזרח ARC).
Materials
| Name | Company | Catalog Number | Comments |
| Diethylpyrocarbonate | Sigma | 40718 | Toxic, open under chemical hood |
| cetrimonium bromide (CTAB) | Sigma | 52365 | Irritant, open under chemical hood |
| potassium phosphate dibasic | Sigma | RES20765 | |
| potassium phosphate monobasic | Sigma | P9791 | |
| Phenol : Chloroform : Isoamylalcohol pH 8 | Sigma | P2069 | Equilibrate at pH 8 before using |
| Chloroform : Isoamylalcohol | Sigma | 25666 | |
| sodium chloride | VWR | 1.06404.0500 | |
| polyethylenglycol 6000 | VWR | 528877-100 | |
| Ethanol Molecular Grade | Sigma | E7148 | |
| Lysing Matrix E tubes | MP Biomedical | 116914050 | |
| Turbo DNAse | Ambion | AM1907 | |
| Taq polymerase | Sigma | D1806 | |
| dNTPs | Bioline | BIO-39028 | |
| SuperScript III | Life Technologies | 18080044 | |
| Rnase Inhibitor | Life Technologies | 10777019 | |
| RNAse/DNAse free 0.2 ml PCR tubes | Sarstedt | 72.985.002 | |
| SureCleanPlus | Bioline | BIO-37047 | |
| pGEM Easy T Vector | Promega | A1360 | |
| E. coli JM109 competent cells | Promega | L2005 | |
| Tryptone | Sigma | T7293 | |
| Yeast Extract | Sigma | 70161 | |
| Ampicillin | Sigma | 59349 | |
| X-Gal | Bioline | BIO-37035 | |
| IPTG | Bioline | BIO-37036 | |
| Agar | VWR | 20768.235 | |
| Plasmid Midi Kit | Qiagen | 12143 | |
| Qubit | Life Technologies | Q33216 | |
| Quant-IT DNA HS Assay | Life Technologies | Q-33120 | |
| Quant-IT RNA HS Assay | Life Technologies | Q32855 | |
| MEGAshortscript kit | Ambion | AM1354 | |
| TaqMan SensiFast Probe Lo-ROX kit | Bioline | BIO-84002 | |
| qPCR machine |
References
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr. Opin. Microbiol. 16 (5), 636-642 (2013).
- Zhou, J., Bruns, M. A., Tiedje, J. M. DNA recovery from soils of diverse composition. Appl. Environ. Microbiol. 62, 316-322 (1996).
- Miller, D. N., Bryant, J. E., Madsen, E. L., Ghiorse, W. C. Evaluation and optimization of DNA extraction and purification procedures from soil and sediments samples. Appl. Environ. Microbiol. 65 (11), 4715-4724 (1999).
- Burgmann, H., Pesaro, M., Widmer, F., Zeyer, J. A strategy for optimizing quality and quantity of DNA extracted from soil. J. Microbiol. Methods. 45 (1), 7-20 (2001).
- Hurt, R. A., et al. Simultaneous recovery of RNA and DNA from soils and sediments. Appl. Environ. Microbiol. 67, 4495-4503 (2001).
- Griffiths, R. I., Whiteley, A. S., O'Donnell, A. G., Bailey, M. J. Rapid method for coextraction of DNA and RNA from natural environments for analysis of ribosomal DNA- and rRNA-based microbial community composition. Appl. Environ. Microbiol. 66 (12), 5488-5491 (2000).
- Martins, G., et al. Structure and activity of lacustrine sediment bacteria involved in nutrient and iron cycles. FEMS Microbiol. Ecol. 77 (3), 666-679 (2011).
- Kuffner, M., et al. Effects of season and experimental warming on the bacterial community in a temperate mountain forest soil assessed by 16S rRNA gene pyrosequencing. FEMS Microbiol. Ecol. 82 (3), 551-562 (2011).
- Tatti, E., et al. Influences of over winter conditions on denitrification and nitrous oxide-producing microorganism abundance and structure in an agricultural soil amended with different nitrogen sources. Agric. Ecosyst . Environ. 183, 47-59 (2014).
- Giovannoni, S. J., Britschgi, T. B., Moyer, C. L., Field, K. G. Genetic diversity in the Sargasso Sea bacterioplankton. Nature. 345, 60-62 (1990).
- Ward, D. W., Weller, R., Bateson, M. M. 16S rRNA sequences reveal numerous uncultured microorganisms a natural community. Nature. 345, 63-65 (1990).
- Suzuki, M. T., Giovannoni, S. J. Bias caused by template annealing in the amplification of mixture of 16S rRNA genes by PCR. Appl. Environ. Microbiol. 62, 625-630 (1996).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous fluorescence monitoring of rapid cycle DNA amplification. Biotechniques. 22, 130-138 (1997).
- Holland, P. M., Abramson, R. D., Watson, R., Gelfand, D. H. Detection of Specific Polymerase Chain Reaction Product by Utilizing the 5' 3' Exonuclease Activity of Thermus aquaticus DNA Polymerase. PNAS. 88, 7276-7280 (1991).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol. Ecol. 67, 2-20 (2009).
- Park, S. J., Park, B. J., Rhee, S. K. Comparative analysis of archaeal 16S rRNA and amoA genes to estimate the abundance and diversity of ammonia-oxidizing archaea in marine sediments. Extremphiles. 12 (4), (2008).
- Urakawa, H., Yoshida, T., Nishimura, M., Ohwada, K. Characterization of depth-related population variation in microbial communities of a coastal marine sediment using 16S rDNA-based approaches and quinone profiling. Environ. Microbiol. 2, 542-554 (2008).
- Smith, C. J., Dong, L. F., Wilson, J., Stott, A., Osborn, A. M., Nedwell, D. B. Seasonal variation in denitrification and dissimilatory nitrate reduction to ammonia process rates and corresponding key functional genes along an estuarine nitrate gradient. Front. Microbiol. 6, 542(2015).
- Sambrook, J., Russell, D. W. Molecular Cloning: a laboratory manual. , Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press. 205(2001).
- Smith, C. J., Nedwell, D. B., Dong, L. F., Osborn, A. M. Evaluation of Quantitative Polymerase Chain Reaction (Q-PCR) based approaches for determining gene copy and gene transcript numbers in environmental samples. Environ. Microbiol. 8 (5), 804-815 (2006).
- Suzuki, M. T., Taylor, L. T. DeLong E.F., Quantitative analysis of small-subunit rRNA genes in mixed microbial population via 5'-nuclease assays. Appl. Environ. Microbiol. 66, 4605-4614 (2000).
- Luna, G. M., Dell'Anno, A., Danovaro, R. DNA extraction procedure: a critical issue for bacterial diversity assessment in marine sediments. Environ. Microbiol. 8 (2), 308-320 (2005).
- Lever, M. A., Torti, A., Eickenbursch, P., Michaud, A. B., Šantl-Temkiv, T., Jørgensen, B. B. A modular method for the extraction of DNA and RNA, and the separation of DNA pools from diverse environmental sample types. Front. Microbiol. 6, 476(2015).
- Lloyd, K. G., MacGregor, B. J., Teske, A. Quantitative PCR methods for RNA and DNA in marine sediments: maximizing yield while overcoming inhibition. FEMS Microbol. Ecol. 72, 144-151 (2010).
- Klappenbach, J. A., Dumbar, J. M., Schmidt, T. M. rRNA Operon copy number reflects ecological strategies of bacteria. App. Environ. Microbiol. 66 (4), 1328-1333 (2000).
- Shi, Y., Tyson, G. W., Eppley, J. M., DeLong, E. F. Integrated metatrascriptomic and metagenomics analyses of stratified microbial assemblages in the open ocean. ISME J. 5, 999-1013 (2011).
- Howe, A. C., Jansson, J. K., Malfatti, S. A. Tringe S.G., Tiedje J.M., & Brown C.T. Tackling soil diversity with the assembly of large, complex metagenomes. PNAS. 111 (13), 4904-4909 (2014).
- Klaedtke, S., et al. Terroir is a key driver of seed-associated microbial assemblages. Environ. Microbiol. , (2015).
- Lueders, T., Wagner, B., Claus, P., Friedrich MW, Stable isotope probing of rRNA and DNA reveals a dynamic methylotroph community and trophic interactions with fungi and protozoa in oxic rice field soil). Environ. Microbiol. 6, 60-72 (2004).
- Kunapuli, U., Lueders, T., Meckenstock, R. U. The use of stable isotope probing to identify key iron-reducing microorganisms involved in anaerobic benzene degradation. ISME J. 1, 643-653 (2007).
- Bürgmann, H., et al. Transcriptional response of Silicibacter pomeroyi DSS-3 to dimethylsulfoniopropionate (DMSP). Environ. Microbiol. 9 (11), 2742-2755 (2007).
- He, J. Z., et al. Geochip: a comprehensive microarray for investigating biogeochemical ecological and environmental processes. ISME J. 1, 67-77 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved