Method Article
リアルタイムPCRによる沿岸堆積物および定量16SのrRNA遺伝子と転写物からの同時DNA-RNA抽出
要約
A protocol to quantify bacterial 16S rRNA genes and transcripts from coastal sediments via real-time PCR is provided. The methodology includes the co-extraction of DNA and RNA; preparation of DNA-free RNA; and 16S rRNA gene and transcript quantification via RT-q-PCR, including standard curve construction.
要約
また、定量的PCR(Q-PCR)として知られているリアルタイムポリメラーゼ連鎖反応は、環境試料中の分類学上の官能基の遺伝子の存在量を定量するための微生物の生態において広く使用されるツールです。逆転写酵素反応(RT-Q-PCR)と組み合わせて使用され、また、遺伝子の転写を定量化するために使用することができます。 Q-PCR反応の指数期の間のPCRアンプリコンの定量化を可能にする高感度な蛍光検出化学物質を利用します。したがって、PCRは、PCR反応のプラトー段階で検出された「エンドポイント」に関連付けられたバイアスが回避されます。 PCRが提供されているリアルタイム経由して沿岸の堆積物からの細菌の16S rRNA遺伝子と転写産物を定量化するためのプロトコル。まず、DNAを含まないRNAを調製するために必要な追加のステップを含む沿岸堆積物からのDNAおよびRNAの共抽出のための方法は、概説されています。第二に、16S rRNA遺伝子とTRの定量化のためのステップバイステップガイドQ-PCR及びRT-Q-PCRを介して抽出された核酸からanscriptsが概説されています。これは、DNAおよびRNA標準曲線を構築するための詳細を含みます。微生物生態学におけるRT-Q-PCRアッセイを使用するための重要な考慮事項が含まれています。
概要
微生物は、生態系の機能を駆動する生物圏の隅石です。微生物の大半は、1培養されていないまま。したがって、分子ベースのアプローチは、環境中の微生物の多様性と機能の我々の理解を進めるために必須です。これらのアプローチの中心環境サンプルからの核酸の抽出およびポリメラーゼ連鎖反応(PCR)を使用して標的遺伝子のその後の増幅です。
DNA / RNA抽出の最初のステップは、微生物群集の存在の細胞壁を溶解し、望ましくない非核酸分子( 例えば 、有機及び無機物質)を除去し、さらに下流の分析のために、溶液中のDNA / RNAを保持することを目指しています。市販の抽出キットの範囲を含む文献2,3,4,5で利用可能ないくつかのオプションのうち、グリフィス法6は、広く7,8,9を採用しています。これは、費用対効果とペンシルバニアそれは細胞を溶解するビーズ破っステップを使用し、同時にDNAやRNAを回収しながら、このようなフミン酸などのPCR阻害剤の同時抽出を最小限にするための手順を組み込んだとしてrticularlyよく堆積物に適しています。
第二段階は、抽出された核酸から、例えば、16S rRNAの分類学的マーカーとして標的遺伝子を増幅するためにポリメラーゼ連鎖反応(PCR)を使用します。このアプローチは、持っていると特徴付けられていない微生物のブラックボックス10,11の探査を促進するために続けています。しかし、エンドポイントPCRベースの方法は、缶バイアス微生物群集12の特性様々な制限に悩まされています。正確に遺伝子/転写産物の存在量を定量化するために、また定量的PCR(定量PCR)として知られているリアルタイムPCRは、使用する必要があります。定量PCRは、PCRの各サイクルの後にアンプリコンの蓄積を追跡する蛍光レポーター色素システムを利用します。それは定量化ではなく、初期の指数期中に発生する可能性があることを意味し、これは重要ですアンプリコンの収率は依然として標的遺伝子の初期存在量に正比例するPCR反応の終点、相より。
二つのレポーター系は、一般的に使用される:挿入核酸染色13及びDNAポリメラーゼ14の5 '→3'エキソヌクレアーゼ活性を。前者のレポーター系は、全ての二本鎖DNAに結合するのでindistinctively副生成物の望ましくない非特異的アンプリコンまたはプライマーダイマーが発生した場合には、標的配列の過大評価をもたらし得ます。これを回避するために、増幅の大規模な最適化が必要とされ得ます。 後者のシステムでは、鋳型の増幅は、内部プローブから開裂し、フルオロフォアを、Taqポリメラーゼの5 'ヌクレアーゼ活性の組み合わせを使用して追跡されます。この機能はのみによる相補的な標的特異的sequencに結合する蛍光プローブの利用にアッセイの特異性を増大させますプライマー対間で電子。両方の化学で定量は蛍光の増加によって測定されるPCRアンプリコンの蓄積は、有意にバックグラウンド蛍光を超えているクロッシングポイント(CP)を決定することによって達成されます。
定量PCRは、広範囲に異なる環境15における遺伝子存在量を決定するために、微生物の生態に使用されています。また、cDNAとRNAの逆転写は、遺伝子発現を定量する定量PCRおよびRT-qPCRを用いて合成されます。したがって、定量PCRおよびRT-qPCRのは、環境試料中の遺伝子および/または転写産物の数の定量化のための高速で、効果的な方法を表します。
沿岸堆積物中の微生物は有機物の無機化、汚染物質の分解や、窒素16,17,18などの主要栄養素の生物地球化学的循環を含む様々な生態系プロセスを駆動します。これらの変換の徹底的な理解は、包括的なaccounが必要です遺伝子と転写産物の存在量に関する定量的データを含む貢献微生物群、のトン。ここでは、沿岸の堆積物中の細菌の16S rRNA遺伝子と転写産物の存在量の定量化のために徹底的にテストされ、合理化および標準化されたプロトコルのシリーズをご紹介します。プロトコルは、サンプル収集、同時DNAとRNA抽出、DNAフリーのRNA調製、抽出された核酸の品質チェック、16S rRNAの-DNAと-RNA標準の発生や環境試料の定量化の概要を説明します。ここで説明する方法に由来し、定量的データは、沿岸生態系を駆動する微生物群集に光を当てるために必要とされます。
プロトコル
海洋沿岸堆積物から1 DNA&RNA抽出
- リボヌクレアーゼ(RNアーゼ)フリー素材とワークスペースの準備
- 0.1%のジエチル(DEPC)で処理することにより無RNAse蒸留水(のdH 2 O)を準備し、37°CO / Nでインキュベートします。 121℃で15分間のdH 2 Oオートクレーブにより残留DEPCを削除します。使用DEPCは、すべてのソリューションを作るためのdH 2 Oを処理しました。
- 180°CO / Nですべてのガラス器具を焼きます。 、プラスチック製のふたとリムを取り外して2 M NaOHでO / Nの溶液中でそれらを浸し、使用前のdH 2 O DEPC処理でそれらを洗い流してください。
- 表1の通りCTABリン酸緩衝溶液を準備します。
- 表2のとおりPEG-のNaCl沈殿溶液を準備します。
- 開始する前に、RNAseを除染液を用いて、すべての作業面( すなわち 、ベンチ、微量、ヒュームフード)とマイクロピペットを清掃してください。全体の手順の間に手袋を着用してください。
- サンプルのクロスコンタミネーションを回避するために、マイクロピペットフィルターチップと一緒に無RNAseプラスチック製品を使用してください。
- 環境サンプルの収集と保管
- トップ2.5センチメートルから滅菌50ミリリットルのファルコンチューブを使用して、干潮時に沿岸の堆積物を収集します。堆積物の15〜20グラム収集します。慎重にサンプル収集後蓋を閉じます。即時処理のために4℃でクーラー内の研究室にすぐに輸送。
- 2ミリリットルビーズビーティングチューブに湿潤重量堆積物の無菌へらや分量0.5グラムと混合することによって、サンプルをホモジナイズします。小分けし、追加の複製、液体窒素中に配置することにより、フラッシュ凍結。 -80℃で保存のアリコート。注意:液体窒素を扱うときは保護手袋及び安全ゴーグルを着用します。
注:現場は現場で2ミリリットル滅菌マイクロチューブに実験室、アリコート堆積物の0.5グラムの近くに配置されていない場合。すぐに李にそれらを置くことによって、チューブをフラッシュ凍結研究室に頭の良い窒素と輸送。処理まで-80℃で保存サンプル。
- 堆積物からのDNAとRNAの同時抽出
注:以下のプロトコルは、グリフィス法6の修正版です。- クロロホルム:イソアミルアルコール(25:24:1)500μlのCTAB-リン酸緩衝液および500μlのフェノール追加2ミリリットルの堆積物の0.5グラムを含むビーズ溶解チューブを、サンプルを均質化するためにチューブを5〜10回転倒。
注意:すべての回で、適切な保護具( すなわち 、実験室のコート、手袋、安全ゴーグル)を着用してヒュームフードでこの手順を実行します。 - 2.5分間フルスピードでボルテックス。 4℃で10分間、16,000×gで遠心分離します。
- ヒュームフードでは上部の水層を抽出し、新しい1.5ミリリットル滅菌マイクロチューブに移します。氷冷クロロホルム500μlのを追加し、氷上でサンプルを維持する:イソアミルアルコール(24:1)。
- エマルジョンがvisibになるまで数回転倒ル。 4℃で16,000×gで10分間遠心分離します。
- ヒュームフードでは上部の水層を抽出し、新しい1.5ミリリットル滅菌チューブに入れてください。 30%PEG-NaCl溶液の2つのボリュームが追加することにより、核酸を沈殿させ、よく混ぜます。
- 2時間氷上でインキュベートまたは4°CO / Nでサンプルを残します。
- 4℃で20分間、16,000×gでのインキュベーション遠心分離機サンプルの終わりに。
注:ペレットは、チューブの底に見えるかもしれません。 - 静かにマイクロピペットを用いて上清を除去し、ペレットを乱さないようにしてください。チューブにPEG溶液の約10μlのままにしておき、氷冷70%エタノール1 mlを追加します。
- 4℃で16,000×gで20分間遠心分離します。ゆっくりペレットに触れないように注意しながらマイクロピペットでエタノールを除去。
- 5秒間遠心分離し、マイクロピペットを用いて、残留エタノールを除去。約5分間空気乾燥さペレットを残します。
- 50μlのDEPC処理中にペレットを再懸濁H 2 O. 1%アガロースゲル19に5μLを実行して、アガロースゲル電気泳動によりDNA / RNAの品質や収量を調べます。 、2つのアリコートに、DNAやRNAのために30μlの15μLを核酸を分割します。
- 必要になるまで-80℃でDNAを保管してください。次のセクションで説明するとおりにRNAを分離します。
- クロロホルム:イソアミルアルコール(25:24:1)500μlのCTAB-リン酸緩衝液および500μlのフェノール追加2ミリリットルの堆積物の0.5グラムを含むビーズ溶解チューブを、サンプルを均質化するためにチューブを5〜10回転倒。
DNAフリーのRNAのRNAと品質チェックの調製
- DNAの消化
- 37℃で30分間インキュベートし、核酸の30μlのボリュームにDNアーゼIバッファーの3μlのおよびDNAse Iの1.5μlを添加します。
- 30秒間ボルテックスすることによってDNアーゼ不活性化試薬を混ぜます。サンプルへの溶液の4.8μLを加えます。時折溶液を混合するためにチューブの底をタップし、室温で5分間インキュベートします。
- RTで90秒間万×gで遠心し、新しいチューブに上清(RNA)を転送、下流の反応を阻害することができ、沈殿物を転送しないように注意しながら。
- RNAの品質チェック
- 1:10に希釈し、滅菌無RNAseのdH 2 Oの18μlに、RNAの2μLを加えます。
- 別々のPCR反応では、滅菌0.5ミリリットルのPCRチューブにそのまま、またはRNAの1:10希釈の1μlを添加します。 。 表3のとおり、16S rRNA遺伝子を増幅するためのPCR反応成分を加える「陽性」( 例えば 、純粋な細菌培養物から抽出したDNA)、「PCR阻害剤」コントロールを含める( すなわち 、正の純粋培養のDNA1μlのと1ニート抽出したRNAのμl)を、滅菌し無RNAseのdH 2 Oの「負」のコントロール
- 次の増幅パラメータでサーモサイクラーを設定:95℃5分、(95℃30秒、57℃30秒、72℃1分)を35サイクル、72℃10分xは。
- 40分間85 Vで1%アガロースゲル上でPCR産物の10%を実行します。 PCR産物は、環境RNに形成されている場合のDNAse I処理を繰り返しますサンプル。
- 1:10に希釈し、滅菌無RNAseのdH 2 Oの18μlに、RNAの2μLを加えます。
RNAからの第一鎖cDNAの3世代
- RNアーゼ/ DNアーゼフリーの0.2ミリリットルのPCRチューブに、表4に従って試薬を追加します。
- 65℃5分でサンプルをインキュベートします。少なくとも1分間氷上に置き、その後10分間25℃でインキュベートします。
- 同じチューブに、表5に列挙した試薬を追加します。
- 50分間55℃でサンプルをインキュベートします。 10分間72℃で反応を不活性化します。
- 使用するまで-20℃で保存するcDNA。
4.定量PCR
- 定量PCRのためのDNA標準の生成
- Q-PCRプライマー(1369F&1492R 21)との純粋培養から抽出したゲノムDNAからの細菌の16S rRNA配列20(または関心の任意の他の配列)を増幅。
- 製造業者の説明書に従って市販のキットを用いてPCR産物を精製します。
- 精製したPCRクローンを作成製造元の指示に従ってベクター系をクローニング3'-Tオーバーハングに製品。
- PCRは、製造業者の説明書に従って大腸菌 JM109コンピテント細胞に挿入を含むベクターを形質転換します。
- プレートアンピシリン(100μg/ ml)、X-galを(20μgの/ ml)およびIPTGへの転換50μlの(0.5 mM)のルリアベルターニプレート(トリプトン10グラム/ L、酵母エキス5グラム/ L、NaClを10グラム寒天/ L、寒天15gの/ Lの)。 37℃でO / Nインキュベートします。
- プレートから単一の正の白色形質転換体を選択します。 100μg/ mlのアンピシリンを含有する50 mlのLBブロス中で陽性の形質転換体を接種し、37℃でO / Nで250 rpmで振とうインキュベートします。
- O / N培養物から、製造業者の説明書に従って市販のキットを用いてプラスミドを単離し、精製します。
- 一度( 例えば 、のEcoRI)プラスミドを切断することのできる適切な制限酵素を用いてプラスミドを線状化。 >吸収比260 / A 280 19を測定することにより、プラスミドの純度を分析します。比が1.8未満であると、フェノール-クロロホルム19プラスミドを再抽出します。注:純粋なDNAは、1.8比を有します。
- 分光光度計19を用いて、260 nmでの吸光度によってプラスミドDNAの濃度を決定します。
注:また、定量の精度は蛍光計と組み合わせて使用される蛍光DNA結合色素を使用することによって改善することができます。 - シーケンスプラスミドを拠点に、ターゲットの大きさを確認するために挿入します。プラスミドのインサートDNAの量( すなわち 、パーセンテージ)を計算する( 例えば 、3,000 bpのベクターにおいて、123 bpのDNA断片は、全DNAの3.9%である)、および4.1.10で得られた濃度を掛け、決定します対象のDNA濃度(挿入)。
- 1μl当たりターゲットのコピーを計算するには、次の式を使用します。
ロード/ 54067 / 54067eq1.jpg "/> - 二本鎖DNA(dsDNAを、塩基対当たり660ダルトン)の平均分子量により、標的DNAの塩基対の数を乗算することにより、ターゲット分子量(TMW)を計算します。
- DNAの2μlをし、無菌のdH 2 O18μlのを使用して10 10 10から2コピーの範囲の標的DNAを希釈各希釈ウェルを混合し、次の希釈を行う前に、簡単にスピンダウン。
- qPCRのためのRNA標準曲線の生成
- ステップ4.1.5からは、ターゲット・リバースプライマー( すなわち 、R1492)と前方ベクタープラスミドプライマーM13Fを用いたコロニーPCR 19によって白色コロニー(約5)の数をスクリーニングします。
注:プライマーのこの組合せは、上流のT7プロモーター部位で正しいセンス方向でクローニングされた挿入物を増幅します。 - 製造業者の指示に従って市販のキットを用いてPCR産物を精製します。精製されたPCR Pを定量化します分光光度計を用いて260nmでroduct。
- インビトロ転写反応のための各リボヌクレオチド、1 T7反応バッファーと1 T7ポリメラーゼ酵素の7.5 mmの20μlの最終容量でPCR産物200ngのを加えます。
- 37℃で4時間反応をインキュベートします。 Iは、DNA鋳型を除去するために37℃で15分間インキュベートし、反応をRNaseフリーのDNaseの1単位を加えます。
- エタノール沈殿19によるin vitroで転写されたRNAを回収し、260 nmで蛍光計または吸光度によってを使用して、RNA収量を定量化します。
- RNAの1μl当たり転写物数を計算します。 T7プロモーター(153塩基)の長さに標的RNA( 例えば 、123塩基)で塩基対の数を追加し、RNAの平均分子量は、塩基当たり340ダルトンを掛けます。
- シリアル部3に概説されるように使用するための4.1.13に概説されたcDNAを希釈し、逆転写酵素(RT)反応を定量化し、標的RNAの500ngのを追加標準曲線として。
- ステップ4.1.5からは、ターゲット・リバースプライマー( すなわち 、R1492)と前方ベクタープラスミドプライマーM13Fを用いたコロニーPCR 19によって白色コロニー(約5)の数をスクリーニングします。
- リアルタイムPCR
- 氷の上で環境DNA / cDNAサンプル、標準および定量PCR試薬を解凍します。光から蛍光プローブを保護します。
- 4.1.14に概説された標準曲線の標準を希釈します。 /ニートから10 -3に対するcDNA無菌のdH 2 Oを18μlに2μlのサンプルを追加することにより、環境DNAの1:10希釈液を作ります
- 6(プライマーおよびプローブ) 表に従って、総反応数+ 10%のためのマスターミックス反応を行い、 表7(反応ミックス)。十分に混合し、短時間遠心。
- 96ウェルの光学定量PCRプレートの各ウェルにマスターミックス19μlのテンプレートの1μlの(標準、負の制御のための環境試料や水)を追加します。三重に各反応(標準、環境試料および陰性対照)を実行します。
- Q-PCR光学カバーで96ウェルプレートをカバーしています。簡単にプレートを遠心。ロード定量PCR機の加熱ブロックにプレートと蓋を閉じます。
- 定量PCRマネージャソフトウェアを開きます。 「プロトコル」タブをクリックし、「新規作成する」をクリックし、以下の増幅条件を追加:3分95℃、(10秒95℃、30秒60℃)を40サイクルxは。 20μlにサンプルボリュームを設定し、「OK」をクリックし、プロトコルを保存します。
- 「プレート」タブをクリックします。 「選択編集」の下で、「フルオロフォアを選択」をクリックし、適切なプローブ、 例えば 、フルオレセインのレポート染料を追加します。 「OK」をクリックします。
- 「サンプルタイプ」メニューから「標準」を選択し、基準を含むウェルを強調表示します。変更3に「サイズを複製」、「適用」をクリックして "シリーズを複製」をクリックします。 、標準的な1のための計算された開始濃度を追加するために「希釈系列」をクリックしてください標準曲線を加えた希釈係数(10)、を選択し、「減少」またはオーダーに応じて「増加」を示しますプレートに。 「適用」をクリックします。
- よく未知のサンプルを含む位置を強調表示し、「サンプルタイプ」ドロップダウンメニューから「不明」を選択します。 」シリーズを複製する」をクリックし、「適用」3.に「サイズを複製する」に変更します。 「サンプル名」の下にサンプル名を編集します。テンプレートなしのコントロール(NTC)のウェルのための手順を繰り返し。
- プレートエディタボックスで「OK」をクリックし、ファイルを保存します。 「実行を開始」をクリックします。
- 実行が完了すると、データ解析ウィンドウが開きます。標準曲線、標準曲線記述子( 表8)および増幅プロットを表示するには、「定量化」タブをクリックします。
- Cp値については、「定量データ」タブをクリックして、未知のサンプルの遺伝子の出発量(SQ)に対応します。表計算ソフトにエクスポートテーブル必要な場合。
- 土砂extracの量、希釈係数によって、未知のサンプルからの遺伝子の存在量を掛けテッド全量核酸は湿重量の堆積物のグラム当たりの遺伝子のコピーを入手するには溶出しました。
注:50μl中堆積物の0.5グラムから溶出DNA、1/10に希釈し、Q-PCR反応にテンプレートとして1μlを添加します。遺伝子コピーをDNAのx希釈第X因子溶出量×2μlの:乗じて遺伝子コピーのグラム-1土砂を計算します。
結果
堆積物から良質のDNAとRNAの抽出は、遺伝子や転写物の存在量を定量化するための最初のステップです。シャープ23Sおよび16S rRNAバンドは、高分子量のゲノムDNAバンドに加えて、表示されたサンプルAC、については、 図1に示すように、成功した抽出収率明確なDNAとRNAのバンド。
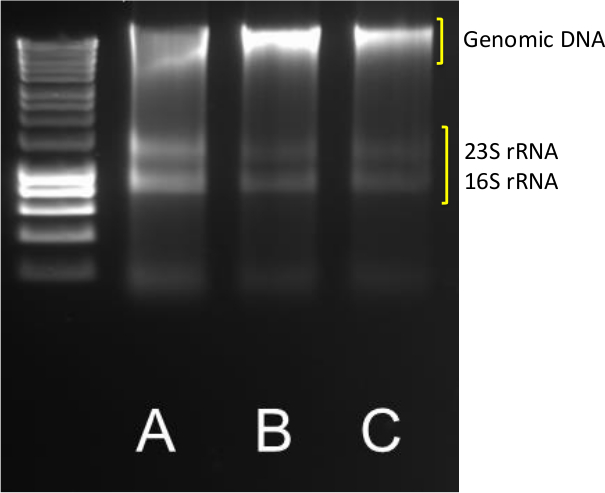
図1. DNA / RNA抽出。三連からのDNA / RNA抽出からの典型的な結果(ABC)0.5グラム沿岸の堆積物。 DNA / RNAの5μlを40分間85 Vで1.4%アガロース上で実行されました。 10,037-200 bpの範囲内の分子マーカーを使用した。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ページを-together.within、共抽出されたDNAの消化は必須です。これは、DNAが正常に削除されたことを確認するために、RNAの16S rRNAのエンドポイントPCRが続かなければなりません。 DNAが完全に除去された場合にのみ陽性対照のバンドが観察されます。阻害剤は、PCR産物の形成を防止されないようにすっきりとRNAの1:10希釈液の両方を使用することが重要です。 RNAは、現在のcDNAに変換するために逆転写酵素反応を受けることができます。これは、遺伝子特異的プライマー、ランダムヘキサマーのいずれかで行うことができます。典型的には、反応は、最適温度プロファイルを確保するために、PCR装置で行われます。このcDNAは、その後の定量PCR反応の鋳型として使用されます。 DNAおよびRNAを各反応に使用されるテンプレートの濃度を決定するために定量化されます。
定量PCR反応プロトコルは、16S rRNAのDNAをターゲットに概説されています。 16SについてのrRNA転写物は、標準立方を代用しますcDNAとRVEとテンプレート。未知の試料を標準曲線に対して定量化されるように、標準曲線が良好な品質であることを保証するために不可欠である。 図2(A)の調製、標準曲線および環境DNAの希釈、(B)、標準曲線の増幅を示します遺伝子コピー数の線形回帰計算に標準曲線の試料および環境試料(C)の変換。 10倍希釈の範囲が正しく準備さと見られている各標準希釈の間に3.3サイクル差を増幅した場合( 図2BI)(それは100%の増幅効率でテンプレート内の10倍の増加のために3.3サイクルを要します)。 90〜110%の範囲( 図2C)内の150のR 2値及びPCR効率を含む標準曲線記述子は、望まれています。存在する場合無鋳型対照(NTC)からCp値を報告することが重要です。これは<標準と未知のサンプル3.3サイクル(のためのCpカットオフを発生した場合em>のすなわち、ログ倍)20( 図2Ciiを課されるNTCのCP値)よりも高いです。沈殿物DNAから抽出された未知のテンプレートは、ニートから10 -1、10 -2、10 -3希釈液(B)を定量しました。きちんとしたサンプルが増幅しなかった、10 -1〜10 -3希釈液のCp値はそれぞれ24.12、26.02と28.40でした。 NTC Cpは27.2のNCTカットオフが課された、30.5でした。グラム-1湿重量の堆積物に遺伝子存在量を変換すると、希釈範囲のために、それぞれ2.5×10 7、10 -1、10 -2 7.1×10 7もたらしました。 10 -3希釈CpはNTCカットオフ未満であったため、使用されませんでした。この場合、10 -2は、最適なテンプレート希釈として選ば れました。

図2. 16S rRNA遺伝子QPCR規格の増幅や沿岸堆積物から抽出された環境DNA。 (A)は、i)DNAの標準曲線の調製および定量PCR増幅のためのii)の環境DNAの希釈液、(B)は、iの定量PCR増幅)DNAの標準曲線およびii)環境サンプルは、各サンプルのCpが示されています。 (C)は、i)標準曲線記述子を持つ標準曲線の線形回帰、環境サンプルからの遺伝子の存在量のii)の計算。 NTC:負テンプレートコントロールこの図の拡大版をご覧になるにはこちらをクリックしてください。
| 試薬 | 量 |
| CTAB | 5グラム |
| K 2 HPO 4 x3H 2 O | 2.58グラム |
| KH 2 PO 4 | 0.1グラム |
| NaClを | 2.05グラム |
| dH 2 O | 100ミリリットルまで |
CTABリン酸緩衝液の表1の調製。
| 試薬 | 量 |
| PEG 6000 | 30グラム |
| NaClを | 9.35グラム |
| dH 2 O | 100ミリリットルまで |
| 注:ゆっくりのdH 2 Oの50ミリリットルの適度な加熱および攪拌下でPEG 6000を追加します。 | |
PEG-NaClを沈殿溶液の表2の調製。
| 試薬 | 量 |
| 希釈していないRNA / 1:10に希釈RNA | 1.0μlの |
| 10×PCRバッファー | 5.0μlの |
| 10ミリメートルのdNTP | 1.0μlの |
| 63Fフォワードプライマー | 1.0μlの |
| CAG GCC TAA CAC ATG CAA GTC | |
| 518Rリバースプライマー | 1.0μlの |
| ATT ACC GCG GCT GCT GG | |
| Taqポリメラーゼ | 0.4μlの |
| 無RNAseのdH 2 O | 40.6μlの |
表3 DNAを含まないRNAの品質チェックPCRマスターミックス。
| 試薬 | 量 |
| RNA | 0.5-5μgの |
| 10ミリメートルのdNTP | 1μlの |
| リバースプライマー、10μM/ランダムヘキサマー、50μM | 1μlの |
| DEPC-H 2 O | 最大13μlの |
表4. cDNA合成反応混合A.
| 試薬 | 量 |
| バッファ5倍 | 4μlの |
| DTT | 1μlの |
| RNase阻害剤 | 1μlの |
| 逆転写酵素 | 1μlの |
表5のcDNA合成反応ミックスB.
| ターゲット | プライマー名 | シーケンス | アニーリングT(℃) | 参照 |
| 16S rRNAの細菌 | Bact1369F | CGG TGA ATA CGT TCY CGG | 56 | 鈴木ら、2000 19 |
| Prok1492R | GGW TAC CTT GTT ACG ACT T | |||
| TM1389F(プローブ) | CTT GTA CAC ACC GCC CGT C |
| 試薬 | 量 |
| 2×マスターミックス | 10μlの |
| フォワードプライマー(10μM) | 0.8μlの |
| リバースプライマー(10μM) | 0.8μlの |
| プローブ(10μM) | 0.4μlの |
| 水 | 7.0μlの |
| 最終体積 | 19μlの |
表7リアルタイムPCR反応ミックス。
| ディスクリプタ | 意義 |
| 相関係数(R 2) | 標準曲線の直線性の尺度。理想的には、R 2 = 1でなければなりません。 R 2 = 0.98から0.99までの値が許容可能です。 |
| 傾き(α) | 反応効率の尺度。理想的には-3.32と同等でなければなりません。 -3.58と-3.1の間の範囲の値。 |
| 効率(= 10(-1 /傾き)-1) | それは100%である場合、テンプレートは、指数関数的増幅中の各熱サイクル後に倍増します。良好な効率の範囲は90から110%の間です。 |
| Yインターセプト(β) | 反応の検出の理論的限界。反応の感度を定量するために使用されていないが、同じ標的の異なる標準曲線を比較した場合、それは重要です。 |
表8. QPCR反応記述子。
ディスカッション
定量PCRによるDNA / RNA抽出の組み合わせは、沿岸の堆積物などの環境試料、の範囲からの遺伝子と転写産物の存在量の高感度定量のための迅速で正確な、比較的費用対効果の高い方法を提供します。
最初の核酸抽出は、22を達成された微生物群集の存在の代表的な視野を確保するための重要なステップです。 D)、C)RNA画分中にDNAを汚染、阻害化合物( 例えば 、フミン酸又はポリフェノール)のb)の共抽出、全細胞溶解のa)の達成:制約の数は、抽出プロトコルのために考慮される必要があります保存された抽出された核酸の急速な分解。使用上の注意は、これらの制限を回避するために注意しなければなりません。例えば、特定の注意は、核酸の抽出は、試料の種類( 例えば 、堆積物、土壌、排水等のために最適化されることを保証するために注意しなければなりません)。 DNAとRNAの両方のための核酸の収量と品質の大幅な改善が予備試験23を実施することによって達成することができます。 PCRベースのアプリケーション24上の阻害化合物の影響を最小限に抑えるために、抽出したDNAとRNAからの希釈液の範囲をテストします。複数の凍結融解サイクルから抽出したDNA / RNAの急速な分解を回避し、-80℃で遺伝情報の損失の可能性、店舗、複数の小容量のアリコートを避けるために。
慎重に、定量PCRを設計するとき 堅牢、高い再現性と感度の高い方法です。注目すべきは、このプロトコルで概説増幅し、標準曲線を構築するための方法は、他の系統学的マーカー( すなわち 、古細菌の16S rRNAを、真菌の18S rRNAの)または環境での重要な機能に関与する遺伝子を含む任意の目的の遺伝子標的のために適合させることができます。定量PCRの使用における既知の制限は以下のとおりです。再現性のある高品質sのa)の生成絶対的定量のためtandard曲線は、b)のプライマー/プローブの選択およびQ-PCRアッセイ条件の最適化、c)低品質/剪断核酸、d)のDNA / RNAの作業希釈液の選択の使用は禁止を避けるために。また、定量PCR法は、カウントをセルに等しくないかもしれない遺伝子/転写物の存在量を提供することを考慮しなければならない:微生物がそれらのゲノム中にリボソーム遺伝子の異なるコピー数を有するので、これは、特に16Sおよび18S rRNA遺伝子を標的とする場合であります25。
品質が悪い標準曲線は、目的の遺伝子の不正確な定量化になります。新鮮な標準曲線を作製することができるから、少量ずつ高濃度基準の在庫を保管することをお勧めします。常に最高濃度のストックたびに新鮮な希釈を行い、標準曲線を保管しないでください。環境試料の正確な定量化のためにスタンの濃度の範囲を確保準の曲線は、未知サンプルの予想Cp値に及びます。転写物を定量する場合は、RNAから標準曲線を構築する二本鎖DNAではありません。可能な場合は、直接比較するサンプルの定量化は、アッセイ間の変動20を回避するために、単一のアッセイ内に完了する必要があります。これは常に可能ではないかもしれません。したがって、アッセイの間に生成された遺伝子の存在量を比較することは、アッセイの間で複製されたサンプルのランダムなサブセットを持つことをお勧めします。プライマーおよびプローブセットの幅広い選択は、現在、定量PCRは、微生物の分類群とターゲットグループのための最大のカバレッジと特異性の両方を確保するために、これらを選択するとき15.慎重に検討が必要とされる官能基を標的とするために利用可能です。定量PCRアッセイの反応効率が十分でない場合、トラブルシューティングは、プライマー-プローブ濃度を変化させる、 例えば 、異なる熱(アニール時間及び/又は温度など)サイクリング条件および/ または反応条件をテストから始まります。 OQ-PCRアッセイおよび反応条件が最適化されたNCE、常に適切なテンプレート濃度を決定するために、DNA / cDNAの希釈の範囲の最初の試験を行います。さらなるアッセイのための最適なテンプレート希釈として最高のコピー数が得られる希釈範囲を選択します。
現在、次世代シーケンシング技術は、効率的な環境26,27,28の過多で微生物群集構造と機能に光を当てます。しかし、これらのデータセットは、多くの場合、エンドポイントPCRアンプリコンライブラリに基づいており、したがって、特定の分類群の存在量のみを半定量的評価を提供します。したがって、リアルタイムPCRベースの技術の能力は、特定の分類学的マーカーを標的とするために(ダウン歪みレベルまで高いドメインから)次世代シーケンシングによって得られた結果の効率的な検証を可能にします。また、定量PCRが正常に安定なイソなどの他の微生物生態学の分子方法と組み合わせて使用されていますトペプロービング(SIP)または系統発生的/機能的なマイクロアレイ。前者のツールと組み合わせて、定量PCRは、代謝的に活性なコミュニティ29,30を定量化するために使用することができます。マイクロアレイ解析と組み合わせることで、定量PCRは、環境31,32の系統学的マーカーベースおよび機能遺伝子の調査の重要な定量的解釈を提供します。
そのため、単独で、または生態系の機能の(多くの場合、プロセスベースの)他の評価と組み合わせて使用するかどうかを、定量的PCRは、微生物群集と生態系機能との間にとらえどころのないリンクの探査における微生物生態学者のための不可欠なツールです。
開示事項
The authors have nothing to disclose.
謝辞
この刊行物は、CJSに認可番号11 / SIRG / B2159awarded下で認可番号NERC NE / JO11959 / 1および科学財団アイルランド&マリー・キュリーアクションCOFUNDの下で自然環境研究会議(NERC)の財政支援を受けて実施した調査から放ちました・東学術研究コンソーシアム(東ARC)。
資料
| Name | Company | Catalog Number | Comments |
| Diethylpyrocarbonate | Sigma | 40718 | Toxic, open under chemical hood |
| cetrimonium bromide (CTAB) | Sigma | 52365 | Irritant, open under chemical hood |
| potassium phosphate dibasic | Sigma | RES20765 | |
| potassium phosphate monobasic | Sigma | P9791 | |
| Phenol : Chloroform : Isoamylalcohol pH 8 | Sigma | P2069 | Equilibrate at pH 8 before using |
| Chloroform : Isoamylalcohol | Sigma | 25666 | |
| sodium chloride | VWR | 1.06404.0500 | |
| polyethylenglycol 6000 | VWR | 528877-100 | |
| Ethanol Molecular Grade | Sigma | E7148 | |
| Lysing Matrix E tubes | MP Biomedical | 116914050 | |
| Turbo DNAse | Ambion | AM1907 | |
| Taq polymerase | Sigma | D1806 | |
| dNTPs | Bioline | BIO-39028 | |
| SuperScript III | Life Technologies | 18080044 | |
| Rnase Inhibitor | Life Technologies | 10777019 | |
| RNAse/DNAse free 0.2 ml PCR tubes | Sarstedt | 72.985.002 | |
| SureCleanPlus | Bioline | BIO-37047 | |
| pGEM Easy T Vector | Promega | A1360 | |
| E. coli JM109 competent cells | Promega | L2005 | |
| Tryptone | Sigma | T7293 | |
| Yeast Extract | Sigma | 70161 | |
| Ampicillin | Sigma | 59349 | |
| X-Gal | Bioline | BIO-37035 | |
| IPTG | Bioline | BIO-37036 | |
| Agar | VWR | 20768.235 | |
| Plasmid Midi Kit | Qiagen | 12143 | |
| Qubit | Life Technologies | Q33216 | |
| Quant-IT DNA HS Assay | Life Technologies | Q-33120 | |
| Quant-IT RNA HS Assay | Life Technologies | Q32855 | |
| MEGAshortscript kit | Ambion | AM1354 | |
| TaqMan SensiFast Probe Lo-ROX kit | Bioline | BIO-84002 | |
| qPCR machine |
参考文献
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr. Opin. Microbiol. 16 (5), 636-642 (2013).
- Zhou, J., Bruns, M. A., Tiedje, J. M. DNA recovery from soils of diverse composition. Appl. Environ. Microbiol. 62, 316-322 (1996).
- Miller, D. N., Bryant, J. E., Madsen, E. L., Ghiorse, W. C. Evaluation and optimization of DNA extraction and purification procedures from soil and sediments samples. Appl. Environ. Microbiol. 65 (11), 4715-4724 (1999).
- Burgmann, H., Pesaro, M., Widmer, F., Zeyer, J. A strategy for optimizing quality and quantity of DNA extracted from soil. J. Microbiol. Methods. 45 (1), 7-20 (2001).
- Hurt, R. A., et al. Simultaneous recovery of RNA and DNA from soils and sediments. Appl. Environ. Microbiol. 67, 4495-4503 (2001).
- Griffiths, R. I., Whiteley, A. S., O'Donnell, A. G., Bailey, M. J. Rapid method for coextraction of DNA and RNA from natural environments for analysis of ribosomal DNA- and rRNA-based microbial community composition. Appl. Environ. Microbiol. 66 (12), 5488-5491 (2000).
- Martins, G., et al. Structure and activity of lacustrine sediment bacteria involved in nutrient and iron cycles. FEMS Microbiol. Ecol. 77 (3), 666-679 (2011).
- Kuffner, M., et al. Effects of season and experimental warming on the bacterial community in a temperate mountain forest soil assessed by 16S rRNA gene pyrosequencing. FEMS Microbiol. Ecol. 82 (3), 551-562 (2011).
- Tatti, E., et al. Influences of over winter conditions on denitrification and nitrous oxide-producing microorganism abundance and structure in an agricultural soil amended with different nitrogen sources. Agric. Ecosyst . Environ. 183, 47-59 (2014).
- Giovannoni, S. J., Britschgi, T. B., Moyer, C. L., Field, K. G. Genetic diversity in the Sargasso Sea bacterioplankton. Nature. 345, 60-62 (1990).
- Ward, D. W., Weller, R., Bateson, M. M. 16S rRNA sequences reveal numerous uncultured microorganisms a natural community. Nature. 345, 63-65 (1990).
- Suzuki, M. T., Giovannoni, S. J. Bias caused by template annealing in the amplification of mixture of 16S rRNA genes by PCR. Appl. Environ. Microbiol. 62, 625-630 (1996).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous fluorescence monitoring of rapid cycle DNA amplification. Biotechniques. 22, 130-138 (1997).
- Holland, P. M., Abramson, R. D., Watson, R., Gelfand, D. H. Detection of Specific Polymerase Chain Reaction Product by Utilizing the 5' 3' Exonuclease Activity of Thermus aquaticus DNA Polymerase. PNAS. 88, 7276-7280 (1991).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol. Ecol. 67, 2-20 (2009).
- Park, S. J., Park, B. J., Rhee, S. K. Comparative analysis of archaeal 16S rRNA and amoA genes to estimate the abundance and diversity of ammonia-oxidizing archaea in marine sediments. Extremphiles. 12 (4), (2008).
- Urakawa, H., Yoshida, T., Nishimura, M., Ohwada, K. Characterization of depth-related population variation in microbial communities of a coastal marine sediment using 16S rDNA-based approaches and quinone profiling. Environ. Microbiol. 2, 542-554 (2008).
- Smith, C. J., Dong, L. F., Wilson, J., Stott, A., Osborn, A. M., Nedwell, D. B. Seasonal variation in denitrification and dissimilatory nitrate reduction to ammonia process rates and corresponding key functional genes along an estuarine nitrate gradient. Front. Microbiol. 6, 542(2015).
- Sambrook, J., Russell, D. W. Molecular Cloning: a laboratory manual. , Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press. 205(2001).
- Smith, C. J., Nedwell, D. B., Dong, L. F., Osborn, A. M. Evaluation of Quantitative Polymerase Chain Reaction (Q-PCR) based approaches for determining gene copy and gene transcript numbers in environmental samples. Environ. Microbiol. 8 (5), 804-815 (2006).
- Suzuki, M. T., Taylor, L. T. DeLong E.F., Quantitative analysis of small-subunit rRNA genes in mixed microbial population via 5'-nuclease assays. Appl. Environ. Microbiol. 66, 4605-4614 (2000).
- Luna, G. M., Dell'Anno, A., Danovaro, R. DNA extraction procedure: a critical issue for bacterial diversity assessment in marine sediments. Environ. Microbiol. 8 (2), 308-320 (2005).
- Lever, M. A., Torti, A., Eickenbursch, P., Michaud, A. B., Šantl-Temkiv, T., Jørgensen, B. B. A modular method for the extraction of DNA and RNA, and the separation of DNA pools from diverse environmental sample types. Front. Microbiol. 6, 476(2015).
- Lloyd, K. G., MacGregor, B. J., Teske, A. Quantitative PCR methods for RNA and DNA in marine sediments: maximizing yield while overcoming inhibition. FEMS Microbol. Ecol. 72, 144-151 (2010).
- Klappenbach, J. A., Dumbar, J. M., Schmidt, T. M. rRNA Operon copy number reflects ecological strategies of bacteria. App. Environ. Microbiol. 66 (4), 1328-1333 (2000).
- Shi, Y., Tyson, G. W., Eppley, J. M., DeLong, E. F. Integrated metatrascriptomic and metagenomics analyses of stratified microbial assemblages in the open ocean. ISME J. 5, 999-1013 (2011).
- Howe, A. C., Jansson, J. K., Malfatti, S. A. Tringe S.G., Tiedje J.M., & Brown C.T. Tackling soil diversity with the assembly of large, complex metagenomes. PNAS. 111 (13), 4904-4909 (2014).
- Klaedtke, S., et al. Terroir is a key driver of seed-associated microbial assemblages. Environ. Microbiol. , (2015).
- Lueders, T., Wagner, B., Claus, P., Friedrich MW, Stable isotope probing of rRNA and DNA reveals a dynamic methylotroph community and trophic interactions with fungi and protozoa in oxic rice field soil). Environ. Microbiol. 6, 60-72 (2004).
- Kunapuli, U., Lueders, T., Meckenstock, R. U. The use of stable isotope probing to identify key iron-reducing microorganisms involved in anaerobic benzene degradation. ISME J. 1, 643-653 (2007).
- Bürgmann, H., et al. Transcriptional response of Silicibacter pomeroyi DSS-3 to dimethylsulfoniopropionate (DMSP). Environ. Microbiol. 9 (11), 2742-2755 (2007).
- He, J. Z., et al. Geochip: a comprehensive microarray for investigating biogeochemical ecological and environmental processes. ISME J. 1, 67-77 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved