Method Article
从海岸沉积物和16S量化的rRNA基因和转录同时DNA-RNA提取采用Real-time PCR检测
摘要
A protocol to quantify bacterial 16S rRNA genes and transcripts from coastal sediments via real-time PCR is provided. The methodology includes the co-extraction of DNA and RNA; preparation of DNA-free RNA; and 16S rRNA gene and transcript quantification via RT-q-PCR, including standard curve construction.
摘要
实时聚合酶链反应也称为定量PCR(Q-PCR)是在微生物生态学一种广泛使用的工具来量化环境样品中的分类和官能团的基因的丰度。组合使用逆转录酶反应(RT-Q-PCR),它也可以被用于量化基因转录。 Q-PCR利用高度敏感的荧光检测化学,允许在反应指数阶段的PCR扩增子的定量。因此,在PCR反应的平台期检测与'终点'的PCR关联的偏差被避免。的协议通过提供PCR实时定量沿海沉积物中的细菌16S rRNA基因和成绩单。首先,用于从海岸沉积物,包括不含DNA的RNA的制备所需的附加步骤的DNA和RNA的共提取的方法,概述。二,一步一步的指导,为16S rRNA基因和TR量化从通过Q-PCR和RT-Q-PCR提取核酸anscripts概述。这包括DNA和RNA标准曲线的构造细节。在微生物生态学采用RT-Q-PCR检测的主要考虑因素包括在内。
引言
微生物是生物圈的驾驶生态系统功能的基石。大多数微生物仍然未培养1。因此,基于分子的方法是从根本上促进我们的环境中微生物的多样性和功能的理解。中央这些方法是核酸从环境样品提取和使用聚合酶链反应(PCR)的靶基因的随后的扩增。
的DNA / RNA提取的第一步骤的目的是裂解微生物群落本的细胞壁,除去不需要的非核酸分子( 例如 ,有机和无机物质),并在进一步的下游分析溶液保留的DNA / RNA。在文献2,3,4,5提供多种选择,包括各种商业提取试剂盒后,格里菲斯方法6,广泛采用7,8,9。它是具有成本效益和巴rticularly非常适合于沉积物,因为它使用珠跳动步骤以裂解细胞,并结合步骤以最小化PCR抑制剂,如腐殖酸这样的共同提取,而同时回收DNA和RNA。
第二步骤利用聚合酶链式反应(PCR)扩增靶基因,如16S rRNA的分类标记,从所提取的核酸。这种方法有,并继续推动未鉴定微生物黑匣子10,11的探索。然而,终点,基于PCR的方法从各种限制的影响,可以偏置微生物群落12的表征。准确量化基因/转录丰度,实时PCR,也称为定量PCR(qPCR的)必须被使用。定量PCR利用了PCR的每个循环之后跟踪扩增子积累荧光报道染料系统。这是显著因为它意味着可以在早期指数阶段发生的量化,而比终点,将PCR反应的阶段中,当扩增子产量仍然是成正比的靶基因的初始丰盈。
两个报告系统是常用的:一个嵌入核酸染色13和DNA聚合酶14的5'3'外切酶活性。因为前者报告系统不显结合到所有双链DNA,它可能会导致与靶序列的过高估计,如果不想要的非特异性扩增或通过产物引物二聚体发生。为了规避这一点,可能需要扩增的广泛的优化。在后者的系统中,模板的扩增是使用Taq聚合酶劈开从内部探针的荧光团的5'核酸酶活性的组合跟踪。此特征增加了测定的特异性由于荧光探针的利用,只有结合于互补的靶特异性sequenc引物对两地电子。与两个化学定量是通过确定交叉点(CP),其中的PCR扩增子作为通过增加荧光测量的积累,是显著高于背景荧光来实现。
的qPCR已在微生物生态学被广泛用来确定在不同的环境中15基因的丰度。此外,为cDNA RNA的逆转录用的qPCR和RT-qPCR的组合来量化基因的表达。因此,定量PCR和RT-qPCR的代表基因和/或转录的数字环境样品中的量化快速,有效的方法。
在海岸沉积物微生物驱动各种生态系统过程,包括有机物矿化,污染物的降解和大量营养素如氮16,17,18的生物地球化学循环。这些转换的详尽的了解需要一个全面有分起作用的微生物种群,包括基因及成绩单丰度的定量数据吨。在这里,我们推出一系列全面测试,简化和标准化的协议细菌16S rRNA基因及成绩单丰度在海岸沉积物进行量化。的协议概括了样品的采集,同时DNA和RNA提取,不含DNA的RNA制备的提取的核酸的质量检查,生成的16S rRNA-DNA和-RNA标准和环境样品的定量。从这里介绍的方法得出量化的数据需要微生物群落驾驶沿海生态系统揭示。
研究方案
1.海洋海岸沉积物DNA和RNA提取
- 核糖核酸(RNA酶) - 免费的材料和工作区的制备
- 通过用0.1%焦碳酸二乙酯(DEPC)处理制备无RNA酶的蒸馏水(DH 2 O),在37℃CO / N孵育。通过在121℃高压灭菌的dh 2 O 15分钟,清除残留的DEPC。使用DEPC处理的dh 2 O,使所有的解决方案。
- 烘烤在180°CO / N的所有玻璃器皿。取下塑料盖和轮辋,在2 M氢氧化钠O / N一溶液浸泡它们,并在使用前处理的dh 2 O DEPC冲洗。
- 制备的CTAB-磷酸盐缓冲溶液按表1中 。
- 制备的PEG-NaCl的沉淀溶液按表2。
- 清理所有的工作表面( 即台,离心,通风橱),并使用启动前RNA酶净化解决方案微量。在整个过程中,应戴上手套。
- 使用无RNase的塑料制品用微量过滤嘴沿避免样品交叉污染。
- 环境样品采集和存储
- 使用无菌50毫升猎鹰管从顶部2.5厘米收集退潮时海岸泥沙。收集约15-20摹沉积物。样品采集后小心地盖上盖子。立即传输到实验室中,在4℃的冷却器以便立即处理。
- 通过用无菌刮铲和等分0.5克湿重沉积物混合到2ml珠打浆管样品均质化。等分试样额外重复,闪冻在液氮放置。商店等分在-80℃。注意:处理液氮时要戴上防护手套和护目镜。
注意:如果在野外现场不靠近实验室,等分0.5克泥沙分为2毫升无菌离心管在外地的网站。通过将它们立即投入到李闪存冻结管镑氮和运送至实验室。样品储存在-80°C,直到处理。
- DNA的共同提取和RNA沉积物
注意:以下协议是格里菲思方法6的修改版本。- 加入500μl的CTAB-磷酸盐缓冲液和500微升酚:氯仿:异戊醇(25:24:1),以含有0.5g沉淀物的2 mL萃取珠裂解管并倒转试管5-10倍,以均质化试样。
注意:执行在通风罩在任何时候都穿着适当的防护装备( 如实验室外套,手套和护目镜)这一步。 - 涡全速2.5分钟。离心机以16,000 xg离心在4℃下10分钟。
- 在一个通风橱中提取的顶部的水层,并转移到一个新的1.5毫升无菌离心管中。保持在冰上的样品中,加入500μl冰冷的氯仿:异戊醇(24:1)。
- 倒置几次直到乳液是visib勒。离心机在4℃下以16,000 xg离心10分钟。
- 在一个通风橱中提取的顶部的水层,并将其放置在一个新的1.5毫升无菌试管中。加入30%的PEG-NaCl溶液两卷沉淀核酸拌匀。
- 在冰上孵育2小时或在4℃CO / N离开样品。
- 在孵育离心机样品以16,000×g离心20分钟结束时,在4℃。
注:沉淀可在管的底部是可见的。 - 轻轻地取出用微量上清,确保不打扰颗粒。离开大约10微升PEG溶液在管和加入1毫升冰冷的70%乙醇。
- 离心16,000 xg离心在4℃下20分钟。慢慢微量小心不要碰到沉淀除去乙醇。
- 离心5秒,并使用微量吸管除去残余的乙醇。离开沉淀空气干燥约5分钟。
- 重悬在50粒料微升DEPC处理过的H 2 O审查通过在1%琼脂糖凝胶19运行5微升琼脂糖凝胶电泳的质量和DNA / RNA的产率。划分核酸分成两等份,15微升为DNA和30微升用于RNA。
- 直到需要在-80存储该DNA℃。分离RNA在下一节中介绍。
- 加入500μl的CTAB-磷酸盐缓冲液和500微升酚:氯仿:异戊醇(25:24:1),以含有0.5g沉淀物的2 mL萃取珠裂解管并倒转试管5-10倍,以均质化试样。
2.游离DNA-RNA的RNA和质量检查的制备
- DNA消化
- 添加DNA酶I缓冲液3微升和1.5微升DNA酶Ⅰ至核酸30微升体积,孵育在37℃下30分钟。
- 通过涡旋30秒混合DNA酶失活试剂。添加4.8微升溶液于样品。孵育在室温5分钟,偶尔挖掘管底部到该溶液中混合。
- 离心在10,000rpm xg离心90秒在RT,将上清液(RNA)转移到新的管中,小心不要转移能够抑制下游反应沉淀物。
- RNA质量检查
- 加入2微升RNA至18微升无菌无RNA酶-DH 2 O的稀释1:10。
- 在单独的PCR反应,加入1微升RNA整齐或1:10的稀释无菌0.5毫升PCR管。添加PCR反应组分以扩增16S rRNA基因按表3。包含一个"正"( 例如 ,从一个纯的细菌培养物中提取的DNA)中,"PCR抑制剂"控制( 即 ,1微升阳性纯培养物DNA的和整齐提取的RNA 1微升)和无菌无RNA酶-DH 2 O.的"负"控制
- 设置进行下述扩增参数热循环:95℃5分钟,(95℃30秒,57℃30秒,72℃1分钟)×35个循环,72℃10分钟。
- 在85 V代表40分钟运行在1%琼脂糖凝胶对PCR产物的10%。重复DNA酶I处理,如果在环境RN形成PCR产物一个样本。
- 加入2微升RNA至18微升无菌无RNA酶-DH 2 O的稀释1:10。
由RNA第一链cDNA的3代
- 按表4添加试剂RNA酶/ DNA酶的免费0.2毫升PCR管。
- 孵育在65℃5分钟的样品。在冰上放置至少1分钟,然后在25℃孵育10分钟。
- 到相同的管中添加表5中列出的试剂。
- 孵育在55℃的样品50分钟。灭活在72℃下进行10分钟的反应。
- 在-20°C,直到使用储存的cDNA。
4.定量PCR
- DNA的标准产生的qPCR
- 扩增来自从纯培养用Q-PCR引物(1369F&1492R 21)提取的基因组DNA的细菌的16S rRNA序列20(或任何其他感兴趣的序列)。
- 纯化使用根据制造商的说明商业试剂盒的PCR产物。
- 克隆纯化的PCR产物成3'-T突出端根据制造商的说明书克隆载体系统。
- 变换含有PCR根据制造商的说明插入到大肠杆菌 JM109感受态细胞的载体。
- 板变换的50微升到氨苄青霉素(100微克/毫升),X-gal的(20微克/毫升)和IPTG(0.5毫摩尔)的Luria BERTANI琼脂板(胰蛋白胨10克/升,酵母提取物5g / L的,氯化钠10克/ L,琼脂15g / L)。孵育O / N在37℃。
- 选择从板单个正白色转化体。接种阳性转化体在含有100μg/ ml氨苄青霉素50毫升LB肉汤和孵育O / N以250rpm振荡在37℃。
- 从O / N培养,分离和使用根据制造商的说明商业试剂盒纯化质粒。
- 线性用适当的限制性内切酶,其能够切割质粒一次( 例如 ,EcoRI)消化的质粒。 >通过测量吸收比A260 / 280 19分析质粒的纯度。如果该比例低于1.8,再提取用苯酚-氯仿19的质粒。注:纯DNA有一个1.8的比例。
- 确定在使用分光光度计19 260nm处吸光度的质粒DNA的浓度。
注意:可替换地,量化的精度可通过使用配合使用荧光计的荧光DNA结合染料进行改进。 - 序列的质粒插入到确认目标的碱基的大小。计算的量在质粒插入DNA( 即 ,百分比)(以一个3000 bp的载体例如 ,一个123 bp的DNA片段是总DNA的3.9%),并通过在4.1.10中得到的浓度相乘,以确定目标(插入)的DNA浓度。
- 使用下面的公式来计算每微升目标的副本:
负载/ 54067 / 54067eq1.jpg"/> - 由平均分子量双链DNA的(双链DNA,每个碱基对660道尔顿)的靶DNA的碱基对的数量乘以计算目标分子量(TMW)。
- 在稀释的范围内的目标DNA利用DNA的2微升和18微升无菌卫生署2 O的10月10日至2月10日份混合每个稀释度良好,在进行下一步之前,稀释后短暂离心。
- 对于RNA定量PCR标准曲线的产生
- 从步骤4.1.5,筛选菌落PCR 19多家白色菌落(大约5)使用目标反向引物( 即 R1492),并正向载体质粒引物M13F。
注意:引物的结合将扩增与T7启动子位点上游的正确义方向克隆的插入物。 - 纯化使用依照制造商的指令的市售试剂盒的PCR产物。量化纯化的PCR pRODUCT在使用分光光度计260纳米。
- 添加200纳克PCR产物中的20微升每核糖核苷酸,1 T7反应缓冲液和1 T7聚合酶酶用于体外转录反应的7.5毫的最终体积。
- 孵育在37℃进行4小时的反应。我添加1个单位不含RNA酶的DNA酶的反应,在37℃下孵育15分钟以除去模板DNA。
- 恢复通过乙醇沉淀19的体外转录的RNA和使用荧光或通过260nm处的吸光度定量RNA产量。
- 计算每个RNA的转录微升数量。添加的碱基对的数目在靶RNA( 例如 ,123个碱基)的T7启动子(153个碱基)的长度和由平均分子量RNA的,每基座340道尔顿繁殖。
- 500ng的量化的靶标RNA的添加逆转录酶(RT)反应在部分3串行稀释的cDNA概述如在4.1.13概述了使用作为标准曲线。
- 从步骤4.1.5,筛选菌落PCR 19多家白色菌落(大约5)使用目标反向引物( 即 R1492),并正向载体质粒引物M13F。
- 实时荧光定量PCR
- 解冻环境的DNA / cDNA样品,标准和在冰上的qPCR试剂。避光的荧光探针。
- 稀释的标准曲线的标准在4.1.14中概述。使环境的DNA的1:10稀释液/加入2微升样品,以18微升无菌卫生署2 O的cDNA的整齐〜10 -3
- 作出反应的总数加上10%按表6(引物和探针)和表7(反应混合物)一个主混合物反应。充分混匀,短暂离心。
- 加主混合物的19微升和1μl模板(标准,环境样品或水为阴性对照)在各孔中96孔光学定量PCR板。执行一式三份各反应(标准,环境样品和阴性对照)。
- 覆盖用Q-PCR光学盖96孔板中。短暂离心板。加载板进入定量PCR机的加热块,并盖上盖子。
- 打开定量PCR管理器软件。点击"协议选项卡",点击"新建",添加以下扩增条件:3分钟95℃(10秒95℃,30秒60℃)×40个循环。设置样本量为20微升,点击"确定"并保存协议。
- 点击"板"标签。在"编辑选择"点击"选择荧光基团"中,增加报告染料合适的探头, 例如 ,荧光素。点击"确定"。
- 突出显示包含标准的井,从"样品类型"菜单中选择"标准"。点击"复制系列"改变"大小复制"到3,点击"应用"。点击"稀释系列",增加了对标准1计算起始浓度,表明加入标准曲线的稀释倍数(10),选择"减少"或根据订单"增加"到板上。点击"应用"。
- 突出以及含有未知样品的位置,从"样品类型"中选择"未知"的下拉菜单。点击"复制系列",变"复制大小"来3.单击"应用"。编辑在"样品名称"样品名称。对于无模板对照(NTC)重复井过程。
- 点击编辑板中的"确定"按钮,保存文件。点击"开始→运行"。
- 在运行完成后,数据分析窗口将打开。点击"量化"标签,显示标准曲线,标准曲线描述符( 表8)和扩增曲线。
- 点击的Cp值"定量数据"选项卡上,并相应启动未知样品的基因(SQ)的数量。如果需要导出表电子表格软件。
- 从未知样品乘法基因的丰度通过稀释因子,沉积物为Extrac的量泰德从与总体积的核酸洗脱到每克湿重的沉积物获得基因拷贝。
注意:洗脱的DNA为0.5g沉积物在50μl,稀释1/10,加入1微升作为模板的q PCR反应;基因拷贝微升DNA×稀释系数x洗脱体积×2的:由相乘计算基因拷贝摹-1沉淀物。
结果
质量好DNA和RNA从沉积物中提取是定量基因和转录物丰度的第一步。一个成功的提取率, 如图1样品交流,其中23S锐和的16S rRNA乐队是除了高分子量的基因组DNA带可视指示清晰的DNA和RNA带。
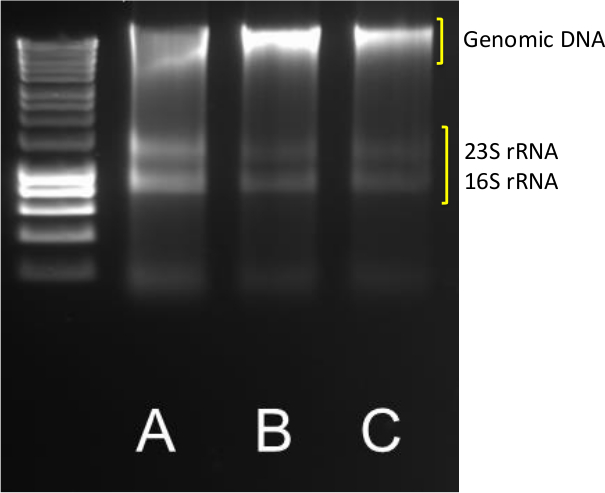
图1. DNA / RNA提取。从DNA / RNA提取的典型结果从一式三份(ABC)将0.5g沿岸沉积物。 5微升的DNA / RNA的混合物在1.4%琼脂糖,在85 V代表40分钟运行。用在10,037-200基点范围内的分子标记。 请点击此处查看该图的放大版本。
要准备RNA,共提取的DNA的消化是强制性的。这必须遵循由RNA的16S rRNA的终点PCR,以确保所述DNA已被成功去除。如果DNA已被完全除去仅在阳性对照的频带被观察到。它是同时使用整齐和1:10稀释的RNA,以确保抑制剂不阻止PCR产物的形成非常重要。 RNA现在可以进行所述逆转录酶反应以将其转化成cDNA。这可与基因特异性引物或随机六聚体进行。典型地,该反应在PCR机器中进行,以确保最佳的温度分布。该cDNA被用作在随后的qPCR反应模板。 DNA和RNA进行定量,以确定在每个反应中使用的模板浓度。
定量PCR反应协议概述针对的16S rRNA的DNA。对于的16S rRNA成绩单替代标准立方米RVE和模板的cDNA。由于未知样品与标准曲线量化,当务之急是确保标准曲线的质量是好的。 图2显示了(A),标准曲线和环境DNA稀释液,(B),标准曲线的扩增准备标准曲线和环境样品(C)的转化率,以基因拷贝数的线性回归和计算。当10倍稀释的范围是正确制备和扩增各标准稀释两者3.3循环差出现时(需要3.3个循环在100%扩增效率在模板十倍增加)( 图2BI)。标准曲线的描述符,包括0.99的90至110%( 图2C)的范围内,R 2的值和PCR效率是期望的。它从没有模板对照(NTC)如本报道的Cp值是重要的。如果发生这种情况的标准和未知样品3.3个周期(Cp为截止即一个日志倍),比NTC的CP值强加20( 图2Cii)高。从沉积物中提取DNA模板未知从整齐的10 -1,10 -2和10 -3稀释液(B)量化。整洁的样品没有放大,10 -1至10 -3稀释度的Cp值分别为24.12,26.02和28.40。该NTC Cp是30.5,27.2的NCT截止被罚款。转换基因的丰度至g -1湿重沉淀导致2.5×10 7和7.1×10 7 10 -1和10 -2分别为稀释范围。 10 -3稀释的Cp是NTC截止以下,并且因此不被使用。在这种情况下,10 -2被选为最优模板稀释。

图2. 16S rRNA基因QPC标准- [R放大和沿海沉积物中提取DNA环境。 (A)ⅰ)的DNA的标准曲线的制备和ii)环境的DNA稀释液定量PCR扩增,(B)中的i定量PCR扩增)的DNA的标准曲线和ii)环境样品中,Cp为各样品被示出。 ( 三 ) 我 )与标准曲线描述标准曲线的线性回归; 二 )从环境样品中基因的丰度的计算。 NTC:负模板控件请点击此处查看该图的放大版本。
| 试剂 | 量 |
| CTAB | 5克 |
| 磷酸氢二钾X3H 2 O | 2.58克 |
| KH 2 PO 4 | 0.1克 |
| 氯化钠 | 2.05克 |
| 卫生署2 O | 至100ml |
表1制备的CTAB-磷酸盐缓冲液中。
| 试剂 | 量 |
| PEG 6000 | 30克 |
| 氯化钠 | 9.35克 |
| 卫生署2 O | 至100ml |
| 注意:添加PEG 6000下慢慢适度加热和为50毫升卫生署2 O的搅拌 | |
表2的PEG-NaCl的沉淀溶液的制备。
| 试剂 | 量 |
| 未稀释的RNA / 1:10稀释RNA | 1.0微升 |
| 10X PCR缓冲液 | 5.0微升 |
| 的dNTP 10毫 | 1.0微升 |
| 63F正向引物 | 1.0微升 |
| CAG GCC TAA CAC ATG CAA GTC | |
| 518R反向引物 | 1.0微升 |
| ATT GCG ACC GCT GCT GG | |
| Taq DNA聚合酶 | 0.4微升 |
| 无RNase-DH 2 O | 40.6微升 |
表3.不含DNA的RNA质量检测PCR反应液。
| 试剂 | 量 |
| RNA | 0.5-5微克 |
| 的dNTP 10毫 | 1微升 |
| 反向引物10微米/随机六聚体50μM | 1微升 |
| DEPC-H 2 O | 截至13微升 |
表4. cDNA合成反应混合物A.
| 试剂 | 量 |
| 缓冲区5X | 4微升 |
| DTT | 1微升 |
| RNA酶抑制剂 | 1微升 |
| 逆转录酶 | 1微升 |
表5. cDNA合成反应混合物B.
| 目标 | 底漆名 | 序列 | 退火温度(℃) | 参考 |
| 的16S rRNA细菌 | Bact1369F | CGG TGA ATA CGT TCY CGG | 56 | 铃木等人,2000 19 |
| Prok1492R | GGW TAC CTT GTT ACG ACTŤ | |||
| TM1389F(探头) | CTT GTA CAC ACC GCC CGTÇ |
| 试剂 | 量 |
| 2X预混 | 10微升 |
| 正向引物(10μM) | 0.8微升 |
| 反向引物(10μM) | 0.8微升 |
| 探针(10μM) | 0.4微升 |
| 水 | 7.0微升 |
| 最终体积 | 19微升 |
表7.实时PCR反应混合物。
| 描述符 | 意义 |
| 相关系数(R 2) | 标准曲线的线性的度量。理想情况下,应该是R 2 = 1。 R 2 = 0.98-0.99的价值是可以接受的。 |
| 斜率(α)的 | 反应效率的措施。理想情况下,应相当于-3.32。值在-3.58和-3.1之间的范围内。 |
| 效率(= 10(-1 /斜率)-1) | 如果它是100%,这些模板指数扩增过程中每个热循环后加倍。一个很好的效率范围为90〜110%之间。 |
| y轴截距(β) | 检测该反应的理论极限。虽然不是用来量化的反应灵敏度,比较同一目标的不同标准曲线时,是非常重要的。 |
表8. QPC- [R反应描述。
讨论
DNA / RNA提取用定量PCR的组合提供了一种快速,准确,比较合算的基因和转录物丰度的从一系列环境样品,如海岸沉积物的敏感定量方法。
初始核酸提取是确保微生物群落本的代表性视图达到22的关键步骤。需要被考虑为提取协议的一些限制:一)总细胞裂解的成就,二)的抑制性化合物( 例如 ,腐殖酸或多酚的共萃取),c)在该RNA部分污染DNA,四)储存时的提取核酸的快速降解。必须采取预防措施,以规避这些限制。例如,特别注意,必须注意确保核酸提取的样品类型( 例如 ,沉淀物,土壤,废水等优化)。在核酸的产量和质量为DNA和RNA显著改进可以通过进行初步实验23来实现。为了最大限度地减少抑制化合物的对基于PCR的应用程序24中的效果,测试的范围内所提取的DNA和RNA稀释液。规避从多个冻融提取的DNA / RNA的快速降解,避免在-80℃的遗传信息可能丢失,存储多个小体积等分试样。
当精心设计,定量PCR 是一个强大的,高度重复性和敏感的方法。值得注意的是,在此协议中概述用于扩增和标准曲线结构的方法可以适用于任何感兴趣的基因靶标,包括其他的系统发育标记物( 即 ,古细菌的16S rRNA,真菌18S rRNA基因)或涉及在环境中的重要功能的基因。在利用定量PCR的已知限制是:a)可重放高质量的S中的代为绝对定量TANDARD曲线,二)引物/探针的选择的Q-PCR测定条件的优化,c)使用低质量/剪切核酸,四)的DNA / RNA的工作稀释度的选择,以避免抑制。此外,它必须被认为是定量PCR技术提供了基因/转录丰度可能不等同于细胞计数:这是特别当16S和18S rRNA基因被定位的情况下,如微生物在它们的基因组具有核糖体基因的不同拷贝数25。
质量差的标准曲线将导致感兴趣的基因的不准确的定量。它是存储高浓度标准小等份从新鲜标准曲线可以由库存好的做法。不要储存标准曲线,总是从每一次最集中的股票做出新的稀释液。环境样品的准确定量保证斯坦的浓度范围准曲线跨越未知样品的预期的Cp值。当量化成绩单,构建从RNA不双链DNA的标准曲线。当可能时,样品可以直接进行比较的量化应单一测定内完成,以避免测定间变异20。这可能不总是可能的。因此,为了比较测定之间产生基因的丰度,最好是具有随机子集测定之间复制样品。的引物和探针集的多种选择目前可用于定量PCR针对微生物类群和官能团选择这些的时候,以确保这两个最大的覆盖面和特异性的目标群体是需要15.仔细考虑。如果一个qPCR分析的反应效率不令人满意的,解决始于测试不同的热循环条件(如退火时间和/或温度)和/或反应条件, 例如 ,不同的引物-探针的浓度。 ØNCE的Q-PCR测定法和反应条件进行了优化,始终进行一系列的DNA / cDNA的稀释物的初始测试以确定适当的模板浓度。选择产生最高拷贝数作为最优模板稀释用于进一步分析的稀释范围。
目前,新一代测序技术,有效地环境26,27,28过多阐明微生物群落结构和功能的光。然而,这些数据集通常是基于终点PCR扩增子库,因此只提供丰特定类群的半定量评估。因此,实时基于PCR的技术的能力,以针对特定分类标记(从高域到应变水平)允许通过新一代测序得到的结果的有效验证。此外,定量PCR已被成功地用于与其它微生物生态学分子的方法,例如稳定异TOPE探测(SIP)或亲缘/功能芯片。与前者的工具相结合,定量PCR可用于定量代谢活性社区29,30。当与微阵列分析相结合,定量PCR提供环境31,32进化标记为基础的功能基因调查的关键定量解释。
因此,无论是单独使用或与生态系统功能的其他(通常基于过程的)评估相结合,定量PCR是在微生物群落和生态系统功能之间的联系难以捉摸的探索微生物生态学家的重要工具。
披露声明
The authors have nothing to disclose.
致谢
本出版物已经从与自然环境研究委员会(NERC)的资助下,授权号NERC NE / JO11959 / 1和爱尔兰科学基金会及以下授权号11 / SIRG / B2159awarded的玛丽 - 居里行动COFUND到CJS进行的研究散发出和东方学术研究联合会(ARC东部)。
材料
| Name | Company | Catalog Number | Comments |
| Diethylpyrocarbonate | Sigma | 40718 | Toxic, open under chemical hood |
| cetrimonium bromide (CTAB) | Sigma | 52365 | Irritant, open under chemical hood |
| potassium phosphate dibasic | Sigma | RES20765 | |
| potassium phosphate monobasic | Sigma | P9791 | |
| Phenol : Chloroform : Isoamylalcohol pH 8 | Sigma | P2069 | Equilibrate at pH 8 before using |
| Chloroform : Isoamylalcohol | Sigma | 25666 | |
| sodium chloride | VWR | 1.06404.0500 | |
| polyethylenglycol 6000 | VWR | 528877-100 | |
| Ethanol Molecular Grade | Sigma | E7148 | |
| Lysing Matrix E tubes | MP Biomedical | 116914050 | |
| Turbo DNAse | Ambion | AM1907 | |
| Taq polymerase | Sigma | D1806 | |
| dNTPs | Bioline | BIO-39028 | |
| SuperScript III | Life Technologies | 18080044 | |
| Rnase Inhibitor | Life Technologies | 10777019 | |
| RNAse/DNAse free 0.2 ml PCR tubes | Sarstedt | 72.985.002 | |
| SureCleanPlus | Bioline | BIO-37047 | |
| pGEM Easy T Vector | Promega | A1360 | |
| E. coli JM109 competent cells | Promega | L2005 | |
| Tryptone | Sigma | T7293 | |
| Yeast Extract | Sigma | 70161 | |
| Ampicillin | Sigma | 59349 | |
| X-Gal | Bioline | BIO-37035 | |
| IPTG | Bioline | BIO-37036 | |
| Agar | VWR | 20768.235 | |
| Plasmid Midi Kit | Qiagen | 12143 | |
| Qubit | Life Technologies | Q33216 | |
| Quant-IT DNA HS Assay | Life Technologies | Q-33120 | |
| Quant-IT RNA HS Assay | Life Technologies | Q32855 | |
| MEGAshortscript kit | Ambion | AM1354 | |
| TaqMan SensiFast Probe Lo-ROX kit | Bioline | BIO-84002 | |
| qPCR machine |
参考文献
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr. Opin. Microbiol. 16 (5), 636-642 (2013).
- Zhou, J., Bruns, M. A., Tiedje, J. M. DNA recovery from soils of diverse composition. Appl. Environ. Microbiol. 62, 316-322 (1996).
- Miller, D. N., Bryant, J. E., Madsen, E. L., Ghiorse, W. C. Evaluation and optimization of DNA extraction and purification procedures from soil and sediments samples. Appl. Environ. Microbiol. 65 (11), 4715-4724 (1999).
- Burgmann, H., Pesaro, M., Widmer, F., Zeyer, J. A strategy for optimizing quality and quantity of DNA extracted from soil. J. Microbiol. Methods. 45 (1), 7-20 (2001).
- Hurt, R. A., et al. Simultaneous recovery of RNA and DNA from soils and sediments. Appl. Environ. Microbiol. 67, 4495-4503 (2001).
- Griffiths, R. I., Whiteley, A. S., O'Donnell, A. G., Bailey, M. J. Rapid method for coextraction of DNA and RNA from natural environments for analysis of ribosomal DNA- and rRNA-based microbial community composition. Appl. Environ. Microbiol. 66 (12), 5488-5491 (2000).
- Martins, G., et al. Structure and activity of lacustrine sediment bacteria involved in nutrient and iron cycles. FEMS Microbiol. Ecol. 77 (3), 666-679 (2011).
- Kuffner, M., et al. Effects of season and experimental warming on the bacterial community in a temperate mountain forest soil assessed by 16S rRNA gene pyrosequencing. FEMS Microbiol. Ecol. 82 (3), 551-562 (2011).
- Tatti, E., et al. Influences of over winter conditions on denitrification and nitrous oxide-producing microorganism abundance and structure in an agricultural soil amended with different nitrogen sources. Agric. Ecosyst . Environ. 183, 47-59 (2014).
- Giovannoni, S. J., Britschgi, T. B., Moyer, C. L., Field, K. G. Genetic diversity in the Sargasso Sea bacterioplankton. Nature. 345, 60-62 (1990).
- Ward, D. W., Weller, R., Bateson, M. M. 16S rRNA sequences reveal numerous uncultured microorganisms a natural community. Nature. 345, 63-65 (1990).
- Suzuki, M. T., Giovannoni, S. J. Bias caused by template annealing in the amplification of mixture of 16S rRNA genes by PCR. Appl. Environ. Microbiol. 62, 625-630 (1996).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous fluorescence monitoring of rapid cycle DNA amplification. Biotechniques. 22, 130-138 (1997).
- Holland, P. M., Abramson, R. D., Watson, R., Gelfand, D. H. Detection of Specific Polymerase Chain Reaction Product by Utilizing the 5' 3' Exonuclease Activity of Thermus aquaticus DNA Polymerase. PNAS. 88, 7276-7280 (1991).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol. Ecol. 67, 2-20 (2009).
- Park, S. J., Park, B. J., Rhee, S. K. Comparative analysis of archaeal 16S rRNA and amoA genes to estimate the abundance and diversity of ammonia-oxidizing archaea in marine sediments. Extremphiles. 12 (4), (2008).
- Urakawa, H., Yoshida, T., Nishimura, M., Ohwada, K. Characterization of depth-related population variation in microbial communities of a coastal marine sediment using 16S rDNA-based approaches and quinone profiling. Environ. Microbiol. 2, 542-554 (2008).
- Smith, C. J., Dong, L. F., Wilson, J., Stott, A., Osborn, A. M., Nedwell, D. B. Seasonal variation in denitrification and dissimilatory nitrate reduction to ammonia process rates and corresponding key functional genes along an estuarine nitrate gradient. Front. Microbiol. 6, 542(2015).
- Sambrook, J., Russell, D. W. Molecular Cloning: a laboratory manual. , Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press. 205(2001).
- Smith, C. J., Nedwell, D. B., Dong, L. F., Osborn, A. M. Evaluation of Quantitative Polymerase Chain Reaction (Q-PCR) based approaches for determining gene copy and gene transcript numbers in environmental samples. Environ. Microbiol. 8 (5), 804-815 (2006).
- Suzuki, M. T., Taylor, L. T. DeLong E.F., Quantitative analysis of small-subunit rRNA genes in mixed microbial population via 5'-nuclease assays. Appl. Environ. Microbiol. 66, 4605-4614 (2000).
- Luna, G. M., Dell'Anno, A., Danovaro, R. DNA extraction procedure: a critical issue for bacterial diversity assessment in marine sediments. Environ. Microbiol. 8 (2), 308-320 (2005).
- Lever, M. A., Torti, A., Eickenbursch, P., Michaud, A. B., Šantl-Temkiv, T., Jørgensen, B. B. A modular method for the extraction of DNA and RNA, and the separation of DNA pools from diverse environmental sample types. Front. Microbiol. 6, 476(2015).
- Lloyd, K. G., MacGregor, B. J., Teske, A. Quantitative PCR methods for RNA and DNA in marine sediments: maximizing yield while overcoming inhibition. FEMS Microbol. Ecol. 72, 144-151 (2010).
- Klappenbach, J. A., Dumbar, J. M., Schmidt, T. M. rRNA Operon copy number reflects ecological strategies of bacteria. App. Environ. Microbiol. 66 (4), 1328-1333 (2000).
- Shi, Y., Tyson, G. W., Eppley, J. M., DeLong, E. F. Integrated metatrascriptomic and metagenomics analyses of stratified microbial assemblages in the open ocean. ISME J. 5, 999-1013 (2011).
- Howe, A. C., Jansson, J. K., Malfatti, S. A. Tringe S.G., Tiedje J.M., & Brown C.T. Tackling soil diversity with the assembly of large, complex metagenomes. PNAS. 111 (13), 4904-4909 (2014).
- Klaedtke, S., et al. Terroir is a key driver of seed-associated microbial assemblages. Environ. Microbiol. , (2015).
- Lueders, T., Wagner, B., Claus, P., Friedrich MW, Stable isotope probing of rRNA and DNA reveals a dynamic methylotroph community and trophic interactions with fungi and protozoa in oxic rice field soil). Environ. Microbiol. 6, 60-72 (2004).
- Kunapuli, U., Lueders, T., Meckenstock, R. U. The use of stable isotope probing to identify key iron-reducing microorganisms involved in anaerobic benzene degradation. ISME J. 1, 643-653 (2007).
- Bürgmann, H., et al. Transcriptional response of Silicibacter pomeroyi DSS-3 to dimethylsulfoniopropionate (DMSP). Environ. Microbiol. 9 (11), 2742-2755 (2007).
- He, J. Z., et al. Geochip: a comprehensive microarray for investigating biogeochemical ecological and environmental processes. ISME J. 1, 67-77 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。