Method Article
הכין ליבות רקמות פרפין מוטבע פורמלין-קבועות הוא רנ"א הפקת DNA
In This Article
Summary
פרוטוקול חילוץ שונה זה משפר תשואות רנ"א ודנ"א מאזורים ממוקדים באופן מדויק יותר של עניין גושי רקמת histopathologic.
Abstract
Formalin-fixed paraffin embedded tissue (FFPET) represents a valuable, well-annotated substrate for molecular investigations. The utility of FFPET in molecular analysis is complicated both by heterogeneous tissue composition and low yields when extracting nucleic acids. A literature search revealed a paucity of protocols addressing these issues, and none that showed a validated method for simultaneous extraction of RNA and DNA from regions of interest in FFPET. This method addresses both issues. Tissue specificity was achieved by mapping cancer areas of interest on microscope slides and transferring annotations onto FFPET blocks. Tissue cores were harvested from areas of interest using 0.6 mm microarray punches. Nucleic acid extraction was performed using a commercial FFPET extraction system, with modifications to homogenization, deparaffinization, and Proteinase K digestion steps to improve tissue digestion and increase nucleic acid yields. The modified protocol yields sufficient quantity and quality of nucleic acids for use in a number of downstream analyses, including a multi-analyte gene expression platform, as well as reverse transcriptase coupled real time PCR analysis of mRNA expression, and methylation-specific PCR (MSP) analysis of DNA methylation.
Introduction
מחקר ביו-מרקר הגנום מבקש לזהות וקושרת מולקולריים בצורה מדויקת ואמין לשקף מצב מחלה, ולעשות זאת בצורה שימושית קליני. 1 פיתוח ביומרקר מסתמך על ניתוח רטרוספקטיבי של דגימות רקמה היטב מבוארות. דגימות רקמה חולות ונורמליות מאוחסנות או כמו רקמה טריה קפוא ב biobanks המיוחד או כרקמת פרפין מוטבע פורמלין-קבוע (FFPET) בלוקים בארכיונים קליניים. רקמה טריה מוקפאת מאפשרת המיצוי של חומצות גרעין איכותית כבר בשימוש נרחב במחקרי גילוי סמן ביולוגי הגנומי. 2,3 עם זאת, פחות דגימות רקמה זמינות biobanks ולומדים רקמות כגון משלבת הטיה לכיוון של מדגמים גדולים, קטגוריות חריגות של מחלה, וחולים לראות במרכזים העוסקים עם יכולות יותר לרקמות בנק. 4 FFPET, לעומת זאת, היא שיטת אחסון ברירת המחדל עבור ברקמות אדם וחי חולות. בעוד בלוקים FFPET לשמור מ הסלולרorphology, מרכיבים סלולריים אחרים צולבים קישורי תהליך הקיבוע כדי חומצות גרעין. צולבי רנ"א ודנ"א הם השבה, אך רק מושפל, מאוד צורות מקוטעות. 5,6 עם זאת, אלה שברי ה- DNA ו- RNA ניתנים לניתוח על ידי מערך הולך וגדל של מבחנים, כוללים ביטוי mRNA, היפר-מתילציה DNA, וממוקדות רצף. 7,8 כדי לנצל הזדמנות זו בכמות הגדולה והמגוונת של FFPET הזמין עבור מחקר, יש צורך בפרוטוקול מיצוי יעיל ואמין.
חלק גדול של מחקר סמן בתוך רקמות מתמקד סרטן. כמו סוגים אחרים של רקמה חולה, רקמת הסרטן לעתים קרובות מראה ההטרוגניות אזורים משמעותית בשימור תא סוג התא. מאז המחקר סמן ביולוגי מסתמך על היכולת לתאם מרכיבים של רקמה חולה עם תכונות מולקולריות, שלב קריטי של תהליך זה הוא הקצירה המדויקת של רקמות כי נשמרה היטב מועשרת עבור דisease נחקרת. בשנת FFPET, שתי טכניקות העשרה לעתים קרובות מנוצלים: microdissection ללכוד לייזר (LCM), חתך microtome. LCM מאפשר מאוד ממוקד קציר רקמות והוא יכול לשמש כדי לבודד ספציפי, סוגי תאים השתמרו היטב ברקמות הטרוגנית. 9,10 עם זאת, LCM דורש ציוד יקר זמן להחריד רב עבור מספר גדול של דגימות. חתך Microtome הוא תהליך בשימוש נרחב יותר שבו חלקים דקים נחתכים מבלוקים FFPET. 11,12 סעיפים Microtome לחתוך לעיתים קרובות כוללים רקמה הטרוגנית בשימור התא (למשל, לעומת נמקי השתמרו היטב) והרכב (למשל, סרטן לעומת parenchyma שפיר), ומכאן עלול להוביל המגון של תכונות מולקולריות נחקרו טוב בנפרד. לפיכך, יש צורך שיטת תפוקה גבוהה המעשיר עבור תאים של עניין. שיטה שלישית, בידוד של חומצות גרעין מ ליבות FFPET, מספקת העשרה זו, הוא מתאים throug הגבוההhput פרוטוקולים, ויש בו נעשה שימוש על ידי אחרים כדי לבודד RNA או DNA מן ליבות רקמות נפרדות. 7,13,14
מספר הפרוטוקולים שפורסמו לציין שיטות חילוץ חומצות גרעין מ FFPET (טבלה 1). עם זאת, פרוטוקולים בם רנ"א ודנ"א מופקים מאותו הרקמות ממוטבים עבור סעיפי רקמות microtome, אבל לא עבור ליבות רקמות. 15,16 באופן דומה, פרוטוקולים שפורסמו אשר מאפשרים להגדיל את סגוליות רקמה, בין אם באמצעות ליבות רקמות או microdissections שקופיות, נהלים להפקת DNA, אך לא RNA. 7,17 כאן, פרוטוקול אופטימיזציה עבור חילוץ כפול של שני ה- DNA ו- RNA מאותו ליבת הרקמות מודגמת. ליבות רקמות נקצרות על ידי החדרת microarray רקמות (TMA) אגרופים לתוך האזורים של עניין ממופה בלוקי FFPET. המיפוי מבוצע באמצעות סימון המיקרוסקופ שקופית עם עט סימון והעברת ביאור אל פני השטח של המקבילה FFPEבלוק T (איור 1).
עבודה לפני שהובילה להתפתחות של פרוטוקול זה נערכה השוואה של מערכות שאיבת חומצה כמה זמינות גרעין מסחרי. לשם השוואה זו, שינויי פרוטוקולים מסחריים, כמפורט להלן ספקו את היבול ואיכות DNA ו- RNA הגבוהים ביותר (Selvarajah et al., הכנה). ליבות רקמות עבות יותר בסעיפי מיקרון 5-10 מיקרומטר משמשים בדרך כלל פרוטוקולי מיצוי FFPET 11,12,14,18 - 20, ועשוי להכיל כמויות משתנות יותר של פרפין. כדי לפצות על זה, deparaffinization הועצם על ידי חזרה על טיפולים קסילן ו אתנול על ידי החדרת צעד המגון ממונע (איור 1). יתר על כן, פעמים עיכול K proteinase היו התארך כדי להגדיל את התשואה DNA. בסך הכל, פרוטוקול זה הוא חסכוני ומאפשר הקמת קשרים בין תכונות מולקולריות histopathologic של מחל large, אוכלוסיות מאופיינות היטב. הפרוטוקול בשלמותו יכול להתבצע באופן מהימן בתוך 2 ימים, כולל 3 שעות של הידיים על הזמן, ללא צורך גדול עבור ציוד מיוחד או יקר.
הצעד אחר צעד הפרוטוקול הוא להלן כגרסה שונה של הפרוטוקול של היצרן. 21 נא ראו טבלה של חומרים / ציוד עבור ריאגנטים ספציפיים, ציוד, ויצרנים.
Protocol
1. רקמות חציבה
- בדוק את השקופית מיקרוסקופ ולהתוות את האזור (ים) של עניין באמצעות סמן קבע קנס נקודות. לגזור קטע סרט פרפין גדול מספיק כדי לכסות את האזור של עניין בשקופית מיקרוסקופ. מניחים סרט בתקיפות בשקופית ועוטפים הסרט מעל לקצוות כדי לשמור את הסרט מפני החלקה. באמצעות סמן קבע קנס נקודות, להתוות את הרקמה כולה ובאזור (ים) של עניין בתוך הרקמה, שמירה על הנגיעה המתארת - אבל מחוץ - באזור (הים).
- הסר את הסרט ולהעביר אותו לבלוק רקמות המקביל. אוריינט סרטם של מרפרף או סיבובו כך את קווי המתאר של הרקמה כולה תואם את הצורה הנצפית של הרקמה בבלוק (איור 1). לחץ על קטע סרט בתקיפות אל פני השטח של הבלוק כדי למנוע החלקה.
- משתמש הקצה של בטוש בלתי מחיק, להפוך רדודים אך גלויים (~ 0.2 מ"מ) חריצים לאורך הקו המתאר של האזור (ים) של בterest, ולאחר מכן הסר את הסרט. טען 1 מ"ל של אקונומיקה, 70% אתנול, ומים לתוך צינורות מ"ל microcentrifuge 1.5 או 2.0 נפרד.
- נקה את אגרוף קולטן (אדום) מן האגרוף 0.6 מ"מ שקבע החלקת האגרוף מעלה ומטה מספר פעמים בעוד טיפ הוא שקוע לתוך האקונומיקה המכיל צינור. חזור על השלב מעל עם 70% אתנול ואז מים (קריטי כדי להבטיח אקונומיקה כי מוסר).
- לחצו על אגרוף לתוך הרקמה, בתוך אזור של אינטרס עד לעומק של 3 מ"מ ו למשוך את האגרוף. שחרר את הליבה לתוך נמוך מחייב צינור מיליליטר 1.5 או 2 על ידי דחיפתו אל מחוץ האגרוף עם העט. אחסן את הליבות ב -20 ° C (לטווח ארוך) או 4 ° C לשימוש לטווח קצר.
- נקה את האגרוף על פי צעד 1.4 ולהמשיך עם האזורים או המדגם הבאים.
2. Deparaffinize ליבות רקמות FFPE
- Carryout deparaffinization ב 1.5 או 2 צינורות מ"ל ידי הוספת קסילן 1 מ"ל עד היסוד רקמות vortexing במרץ למשך 10 שניות. heat 3 דקות ב 50 מעלות צלזיוס.
- צנטריפוגה במשך 2 דקות בטמפרטורת החדר (RT) ומהירות מרבית (21,130 XG) צינור ומניחים על קרח למשך 5 דקות (מאפשר משקע שעוותי לגבש על החלק העליון).
- מוציאים בזהירות פרפין שנצברו סביב המניסקוס עם supernatant באמצעות קצה פיפטה וחזור טיפול קסילן (שלבים 2.1-2.2).
- הוסף 1 מ"ל של אתנול (100%) ו מערבולת במרץ למשך 10 שניות. צנטריפוגות במשך 2 דקות ב RT (מהירות מרבית), ובזהירות להתעלמות אתנול. חזור על השלב הנ"ל פעם.
3. המגון של ליבות Deparaffinized
- Resuspend ליבות 700 μl של אתנול (100%) לפני המגון. באמצעות homogenizer רקמות ממונע, לטחון את הליבות לחלקיקי רקמות קנס (~ 1 דק 'על הגדרה בינונית). נקו את החללית homogenizer בין כל דגימה כדי למזער לשאת על זיהום.
- מלאו 15 מ"ל צינורות עם ~ 10 מ"ל של אקונומיקה, פתרון נטרול RNase ואתנול 70%. לאחר הומו המדגםgenization, לשטוף את החללית homogenizer בכל אחד הפתרונות ניקוי בסדר כאמור לעיל. הפעל את homogenizer על המהירות הגבוהה ביותר בשלב הכביסה.
- נגב את החללית עם רקמה ולאפשר בדיקה להתייבש לחלוטין לפני שמאחד את המדגם הבא. ראייה לבדוק את להבי בדיקה עבור פיסות רקמה שיורית. אם נמצא, לנקות את החללית שוב. שנה את פתרונות ניקוי (אקונומיקה, אתנול, והפתרון מנטרל RNase) יומי.
- בעקבות המגון, להביא את נפח דגימה עד 1 מ"ל על ידי הוספת אתנול 100% יותר (~ 300 μl). צנטריפוגה במהירות המרבית למשך 15 דקות, בזהירות לשאוב אתנול גלולה אוויר יבש למשך כ 15-20 דקות לפני שאתה ממשיך עם מיצוי RNA.
4. עיכול עם proteinase K
- Resuspend גלולה ב 150 μl proteinase K עיכול הצפת צינור קפיצי לשחרר את הכדור. הוסף 10 μl של K proteinase טמפרטורה יציבה ומערבבים ידי מצליף (לאמערבולת הצינור). דגירת התוכן בצינור ב 56 מעלות צלזיוס למשך 15 דקות עם תסיסה עדינה.
- אפשר צינור כדי לדגור על קרח למשך 3 דקות. קירור שלם חשוב ממטרים יעילים בשלב הבא. צנטריפוגה במשך 15 דקות במהירות מרבית.
5. RNA נפרד מ- DNA
- להעביר בזהירות את supernatant, מבלי להפריע גלולה, על 1.5 מ"ל חדש לטיהור RNA.
- שמור גלולה עבור טיהור DNA (גלולה יכול להיות מאוחסן במשך שעה 2 ב RT, עד 1 יום ב 2-8 מעלות צלזיוס, או לפרקי זמן ארוכים יותר ב -20 ° C).
6. RNA טיהור
- דגירת supernatant המכיל RNA על 80 מעלות צלזיוס למשך 15 דקות (לא תעלה הפעם). לאחר מכן, בקצרה צנטריפוגות צינור לאסוף טיפות מן הפנימי של המכסה.
- להוסיף 320 μl הצפת RLT להתאים תנאים מחייבים, ומערבבים על ידי pipetting. לאחר מכן, להוסיף 720 אתנול μl (100%), ו מערבולת.
- העברת 600 μl של המדגם, לרבות כל משקע שאולי נוצר, לעמודת ספין RNA (מסופקת בערכה) להציב צינור איסוף 2 מיליליטר ומניח בצד התוכן הנותר. צנטריפוגה במשך 15 שניות ≥8,000 XG, להשליך את הזרימה דרך שימוש חוזר צינור איסוף.
- העברת יתרת מדגם על טור, כולל טיפות שעשויות צברו במכסה של הצינור, צנטריפוגות במשך 15 שניות ≥8,000 XG, וזורקת את הזרימה דרך.
- להוסיף 350 μl הצפת FRN לעמודה ספין צנטריפוגות במשך 15 שניות ב ≥8,000 XG, להשליך את הזרימה דרך ושימוש חוזר צינור איסוף.
- בעדינות מערבבים 10 μl DNase אני פתרון מניות עם 70 μl הצפת RDD, להוסיף ישירות הממברנה טור ספין, לדגור על RT במשך 15 דקות.
- הוסף 500 μl מאגר FRN לעמודה ספין, צנטריפוגות במשך 15 שניות ≥8,000 XG ולשמור את הזרימה דרך לשימוש בשלב הבא. כדי לשפר את ההתאוששות של RNA הקטן, מניח את טור הספין בתוך 2 מ"ל חדש צינור איסוף ולהחיל את הזרימה דרך מהשלב הקודם לעמודה ספין.
- צנטריפוגה במשך 15 שניות ≥8,000 XG, להשליך את הזרימה דרך שימוש חוזר צינור איסוף בשלב הבא. הוסף 500 μl הצפת הרשתית לעמודה ספין צנטריפוגות במשך 15 שניות ב ≥8,000 XG, להשליך את הזרימה דרך שימוש חוזר צינור איסוף בשלב הבא.
- הוסף 500 μl הצפת רשתית לעמודת הספין צנטריפוגות במשך 15 שניות ב ≥8,000 XG וזורק את צינור האיסוף עם זרימת הדרך.
- מניח את טור ספין צינור איסוף חדש 2 מיליליטר, פותח את המכסה צנטריפוגות במהירות מרבית למשך 5 דקות. מחק את צינור האיסוף עם זרימת הדרך.
- מניחים את הטור ספין אוסף חדש 1.5 מ"ל, להוסיף 20 μl של RNase ללא מים ישירות על גבי הממברנה טור ספין, דגירה הצינור דקות 1 ב RT. צנטריפוגה במהירות המרבית למשך 1 דקות כדי elute רנ"א. חנות מדגם RNA eluted ב -80 מעלות צלזיוס.
ילדה = "jove_title"> 7. טיהור DNA
- Resuspend גלולה שהושגו במהלך החילוץ RNA על ידי תוספת בשלבים של 45 μl של חיץ proteinase K (400 מ"מ טריס 7.5, 400 מ"מ NaCl, 3 מ"מ MgCl 2, 4% SDS); 45 μl H 2 O; ו -400 מיקרוגרם של עוצמה גבוהה proteinase ק
- דגירת הפתרון הנ"ל ב 56 מעלות צלזיוס למשך 24 שעות (מומלץ) או הלילה. Performincubation ב 90 מעלות צלזיוס למשך 2 שעות ללא תסיסה בקצרה צנטריפוגות צינור microcentrifuge לאסוף טיפות מן הפנימי של המכסה.
- אפשר המדגם להתקרר RT ואז להוסיף 4 μl RNase A (100 מ"ג / מ"ל). דגירה המדגם למשך 2 דקות ב RT.
- הוסף 200 μl מאגר AL המדגם, ומערבבים היטב על ידי vortexing. לאחר מכן, הוסף 200 μl של 100% אתנול, ומערבבים היטב על ידי vortexing. מעבירים את המדגם לעמודה ספין סיפק, מקום בצינור אוסף 2 מ"ל, ו צנטריפוגות דקות 1 ב x ז ≥8,000.
- מחק את צינור האיסוף עםהזרימה דרך למקם את טור ספין צינור איסוף חדש 2 מיליליטר. הוסף 700 μl מאגר AW1 לעמודה ספין, צנטריפוגות במשך 15 שניות ≥8,000 XG, להשליך את הזרימה דרך שימוש חוזר צינור איסוף.
- הוסף 700 μl מאגר AW2 לעמודה ספין, צנטריפוגות במשך 15 שניות ≥8,000 XG, להשליך את הזרימה דרך שימוש חוזר צינור איסוף. לאחר מכן, הוסף 700 μl של אתנול 100% לעמודת הספין, צנטריפוגות במשך 15 שניות ≥8,000 XG וזורק את צינור האיסוף עם זרימת הדרך.
- מניח את טור ספין צינור איסוף חדש 2 מיליליטר, פתח את המכסה של טור הספין, צנטריפוגות במלוא מהירות במשך 5 דקות. מחק את צינור האיסוף עם זרימת הדרך.
- מניח את טור ספין צינור 1.5 מיליליטר אוסף חדש ולהוסיף 25 μl מי nuclease חינם מחוממים (50 מעלות צלזיוס). דגירת עמודת צינור ב 50 מעלות צלזיוס למשך 10 דקות. צנטריפוגה 1 דקות במהירות מרבית, להוסיף 25 μl מי nuclease חינם (RT) לעמודת דגירה1 דקות ב RT.
- צנטריפוגה 1 דקות במהירות המרבית (21,130 XG) וקציר הזרימה דרך המכילים DNA הגנומי (כ 50 μl של ה- DNA בסך הכל). אחסן את הטור ב -20 ° C (במקרה elution אחר נדרש בהמשך).
תוצאות
פרוטוקול זה מהווה שיטת אופטימיזציה עבור מחל DNA ו- RNA ליבות רקמות, באמצעות שינויים של מערכת הפקה מסחרית מיועדת סעיפי רקמות. אופטימיזציה כללה את כניסתה של הומוגניזציה רקמות, ניצול של K proteinase חזק יותר להפקת DNA, והארכת זמן עיכול רקמות. גרפי ניתוחים סטטיסטיים כלולים 2-way ANOVA, רגרסיה ליניארית וקורלציה.
אופטימיזציות של עיכול proteinase
ההערכה המסחרית כללה פתרון K proteinase היציב בטמפרטורת חדר אשר הוחלף עם K proteinase החזק יותר, וכתוצאה מכך תשואת DNA גבוהה יותר (איור 2 א). כדי להגביר עוד יותר את תשואות DNA, עיכול הוארך מעת 2 כדי 24 שעות. לא נמצא הבדלים משמעותיים נראו בין שתי נקודות הזמן, אבל לעיכול 24 השעות הופיע לספק תשואות עקביות יותר samples. עם זאת, דגירה נוספת ל -48 שעות לא לשפר עוד יותר את התאוששות DNA (איור 2B; p = 0.74).
שחזור DNA אופייני של דגימות רקמת סרטן ערמונית FFPE
באמצעות פרוטוקול מותאם, רנ"א ודנ"א היו שיתוף שחולצו מן דגימות FFPET סרטן הערמונית 333 הנעים בין 3 ל -14 שנות גיל מדגם. מכל מדגם, 3 ליבות רקמות (נפח רקמה כולל ממוצע של 0.95 ± 0.13 מ"מ 3) שמשו כקלט. אמנם יש שיטות ג'ל אלקטרופורזה אחרות המבוססות microfluidic שיכול להעריך ריכוזים ולספק הערכות של התפלגות הגודל של מולקולות חומצות גרעין, שיטות כאלה אינן מספקים כימות חומצות גרעין לשחזור, ולא ניתן להבחין בין רנ"א ודנ"א כמו לעשות מבחנים מבוססים flourometrically. 22 ו, כי תוצאות ג'ל אלקטרופורזה מבוסס microfluidic אינן אמינות מקוטעחומצות גרעין נגזרות FFPET, 23 תשואות חומצות גרעין נמדדו fluorometrically (ראה רשימה מגיב לפרטים נוספים). התשואה הממוצעת היתה 2,270 ננוגרם של רנ"א 820 ng של DNA (איור 3 א). כ 90% מכלל הדגימות FFPET ניתח במחקר זה הניב ≥100 ng של DNA ו- ≥ 500 ננוגרם של רנ"א. מעניין לציין, כי לא נמצא קשר משמעותי בין גיל מדגם FFPET והתאוששות חומצות גרעין (איור 3 ב). בסך הכל, תשואות RNA ו- DNA היו בקורלציה ברחבי דגימות (R 2 = 0.39, p <0.0001), אם כי יותר מכפליים RNA בהרבה DNA נמצא מכל מדגם (איור 3 ג).
כאשר עבודת הטייס ואופטימיזציה בוצעה על רקמות ערמונית, הצעד הבא היה לבדוק את הביצועים של פרוטוקול זה על כמה סוגים נוספים של רקמת ארכיונים. החל הוסר בניתוח דגימות FFPET נתיחה מייצגת להיותכבד nign (1 מדגם מ -1 במקרה), סרטן המוח (8 דגימות מ -1 במקרה), שלפוחית שתן (2 דגימות 2 מקרים), וסרטן השד (3 דגימות 3 מקרים), פרוטוקול ניב> 100 ng של DNA ו- RNA 90% מהדגימות (איור 3D). בעוד תשואות חומצות גרעין היו נמוכות ברקמות נתיחה מאשר ברקמות כירורגית, תוצאות נציג עולות כי בפרוטוקול מייצר תשואות דומות ברחבי הסרטן נגזר מאתרים שונים.
הערכת רנ"א ודנ"א Integrity וביצועים לנציגם ב ניתוח Downstream
ניתוח ביטוי RNA של 47 גנים ב -8 דגימות סרטן ערמונית FFPET נבחרות מדגם שורת תאי סרטן ערמונית PC-3 טרי (כביקורת חיובית) בוצע באמצעות פלטפורמת ביטוי מסחרית multianalyte הגן כי הוא מותאם במיוחד FFPET. ספירת mRNA ב PC3 הייתה בדרך כלל גבוהה יותר מאשר אלו של FFPET samples (איור 4 א). עם זאת, השוואת הביטוי היחסי של כל הגנים, דגימות סרטן הערמונית FFPET הראה פרופילים ביטוי דומה RNA PC-3, בה צוין כי שני מקורות RNA מתאימים פרופיל ביטוי RNA.
כדי להפגין ביצועים של הדנ"א הגנומי חילוץ עם פרוטוקול זה, תמציות DNA-המרה bisulfite ממדגמים FFPET היו מוגבר על ידי מתילציה ספציפי PCR (MSP). 24 ניתוח MSP של אלמנטים חוזרים ALU, מפוגל מאוד אזורים הנוכחי במיליוני עותקים בגנום האנושי, 25 שמשו כביקורת מתילציה גנומית, וצפויים להראות וריאציות מינימאליות בין דגימות. כפי שניתן לראות בתרשים 4 ב ', שם היה לא מעט על מנת וריאציה לראות בין מדגמים שונים ברמות מתילציה ALU MSP. יתר על כן, מבחני MSP מבוסס על GSTP1, בגן הידוע להיות hypermethylated בסרטן הערמונית אך לא בדגימות שפירות, 26 הראה שום ומחזקים לזיהויications ב דנ"א מדגימות שפיר. כצפוי, ערכי סף מחזור qPCR נמוכים אותרו DNA מרקמות הסרטן, המציין העשרה של עותקי GSTP1 מפוגלים. השירות של חומצות גרעין התאוששו על ידי פרוטוקול זה נבדק נוסף מבחנים במורד זרם טיפוסיים, באמצעות חומצות גרעין התאוששו מכבד שפירים מתוך מוח (פוסט-מורטם) וממנה שתי דגימות בניתוח סרטן השד. שני מבחני RT-qPCR מבוסס הביטוי MSP בצורה טובה על סרטן השד והכבד FFPET, אך assay RT-PCR לא להגביר mRNA מבוטא בכמות גבוהה מן המדגם גידול במוח שלאחר המוות (4C איור), דבר המצביע על כך RNA צנחה, הנראה בשל קיבעון רקמות מתעכב.
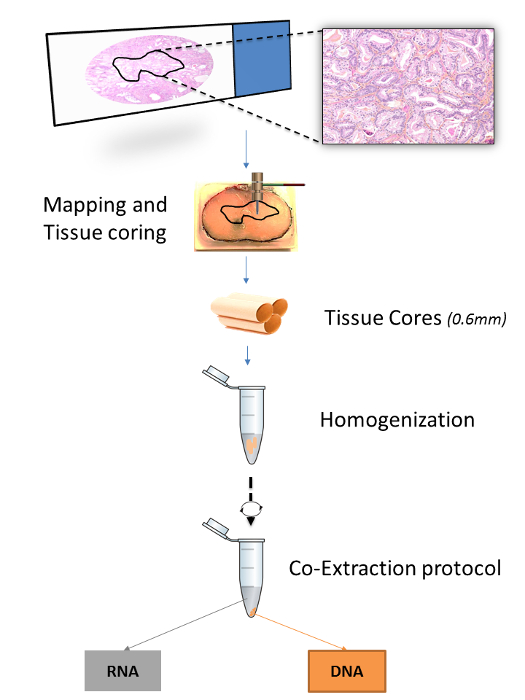
איור 1:. סקירה כללית של הליך ההפקה עבור דוגמאות FFPET הדמות מדגימה כיצד שטח של ענייןלחסום רקמות ממופות מבוסס על מבחר histopathologic של שקופית מיקרוסקופ. שלוש 0.6 ליבות רקמות מ"מ מתקבלות ומכל אחד באזור רקמות באמצעות אגרופי ביופסיה, הומוגני יחד ולאחר מכן נתון חילוץ הן של RNA ו- DNA. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
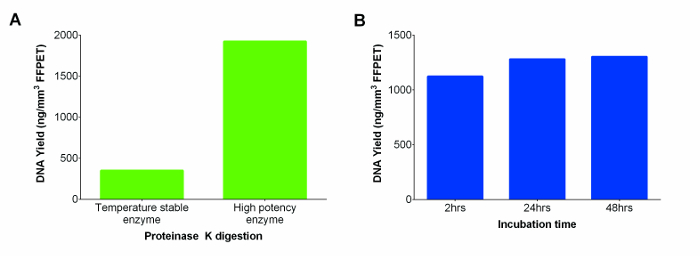
איור 2: חומצות גרעין (DNA ו- RNA) תשואות ng / מ"מ 3 של FFPET משני proteinase K (טמפרטורה יציבה אנזימי עצמה גבוהות), ונבדק על פני טווח של דגירת טיימס עבור האחרית (א) ביצועים של proteinase K. בין ספקים שונים. עקירות DNA בוצעו על מדגם FFPET נציג באמצעות אנזים יציב בטמפרטורה שמצורף kזה לעומת אנזים חזק יותר מיצרן אחר. (ב) קביעת זמן דגירה אופטימלי proteinase K על מנת למקסם את התשואה ה- DNA. ביצועים של ריכוז גבוה עיכול proteinase K הוערכו בשלוש תקופות דגירה שונות באמצעות 3 דגימות FFPET. ברי שגיאה מייצגים סטיית התקן של ממוצע (SEM). נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
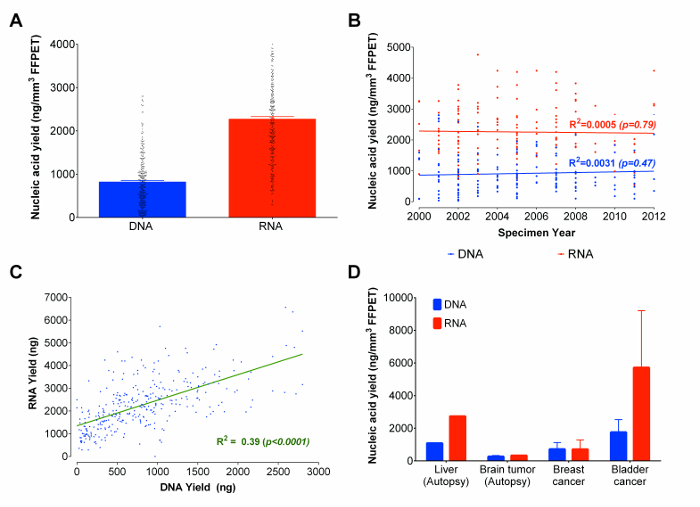
איור 3:. חומצות גרעין (DNA ו- RNA) תשואות ng / מ"מ 3 של FFPET בסך הכל, על פני שנים לדוגמא סוגי רקמות נציג (א) סך התאושש חומצות גרעין מרקמות פורמלין-קבוע מוטבע פרפין. כמויות חומצות הגרעין שהוצגו מבוססות על העקירות של 333 דגימות FFPET uלשיר את פרוטוקול אופטימיזציה. (ב) העלילה מתאם בין ה- DNA הכולל התאושש RNA והגיל של דגימות FFPET. הדגימות FFPET חילוץ המשמש התקבלו בשנים 2000 עד 2012. (ג) מתאם בין תשואות מן במקביל חילוץ DNA ו- RNA 333 דגימות הערמונית. קיים מתאם חיובי בין תשואות DNA ו- RNA. (ד) הפגנה של הפרוטוקול באמצעות סוגי רקמות ארכיונים נוספים. פרוטוקול אופטימיזציה שמש לחלץ חומצות גרעין מ -14 סרטן (שד, שלפוחית שתן ומוח) ונורמליות דגימות (כבדה). ברי שגיאה מייצגים SEM. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
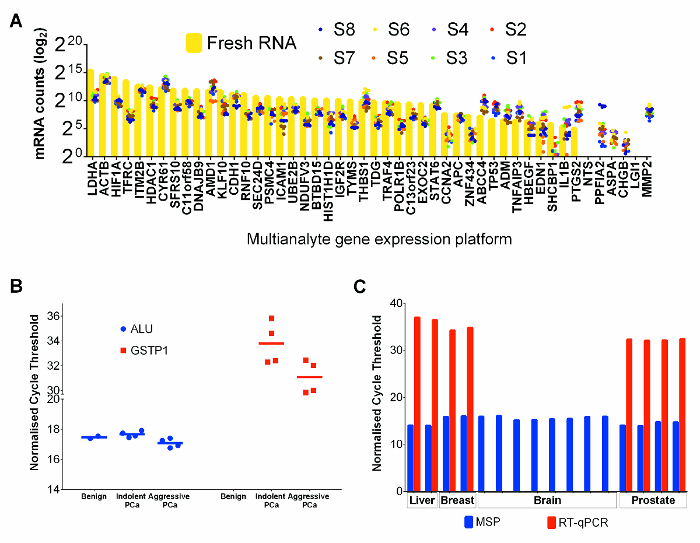
איור 4: ביצועים של רנ"א ודנ"א Co-מופק TissuCores הדואר ב Downstream יישומים. (א) mRNA נחשב עבור רקמות סרטן ערמונית FFPE ולשליטה PC-3 תאי קו טרי. כל נקודה מייצגת הממוצעת של 3 משכפל טכני חילוץ בנפרד. ערכי RNA תא PC-3 קו טרי מומחשים על ידי פסים צהובים וערכי רקמות FFPET מיוצגים על ידי נקודות צבעוניות. (ב) מבחני PCR ספציפיים מתילציה על DNA של דגימות סרטן הערמונית FFPET. ערכי מחזור סף התקבלו עבור 10 דגימות בצעו כצפוי באמצעות 50 ng / תגובה של DNA מרת bisulfite. מבחני MSP ALU מכל מדגם FFPET היו ערכי סף מחזור דומים (p> 0.67). מבחני GSTP1 MSP הראו מתילציה גבוהה (סף מחזור נמוך) רמות בסרטן הערמונית מאשר ערמונית שפירה. (ג) הערכת איכות DNA ו- RNA סוגי רקמות נוספות. HPRT1 ביטוי גני Alu גן מבחני מתילציה בוצעו על חומצות גרעין (RNA ו- DNA בהתאמה) מופקות ולאmal כבד (נתיחה), והמוח (נתיחה) וסרטן השד (כל FFPET). תוצאות הערמונית מוצגות להשוואה. הערה: תוצאות דומות נצפו מכל סוג רקמה, למעט הגברת mRNA נכשלה ממדגם אחד נתיחה. כל נקודה או עמודה מייצגת דגימה, ברי שגיאה מייצגים SEM. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| ליבות רקמות | |||||||||||
| קלט רקמות (mm3) | שחולץ חומצות גרעין | מַתַן תוֹקֵף | בדיקת איכות: DNA | בדיקת איכות: RNA | |||||||
| (# של דגימות) | גיל של המדגם (שנים) | תשואה סה"כ (נ"ג) | גודל שבר (נ"ב) | PCR | תשואה סה"כ (נ"ג) | גודל שבר (נ"ב) | PCR | NanoString | |||
| Pikor et al. | 43 - 129 | DNA | אין מידע | אין מידע | אין מידע | אין מידע | |||||
| Montaser-Kouhsari et al. | 18 - 29.5 | RNA | 763 | 0-25 | 843 | אין מידע | |||||
| מאמר זה | 1.71 | DNA ו- RNA | > 350 | 3-12 | 820 | 100 - 500 |  | 2270 | 100-500 |  | /ftp_upload/54299/check_mark.jpg "/> |
| סעיפי רקמות | |||||||||||
| קלט רקמות | שחולץ חומצות גרעין | מַתַן תוֹקֵף | בדיקת איכות: DNA | בדיקת איכות: RNA | |||||||
| (# של דגימות) | גיל של המדגם (שנים) | תשואה סה"כ (נ"ג) | גודל שבר (נ"ב) | PCR | תשואה סה"כ (נ"ג) | גודל שבר (נ"ב) | PCR | NanoString | |||
| הייכל et al. | 5 x 5 מיקרומטר | DNA | 12 | 7-22 | 88-300 | 103-351 |  | ||||
| צ'ונג et al. | 1 x 20 um | RNA | 9 | > 5 | 16,000- 23,000 | 100-200 |  | ||||
| Antica et al. | 2 x 4 מיקרומטר | RNA | 18 | אין מידע | לא ידוע (621 ng / μl) | 80-202 + |  | ||||
| Ghatak et al. | 5 x 5 מיקרומטר | DNA ו- RNA | 5 | 1 | 14 256 | <1030 |  | 16 000 | 109-400 + |  | |
| הניג et al. | 1 x 10 מיקרומטר | DNA ו- RNA | 210 | 1-25 | אין מידע | אין מידע |  | אין מידע | אין מידע |  | |
| Microdissection לכידת לייזר | |||||||||||
| קלט רקמות (mm2) | שחולץ חומצות גרעין | מַתַן תוֹקֵף | בדיקת איכות: DNA | בדיקת איכות: RNA | |||||||
| (# של דגימות) | גיל של המדגם (שנים) | תשואה סה"כ (נ"ג) | גודל שבר (נ"ב) | PCR | תשואה סה"כ (נ"ג) | גודל שבר (נ"ב) | PCR | NanoString | |||
| שלג et al. | 1-2 | DNA | 110 | 0-2 | 430 | אין מידע |  | ||||
הטבלה 1:. מהשוואת פרוטוקולי מיצוי RNA DNA ופורסמו עבור ליבות רקמות, מדורי ליזר ללכוד microdissections כללה גם כמה הערכת נקודות קצה המולקולרית של שיטות אלה תוך שימוש ב- PCR Nanostring מבחנים.
Discussion
עבור חילוץ מוצלח של DNA ו- RNA מאזורים רקמה של עניין, coring המדויק הוא קריטי. פרוטוקול זה מתאר את השימוש של אגרוף רקמות לבודד 0.6 מ"מ ליבות בקוטר מתאר את התהליך להעברת סימונים מן מיקרוסקופ שקופיות המקביל בלוקים FFPET. עשיית שינויי הפרוטוקול של היצרן נדרשו לחלץ חומצות גרעין ביעילות ליבות, שהן כ 50 פעמים עבות יותר בסעיפי microtome עבורו הפרוטוקול נועד. מאז הליבות עשויות להכיל ביחס שעווה פרפין יותר סעיפי רקמות, deparaffinization היעיל של ליבות באמצעות צעדי טיפול קסילן ואתנול חזרו נדרשו. הצלחת השלבים שלאחר deparaffinization תלוי המגון ליבות רקמה מכנית מתאימה ועיכול K proteinase ביותר. אופטימיזציה נוספת של דרכי העיכול proteinase K יכול להתבצע.
ראוי להזכיר כי זיהו שיטה זובאזורי fies עניין על פני השטח של הבלוק, כפי שהוגדרו מקביל שקופיות histopathology. כמו רקמת יבולי ליבה שעשויה להיות 3 או 4 מ"מ עמוק, משתמשים של פרוטוקול זה יכול להיות מודאגים לגבי מה תאים או רקמות הנח מתחת לפני שטח הבלוק. אמנם זה דאגה מוצדקת, מחקרים רבים (הנסקרת תוך התייחסות 27) הוכיחו כי ליבות רקמות מייצגים נאמנה את היסטולוגית ותכונות המולקולרי של גושי רקמה פתולוגית, במיוחד כאשר ליבות כפולות או בשלושה עותקים נדגמים מאזור עניין.
כמו ערכת החילוץ המסחרית השונה אמצה בפרוטוקול זה מאפשרת מיצוי במקביל של שני ה- DNA ו- RNA מאותו הרקמות, הפרוטוקול חוסך חומר ביולוגי יקר ומאפשר השוואה ישירה בין שתי חומצות גרעין נובע המדגם הזהה. מיצוי במקביל של רנ"א ודנ"א יצמצם דלדול עבודה ורקמות בחצי, ומאפשר ניתוח משולב מדויק של הגן expression, כמו גם תכונות אפיגנטיים וגנטיות מצויים בדנ"א. מאז התשואות של שניהם RNA ו- DNA מן ליבות רקמות נציגים אלה בדרך כלל יעלו על 600 ו -300 ng, בהתאמה, ומכיוון שרוב רצף הדור הנוכחי PCR וליד יישומים בדרך כלל דורשים 10-100 ng, רוב הדגימות המטוהרות על ידי פרוטוקול זה אמור לספק מספיק חומר במשך כמה מבחנים במורד זרם. פרוטוקול זה הוכח להיות לשחזור פני מעבדות עצמאיות (Selvarajah et al., הכנה.). RNA מן בפרוטוקול זה היה באיכות מספקת עבור ניתוח ביטוי גנים או באמצעות RT-PCR או פלטפורמה multianalyte פופולרי, ו- DNA ביצע היטב מבחני PCR ספציפיים מתילציה. מחקרים עתידיים שמטרתם להעריך את התועלת של חומצות גרעין התאוששו רצף הדור הבא הם מוצדקים.
לפיכך, מספר שינויים נעשו פרוטוקול זמין מסחרי, המיועד סעיפי FFPET דקים, טיוח זה מתאים FOr שיתוף החילוץ של RNA ו- DNA מ -0.6 מ"מ ליבות FFPET. הפרוטוקול הפגין תשואות גבוהות באופן עקבי במדגם גדול של דגימות סרטן ערמונית במספר מוגבל של דגימות סרטן השד, המוח ושלפוחית השתן. באופן כללי, הפרוטוקול אמור לאפשר למשתמשים לבצע ניתוחים ממוקדים מבוסס גנים של אוספים גדולים של רקמה היטב מבוארים. חשוב לציין כי הפרוטוקול מאפשר דגימה ממוקדת ויעילה של אזורים של עניין FFPET, ידות על מעט יחסית זמן, תשואות גבוהות מספיק עבור מרבית היישומים במורד הזרם.
Disclosures
The authors declare no competing financial interests.
Acknowledgements
This research was supported by a team grant from Movember/Prostate Cancer Canada to JMSB, DMB, PCP, and JL, and by the Ontario Institute of Cancer Research (JMSB, DMB, and PCP) and Motorcycle Ride for Dad Kingston/University Hospitals Kingston Foundation/Kingston General Hospital (DMB, PCP).
Materials
| Name | Company | Catalog Number | Comments |
| Plastic paraffin film, "Parafilm 'M'" | Bemis | RK-06720-40 | Any generic paraffin film will work as a substitute |
| Sodium Hypochlorite, "Ultra Bleach" | Likewise | 53-2879-2 | Any generic bleach will work as a substitute. Hazardous material that can cause burns on contact. |
| Molecular biology grade absolute ethanol | Fisher BioReagents | BP2818-500 | Sigma-Aldrich E7023 suffices as a substitute |
| Molecular Grade H2O | G-Biosciences | 786-293 | Sigma W4502 suffices, as well as any other brand of molecular grade H2O |
| 0.6 mm Punch Set for Beecher Instruments | Estigen | MPO6[Yellow] | Make sure to use the red receiver punch from the set |
| Fine point permanent marker | Sharpie | 10365796S | Using the marker on FFPE tissues causes it to dry out quickly, so several may be required |
| FFPE tissue block | |||
| Stained tissue slide corresponding to FFPE block | |||
| 1.5 ml Micro-Centrifuge Tubes | Fisher BioReagents | 05-408-137 | |
| 2.0 ml Low binding tubes (LoBind Micro-Centrifuge Tubes) | Eppendorf | 22431048 | |
| 1.5 ml Low binding tubes (LoBind Micro-Centrifuge Tubes) | Eppendorf | 22431021 | |
| Histology Xylene | VWR | CA 95057-822 | Fisher Scientific X5-500 suffices as a substitute |
| Molecular Biology Grade 2-Propanol | Sigma | I9516 | |
| AllPrep FFPE DNA/RNA Kit | Qiagen | 80234 | Prepare buffers accodring to the AllPrep DNA/RNA FFPE Handbook21 |
| Buffers: RLT, FRN, RPE, ATL, AL, AW1, AW2, DNaseI solution | Qiagen | 80234 | Prepare buffers accodring to the AllPrep DNA/RNA FFPE Handbook21 |
| Temperature stable proteinase K | Qiagen | 80234 | |
| High potency proteinase K | Invitrogen | 25530-049 | Invitrogen 25530-015 suffices as a substitute |
| RNAse neutralizing solution (Rnase AWAY) | Molecular BioProducts | 7003 | |
| RNaseA 100 mg/ml | Qiagen | 19101 | |
| BD Integra Syringe 3 ml 21G x 1/2 | BD | 305274 | |
| Motorized tissue homogenizer (TissueRuptor) | Qiagen | 9001271 | Fisher Scientific 14-261-29 suffices as a substitute |
| -20 °C and -80 °C Laboratory Freezer | |||
| Micro-Centrifuge with rotor for 2 ml tubes | |||
| Digital Vortex Mixer | |||
| Pipettes and filter tips | |||
| Heating blocks or water baths | |||
| Tris Hydrochloride | Amresco | 0234 | |
| Sodium Chloride | Amresco | 0241 | |
| Anhydrous Magnesium Chloride | Sigma | M8266 | |
| Sodium Dodecyl Sulfate | Sigma | L4509 | |
| Acrodisc 25 mm syring filters with 0.45 µm Supor membrane | Pall | PN 4614 | |
| Syringe with retracting BD PrecisionGlide needle 3 ml | BD Integra | 305274 | |
| Hydrochloric Acid | BDH | 3026 | |
| Multianalyte gene expression platfrom (nCounter ® CAE codeset and Nanostring nCounter platform) | Nanostring nCounter platform, Nanostring | ||
| Fluorometric nucleic acid quantification (Qubit dsDNA HS Assay Kit and Qubit® RNA BR Assay Kit) | Invitrogen |
References
- Kern, S. E. Why your new cancer biomarker may never work: recurrent patterns and remarkable diversity in biomarker failures. Cancer Res. 72 (23), 6097-6101 (2012).
- Klopfleisch, R., Weiss, A. T. A., Gruber, A. D. Excavation of a buried treasure--DNA, mRNA, miRNA and protein analysis in formalin fixed, paraffin embedded tissues. Histol. Histopathol. 26 (6), 797-810 (2011).
- Beltran, H., et al. Targeted Next-generation Sequencing of Advanced Prostate Cancer Identifies Potential Therapeutic Targets and Disease Heterogeneity. Eur. Urol. 63 (5), 920-926 (2013).
- Hoppin, J. A., Tolbert, P. E., Taylor, J. A., Schroeder, J. C., Holly, E. A. Potential for selection bias with tumor tissue retrieval in molecular epidemiology studies. Ann. Epidemiol. 12 (1), 1-6 (2002).
- von Ahlfen, S., Missel, A., Bendrat, K., Schlumpberger, M. Determinants of RNA quality from FFPE samples. PLOS ONE. 2 (12), e1261 (2007).
- Masuda, N., Ohnishi, T., Kawamoto, S., Monden, M., Okubo, K. Analysis of chemical modification of RNA from formalin-fixed samples and optimization of molecular biology applications for such samples. Nucleic Acids Res. 27 (22), 4436-4443 (1999).
- Pikor, L. A., Enfield, K. S. S., Cameron, H., Lam, W. L. DNA extraction from paraffin embedded material for genetic and epigenetic analyses. J. Vis. Exp. (49), (2011).
- Turashvili, G., et al. Nucleic acid quantity and quality from paraffin blocks: defining optimal fixation, processing and DNA/RNA extraction techniques. Exp. Mol. Pathol. 92 (1), 33-43 (2012).
- Espina, V., et al. Laser-capture microdissection. Nat. Protoc. 1 (2), 586-603 (2006).
- Hackler, L., Masuda, T., Oliver, V. F., Merbs, S. L., Zack, D. J. Use of laser capture microdissection for analysis of retinal mRNA/miRNA expression and DNA methylation. Methods Mol. Biol. 884, 289-304 (2012).
- Bonin, S., Stanta, G. Nucleic acid extraction methods from fixed and paraffin-embedded tissues in cancer diagnostics. Expert Rev. Mol. Diagn. 13 (3), 271-282 (2013).
- Bonin, S., et al. Multicentre validation study of nucleic acids extraction from FFPE tissues. Virchows Archiv. 457 (3), 309-317 (2010).
- Montaser-Kouhsari, L., et al. Image-guided Coring for Large-scale Studies in Molecular Pathology. Appl. Immunohistochem. Mol. Morphol. , (2015).
- van Eijk, R., Stevens, L., Morreau, H., van Wezel, T. Assessment of a fully automated high-throughput DNA extraction method from formalin-fixed, paraffin-embedded tissue for KRAS, and BRAF somatic mutation analysis. Exp. Mol. Pathol. 94 (1), 121-125 (2013).
- Ghatak, S., Sanga, Z., Pautu, J. L., Kumar, N. S. Coextraction and PCR Based Analysis of Nucleic Acids From Formalin-Fixed Paraffin-Embedded Specimens. J. Clin. Lab. Anal. , (2014).
- Hennig, G., et al. Automated extraction of DNA and RNA from a single formalin-fixed paraffin-embedded tissue section for analysis of both single-nucleotide polymorphisms and mRNA expression. Clin. Chem. 56 (12), 1845-1853 (2010).
- Snow, A. N., Stence, A. A., Pruessner, J. A., Bossler, A. D., Ma, D. A simple and cost-effective method of DNA extraction from small formalin-fixed paraffin-embedded tissue for molecular oncologic testing. BMC Clin. Pathol. 14 (1), 30 (2014).
- Torrente, M. C., et al. DNA extraction from formalin-fixed laryngeal biopsies: Comparison of techniques. Acta Otolaryngol. 131 (3), 330-333 (2011).
- Okello, J. B. A., et al. Comparison of methods in the recovery of nucleic acids from archival formalin-fixed paraffin-embedded autopsy tissues. Anal. Bochem. 400 (1), 110-117 (2010).
- Abramovitz, M., et al. Optimization of RNA extraction from FFPE tissues for expression profiling in the DASL assay. BioTechniques. 44 (3), 417-423 (2008).
- . . AllPrep DNA/RNA FFPE Handbook. , (2012).
- Laurent, L. C., et al. Meeting report: discussions and preliminary findings on extracellular RNA measurement methods from laboratories in the NIH Extracellular RNA Communication Consortium. Journal of Extracell. Vesicles. 4, (2015).
- . Methods of RNA Quality Assessment Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment (2012)
- Herman, J. G., Graff, J. R., Myohanen, S., Nelkin, B. D., Baylin, S. B. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc. Natl. Acad. Sci. U.S.A. 93 (18), 9821-9826 (1996).
- Weisenberger, D. J., Campan, M., et al. Analysis of repetitive element DNA methylation by MethyLight. Nucleic acids research. 33 (21), 6823-6836 (2005).
- Yegnasubramanian, S. Hypermethylation of CpG Islands in Primary and Metastatic Human Prostate Cancer. Cancer Res. 64 (6), 1975-1986 (2004).
- Parsons, M., Grabsch, H. How to make tissue microarrays. Diagn. Histopathol. 15 (3), 142-150 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved