Method Article
Herstellung von Formalin-fixierten und in Paraffin eingebetteten Gewebe Cores sowohl für RNA und DNA-Extraktion
In diesem Artikel
Zusammenfassung
Dieses modifizierte Extraktionsprotokoll verbessert RNA und DNA-Ausbeuten von gezieltere Regionen von Interesse in histopathologischen Gewebeblöcke.
Zusammenfassung
Formalin-fixed paraffin embedded tissue (FFPET) represents a valuable, well-annotated substrate for molecular investigations. The utility of FFPET in molecular analysis is complicated both by heterogeneous tissue composition and low yields when extracting nucleic acids. A literature search revealed a paucity of protocols addressing these issues, and none that showed a validated method for simultaneous extraction of RNA and DNA from regions of interest in FFPET. This method addresses both issues. Tissue specificity was achieved by mapping cancer areas of interest on microscope slides and transferring annotations onto FFPET blocks. Tissue cores were harvested from areas of interest using 0.6 mm microarray punches. Nucleic acid extraction was performed using a commercial FFPET extraction system, with modifications to homogenization, deparaffinization, and Proteinase K digestion steps to improve tissue digestion and increase nucleic acid yields. The modified protocol yields sufficient quantity and quality of nucleic acids for use in a number of downstream analyses, including a multi-analyte gene expression platform, as well as reverse transcriptase coupled real time PCR analysis of mRNA expression, and methylation-specific PCR (MSP) analysis of DNA methylation.
Einleitung
Genomische Biomarker - Forschung sucht molekulare Korrelate zu identifizieren , die genau und zuverlässig Krankheitszustand widerspiegeln, und zwar in einer klinisch nützliche Art und Weise. 1 Biomarkerentwicklung auf retrospektive Analyse von gut kommentierten Gewebeproben angewiesen ist. Erkrankte und Normalgewebeproben werden entweder als gefrorenes Frischgewebe in spezialisierten Biobanken oder als Formalin-fixierten und in Paraffin eingebetteten Gewebeproben (FFPET) Blöcke in klinischen Archiven gespeichert. Frisch gefrorene Gewebe ermöglicht die Gewinnung von hochwertigen Nukleinsäuren und wurde in der genomischen Entdeckung neuer Biomarker - Studien weit verbreitet. 2,3 jedoch weniger Gewebeproben in Biobanken zur Verfügung stehen und so Gewebe studieren führt eine Tendenz zu größeren Proben, ungewöhnliche Kategorien erkrankter Mensch und Tier die Standard - Speicherverfahren für Gewebe von Krankheit und Patienten in spezialisierten Zentren mit mehr Fähigkeiten zu Bank Gewebe 4 FFPET, ist dagegen gesehen.. Während FFPET Blöcke halten zelluläre morphology, die Fixierung Prozess Vernetzungen andere zelluläre Bestandteile zu Nukleinsäuren. Vernetzte RNA und DNA sind erzielbare, aber nur in verschlechterten, stark fragmentierten Formen. 5,6 Allerdings sind diese DNA und RNA - Fragmente sind zugänglich für die Analyse durch eine zunehmend größere Anzahl von Assays, einschließlich der mRNA - Expression, DNA hypermethylation und gezielte Sequenzierung. 7,8 Um diese Möglichkeit in der großen Menge und Vielfalt der FFPET für die Forschung zur Verfügung ausnutzen, gibt es einen Bedarf für eine effiziente und zuverlässige Extraktion Protokoll.
Ein großer Teil der Biomarker-Forschung in Gewebe konzentriert sich auf Krebs. Wie andere Arten von erkranktem Gewebe, Krebsgewebe zeigt oft erhebliche regionale Heterogenität in Zellkonservierung und Zelltyp. Da biomarker Forschung auf der Fähigkeit beruht Bestandteile von erkranktem Gewebe mit molekularen Eigenschaften zu korrelieren, ein kritischer Schritt dieses Verfahrens ist die genaue Entnahme von Gewebe, das für die d gut erhalten und angereichert wirdisease untersucht. In FFPET werden zwei Anreicherungstechniken oft in Anspruch genommen: Laser Mikrodissektion (LCM) und Mikrotom Schnitte. LCM ermöglicht eine hochGewebeErnte fokussiert und verwendet werden können , spezifische, gut erhaltene Zelltypen in heterogenen Geweben zu isolieren. 9,10 erfordert jedoch LCM teure Ausrüstung und ist prohibitiv Zeit für eine große Anzahl von Proben raubend. Mikrotom Schnitte ist ein weit verbreitetes Verfahren , bei dem dünne Schnitte von FFPET Blöcke geschnitten werden. 11,12 Mikrotom geschnittenen Abschnitte oft Gewebe enthalten, die in Zellkonservierung (zB nekrotischen vs. gut erhaltene) und Zusammensetzung heterogen ist (zB Krebs vs. gutartigen Parenchym) und somit kann am besten getrennt untersucht, um die Homogenisierung der molekularen Eigenschaften führen. Somit besteht ein Bedarf für ein Hochdurchsatzverfahren, das für die Zellen von Interesse anreichert. Eine dritte Methode, die Isolierung von Nukleinsäuren aus FFPET Kerne, stellt diese Anreicherung ist geeignet für hohe throughput Protokolle und wurde von anderen benutzt worden RNA oder DNA aus einzelnen Gewebekerne zu isolieren. 7,13,14
Eine Reihe von veröffentlichten Protokolle spezifizieren Methoden von Nukleinsäuren aus FFPET (Tabelle 1) zu extrahieren. Allerdings Protokolle , bei denen RNA und DNA aus dem gleichen Gewebe extrahiert wurden für Mikrotom Gewebeschnitte optimiert worden, aber nicht für die Gewebekerne. 15,16 Ebenso veröffentlichte Protokolle , die Gewebespezifität erhöht bieten, entweder durch das Gewebe Kerne oder Schieber Mikrodissektionen, geben Verfahren zur Extraktion von DNA, aber nicht RNA. 7,17 Hier wird ein optimiertes Protokoll für Dual Extraktion von sowohl DNA als auch RNA , die aus dem gleichen Gewebekern demonstriert. Tissue-Kerne werden durch das Einfügen Tissue Microarray (TMA) Schläge in Regionen von Interesse gemappt FFPET Blöcke geerntet. Das Mapping wird durch Kommentierung einen Objektträger mit einem Markierungsstift und Übertragen der Annotation zu der Oberfläche des entsprechenden FFPE ausgeführtT - Block (Abbildung 1).
Frühere Arbeiten, die zur Entwicklung dieses Protokolls führte, einen Vergleich von mehreren kommerziell erhältlichen Nukleinsäureextraktion Systeme. In diesem Vergleich Modifikationen an kommerziellen Protokolle, wie unten beschrieben , die höchsten DNA und RNA - Ausbeuten und Qualität zur Verfügung gestellt (Selvarajah et al., In Vorbereitung). Gewebekerne sind dicker als die 5-10 um micron Abschnitte verwendet typischerweise in FFPET Extraktionsprotokolle 11,12,14,18 - 20, und kann mehr variable Mengen an Paraffin enthalten. Um dies auszugleichen, wurde verstärkt durch Entparaffinierung Xylol und Ethanol Behandlungen zu wiederholen und durch eine motorisierte Homogenisierungsschritt (Figur 1) eingeführt wird . Weiterhin wurden Proteinase K Verdau Zeiten verlängert DNA-Ausbeute zu erhöhen. Insgesamt ist dieses Protokoll ist kostengünstig und ermöglicht die Einrichtung von Verbindungen zwischen den molekularen und histopathologischen Merkmale der Krankheit in large, gut charakterisierten Populationen. Das Protokoll in seiner Gesamtheit kann innerhalb von 2 Tagen zuverlässig durchgeführt werden, einschließlich 3 Stunden von Hands-on-Zeit, mit wenig Bedarf an spezialisierten oder teure Ausrüstung.
Die Schritt- für -Schritt - Protokoll ist im Folgenden als eine modifizierte Version des Protokolls des Herstellers. 21 entnehmen Sie bitte Tabelle der Materialien / Ausrüstung für spezifische Reagenzien, Geräte und Hersteller.
Protokoll
1. Die Gewebe Coring
- Überprüfen Sie die Mikroskop-Objektträger und skizzieren die Region (en) von Interesse ein fein Punkt permanent Marker. Schneiden Sie einen Abschnitt von Paraffinfilm aus groß genug, um den Bereich von Interesse auf dem Objektträger zu decken. Legen Film fest auf Folie und Wrap Folie über Kanten der Film rutscht zu halten. Mit einem feinen Punktpermanentmarker, skizzieren das gesamte Gewebe und die Region (en) von Interesse innerhalb des Gewebes, die Umrisse berühren zu halten - aber außerhalb - die Region (en).
- Entfernen Sie den Film und übertragen sie an den entsprechenden Gewebeblock. Orientieren der Folie durch Umklappen oder Drehen , so daß der Umriß des gesamten Gewebes entspricht der beobachteten Form des Gewebes in dem Block (Abbildung 1). Drücken der Abschnitt der Folie fest an der Oberfläche des Blocks zu Verrutschen verhindern.
- Mit der Spitze des Permanentmarker machen flach aber sichtbar (~ 0,2 mm) Vertiefungen entlang der Kontur der Region (en) inInteresse, dann den Film zu entfernen. Last 1 ml Bleichmittel, 70% Ethanol und Wasser in separate 1,5 oder 2,0 ml Mikrozentrifugenröhrchen.
- Reinigen Sie den Rezeptor (rot) Stempel aus dem 0,6 mm Stempel gesetzt durch den Stempel nach oben schieben und nach unten mehrmals während die Spitze in das Rohr mit Bleichlauge eingetaucht ist. Wiederholen der obigen Schritt mit 70% Ethanol und dann Wasser (wichtig, dass Bleichmittel, um sicherzustellen, entfernt wird).
- Drücken Sie den Stempel in das Gewebe, in der Region von Interesse zu einer Tiefe von 3 mm und den Stempel zurückzuziehen. Lassen Sie den Kern in eine niedrige Bindungs 1,5 oder 2-ml-Röhrchen, indem sie es aus dem Schlag mit dem Stift drücken. Lagern Sie die Kerne bei -20 ° C (langfristige) oder 4 ° C für den kurzfristigen Einsatz.
- Reinigen Sie den Stempel nach 1.4 zu treten und mit den nächsten Regionen oder Probe fortzusetzen.
2. entparaffinieren FFPE Gewebe Cores
- Carryout Entparaffinisierung in 1,5 oder 2-ml-Röhrchen durch Zugabe von 1 ml Xylol zu dem Gewebe Kern und Verwirbelung kräftig für 10 Sekunden. Heat für 3 min bei 50 ° C.
- Zentrifuge für 2 min bei Raumtemperatur (RT) und maximale Geschwindigkeit (21.130 xg) und das Röhrchen auf Eis für 5 min (ermöglicht dem waxy Rückstand auf dem oberen zu verfestigen).
- mit einer Pipettenspitze und wiederholen Xylol Behandlung (Schritte 2,1-2,2) vorsichtig Paraffin um Meniskus mit Überstand angesammelt entfernen.
- 1 ml Ethanol (100%) und Vortex kräftig für 10 Sekunden. Zentrifuge für 2 min bei RT (maximale Drehzahl) und vorsichtig Ethanol verwerfen. Wiederholen Sie einmal den obigen Schritt.
3. Homogenisieren der Entparaffinierte Cores
- Resuspendieren der Kerne in 700 ul Ethanol (100%) vor der Homogenisierung. Mit Hilfe eines motorisierten Gewebe-Homogenisator, mahlen die Kerne in feine Gewebepartikel (~ 1 min auf mittlere Einstellung). Reinigen Sie den Homogenisator Sonde zwischen den einzelnen Proben-Verschleppung zu minimieren Kontamination.
- Füllen Sie 15 ml-Röhrchen mit ~ 10 ml Bleiche, RNase Neutralisierungslösung und 70% Ethanol. Nach der Proben homogenisierung, waschen Sie die Homogenisator Sonde in jedem der Reinigungslösungen in der Reihenfolge wie oben angegeben. Führen Sie den Homogenisator auf der höchsten Geschwindigkeit während der Waschstufe.
- Wischen Sie die Sonde mit dem Gewebe und lassen Sonde vollständig trocknen, bevor die nächste Probe homogenisiert. Sichtprüfung der Sonde Klingen für Restgewebestücke. Wenn sie gefunden wurde, reinigen Sie die Sonde erneut. Ändern Sie die Reinigungslösungen (Bleichmittel, Ethanol und RNase neutralisierende Lösung) täglich.
- Nach der Homogenisierung bringen das Probenvolumen auf 1 ml von mehr 100% Ethanol Zugabe (~ 300 ul). Zentrifuge bei maximaler Geschwindigkeit für 15 min, aspirieren sorgfältig das Ethanol und Luft trocknen Pellets für ca. 15-20 Minuten, bevor sie mit RNA-Extraktion fortfahren.
4. Verdauung mit Proteinase K
- Das Pellet in 150 & mgr; l Proteinase K Digestion Buffer und das Röhrchen klopfen, um das Pellet zu lösen. In 10 ul temperaturstabile Proteinase K und mischen durch Ausklopfen (nichtverwirbeln das Rohr). Inkubieren des Inhalts in das Rohr bei 56 ° C für 15 min unter leichtem Rühren.
- Erlauben Röhrchen auf Eis für 3 min inkubiert. Vollständige Kühlung ist wichtig für eine effiziente Fällung im folgenden Schritt. Zentrifuge für 15 min bei maximaler Geschwindigkeit.
5. Separate RNA aus DNA
- übertragen vorsichtig den Überstand, ohne das Pellet zu stören, in ein neues 1,5 ml für RNA-Reinigung.
- Halten Sie das Pellet für die DNA-Reinigung (Pellet kann für 2 Stunden bei RT gelagert werden, bis zu 1 Tag bei 2-8 ° C oder für längere Zeit bei -20 ° C).
6. RNA-Reinigung
- Inkubieren der RNA enthaltende Überstand bei 80 ° C für 15 min (übersteigen diese Zeit nicht). Als nächstes wird kurz zentrifugieren das Rohr Tropfen aus der Innenseite des Deckels zu sammeln.
- In 320 & mgr; l Puffer RLT Bindungsbedingungen einzustellen, und mischen durch Pipettieren. Als Nächstes fügen Sie 720 ul Ethanol (100%) und Wirbel.
- Transfer 600 ul der Probe, einschließlich der Niederschlag, der sich gebildet haben können, zu RNA-Spin-Säule (im Kit mitgeliefert) in ein 2 ml Sammelröhrchen gelegt und der übrige Inhalt beiseite stellen. Zentrifuge für 15 Sekunden bei ≥8,000 xg, entsorgen Sie die Flow-Through und Wiederverwendung der Sammelröhrchen.
- Transfer verbleibende Probe auf eine Säule, einschließlich der Tröpfchen, die bei ≥8,000 xg für 15 sec im Deckel des Röhrchens, Zentrifuge angesammelt haben können, und den Durchlauf verwerfen.
- In 350 & mgr; l Puffer FRN auf die Spinsäule und Zentrifuge für 15 Sekunden bei ≥8,000 xg, entsorgen Sie die Flow-Through-und Sammelrohr wieder verwenden.
- Sanft 10 ul DNase I-Stammlösung für 15 Minuten mit 70 & mgr; l Puffer RDD, fügen Sie direkt auf die Spinsäule Membran und Inkubation bei RT mischen.
- In 500 ul Puffer FRN auf die Spinsäule, Zentrifuge für 15 Sekunden bei ≥8,000 xg und speichern Sie die Flow-Through für die Verwendung im nächsten Schritt. Zur Verbesserung der Rückgewinnung von kleinen RNAs, legen Sie die Spin-Säule in einSammelröhrchen neues 2 ml und Anwendung der Durchfluss aus dem vorherigen Schritt auf die Spin-Säule.
- Zentrifuge für 15 Sekunden bei ≥8,000 xg, den Durchlauf verwerfen und das Sammelrohr im nächsten Schritt wieder verwenden. In 500 ul Puffer RPE in die Spin-Säule und Zentrifuge für 15 Sekunden bei ≥8,000 xg, entsorgen Sie die Flow-Through und Wiederverwendung der Sammelröhre in den nächsten Schritt.
- In 500 ul Puffer RPE in die Spin-Säule und Zentrifuge für 15 Sekunden bei ≥8,000 xg und entsorgen Sie die Sammelröhrchen mit dem Flow-Through.
- Legen Sie die Spin-Säule in ein neues 2 ml Sammelröhrchen, öffnen Sie den Deckel und Zentrifuge bei maximaler Geschwindigkeit für 5 min. Entsorgen Sie das Sammelrohr mit dem Flow-Through.
- Legen Sie die Spin-Säule in ein neues 1,5-ml-Collection, fügen Sie 20 ul RNase-freies Wasser direkt auf die Spinsäule Membran und inkubieren Sie die Röhrchen für 1 min bei RT. Zentrifuge bei maximaler Geschwindigkeit für 1 Minute um die RNA zu eluieren. Lagern Sie die eluierte RNA-Probe bei -80 ° C.
7. DNA Purification
- Das Pellet während der RNA - Extraktion durch schrittweise Zugabe von 45 & mgr; l Proteinase K Puffer erhalten (400 mM Tris 7,5, 400 mM NaCl, 3 mM MgCl 2, 4% SDS); 45 & mgr; l H 2 O; und 400 ug hoher Potenz Proteinase K.
- Inkubieren der obigen Lösung bei 56 ° C für 24 h (empfohlen) oder über Nacht. Performincubation bei 90 ° C für 2 Stunden ohne Rühren und kurz zentrifugieren das Mikrozentrifugenröhrchen Tropfen aus dem Inneren des Deckels zu sammeln.
- Lassen Sie die Probe auf RT abkühlen und fügen Sie dann 4 ul RNase A (100 mg / ml). Inkubieren der Probe für 2 min bei RT.
- In 200 ul Puffer AL auf die Probe und mischen gründlich durch Vortexen. Als nächstes wurden 200 ul 100% Ethanol hinzufügen und gründlich durch Vortexen mischen. Übertragen Sie die gesamte Probe zur Verfügung gestellt Spin-Säule, in einem 2 ml Sammelröhrchen und Zentrifuge für 1 min bei ≥8,000 x g.
- Entsorgen Sie das Sammelrohr mit derDurchfluss- und legen Sie die Spin-Säule in ein neues 2 ml Sammelröhrchen. In 700 & mgr; l Puffer AW1 auf die Spinsäule, Zentrifuge für 15 Sekunden bei ≥8,000 xg, entsorgen Sie die Flow-Through und Wiederverwendung der Sammelröhrchen.
- In 700 & mgr; l Puffer AW2 auf die Spinsäule, Zentrifuge für 15 Sekunden bei ≥8,000 xg, entsorgen Sie die Flow-Through und Wiederverwendung der Sammelröhrchen. Als nächstes wird bei ≥8,000 xg 700 ul 100% Ethanol auf die spin-Säule zentrifugieren für 15 Sekunden hinzuzufügen und das Sammelrohr mit dem Durchlauf verwerfen.
- Legen Sie die Spin-Säule in ein neues 2 ml Sammelröhrchen, öffnen Sie den Deckel der Spin-Säule und Zentrifuge bei voller Geschwindigkeit für 5 min. Entsorgen Sie das Sammelrohr mit dem Flow-Through.
- Legen Sie die Spin-Säule in ein neues 1,5 ml Sammelröhrchen und fügen Sie 25 ul erhitzten Nuklease-freies Wasser (50 ° C). Inkubieren Säule und Rohr bei 50 ° C für 10 min. Zentrifuge für 1 min bei maximaler Geschwindigkeit, fügen Sie 25 ul Nuklease-freies Wasser (RT) zu Säule und Inkubation für1 min bei RT.
- Zentrifuge für 1 min bei Maximalgeschwindigkeit (21.130 xg) und Erntedurchfluss enthaltend genomische DNA (etwa 50 & mgr; l DNA insgesamt). Lagern Sie die Säule bei -20 ° C (im Fall, dass ein weiterer Elution erforderlich war später).
Ergebnisse
Dieses Protokoll stellt ein optimiertes Verfahren für die DNA und RNA aus Gewebekerne Rückgewinnung unter Verwendung von Änderungen eines handelsüblichen Extraktionssystem für Gewebeschnitten ausgelegt. Optimierung umfassten die Einführung von Gewebehomogenisierung einem Einsatz von mehr potent Proteinase K für die DNA-Extraktion und Erweiterung der Gewebeaufschlusszeit. Grafiken und statistische Analysen inklusive 2-Wege-ANOVA, lineare Regression und Korrelation.
Optimierungen von Proteinase Verdauung
Die kommerzielle Kit enthalten eine bei Raumtemperatur stabile Proteinase K - Lösung , die mit einer potenteren Proteinase K ersetzt wurde, was zu einer höheren Ausbeute DNA (2A). Um die DNA-Ausbeuten zu erhöhen, Verdau wurde 2-24 h verlängert. Keine signifikanten Unterschiede wurden zwischen den zwei Zeitpunkten zu sehen ist, aber die 24-Stunden-Verdau erschien konsistentere Ausbeuten über sa bereitzustellenmples. Allerdings sind weitere Inkubation bis 48 Stunden nicht weiter DNA - Ausbeute verbessert hat (2B; p = 0,74).
Typische DNA-Ausbeute von FFPE Prostatakrebs-Gewebeproben
Unter Verwendung des optimierten Protokolls, RNA und DNA wurden mitextrahierten von 333 Prostatakrebs FFPET Proben von 3 bis 14 Jahren im Probenalter. Von jeder Probe, 3 Gewebekerne (durchschnittliche Gesamtgewebevolumen von 0,95 ± 0,13 mm 3) wurden als Eingabe verwendet. Während es andere mikrofluidische basierte Gel-Elektrophorese Verfahren sind die Konzentrationen und liefern Auswertungen der Größenverteilung von Nukleinsäuren Moleküle abschätzen kann, solche Verfahren bieten keine reproduzierbare Quantifizierung von Nukleinsäuren und zwischen RNA und DNA nicht unterscheiden kann flourometrically basierte Assays tun. 22 Und weil mikrofluidischen basierte Gel-Elektrophorese Ergebnisse für fragmentierte nicht zuverlässigNukleinsäuren , die aus FFPET, 23 Nukleinsäure-Ausbeuten wurden gemessen fluorometrisch (Reagenzienliste für Details). Die durchschnittliche Ausbeute betrug 2,270 ng von RNA und 820 ng DNA (Figur 3A). Etwa 90% aller FFPET Proben in dieser Studie analysiert ergab ≥100 ng DNA und ≥ 500 ng RNA. Interessanterweise gab es keine signifikante Korrelation zwischen dem Alter der FFPET Probe und Nukleinsäurerückgewinnung (3B). Gesamt - RNA und DNA - Ausbeuten wurden auf Proben korreliert (R 2 = 0,39; p <0,0001), obwohl mehr als doppelt so viel RNA als DNA aus jeder Probe gewonnen wurde (3C).
Da der Pilot und Optimierungsarbeiten an Prostatagewebe durchgeführt wurde, war der nächste Schritt die Leistung dieses Protokoll auf ein paar zusätzliche Arten von Archivierungs Gewebe zu untersuchen. sein Beginnend mit chirurgisch entfernt und Autopsie FFPET Proben mitnign Leber (1 Probe 1 Fall), Krebserkrankungen des Gehirns (8 Proben von 1 Fall), der Harnblase (2 Proben aus 2 Fälle) und Brust (3 Proben von 3 Fälle), ergab das Protokoll> 100 ng DNA und RNA von 90% der Proben (Figur 3D). Während Nukleinsäure Ausbeuten in der Autopsie Gewebe niedriger waren als in der chirurgischen Gewebe, zeigen repräsentative Ergebnisse, dass das Protokoll ähnlichen Ausbeuten über Krebserkrankungen von verschiedenen Standorten abgeleitet produziert.
Bewertung von RNA und DNA-Integrität und ihre Vertreter Leistung in Downstream-Analyse
RNA-Expressionsanalyse von 47 Genen in 8 ausgewählt FFPET Prostatakrebsproben und einem frischen PC-3-Prostatakrebs-Zelllinie Probe (als positive Kontrolle) wurde unter Verwendung eines kommerziellen Multianalyt Genexpressionsplattform durchgeführt, die für FFPET optimiert ist. Die mRNA-Zählungen in PC3 waren in der Regel höher als die von FFPET samples (4A). Allerdings relativen Expression aller Gene verglichen, FFPET Prostatakrebs Proben zeigten ähnliche Expressionsprofile zu PC-3-RNA, was darauf hinweist, dass beide Quellen von RNA geeignet sind für die RNA-Expressionsprofilen.
Um die Leistung der genomischen DNA mit diesem Protokoll, Bisulfit-umgewandelte DNA Extrakte aus FFPET Proben wurden amplifiziert durch methylierungsspezifische PCR (MSP). 24 MSP - Analyse von ALU repetitiven Elementen, hoch methylierte Regionen in Millionen von Kopien im menschlichen Genom extrahiert demonstrieren, 25 wurde als genomische Methylierungskontrolle verwendet, und erwartet , dass minimale Unterschiede zwischen den Proben zu zeigen. Wie in 4B gezeigt, war es zu keiner Variation wenig zwischen verschiedenen Proben in ALU MSP Methylierungsspiegel gesehen. Ferner MSP - Assays auf Basis von GSTP1, ein Gen bekannt bei Prostatakrebs hypermethyliert zu werden , aber nicht in gutartigen Proben zeigten 26 keine nachweisbare Amplifikationen in DNA aus gutartigen Proben. Wie erwartet, niedrigere Werte qPCR cycle threshold wurden in DNA aus Krebsgewebe nachgewiesen, Anreicherung von methylierten GSTP1 Kopien angibt. Die Nützlichkeit von Nukleinsäuren durch dieses Protokoll gewonnenen Säuren wurde weiter in typischen Downstream-Tests getestet, Nukleinsäuren aus gutartigen Leber und von einem Gehirn (post-mortem) und von zwei operativ entfernt Brustkrebsproben gewonnen werden. Beide RT-qPCR basierenden Expressions und MSP - Assays durchgeführt und auf Brustkrebs und Leber FFPET, aber die RT-PCR - Test konnte eine stark exprimiert mRNA aus dem Post-mortem - Gehirntumorprobe (4C) zu verstärken, was darauf hindeutet , dass die RNA abgebaut hatte, wahrscheinlich aufgrund verzögerter Gewebefixierung.
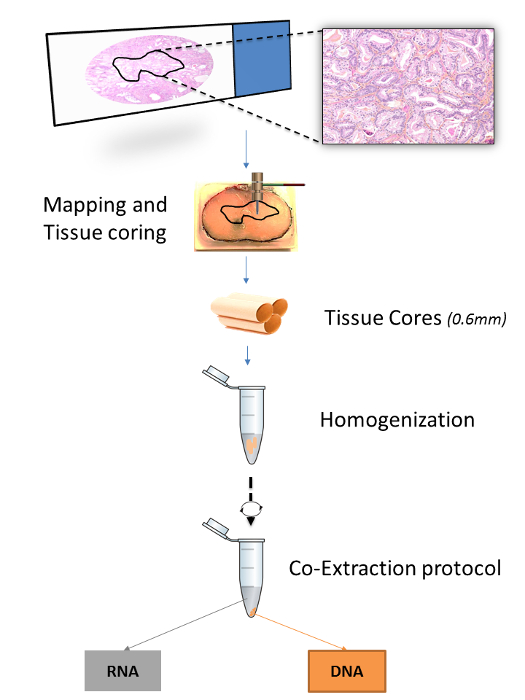
Abb . 1: Übersicht über die Extraktionsverfahren für FFPET Proben Die Abbildung zeigt , wie ein Gebiet von Interesse in einGewebeblock basiert auf histopathologische Auswahl aus einem Objektträger abgebildet. Drei 0,6 - mm - Gewebekerne werden dann von jedem Gewebebereich erhalten durch Biopsieausstanzungen mit, homogenisiert zusammen und dann zur Extraktion von sowohl RNA als auch DNA unterzogen. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
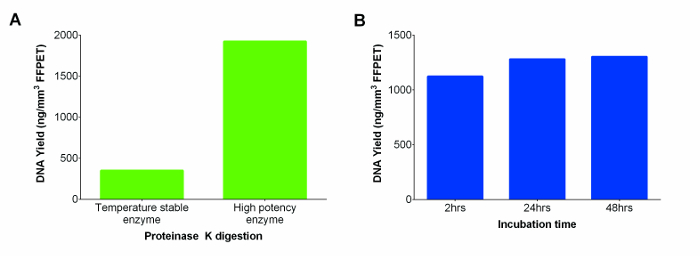
Abbildung 2: Nukleinsäuren (DNA und RNA) Ausbeuten in ng / mm 3 von FFPET aus zwei Proteinase K (Temperatur stabil und hochwirksamer Enzyme), und in einer Reihe von Inkubationszeiten für die Letzten Getestet (A) Leistung von Proteinase K. von verschiedenen Lieferanten. DNA-Extraktionen wurden auf einer repräsentativen FFPET Probe durchgeführt stabile Enzym Temperatur mit dem k geliefertes im Vergleich zu einem stärker Enzym von einem anderen Hersteller. (B) Bestimmung optimaler Proteinase K Inkubationszeit die DNA - Ausbeute zu maximieren. Leistung der hohen Konzentration Proteinase K Verdau wurde bei drei verschiedenen Inkubationszeiten ausgewertet unter Verwendung von 3 FFPET Proben. Die Fehlerbalken stellen die Standardabweichung vom Mittelwert (SEM). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
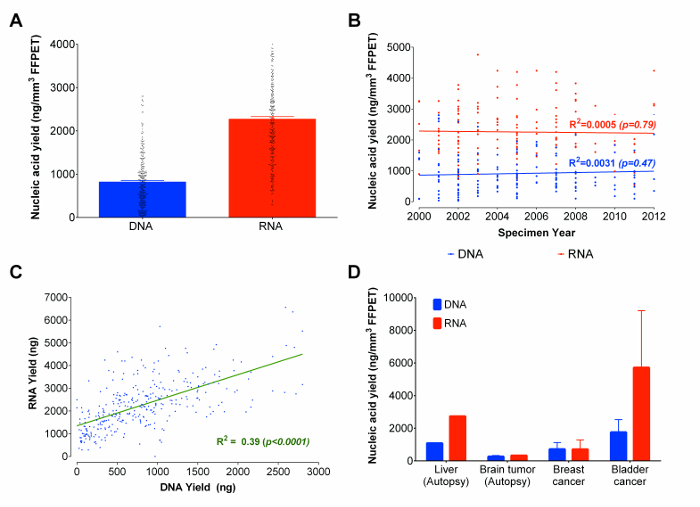
Abbildung 3:. Nukleinsäuren (DNA und RNA) Ausbeuten in ng / mm 3 von FFPET insgesamt über Proben Jahre und Vertreter Gewebetypen (A) Gesamt gewonnenen Nukleinsäuren aus Formalin fixierten und in Paraffin eingebetteten Gewebeproben. Die Nukleinsäuren Mengen vorgelegt werden auf der Gewinnung von 333 FFPET Proben basierend usingen das optimierte Protokoll. (B) Korrelationsplot zwischen gewonnenen Gesamt - DNA und RNA und das Alter der FFPET Proben. Die extrahierten FFPET Proben wurden verwendet , um aus den Jahren 2000 bis 2012 erhalten wird (C) Korrelation zwischen Ausbeuten aus gleichzeitig extrahierten DNA und RNA aus 333 Prostataproben. Es besteht eine positive Korrelation zwischen der DNA und RNA-Ausbeuten. (D) Demonstration des Protokolls zusätzliche Archivgewebetypen verwenden. Das optimierte Protokoll wurde verwendet, um Nukleinsäuren aus 14 Krebs (Brust-, Blasen- und Gehirn) und normal (Leber) Proben zu extrahieren. Die Fehlerbalken stellen SEM. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
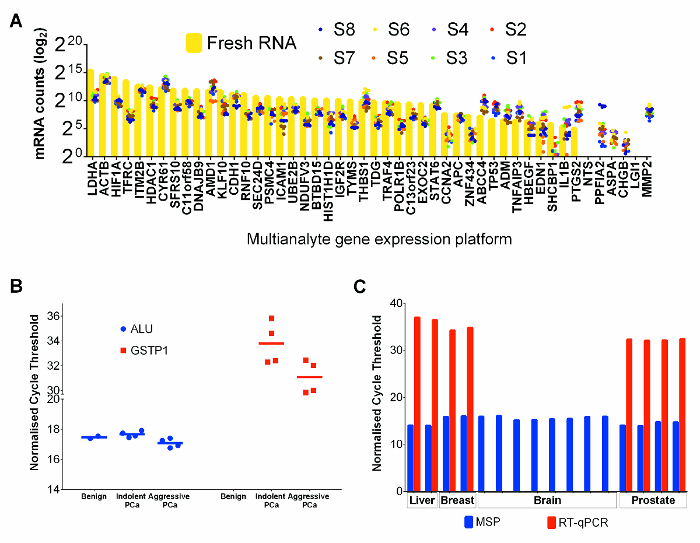
Abbildung 4: Leistung der RNA und DNA-Co , extrahiert aus TissuE - Kerne in Downstream - Anwendungen. (A) mRNA zählt für Krebsgewebe FFPE Prostata und für frischen PC-3 - Zelllinie Kontrolle. Jeder Punkt stellt den Durchschnitt von 3 technische Replikate separat extrahiert. Frische PC-3-Zelllinie RNA-Werte dargestellt werden durch gelbe Balken und FFPET Gewebe Werte sind durch farbige Punkte dargestellt. (B) methylierungsspezifische PCR - Assays auf die DNA von FFPET Prostatakrebsproben. unter Verwendung von 50 ng / Reaktion von Bisulfit-konvertierter DNA wie erwartet durchgeführt Zyklus Schwellenwerte wurden 10 Proben erhalten. ALU MSP - Assays von allen FFPET Proben hatten ähnliche Zyklus Schwellenwerte (p> 0,67). GSTP1 MSP-Assays zeigten höhere Methylierung (untere cycle threshold) -Spiegel bei Prostatakrebs als in gutartigen Prostata. (C) Bewertung der DNA- und RNA - Qualität von zusätzlichen Gewebetypen. HPRT1 Genexpression und Alu-Methylierung Tests wurden auf Nukleinsäuren durchgeführt (RNA und DNA jeweils) extrahiert aus nochmal Leber (Autopsie) und Gehirn (Autopsie) und Brustkrebs (alle FFPET). Ergebnisse von Prostata sind zum Vergleich gezeigt. Hinweis: ähnliche Ergebnisse wurden von jedem Gewebetyp beobachtet, mit Ausnahme für gescheiterte mRNA-Amplifikation aus einer Autopsie Probe. Jeder Punkt oder Balken steht für eine Probe und Fehlerbalken SEM dar. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
| Tissue Cores | |||||||||||
| Tissue Input (mm3) | Extrahierte Nukleinsäure | Bestätigung | Qualitätskontrolle: DNA | Qualitätskontrolle: RNA | |||||||
| (Anzahl der Proben) | Alter der Probe (Jahre) | Gesamtertrag (ng) | Fragmentgröße (bp) | PCR | Gesamtertrag (ng) | Fragmentgröße (bp) | PCR | nano | |||
| Pikor et al. | 43-129 | DNA | Keine Daten | Keine Daten | Keine Daten | Keine Daten | |||||
| Montaser-Kouhsari et al. | 18-29,5 | RNA | 763 | 0-25 | 843 | Keine Daten | |||||
| Dieses Papier | 1,71 | DNA und RNA | > 350 | 3-12 | 820 | 100-500 |  | 2270 | 100-500 |  | /ftp_upload/54299/check_mark.jpg "/> |
| Gewebeschnitte | |||||||||||
| Tissue-Eingang | Extrahierte Nukleinsäure | Bestätigung | Qualitätskontrolle: DNA | Qualitätskontrolle: RNA | |||||||
| (Anzahl der Proben) | Alter der Probe (Jahre) | Gesamtertrag (ng) | Fragmentgröße (bp) | PCR | Gesamtertrag (ng) | Fragmentgröße (bp) | PCR | nano | |||
| Heikal et al. | 5 x 5 um | DNA | 12 | 22.07 | 88-300 | 103-351 |  | ||||
| Chung et al. | 1 x 20 & mgr; m | RNA | 9 | > 5 | 16,000- 23,000 | 100-200 |  | ||||
| Antica et al. | 2 x 4 um | RNA | 18 | Keine Daten | Unknown (621 ng / ul) | 80-202 + |  | ||||
| Ghatak et al. | 5 x 5 um | DNA und RNA | 5 | 1 | 14 256 | <1030 |  | 16 000 | 109-400 + |  | |
| Hennig et al. | 1 x 10 & mgr; m | DNA und RNA | 210 | 1-25 | Keine Daten | Keine Daten |  | Keine Daten | Keine Daten |  | |
| Laser Mikrodissektion | |||||||||||
| Tissue-Eingang (mm2) | Extrahierte Nukleinsäure | Bestätigung | Qualitätskontrolle: DNA | Qualitätskontrolle: RNA | |||||||
| (Anzahl der Proben) | Alter der Probe (Jahre) | Gesamtertrag (ng) | Fragmentgröße (bp) | PCR | Gesamtertrag (ng) | Fragmentgröße (bp) | PCR | nano | |||
| Snow et al. | 1-2 | DNA | 110 | 0-2 | 430 | Keine Daten |  | ||||
Tabelle 1:. Ein Vergleich der veröffentlichten DNA und RNA - Extraktion Protokolle für die Gewebekerne, Schnitte und Laser Capture Mikrodissektionen Ebenfalls enthalten sind mehrere molekulare Endpunkte Bewertung dieser Methoden unter Verwendung von PCR und nano Assays.
Diskussion
Für eine erfolgreiche Extraktion von DNA und RNA aus Geweberegionen von Interesse ist eine genaue Entkernungs kritisch. Dieses Protokoll beschreibt die Verwendung einer Gewebestanze 0,6 mm Durchmesser Kerne zu isolieren und beschreibt den Prozess für Einträge von Mikroskop übertragen Folien FFPET Blöcken entsprechen. Modifikationen des Protokolls des Herstellers waren erforderlich, um effizient Nukleinsäuren aus Kernen extrahiert, die ungefähr 50-mal dicker als die Mikrotomschnitte, für die das Protokoll bestimmt wurde. Da die Kerne mehr Paraffinwachs in Bezug auf Gewebeschnitten, wirksame Entparaffinierung von Kernen durch wiederholte Xylol und Ethanol Behandlungsschritte enthalten waren erforderlich. Der Erfolg der Post-Entparaffinierung Schritte abhängig von der richtigen mechanischen Gewebekerne Homogenisierung und effiziente Proteinase K Verdauung. Eine weitere Optimierung der Proteinase K Verdau durchgeführt werden.
Es ist erwähnenswert, dass dieses Verfahren identiFIE Bereiche auf der Oberfläche des Blocks von Interesse, wie in der entsprechenden Histopathologie Folien identifiziert. Als Kern Ernten Gewebe, das 3 oder 4 mm tief, Nutzer dieses Protokoll kann sein besorgt sein, was unter der Blockoberfläche legen Zellen oder Gewebe. Dies ist zwar ein berechtigtes Anliegen ist, haben mehrere Studien (besprochen in Bezug 27) gezeigt , dass Gewebe Kerne getreulich die histologischen und molekularen Eigenschaften von pathologischer Gewebeblöcke darstellen, insbesondere wenn doppelte oder dreifache Kerne aus dem Bereich von Interesse abgetastet werden.
Als modifiziertes commercial in diesem Protokoll erlassen Extraktionskit gleichzeitige Extraktion von sowohl DNA als auch RNA, die aus dem gleichen Gewebe ermöglicht, speichert das Protokoll wertvolle biologische Material und ermöglicht einen direkten Vergleich zwischen den beiden resultierenden Nukleinsäuren aus derselben Probe. Die gleichzeitige Extraktion von RNA und DNA schneidet sich Arbeit und Gewebeabbau um die Hälfte, und ermöglicht eine präzise integrierte Analyse von Gen-ausdrITZUNG sowie epigenetischen und genetischen DNA-Merkmale aufweisen. Da die Erträge sowohl RNA und DNA aus diesen repräsentativen Gewebekerne übersteigen in der Regel 600 bis 300 ng, bzw., und da die meisten aktuellen PCR und Sequenzierung der nächsten Generation typischerweise Anwendungen 10-100 ng erfordern, die meisten Proben durch dieses Protokoll gereinigt sollte ausreichend liefern Material für mehrere Downstream-Assays. Dieses Protokoll wurde in unabhängigen Labors (Selvarajah et al., In Vorb.) Reproduzierbar erwiesen. RNA aus diesem Protokoll war von ausreichender Qualität zur Analyse der Genexpression entweder RT-PCR oder eine populäre Multianalyt-Plattform und DNA durchgeführt gut in Methylierung spezifischer PCR-Assays verwendet. Zukünftige Studien darauf abzielen, die Nützlichkeit der gewonnenen Nukleinsäuren in Sequenzierung der nächsten Generation sind die Beurteilung gerechtfertigt.
So wurden mehrere Modifikationen an einem handelsüblichen Protokoll hergestellt, für dünne Abschnitte FFPET ausgelegt, wodurch es geeignet for die Co-Extraktion von RNA und DNA von 0,6 mm FFPET Kerne. Das Protokoll zeigte durchweg hohen Ausbeuten in einer großen Kohorte von Prostatakrebs Proben und in einer begrenzten Anzahl von Proben von Krebserkrankungen der Brust, Gehirn und Blase. Insgesamt sollte das Protokoll Benutzern ermöglichen, Analysen von großen, gut kommentierten Gewebesammlungen durchzuführen gezielte Gen-basiert. Wichtig ist, ermöglicht das Protokoll effiziente fokussierte Probenahme von Regionen von Interesse in FFPET, relativ wenig Hands-on-Zeit, und hoch genug Erträge bei den meisten Downstream-Anwendungen.
Offenlegungen
The authors declare no competing financial interests.
Danksagungen
This research was supported by a team grant from Movember/Prostate Cancer Canada to JMSB, DMB, PCP, and JL, and by the Ontario Institute of Cancer Research (JMSB, DMB, and PCP) and Motorcycle Ride for Dad Kingston/University Hospitals Kingston Foundation/Kingston General Hospital (DMB, PCP).
Materialien
| Name | Company | Catalog Number | Comments |
| Plastic paraffin film, "Parafilm 'M'" | Bemis | RK-06720-40 | Any generic paraffin film will work as a substitute |
| Sodium Hypochlorite, "Ultra Bleach" | Likewise | 53-2879-2 | Any generic bleach will work as a substitute. Hazardous material that can cause burns on contact. |
| Molecular biology grade absolute ethanol | Fisher BioReagents | BP2818-500 | Sigma-Aldrich E7023 suffices as a substitute |
| Molecular Grade H2O | G-Biosciences | 786-293 | Sigma W4502 suffices, as well as any other brand of molecular grade H2O |
| 0.6 mm Punch Set for Beecher Instruments | Estigen | MPO6[Yellow] | Make sure to use the red receiver punch from the set |
| Fine point permanent marker | Sharpie | 10365796S | Using the marker on FFPE tissues causes it to dry out quickly, so several may be required |
| FFPE tissue block | |||
| Stained tissue slide corresponding to FFPE block | |||
| 1.5 ml Micro-Centrifuge Tubes | Fisher BioReagents | 05-408-137 | |
| 2.0 ml Low binding tubes (LoBind Micro-Centrifuge Tubes) | Eppendorf | 22431048 | |
| 1.5 ml Low binding tubes (LoBind Micro-Centrifuge Tubes) | Eppendorf | 22431021 | |
| Histology Xylene | VWR | CA 95057-822 | Fisher Scientific X5-500 suffices as a substitute |
| Molecular Biology Grade 2-Propanol | Sigma | I9516 | |
| AllPrep FFPE DNA/RNA Kit | Qiagen | 80234 | Prepare buffers accodring to the AllPrep DNA/RNA FFPE Handbook21 |
| Buffers: RLT, FRN, RPE, ATL, AL, AW1, AW2, DNaseI solution | Qiagen | 80234 | Prepare buffers accodring to the AllPrep DNA/RNA FFPE Handbook21 |
| Temperature stable proteinase K | Qiagen | 80234 | |
| High potency proteinase K | Invitrogen | 25530-049 | Invitrogen 25530-015 suffices as a substitute |
| RNAse neutralizing solution (Rnase AWAY) | Molecular BioProducts | 7003 | |
| RNaseA 100 mg/ml | Qiagen | 19101 | |
| BD Integra Syringe 3 ml 21G x 1/2 | BD | 305274 | |
| Motorized tissue homogenizer (TissueRuptor) | Qiagen | 9001271 | Fisher Scientific 14-261-29 suffices as a substitute |
| -20 °C and -80 °C Laboratory Freezer | |||
| Micro-Centrifuge with rotor for 2 ml tubes | |||
| Digital Vortex Mixer | |||
| Pipettes and filter tips | |||
| Heating blocks or water baths | |||
| Tris Hydrochloride | Amresco | 0234 | |
| Sodium Chloride | Amresco | 0241 | |
| Anhydrous Magnesium Chloride | Sigma | M8266 | |
| Sodium Dodecyl Sulfate | Sigma | L4509 | |
| Acrodisc 25 mm syring filters with 0.45 µm Supor membrane | Pall | PN 4614 | |
| Syringe with retracting BD PrecisionGlide needle 3 ml | BD Integra | 305274 | |
| Hydrochloric Acid | BDH | 3026 | |
| Multianalyte gene expression platfrom (nCounter ® CAE codeset and Nanostring nCounter platform) | Nanostring nCounter platform, Nanostring | ||
| Fluorometric nucleic acid quantification (Qubit dsDNA HS Assay Kit and Qubit® RNA BR Assay Kit) | Invitrogen |
Referenzen
- Kern, S. E. Why your new cancer biomarker may never work: recurrent patterns and remarkable diversity in biomarker failures. Cancer Res. 72 (23), 6097-6101 (2012).
- Klopfleisch, R., Weiss, A. T. A., Gruber, A. D. Excavation of a buried treasure--DNA, mRNA, miRNA and protein analysis in formalin fixed, paraffin embedded tissues. Histol. Histopathol. 26 (6), 797-810 (2011).
- Beltran, H., et al. Targeted Next-generation Sequencing of Advanced Prostate Cancer Identifies Potential Therapeutic Targets and Disease Heterogeneity. Eur. Urol. 63 (5), 920-926 (2013).
- Hoppin, J. A., Tolbert, P. E., Taylor, J. A., Schroeder, J. C., Holly, E. A. Potential for selection bias with tumor tissue retrieval in molecular epidemiology studies. Ann. Epidemiol. 12 (1), 1-6 (2002).
- von Ahlfen, S., Missel, A., Bendrat, K., Schlumpberger, M. Determinants of RNA quality from FFPE samples. PLOS ONE. 2 (12), e1261 (2007).
- Masuda, N., Ohnishi, T., Kawamoto, S., Monden, M., Okubo, K. Analysis of chemical modification of RNA from formalin-fixed samples and optimization of molecular biology applications for such samples. Nucleic Acids Res. 27 (22), 4436-4443 (1999).
- Pikor, L. A., Enfield, K. S. S., Cameron, H., Lam, W. L. DNA extraction from paraffin embedded material for genetic and epigenetic analyses. J. Vis. Exp. (49), (2011).
- Turashvili, G., et al. Nucleic acid quantity and quality from paraffin blocks: defining optimal fixation, processing and DNA/RNA extraction techniques. Exp. Mol. Pathol. 92 (1), 33-43 (2012).
- Espina, V., et al. Laser-capture microdissection. Nat. Protoc. 1 (2), 586-603 (2006).
- Hackler, L., Masuda, T., Oliver, V. F., Merbs, S. L., Zack, D. J. Use of laser capture microdissection for analysis of retinal mRNA/miRNA expression and DNA methylation. Methods Mol. Biol. 884, 289-304 (2012).
- Bonin, S., Stanta, G. Nucleic acid extraction methods from fixed and paraffin-embedded tissues in cancer diagnostics. Expert Rev. Mol. Diagn. 13 (3), 271-282 (2013).
- Bonin, S., et al. Multicentre validation study of nucleic acids extraction from FFPE tissues. Virchows Archiv. 457 (3), 309-317 (2010).
- Montaser-Kouhsari, L., et al. Image-guided Coring for Large-scale Studies in Molecular Pathology. Appl. Immunohistochem. Mol. Morphol. , (2015).
- van Eijk, R., Stevens, L., Morreau, H., van Wezel, T. Assessment of a fully automated high-throughput DNA extraction method from formalin-fixed, paraffin-embedded tissue for KRAS, and BRAF somatic mutation analysis. Exp. Mol. Pathol. 94 (1), 121-125 (2013).
- Ghatak, S., Sanga, Z., Pautu, J. L., Kumar, N. S. Coextraction and PCR Based Analysis of Nucleic Acids From Formalin-Fixed Paraffin-Embedded Specimens. J. Clin. Lab. Anal. , (2014).
- Hennig, G., et al. Automated extraction of DNA and RNA from a single formalin-fixed paraffin-embedded tissue section for analysis of both single-nucleotide polymorphisms and mRNA expression. Clin. Chem. 56 (12), 1845-1853 (2010).
- Snow, A. N., Stence, A. A., Pruessner, J. A., Bossler, A. D., Ma, D. A simple and cost-effective method of DNA extraction from small formalin-fixed paraffin-embedded tissue for molecular oncologic testing. BMC Clin. Pathol. 14 (1), 30 (2014).
- Torrente, M. C., et al. DNA extraction from formalin-fixed laryngeal biopsies: Comparison of techniques. Acta Otolaryngol. 131 (3), 330-333 (2011).
- Okello, J. B. A., et al. Comparison of methods in the recovery of nucleic acids from archival formalin-fixed paraffin-embedded autopsy tissues. Anal. Bochem. 400 (1), 110-117 (2010).
- Abramovitz, M., et al. Optimization of RNA extraction from FFPE tissues for expression profiling in the DASL assay. BioTechniques. 44 (3), 417-423 (2008).
- . . AllPrep DNA/RNA FFPE Handbook. , (2012).
- Laurent, L. C., et al. Meeting report: discussions and preliminary findings on extracellular RNA measurement methods from laboratories in the NIH Extracellular RNA Communication Consortium. Journal of Extracell. Vesicles. 4, (2015).
- . Methods of RNA Quality Assessment Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment (2012)
- Herman, J. G., Graff, J. R., Myohanen, S., Nelkin, B. D., Baylin, S. B. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc. Natl. Acad. Sci. U.S.A. 93 (18), 9821-9826 (1996).
- Weisenberger, D. J., Campan, M., et al. Analysis of repetitive element DNA methylation by MethyLight. Nucleic acids research. 33 (21), 6823-6836 (2005).
- Yegnasubramanian, S. Hypermethylation of CpG Islands in Primary and Metastatic Human Prostate Cancer. Cancer Res. 64 (6), 1975-1986 (2004).
- Parsons, M., Grabsch, H. How to make tissue microarrays. Diagn. Histopathol. 15 (3), 142-150 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten