Method Article
Nanopore רצפי DNA לניתוח Metagenomic אדמה
In This Article
Summary
Nanopore טכנולוגיה עבור רצפי מולקולות יש אפליקציות רחב במדעי החיים, כולל זיהוי של פתוגנים, ניטור בטיחות מזון, אנליזה גנומית, ניטור סביבתי metagenomic אפיון בקטריאלי עמידות לאנטיביוטיקה. במאמר זה, הוכח נוהל metagenomic אדמה רצפי DNA לצורך זיהוי מינים בטכנולוגיה רצף nanopore.
Abstract
מאמר זה מתאר את השלבים להקמת ספרייה דנ א אדמה, הכנה, השימוש התא זרימה nanopore, וניתוח של רצפי דנ א מזוהה באמצעות תוכנת מחשב. רצפי Nanopore DNA היא טכניקה גמיש המאפשר רצף הגנום מיקרוביאלי מהירה לזהות מינים בקטריאליים וויראליים, כדי לאפיין את זני חיידקים, וכדי לזהות מוטציות גנטיות המקנים עמידות לאנטיביוטיקה. היתרונות של nanopore רצף (NS) עבור מדעי החיים כוללים שלו מורכבות נמוכה, עלות מופחתת, ורצף מהיר בזמן אמת של דנ א גנומי מטוהרים, PCR amplicons, דגימות cDNA או RNA. NS הוא דוגמה "רצף strand" הכוללת רצף ה-DNA על ידי המנחה מולקולה בודדת DNA נטושים דרך nanopore זה מוכנס לתוך קרום פולימר סינתטי. הקרום יש זרם חשמלי להחילה אז בסיסי בודדות עוברים דרך nanopore הזרם החשמלי מופרת בדרגות על ידי היסודות נוקלאוטיד ארבע. הזיהוי של כל נוקלאוטיד מתרחשת על ידי גילוי של אפנון האופיינית של הזרם החשמלי על ידי היסודות השונים כפי שהם עוברים nanopore. מערכת NS מורכב כף יד, USB מופעל התקן נייד, תא זרימה חד פעמיות המכיל מגוון nanopore. המכשיר הנייד מחובר מחשב נייד סטנדרטי קורא ומתעדת את רצף ה-DNA באמצעות תוכנת מחשב.
Introduction
המטרה של הליך זה הוא להדגים את השלבים הדרושים עבור הכנה של ספריית ה-DNA סביבתיים רצף, ניצול של nanopore זרימה תא רצף התקן, וכדי לבצע אנליזה של רצפי דנ א שנוצר באמצעות מערכת תוכנה, המרכז הלאומי לביוטכנולוגיה מידע (NCBI) ושפור לזהות מינים מיקרוביאלית בקרקע. כיום, רוב פלטפורמות רצפי DNA דורשות השקעה גדולה ב מכשור מורכב, שאינו ריאלי בסביבות המסכן משאבים או ביישומים שדה והדרכה טכנית. פלטפורמת רצף (NS) nanopore מבטלת נושאים אלה עם העלות האפקטיבית, פשוט להשתמש ספריית הכנת פרוטוקול, ואת מכשיר נייד רצף ולנתח מגוון רחב של סוגים שונים של חומצות גרעין1,3. לנו יש שולבו פלטפורמת NS בכמה כיתות המעבדה לתלמידי תואר שני.
Nanopore טכנולוגיה עבור רצפי מולקולות הוכיחה יישומים רחב במדעי החיים, כולל זיהוי של פתוגנים בקטריאליים וויראליים1,2,6, המגוון הביולוגי סביבתיים מחקרים, ניטור בטיחות מזון, אנליזה גנומית3,5, ואפיון של חיידקים עמידות לאנטיביוטיקה4. NS היא שיטה מהירה ומדויקת על רצף חומצות גרעין המבוסס על העיקרון של "לנטישה רצף" על ידי גילוי הפרעות חשמל על ידי בסיסים נוקלאוטיד בודד כאשר יחיד DNA נטוש עוברת דרך nanopore מוכנס לתוך חשמלית ממברנות פולימר סינתטי. השלבים הכרוכים בהכנת DNA NS כוללות פיצול הגנום, ותיקון קצה 3' דה-עוקב של שברי גנומית, מתאם, הקשר חישול ל- DNA, טיהור ספריית DNA ולאחר טעינת הספריה לתוך המכשיר הסלולרי זרימה nanopore. פיצול של הגנום ~ 8 kb מידות מושגת על ידי צריך שתוציאו µg 1-2 של ה-DNA גנומי דרך צינור פיצול גרם-צינור. הקצוות המפוצלים גנומית מכן תוקן, זנב עם dA פוליפוני באמצעות ערכת זמינים מסחרית. רצפים במתאם יחיד נטושים, אשר תואמים עם החלבון מנוע nanopore, מתווספים לקצוות דנ א אשר משמשים כדי להנחות את רצף ה-DNA דרך nanopore (איור 1). רצפי הקשר נדרשים עבור טיהור DNA, לוקליזציה מולקולות DNA קרום הנקבובית. הסיכה נוצר על ידי נבדק מתאם סיכת ראש בקצה אחד של הספרייה זנב dA. המבנים מכבנה בקצוות ה-DNA מאפשר קריאה הגדילים antisense על תבונה כמו ה-DNA עובר nanopore (איור 2). בספרייה גנומית מוכן ואז מטוהרים מן התגובה באמצעות חרוזים streptavidin באמצעות שדה מגנטי, ואחריה טעינה הדגימה לתוך התא זרימת nanopore לניתוח.
ברצף הדנ א שקובעת איכות, קריאות רצף המקובלות לניתוח. ואז נתונים מספר כלים ביואינפורמטיקה לזהות חיידקים. הרצף "מתורגמים" לתוך FASTQ של תבנית FAST5. בפורמט FASTQ, רצפי ואז ניתן להשתמש בניתוח הפיצוץ.
Protocol
הערה: ה-DNA Metagenomic מטוהר מן האדמה (במחוז בולטימור, מרילנד) באמצעות בידוד גנומית של קרקע זמינים מסחרית קיט (ראה טבלה של חומרים). שימוש ספקטרופוטומטרים UV (ראה טבלה של חומרים), ה-DNA גנומי מטוהרים צריך להיות יחס (ננומטר) 260/280 > 1.8 ו- 260/230 יחס בין 2.0-2.2 להבטיח כי המדגם הינו ללא מזהמים. כמות ה-DNA גנומי הדרושות NS נע בין 200 ng כדי 2 µg.
1. שיטת הכנה מהירה ספריה (פרוטוקול קצר)
הערה: הוא זה פרוטוקול קצר. ראה טבלה של חומרים עבור ערכת רצף מהיר.
- ב- mL 0.2 צינור דק עם קירות (ראה טבלה של חומרים) להוסיף 200 ng של משקל מולקולרי גבוה ה-DNA הגמר נפח של 7.5 µL של מים מזוקקים.
- להוסיף 2.5 µL של פיצול מיקס (FM) ערכת הכנה מהירה לספריה, לערבב בעדינות על-ידי היפוך. הדופק צנטריפוגה ספין למטה (1,000 x g עבור 5 s).
- הכנס את הדגימה thermocycler (ראה טבלה של חומרים) עבור סיבוב אחד ב 30 ° C עבור 1 דקות ולאחריה 75 ° C עבור מינימלית 1 להסיר לצנטריפוגה צינור ודופק על תוצאה לדגימה.
- הוסף µL 1 של מתאם מהירה ו- 0.2 µL של בלאנט/ת א ליגאז מיקס מאסטר ולאחר מכן לאחסן בספריה מותאמת, קשור בקרח.
הערה: אם באמצעות פרוטוקול קצר והמשך לשלב 8
2. מצדו רצף פרוטוקול (פרוטוקול ארוך): Metagenomic DNA פיצול
הערה: ראה טבלה של חומרים עבור ערכת רצף מצדו.
- למהול 1 µg של דנ א גנומי מטוהרים נפח סופי של 46 µL במים יונים.
- העברת הדגימה כדי פיצול ה-DNA צינור (ראה טבלה של חומרים), צנטריפוגה עבור 1 דקות בטמפרטורת החדר ב 8000 g x באמצעות microcentrifuge (ראה טבלה של חומרים) כדי לייצר שברי גנומית ~ 8 kb. ודא כי כל הנוזל עבר לתוך הצינור אוסף.
- היפוך הצינור פיצול, לחזור צנטריפוגה ולאחר צנטריפוגה עבור 1 דקות לאסוף את הדנ א הגנומי מפוצלים אל החדר התחתון.
- להעביר את ה-DNA גנומי מפוצלים צינור 1.5 מ ל איגוד נמוך-DNA סטרילי, ולנתח חלק על ג'ל agarose אחוזים נמוכים (0.6%) כדי לאשר פיצול כדי > 30 kb.
3. מפוצלים הדנ א סוף-אופן ההכנה
- באמצעות 800 ng של דנ א גנומי מפוצלים ב 45 µL של מים יונים, להוסיף תגובה סוף-פרפ ' µL 7 ' מאגר (ראה טבלה של חומרים), 3 µL אנזימים מיקס (ראה טבלה של חומרים), ו- 5 µL של נוקלאז ללא מים.
- מיקס על ידי צנטריפוגה היפוך ודופק (1,000 x g עבור 5 s).
- להעביר מדגם צינור דק עם קירות 0.2 מ"ל, דגירה דוגמת עבור 5 דקות ב- 20 ° C, ואחריו דגירה-65 מעלות צלזיוס במשך 5 דקות.
- צנטריפוגה הדופק להביא תוכן לתחתית הצינור.
- העברה מדגם צינור 1.5 מ ל נמוכה-DNA איגוד microcentrifuge.
- להכין beads מגנטי (ראה טבלה של חומרים). להוסיף 60 µL של חרוזים resuspended התגובה הכנה סוף שלב 3.5, לערבב על-ידי pipetting.
- דגירה על מערבל מסובב ב 100 סל"ד (ראה טבלה של חומרים) עבור 5 דקות בטמפרטורת החדר.
- דופק centrifuge המדגם הצניפה החרוזים, ולאחר מכן למקם את הדגימה ליד מגנט.
- פיפטה ברגע החרוזים הם דבקו המגנט, תגובת שיקוע, להשליך.
- הסר הצינור החרוזים מגנט ושטוף עם 200 µL של אתנול 70% שזה עתה הוכנו על ידי בעדינות pipetting שיהיה המדגם.
- צנטריפוגה הדופק הצניפה החרוזים ולחזור המגנט.
- הסר את תגובת שיקוע וזורקים. חזור על השלב כביסה שוב באמצעות µL 200 של 70% אתנול.
- דופק centrifuge ברכבת התחתית, לחזור המגנט ולהסיר כל בכביסה אתנול 70%.
- עם המכסה צינור microcentrifuge פתוחה, לאפשר את החרוזים מהאוויר להתייבש במשך 5 דקות בטמפרטורת החדר.
- להסיר את הצינורית של מגנט, להשעות את צניפה ב µL 31 סטרילי, נטולת נוקלאז מים. תקופת דגירה של 2 דקות בטמפרטורת החדר.
- להחזיר את הצינור המגנט עד כל החרוזים יש מגורען, eluate ברור.
- להעביר את eluate צינור microcentrifuge 1.5 mL. שימוש µL 1 מדגם, לכמת את הדנ א. נכין קצה באמצעות ספקטרופוטומטרים UV ב-260 nm (ראה טבלה של חומרים).
הערה: אחוז ההחלמה צריך להיות ~ 70% (700 ng) של ריכוז ההתחלתי של µg 1 של הדנ א.
4. מתאם, בנוסף הקשר שברי DNA גנומי מוכנה סוף
- לערבב את כל ליגאז בלאנט/ת צינורות מיקס מאסטר על ידי היפוך ולאחר מכן דופק צנטריפוגה כדי להביא תוכן התחתון.
- באמצעות 30 µL של DNA מוכנה סוף, להוסיף 20 µL של מתאם שילוב, µL 50 של בלאנט/ת א מצדו מיקס מאסטר.
- לערבב על-ידי היפוך, הדופק צנטריפוגה, תקופת דגירה של 10 דקות בטמפרטורת החדר.
5. מגנטי Bלראש הכנה
- מערבולת החרוזים, ואז העברת µL 50 של חרוזים על תנאי כדי microfuge 1.5 mL צינור.
- מניחים את הצינורית על המגנט על גלולה החרוזים עד eluate.
- הסר את תגובת שיקוע וזורקים.
- להוסיף 100 µL חרוז איגוד מאגר (ראה טבלה של חומרים עבור ערכת חרוז) החרוזים, מערבולת כדי resuspend את החרוזים, גלולה החרוזים על המגנט, להסיר את תגובת שיקוע ולמחוק. חזור על השטיפה עם 100 µL מחייב המאגר.
- להוסיף 100 µL חרוז איגוד המאגר החרוזים שטף. מערבולת כדי resuspend את החרוזים.
6. ספריית טיהור
- שימוש micropipette P200, להוסיף µL 40 של חרוזים מהשלב הקודם גנומית הותאם/קשור לדנ א. מערבבים את הדגימה בקפידה על-ידי pipetting.
- דגירה המדגם על מערבל מסובב ב 100 סל"ד למשך 5 דקות בטמפרטורת החדר.
- שים המדגם על המגנט ולאפשר את החרוזים להתיישב. פיפטה הנחה תגובת שיקוע וזורקים.
- Resuspend את החרוזים ב 140 µL חרוז איגוד מאגר על-ידי pipetting. מקם את הדגימות המגנט, למחוק את תגובת שיקוע ולאחר לשטוף שוב עם עוד 140 µL חרוז איגוד מאגר.
- דופק centrifuge ברכבת התחתית, במקום את הצינורית על המגנט עבור 2 דק. להסיר את המאגר איגוד חרוז נותרת בגדר.
7. • תנאי של ספריה של חרוזי מגנט
- Resuspend את החרוזים מגנטי ב 15 µL • תנאי מאגר על-ידי pipetting. דגירה המדגם 10 דקות בטמפרטורת החדר.
- מקום ברכבת התחתית נגד המגנט הצניפה החרוזים.
הערה: אחוז ההחלמה צריך להיות ~ 25% (250 ng) של ריכוז ההתחלתי של µg 1 של הדנ א.
8. החל פקד בניהול/איכות
- להסיר את התא זרימה אריזה ולצרף את התא זרימה הרצפים נייד, בזמן אמת (ראה טבלה של חומרים).
- לצרף את הרצפים (sequencer) למחשב באמצעות כבל USB ולהתחיל את התוכנה רצף (ראה טבלה של חומרים).
- לחץ על "התחבר" התקן דרך התוכנה רצף, בחר "NC_Platform_QC.py" ולאחר לחץ על "התחל".
- בקרת איכות (QC) אורכת כ 6-7 דקות כדי להשלים; חפשו 'ים של ירוק' (שטחים גדולים של פלט) על הנתונים QC כדי לאשר כי ישנם מספיק נקבוביות פעילה (> 800) עבור רצפי DNA.
9. מתחיל רצף ריצה
- פתח את התוכנית רצף; תיבת דו-שיח תופיע. בתיבה מזהה לדוגמה, שם את הדגימה.
- לחץ על התיבה "זרימה Cell ID" והזן את הקוד נמצא על-גבי המדבקה על גג התא זרימה.
- פותחים את המכסה תא זרימה והחלק ואז את המכסה מדגם ימינה (בכיוון השעון) כך שגלוי הנמל הדגימה. ברגע היציאה פתוח, בדוק בועה קטנה. הסר microliters כמה המאגר כולל את הבועה.
- בדוק כדי לוודא כי יש מאגר על פני מערך חיישן. להפוך את המאגר לקרקע על ידי ערבוב 480 µL של הפעלת מאגר עם תערובת דלק (RBF-1, ראה את הטבלה של חומרים) (יש להקפיד לערבב ביסודיות הראשון) עם מים ללא נוקלאז µL 520.
- להוסיף µL 800 המאגר לקרקע הנמל לקרקע. בזהירות הרימו את המכסה "" כדי להפוך אותו לנגיש.
- לאחר 5 דקות, להוסיף µL 200 נוספים של מאגר לקרקע הנמל לקרקע.
10. טעינת הספריה
- לפני הכנת המקום בספריה של ריאגנטים הבאים על הקרח: RBF-1 (מתוך ערכת רצף), הותאם ו קשורה ספריה, ספריה טעינת חרוזים (LLB; הערכה רצף).
- כדי שפופרת צנטרפוגה 0.2 מ"ל, להוסיף µL 25.5 של RBF ו 12 µL של מים נטולי נוקלאז לשמור בטמפרטורת החדר.
- מערבבים את תואר ראשון במשפטים מאת pipetting שיהיה. להוסיף µL 26.5 של LLB הצינור 0.2 µL. מערבבים את ריאגנטים מאת pipetting שיהיה.
הערה: LLB נוטה הסדר מחוץ התערובת. - להוסיף µL 11 של הספריה מותאם, קשור הצינור 0.2 µL. לערבב על-ידי היפוך ו ספין על ידי צנטריפוגה דופק.
- הוסף µL 75 מדגם מהשלב 9.4 ליציאת ה-"" dropwise. ודא כי כל טיפה זורם לתוך הנמל לפני הוספת הבא.
- בזהירות להחליף את הדגימה המכסה כך bung מזין את היציאה, ולהחליף את המכסה התקן.
11. הפעלת קובץ Script פרוטוקול תוכנה רצף
- פתח את התפריט הנפתח תחת "בחר תוכנית" ובחר "רצף של 48 שעות."
- לחץ על "לבצע" כפתור כדי להפעיל את קובץ ה-script.
- בדקו את התוצאות של Mux לסרוק דיווח בזרם ההודעות עבור מספר הנקבוביות פעיל ב MUX לסרוק.
הערה: מספר זה עשוי להיות שונה מספר הנקבוביות ב- QC לרוץ, הבדל גדול עשוי להצביע על בעיה.- אם יש ירידה משמעותית של הנקבוביות פעיל רץ QC, להפעיל מחדש את התוכנה, אם עדיין יש הבדל משמעותי, לאתחל מחדש את המחשב.
- בדוק טמפרטורת הקירור היא 34 מעלות צלזיוס כפי שדווח על ידי סקריפט תוכנה רצף.
הערה: אם הטמפרטורה היא לא ב 34 ° C ואז לא יהיו מספיק נקבוביות פעיל לריצה. - בדוק ההיסטוגרמה באורכים קריאה כדי לקבוע אם המרחק רצף הצפוי ומתאימה הנבחנים.
- סגור את הסוכן בשולחן העבודה באמצעות 'סגור x.' להפסיק תוכנת עריכה ברצף על ידי סוגר את אינטרנט GUI. לנתק את המכשיר מהמחשב.
12. ניתוח של ריצה ותוצאות
הערה: במהלך רצף ההפעלה, הנתונים ניתן לנטר שימוש בתוכנית "תצוגה ח באמצעות הסוכן של שולחן העבודה. במהלך או לאחר ההפעלה הושלמה, קבצים זמינים ב FAST5 קובץ בפורמט ה ← נתונים קורא ← להעביר תיקיה (ברירת המחדל). אם תיקיה זו פתוחה כאשר רצף פעילה, המחשב עשוי להקפיא.
- כדי להציג ולטפל רצפים, להוריד "HDFViewer".
- כדי להציג את הקבצים רצף, ישירות פתוח HDF הצופה ← פתוח נתונים בתיקיה ← בחר שכל הקבצים סוגי ← בחר c: כונן (ברירת מחדל) ← בבחירת נתונים ← בחר קריאות ← בחר לעבור.
- בחר את הקובץ FASTQ בתפריט הנפתח השמאלי; קבצים יפתח, ניתן לשמור בפורמט FASTQ.
הערה: לצורך הניסוי התלמיד, רצפים של נוקלאוטידים 350 או יותר נבחרו לצורך ניתוח נוסף. פלט הטלפונים הבסיס מאפשר למשתמש למיין רצף פעולות הקריאה בהתאם לגודל. FASTQ קבצים ניתן לנתח באמצעות פיצוץ (NCBI) או תוכניות אחרות ביואינפורמטיקה.
תוצאות
הטכנולוגיה nanopore ועיצוב ניסיוני מספק שיטה מהירה וזולה עבור תלמידים על רצף אדמה דנ א. הפעל נציג עבר את הפרמטרים בקרת איכות עם יותר מ-800 נקבוביות זמין עבור רצף. הפעל כתוצאה מ 125,000 קריאות זמינה למחקר באורך רצף החציוני של 5.38 kb. רצפים מקבלים ניקוד האיכות, רק אלה רצפים עם ציונים מקובל מכן נותחו. כמו הפלט של הרצף התגובה הוא בתבנית FAST5, שאינה מקובלת על-ידי תוכניות כגון הפיצוץ (NCBI), רצפי נתפסו במציג HDF אשר ממירה את רצפי לתבנית FASTQ, אשר תואם לניתוח הפיצוץ. בעתיד חזרות של התוכנה, הנתונים יהיה זמין כתבנית FASTQ, ומבטלת את הצורך עבור הצופה HDF. לראות משלים המספרים 1, 2 ו- 3-
חמישים רצף נוקלאוטידים מעל 350 אורך נידונו ניתוח לפי BLASTN (נוקלאוטידים נגד נוקלאוטיד מסד הנתונים) או BLASTX (מתורגם חומצת אמינו לרצף נוקלאוטידים) (NCBI) כדי לזהות אורגניזמים במדגם קרקע. בהתחשב בתקופה אילוצים של המחלקה, כי המוקד של הקורס הוא על שיטות מעבדה, לא ביואינפורמטיקה, יש לנו את התלמידים לנתח את רצפי בתוך הפרמטרים האלה. מחלקות ביואינפורמטיקה שלנו יכול להשתמש בנתונים לפיתוח של כלי ניתוח צינור. רמה זו של ביואינפורמטיקה ניתוח אינו מכוסה בכיתה מבוסס מעבדה. טבלה 1 היא רשימה של האורגניזמים שזוהו עם הערכים e של פחות מ-4 e-04. באופן כללי, ערכי e-פחות מ- 4 e-04 מצביעים על דמיון חזק בין הרצף בהתאמה לרצף הקלט (שאילתת). האורגניזמים המזוהה על-ידי BLASTN (נגד מסד הנתונים לא יתיר (ע נ)) מתוך מגוון רחב של מינים, זמינים גנומית נוסף וניתוחים חלבון. דוגמה זו של האדמה, אשר באו גן במחוז בולטימור, MD היה מעולם לא נוסה, כך היו נתונים קודמים כדי לקבוע מה יהיה האורגניזמים בקרקע. BLASTN ניתוח (נגד מסד הנתונים nr) ניתוח גם ציין היו רצפי עם אין התאמה במסד הנתונים של נאט ו. כדי לקבוע אם אלו באמת הרומן רצפים, נדרש מחקר נוסף. ניתוח של מספר רצפי מאת BLASTX גילה חלבונים שונים של אורגניזמים אינו מיוצג בניתוח BLASTN. בטבלה 2 פירוט מספר חלבונים אפשרי כולל endopeptidases ו- ATPases כמו חלבונים. שימוש בספריה זו של רצפים, תלמידים יש ההזדמנות להשתמש בכלים לביואינפורמטיקה מתוחכמים יותר לבצע ניתוח נוסף, אם בבימויו של המדריך.
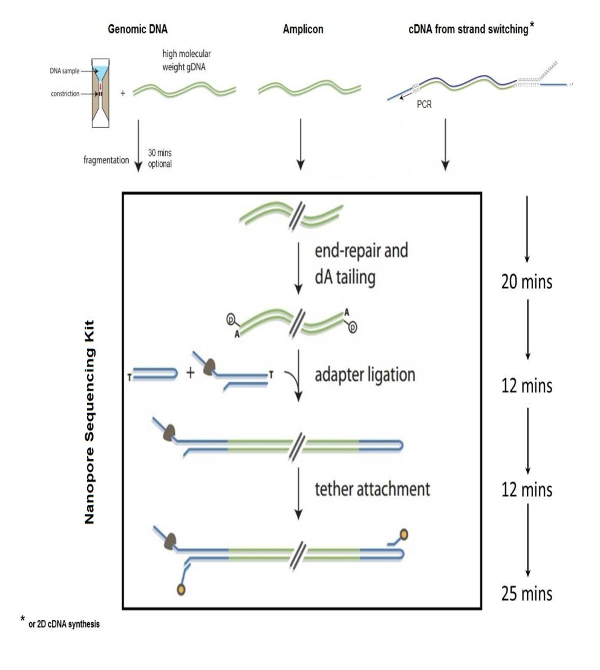
איור 1 : הכנה ספרייה גנומית. פעולות עבור הכנה של ספריית דנ א גנומי רצף באמצעות טכנולוגיית nanopore. איור זה השתנה עם ההרשאות הדרושות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2 . סכמטי של רצפי DNA באמצעות nanopore. DNA עובר דרך קרום nanopore בזיהוי הדור ובסיס האות החשמלי. איור זה השתנה עם ההרשאות הדרושות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| אורגניזם | ערך e |
| Streptomyces sp. | 3.00E-06 |
| Nocardoides sp. | 5.00E-04 |
| גורדוניה sp. | 5.00E-04 |
| Hyphomicrobium nitrativorens | 1.00E-139 |
| נובלה Starkeya | 1.00E-31 |
| Hyphomicrobium denitrificans | 1.00E-30 |
| Fibomicrobium sp. | 5.00E-17 |
| Pseudomonas sp. | 2.00E-15 |
| מרינוס Rhodothemus | 1.00E-17 |
| Turneiella parva | 2.00E-24 |
| E. coli | 0.00E + 00 |
| Sp מקבעי חנקן ופטריות. | 3.00E-30 |
| Gemmatirosa kalamazoonesis | 1.00E-20 |
| Burkholderia sp. | 8.00E-10 |
| Sphingomonas sp. | 8.00E-10 |
| Cellumonas sp. | 6.00E-11 |
טבלה 1: נבחר את התוצאות של ניתוח BLASTN. אדמה רצפי DNA metagenomics נתון BLASTN דמיון חיפוש במסד הנתונים של נוקלאוטיד יתירים NCBI. המידע המדווח להיות e-ערכי של 4x e-4 או פחות.
| חלבון | אורגניזם |
| חלבון היפותטי | חיידק Acidobacter |
| Methyltransferase תלויים סאם | ה denitrificans |
| ATPase | Jannaschia sp. |
| Endopeptidase | שיגלה sonnei |
| פירוק חלבונים | E. coli |
בטבלה 2: נבחר את התוצאות של ניתוח BLASTX. אדמה רצפי DNA metagenomics נתון BLASTX חיפוש דמיון נגד המאגר חלבון לא יתיר NCBI.
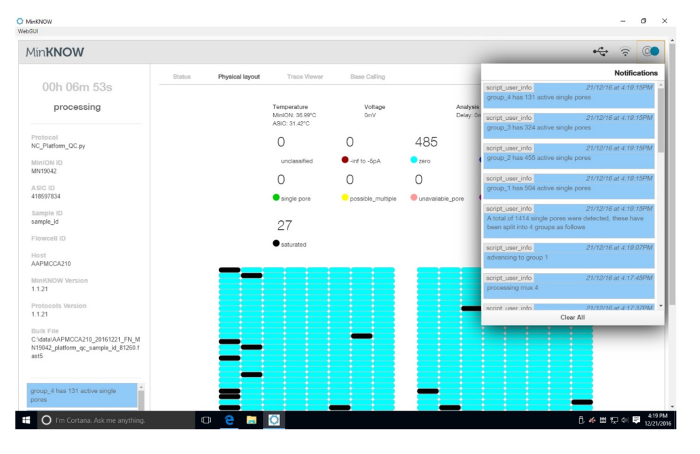
משלימה איור 1: התוצאה היא פלטפורמה QC. התוצאות של פלטפורמת QC מוצגים. בפינה הימנית העליונה הן התוצאות של QC של הנקבוביות פעיל. כל רביע של התא זרימה נבחנה על הנקבוביות פעיל. הדף הראשי הוא דינמי ומשתנה כמו כל רביע נבדק. בהפעלה זו, אותרו 1,414 נקבוביות יחיד.אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
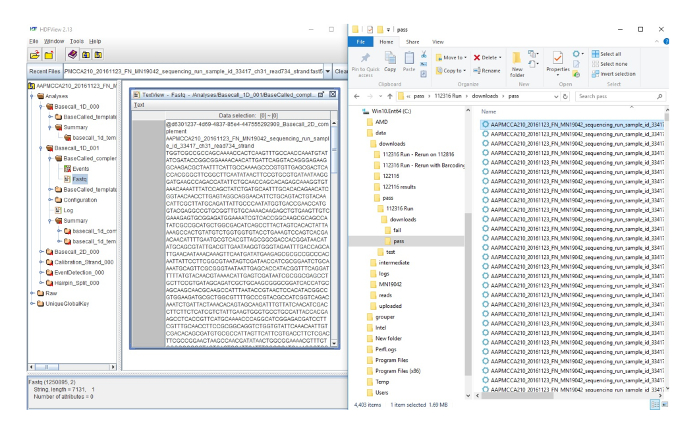
משלימה איור 2: FAST5 המרת קבצים לקבצים FASTQ. מצד ימין, הנה הפלט של נתונים מ הפעל רצף. כל שורה מייצגת מרצף בודדים. הצד השמאלי של האיור מציג את רצף המסומן מצד ימין, הצופה HDF הממירה את הרצף בקובץ FASTQ אשר יכול לשמש לצורך ניתוח נוסף. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
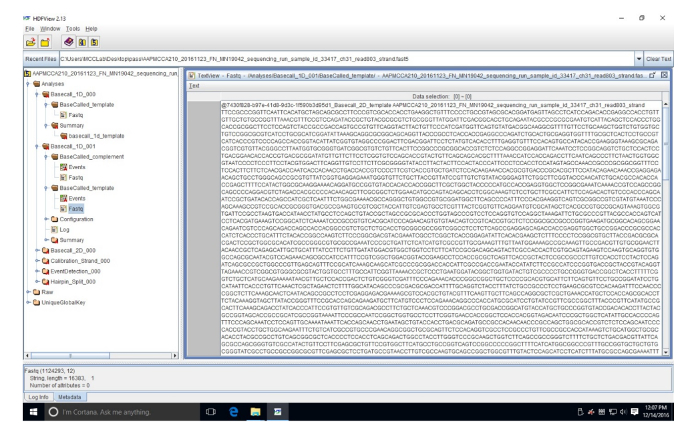
משלימה איור 3: דוגמה של FASTQ רצף במציג HDF. רצף זה (קריאה 803) הוא בקובץ FASTQ אשר ממירה את נתוני FAST5 נוקלאוטידים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
שיטות רצף הדור הבא רבות נוצרו כל אחד תלוי רצף על ידי סינתזה אבל הפלטפורמות זיהוי לזיהוי נוקלאוטידים שונים. NS, התוספת האחרונה לשוק, משתמש בשיטה שונה לחלוטין, אשר אינה דורשת תגובה רצף או תווית נוקלאוטידים. שיטה זו מנצלת תשלום דיפרנציאלי של נוקלאוטידים כבר incorporated כפי שהם עוברים הנקבובית מחושמלת. הזיהוי של כל נוקלאוטיד מתרחשת על ידי אפנון של הזרם החשמלי על ידי היסודות השונים כפי שהם עוברים nanopore. מערכת מרובבת זו מאפשר למשתמש רצף קטעים רבים בכל פעם. על ידי קביעת רצף שני גדילי ה-dna, גדל באופן משמעותי הדיוק של הרצף, התוכנה רצף, אשר ניתן לטעון על גבי מחשב נייד, מעבד את האותות, מספק את המידע רצף מסוגל לנתח.
ב- pyrosequencing, רצף לפי שיטת סינתזה (SBS), זיהוי של בסיס ספציפי שולבו התבנית תלוי וזמינותו לוציפראז הדור של אותות chemiluminescent8. ב רצף מוליכים למחצה יון, יון מימן שפורסמו, אשר מפחית את רמת החומציות מזוהה על ידי חיישן יון9. מולקולה בודדת רצף בזמן אמת תלויה אפס במצב גל מדריך (ZMW)10, אשר מאירה לגילוי מולקולה נמכרות מתויג נוקלאוטיד incorporated. SBS משתמש בשיטה ייחודית כדי להגביר את המטרה DNA כך אשכולות הרצפים הייחודיים הם שנוצר13. זיהוי של נוקלאוטיד הוסיף מושגת כאשר זריחה של נוקלאוטיד מתויג נרשם. NS מאידך גיסא יש יתרון ייחודי על פני שיטות אחרות בכך שזה דורש משאבים טכניים מוגבל, נייד, מייצרת קריאות רצף ארוך, דורש אין הגברה DNA מוקדמת, יכול להיות מופעל עם עלות מופחתת בהשוואה לשיטות אחרות. התלמידים שלנו למצוא פרוטוקול הכנה ספריה חדשה, מהירה פשוטה, נוטה שיעור מעבדה 3 שעות. בחלק מן הנושאים נתקלנו היו בועות בתא זרימה אשר היו קשה להסיר, זה נדרש כוח משמעותי המחשב (טראבייט אחד של אחסון), הפלט הנוכחי של הנתונים בקובץ FAST5, רצף זרימה התא יש חיי מדף מוגבלים לפני זה מתדרדר. בנוסף, חסרונות אחרים של פרוטוקול רצף מצדו NS (פרוטוקול ארוך) הוא כי זה דורש מספר צעדים הכנה ספריה, דורש התמחות בטכניקות ביולוגיה מולקולרית, יוצר נאמנות רצף מופחתת בהשוואה לחלק רצף מתודולוגיות3. אולם, ההתקדמות האחרונה עם ערכת הכנה מהירה ספריה חדשה דורשת רק 10 דקות הכנה ספריה, הוכיח שיעור שגיאה רצף מופחת. השיטה החדשה של הכנת ספריה לא התקשה מאוד לשימוש בכיתה במעבדה.
ישנם מספר שלבים קריטיים בפרוטוקול, בפרט QC של התא זרימה. זה כולל ביצוע של QC הראשונית תוך חמישה ימים של קבלת של התא זרימה ושימוש בהם בתוך 8 שבועות. למרות השתמשנו תאים זרימה שהיו מעבר 8 שבועות, הקטינה באופן משמעותי מספר הנקבוביות פתוח/פעיל. חשוב כי ניסויים מתוכננים כדי להתאים ציר זמן שבו שימוש מרבי של תאי זרימה מושגת. יש להשתמש בפרוטוקול ניקוי ואנו תאי זרימה עם הצלחה לעשות בה שימוש חוזר.
חוקרים את metagenomics של אדמת מייצגים מאגר גנטי עצום של גיוון מיקרוביאלי. כך למשל, גרם אחד של אדמת מוערך מכילים בין 107 -9 10 תאים prokaryotic14. יתר על כן, אדמת אורגניזמים הם המקור העיקרי של מוצרים טבעיים חדשניים, אנזימים אנטיביוטיקה. לפיכך, ניתוח רצף הדנ א metagenomics אדמה מייצגת כלי הדרכה חשוב לסטודנטים בכל רמה של חינוך. של הטכנולוגיה NS להקל של שימוש ולהפוך בעלות נמוכה זו מערכת כלי הוראה יעילה מאוד. סטודנטים יכולים רצף דגימות סביבתי ולאחר עם סיומו של הרצף להשתמש בכלים לביואינפורמטיקה זמין כדי לזהות ולאפיין רצפי חיידקים, metagenomics הבדיקה דוגמאות. באמצעות הטכנולוגיה NS, סטודנטים יש התנסות אמיתית, יש עד עכשיו היה מתוך להגיע לשימוש בקורסים מעבדה בשל מומחיות טכנית מתקדמת עלויות גבוהות ריאגנט, ציוד ותחזוקה של פלטפורמות אחרות רצף. לאחרונה, אחד הסטודנטים שלנו (הריסון ג', תקשורת אישית) דיווחו על השימוש בטכנולוגיה זו בפרוייקט ניטור סביבתי של החווה קרקעות. אנו מצפים כי יהיו הרבה יותר יישומים לטכנולוגיה הזו במרחב החינוך.
Disclosures
המחברים יש אין גילויים.
Acknowledgements
פרויקט זה היה תמיכה חלקית על ידי אוניברסיטת ג'ונס הופקינס, משרד פרווסט דרך שער מתווה.
Materials
| Name | Company | Catalog Number | Comments |
| Thermal Cycler | LifeECO | BTC42096 | |
| Covaris g-TUBE | Covaris | 520079 | |
| NEBNext End Repair Module | New England BioLab | E7546 | |
| Eppendorf LoBind Centrifuge Tubes | Sigma-Aldrich | Z666505 | |
| NEB Blunt/TA Ligase Master Mix | New England BioLab | MO367 | |
| MyOne C1 Strepavidin Beads | Thermo Fisher | 65001 | |
| Eppendorf microcentrifuge | Eppendorf | Model 5420 | |
| Nanodrop UV spectrophotometer | Thermo Fisher | ND-2000 | Model 2000 |
| Belly Dancer Orbital Shaker | Sigma-Aldrich | Z768499 | |
| Power Soil DNA Isolation Kit | MO BIO | 12888-50 | |
| Ligation Sequencing Kit 2D | Oxford Nanopore | SQK-LSK208 | |
| Rapid Sequencing Kit for Genomic DNA | Oxford Nanopore | SQK-RAD002 | |
| End-Prep Reaction Buffer and enzyme mix (NEB Blunt/TA Ligase Naster Mix) | New England BioLab | MO367 | |
| AmPure XP Magnetic Beads | Beckman Coulter | A63880 | |
| Basic Starter Pack | Oxford Nanopore | Includes MinION Sequencing Device and Flow Cell | |
| MinKNOW software | Oxford Nanopore | ||
| Rapid Sequencing Kit for Genomic DNA | Oxford Nanopore | SQK-RAD002 | Includes Running Buffer with Fuel (RBF), Fragmentation Mix (FRM) Rapid Adapter (RAD) Library Lodaing Beads (LLB) |
References
- Quick, J., et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
- Greninger, A. L., et al. Rapid metagenomics identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome Med. 7, 99 (2015).
- Quick, J., et al. Rapid draft sequencing and real-time nanopore sequencing in a hospital outbreak of Salmonella. Genome Biology. 16, 114 (2015).
- Ashton, P. M., et al. MinION nanopore sequencing identifies the position and structure of a bacterial antibiotic resistance island. Nat Biotechnol. 33, 296-300 (2015).
- Loman, N. J., Quick, J., Simpson, J. T. A complete bacterial genome assembled de novo. using only nanopore sequencing data. Nat Method. 12, 733-735 (2015).
- Hoenen, T., et al. Nanopore Sequencing as a Rapidly Deployable Outbreak Tool. Emerg Infect Dis. 22 (2), 331-334 (2016).
- Petrosino, J. F., Highlander, S., Luna, R. A., Gibbs, R. A., Versalovic, J. Metagenomic pyrosequencing and microbial identification. Clin Chem. 55 (5), 856-866 (2009).
- Cummings, P. J., Ahmed, R., Durocher, J. A., Jessen, A., Vardi, T., Obom, K. M. Pyrosequencing for Microbial Identification and Characterization. J. Vis. Exp. (78), e50405 (2013).
- Rothberg, J. W., et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., et al. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Stoddart, D., Heron, A. J., Mikhailova, E., Maglia, G., Bayley, H. Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. PNAS. 106, 7702-7707 (2009).
- Shendure, J., Hanlee, J. Next generation DNA sequencing. Nat Biotechnol. 26, 1135-1145 (2008).
- Daniel, R. The Metagenomics of Soil. Nat Rev Microbiol. 3, 470-478 (2005).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved