Method Article
Nanopore DNA sıralama Metagenomic toprak analizleri için
Bu Makalede
Özet
Nanopore teknoloji sıralama biomolecules için geniş uygulamalar patojenler, gıda güvenliği izleme, genom analizi, metagenomic çevre izleme ve bakteriyel karakterizasyonu kimliği de dahil olmak üzere Yaşam Bilimleri içinde vardır. antibiyotik direnci. Bu makalede, metagenomic toprak DNA sıralama nanopore sıralama teknolojisini kullanarak tür tanımlayıcısı için yordam gösterilmiştir.
Özet
Bu makalede toprak, hazırlık ve nanopore akışı hücre kullanımı ve çözümleme bilgisayar yazılımı kullanarak tanımlanan DNA dizilerinin bir DNA Kütüphane inşaatı için adımları açıklar. Nanopore DNA sıralama bakteri suşları karakterize ve antibiyotik direnç görüşmek genetik mutasyonlar algılamak için hızlı mikrobiyal genom sıralama bakteriyel ve viral türleri, belirlemek sağlayan esnek bir tekniktir. (NS) yaşam bilimleri için sıralama nanopore avantajı, düşük karmaşıklık, düşük maliyet ve hızlı gerçek zamanlı sıralama arıtılmış genomik DNA, PCR amplicons, cDNA örnekleri veya RNA içerir. NS "tek telli DNA molekülünün bir sentetik polimer membran eklenen bir nanopore aracılığıyla rehberlik ederek sıralama DNA içeren strand sıralama" örneğidir. Membran o, bireysel üsleri ile nanopore elektrik akımı geçerken farklı derecelerde dört nükleotit bazlar tarafından bozulur öylesine genelinde uygulanan bir elektrik akımı vardır. Her nükleotit tanımlaması nanopore geçerken elektrik akımı karakteristik modülasyon tarafından farklı esaslarını tespit ederek oluşur. NS sistemi el ile oluşur, USB taşınabilir aygıt ve nanopore dizi içeren bir tek kullanımlık akışı hücre. Taşınabilir aygıt içine a standart dizüstü bilgisayar okur ve bilgisayar yazılımı kullanarak DNA dizisi kaydeden fişler.
Giriş
Bu yordamı, sıralama nanopore akışı hücre sıralama cihazın kullanımı için bir çevre DNA Kütüphane hazırlanması için gereken adımları göstermek için ve sistem yazılımı kullanarak oluşturulan DNA dizilerinin analiz gerçekleştirmek için hedeftir ve Merkezi Ulusal toprak mikrobiyal türler tanımlamak için biyoteknoloji bilgi (NCBI) Biyoinformatik araçları için. Şu anda, çoğu DNA sıralama platformlar teknik eğitim ve kaynak zavallı ortamlarda veya alan uygulamaları mümkün değildir karmaşık araçları büyük bir yatırım gerektirir. Nanopore sıralama (NS) platformu ile bir maliyet etkin, bu sorunları ortadan kaldırır Kütüphane hazırlık Protokolü ve taşınabilir bir aygıta sıra ve nükleik asitler1,3farklı türleri çeşitli analiz etmek için kullanımı kolay. Yüksek lisans öğrencileri için birkaç laboratuar dersleri NS platform dahil olması.
Nanopore teknoloji sıralama biomolecules için yaşam bilimleri, bakteriyel ve viral patojenler1,2,6, çevre biyolojik çeşitlilik tanımlamasını içeren geniş uygulamalarda göstermiştir çalışmalar, gıda güvenliği izleme, genom analizi3,5ve bakteriyel antibiyotik direnci4karakterizasyonu. NS bir elektrikli eklenen bir nanopore tek iplikçikli DNA geçtiği zaman prensibi üzerine "sıralama elektrik kesintileri algılayarak strand" bireysel nükleotit bazlar tarafından dayanır bir hızlı ve doğru sıra nükleik asitler için yöntemidir Sentetik polimer membran. DNA genom parçalanma, sonunda tamir ve 3' genomik parçaları, adaptör ve DNA'yı, urgan tavlama dA-takip NS eklemek için hazırlanmasında ilgili adımlar DNA Kütüphane arıtma ve Kütüphane nanopore akışı hücre cihazın içine yükleme. Genom parçalanma ~ 8 kb boyutları içine 1-2 µg bir g-tüp parçalanma tüpünden genomik DNA'ın centrifuging tarafından gerçekleştirilir. Parçalanmış genomik biter sonra tamir ve piyasada bulunan bir seti kullanarak Poli dA ile izledi. Nanopore motor protein ile uyumlu olan, tek telli bağdaştırıcı dizileri eklenir DNA dizisi nanopore (şekil 1) aracılığıyla rehberlik için kullanılan DNA biter. Urgan dizileri DNA arıtma ve DNA molekülleri gözenek membran için yerelleştirme için gereklidir. Saç tokası dA kuyruklu kütüphane bir ucunu bir saç tokası bağdaştırıcısına birleştirilmesi (ligasyon) tarafından oluşturulur. Saç tokası yapılar DNA ucunda DNA nanopore (Şekil 2) geçerken anlam ve Antisens iplikçiklerinin okuma sağlar. Hazırlanan genomik Kütüphanesi sonra içine nanopore akışı hücre analizi için örnek yükleme tarafından izlenen bir manyetik alanını kullanarak streptavidin boncuk kullanarak gelen tepki saf.
Sıralı DNA kalitesi için değerlendirilir ve analiz için kabul edilebilir sıralama okuma sonra mikrop tanımlamak için çeşitli Biyoinformatik araçlar için tabi. Dizileri "bir FASTQ FAST5 biçiminden çevrilir". FASTQ biçiminde dizileri sonra patlama analizde kullanılabilir.
Protokol
Not: Piyasada bulunan toprak genomik yalıtım kullanarak topraktan (Baltimore İlçesi, Maryland) Metagenomic DNA saflaştırılmış ( Tablo malzemelerigörmek) kit. Bir UV Spektrofotometre kullanarak (bkz. Tablo malzemeler), arıtılmış genomik DNA 260/280 (nm) oranına sahip olması > 1.8 ve 260/230 oranı örnek temin için 2.0-2.2 arasında kirleticiler ücretsizdir. Genomik DNA 200 NS aralıkları için gerekli miktarı 2 µg ng.
1. hızlı Kütüphane hazırlama yöntemi (kısa protokol)
Not: Bu kısa bir protokoldür. Malzemeler tablo için hızlı sıralama kit'e bakın.
- Bir 0.2 mL tüp ince duvarlı ( Tablo malzemelerigörmek) ileri 200 ng yüksek molekül ağırlıklı DNA 7.5 µL distile su hacminin bir final.
- Parçalanma mix (FM) 2.5 µL hızlı Kütüphane hazırlık seti ve karışımı yavaşça INVERSION tarafından ekleyin. Spin aşağı darbe santrifüj (1000 x g 5 için s).
- Örnek bir thermocycler yerleştirin (bkz. Tablo reçetesi) 30 ° c için 1 dk 1 dk. 75 ° C ardından bir tur için örnek spin tüp ve nabız santrifüj kaldırmak.
- 1 µL hızlı bağdaştırıcısının ve 0.2 µL blunt/TA ligaz ana karışımı ekleyin ve sonra adapte ve gergin Kütüphane buza saklayın.
Not: kısa protokolünü kullanarak adım 8 devam ederseniz
2. tüp ligasyonu sıralama Protokolü (uzun protokol): Metagenomic DNA fragmantasyonu
Not: Bkz: tüp ligasyonu sıralama kit için Malzemeler tablo .
- Son hacmiyle 46 µL deiyonize su arıtılmış genomik DNA'ın 1 µg sulandırmak.
- Bir DNA fragmantasyonu örneğe ( Tablo malzemelerigörmek) tüp ve santrifüj kapasitesi 8000 x g bir microcentrifuge kullanarak, oda sıcaklığında 1 dk. için transfer (~ 8 kb genomik parçaları üretmek için Malzemeler tablobkz:). Tüm sıvı koleksiyonu tüp içine geçti emin olun.
- Parçalanma tüp, santrifüj ve alt odasına parçalanmış genomik DNA toplamak 1 dk santrifüj dönüş ters çevir.
- Parçalanmış genomik DNA steril düşük-DNA bağlayıcı 1,5 mL tüp aktarmak ve parçalanma onaylamak için bir düşük yüzde özel jel (%0.6) bir kısmı için analiz > 30 kb.
3. parçalanmış genomik DNA sonu-hazırlık
- 800 kullanarak ng deiyonize su, 45 µL içinde parçalanmış genomik DNA'ın eklemek 7 µL son hazırlık tepki tampon (bakınız Tablo malzemeler), 3 µL enzim karışımı (bkz. Tablo reçetesi) ve su Ücretsiz 5 µL nükleaz.
- Karıştırmak yanında inversiyon ve nabız santrifüj (1000 x g 5 için s).
- Örnek bir 0.2 mL tüp ince duvarlı aktarmak ve örnek 65 ° c 5 min için kuluçka ardından 20 ° C'de 5 min için kuluçkaya.
- Nabız santrifüj tüpü dibine içeriği getirmek.
- Örnek bir 1,5 mL düşük-DNA bağlayıcı microcentrifuge tüp aktarın.
- Manyetik boncuklar ( malzemelerin tabloyabakın) hazırlayın. Resuspended boncuk 60 µL son hazırlık tepki adım 3.5 pipetting tarafından ekleyip karıştırın.
- Oda sıcaklığında 5 min için 100 RPM ( Tablo malzemelerigörmek) bir rotator Mikser üzerinde kuluçkaya.
- Nabız boncuk cips, sonra örnek mıknatıs yanında yer için örnek santrifüj kapasitesi.
- Bir kez boncuk için mıknatıs yapıştırılır vardır, süpernatant ve atma kapalı pipet.
- Tüp ile taze hazırlanmış % 70 etanol 200 µL mıknatıs ve yıkama boncuk hafifçe alçalarak örnek pipetting tarafından kaldırın.
- Nabız santrifüj boncuk cips ve mıknatıs için dönmek için.
- Süpernatant kaldırmak ve atın. 200 µL % 70 etanol kullanarak yeniden çamaşır adımı yineleyin.
- Nabız tüp mıknatıs için dönüş santrifüj kapasitesi ve tüm % 70 etanol yıkama kaldırın.
- Microcentrifuge tüp kapağı açıkken, hava boncuk oda sıcaklığında 5 min için Kuru izin.
- Tüp mıknatıs kaldır ve Pelet steril, nükleaz ücretsiz su 31 µL içinde askıya alma. Oda sıcaklığında 2 min için kuluçkaya.
- Tüp mıknatıs için tüm boncuk pelleted ve eluate açıktır kadar döndürür.
- Eluate 1.5 mL microcentrifuge tüp aktarın. Örnek, 1 µL kullanarak ölçmek bir UV Spektrofotometre 260 kullanarak bitiş prepped DNA nm ( Tablo malzemelerigörmek).
Not: ~ %70 yüzde kurtarma olmalıdır (700 ng) üzerinden 1 µg genomik DNA'ın bir başlangıç konsantrasyonu.
4. adaptör ve sonunda hazırlandı genomik DNA parçalarının urgan ek
- Blunt'ın / TA ligaz ana mix tüpler INVERSION tarafından mix ve içeriği altına getirmek için santrifüj darbe.
- Sonunda hazırlandı DNA'ın 30 µL kullanarak, 20 µL adaptör karışımı ve blunt/TA ligasyonu ana Mix 50 µL ekleyin.
- İnversiyon, nabız santrifüj, mix ve oda sıcaklığında 10 dakika için kuluçkaya.
5. manyetik Bead hazırlık
- Girdap tüp boncuk ve daha sonra transfer 50 µL 1.5 mL microfuge için askıya alınmış boncuk.
- Eluate temizlenene kadar boncukları cips için yer, mıknatıs tüp.
- Süpernatant kaldırmak ve atın.
- Boncuk bağlama arabellek 100 µL ekleyin ( Tablo malzemeleri boncuk kiti için görmek) boncuklar, boncuklar, resuspend girdap mıknatıs üstündeki cips, süpernatant kaldırmak ve atmak. Yıkama 100 µL bağlama arabellek ile yineleyin.
- Yıkanmış boncuk boncuk bağlama arabellek 100 µL ekleyin. Girdap boncuk resuspend.
6. Kütüphane arıtma
- P200 micropipette kullanarak, boncuk 40 µL adapte/gergin genomik DNA için önceki adımda ekleyin. Örnek pipetting tarafından dikkatlice karıştırın.
- Örnek oda sıcaklığında 5 min için 100 RPM bir rotator Mikser üzerinde kuluçkaya.
- Örnek üzerinde mıknatıs yerleştirmek ve boncuk yerleşmek izin verir. Süpernatant pipet ve atın.
- Boncuk boncuk bağlama arabellek 140 µL içinde pipetting tarafından resuspend. Örnek karşı mıknatıs yerleştirin, süpernatant atın ve tekrar boncuk bağlama arabellek başka bir 140 µL ile yıkayın.
- Nabız tüp santrifüj kapasitesi, pelet kalan boncuk bağlama arabellek 2 dk. kaldırmak için tüp üzerinde mıknatıs yerleştirin.
7. mıknatıs boncuk kitaplığından elüsyon
- Manyetik boncuklar elüsyon arabellek 15 µL içinde pipetting tarafından resuspend. Örnek oda sıcaklığında 10 dakika için kuluçkaya.
- Boru boncuk cips mıknatıs karşı koyun.
Not: ~ % 25 yüzde kurtarma olmalıdır (250 ng) üzerinden 1 µg genomik DNA'ın bir başlangıç konsantrasyonu.
8. başlayan bir çalışma/kalite kontrol
- Akış hücre paketinden çıkarın ve akış hücre için taşınabilir, gerçek zamanlı sequencer ekleyebilirsiniz (bkz. Tablo malzeme).
- Sequencer'ı bir bilgisayar yolu ile USB tel kablo ekleyin ve sıralama yazılımını başlatın ( Tablo malzemelerigörmek).
- Tıkırtı "Bağlan" aygıt sıralama yazılımı aracılığıyla, "NC_Platform_QC.py" seçin ve "Başlat".
- Kalite kontrol (QC) tamamlamak için yaklaşık 6-7 dakika sürer; Bak bir 'Yeşil deniz' için (çıkış geniş alanlar) üzerinde yeterince etkin gözenekleri onaylamak için QC okuma (> 800) DNA sıralama için.
9. bir sıralama Çalıştır başlatma
- Sıralama programda açın. bir iletişim kutusu görüntülenir. Örnek kimliği kutusuna örnek adını verin.
- "Akış hücre kimliği" kutusunu tıklatın ve etiket akışı hücrenin üst kısmında bulunan kodları girin.
- Akış cep kapağı açın ve örnek bağlantı noktası kapak sağa kaydırın (saat yönünde) örneği bağlantı noktası görünür olmasını sağlayın. Bağlantı noktası kontrol için küçük bir kabarcık, açıldıktan sonra. Arabellek kabarcık dahil olmak üzere birkaç microliters kaldırın.
- Emin olmak için sensör üzerinde arabellek olup olmadığını denetleyin. Arabellek yakıt karışımı ile çalışan 480 µL karıştırılarak macun tampon yapmak (RBF-1, Tablo reçetesigörmek) (mix emin olun iyice ilk) 520 µL nükleaz ücretsiz su ile.
- Astar arabelleğini 800 µL astar bağlantı noktasına ekleyin. Dikkatli bir şekilde erişilebilir olması için "Nokta" kapağını kaldırın.
- 5 dk sonra ek bir 200 µL astar arabelleğini astar bağlantı noktasına ekleyin.
10. Kütüphane yükleniyor
- Kütüphane yer buz üzerinde aşağıdaki reaktifler hazırlamadan önce: RBF-1 (sıralama takımı)'ndan uyarlanmış ve kaşif Balonlu keşif kitaplığı ve boncuk yükleme kitaplığı (LLB; sıralama kitinden).
- 0.2 mL santrifüj tüpü için RBF 25,5 µL ve 12 µL nükleaz ücretsiz su oda sıcaklığında muhafaza ekleyin.
- LLB alçalarak pipetting tarafından karıştırın. LLB 26.5 µL 0.2 µL tüp ekleyin. Reaktifleri alçalarak pipetting tarafından karıştırın.
Not: LLB karışımı dışarı yerleşmek eğilimindedir. - 11 µL adapte ve gergin kitaplığının 0.2 µL tüp ekleyin. İnversiyon ve spin tarafından darbe Santrifüjü tarafından karıştırın.
- 75 µL örnek--dan adım 9,4 dropwise "Nokta" bağlantı noktasına ekleyin. Her damla sonraki eklemeden önce bağlantı noktasına akar emin olun.
- Dikkatle tıkamak bağlantı noktası girer böylece örnek bağlantı noktası kapağı yerine takın ve cihaz kapağı değiştirin.
11. bir sıralama yazılımı iletişim kuralı komut dosyası başlatma
- 'Seçin programı' kapsamında açılan menüsünü açın ve "48 saat sıralama." seçin
- Komut dosyasını başlatmak için "Çalıştır" butonuna tıklayın.
- Bildirim akışı etkin gözenekleri MUX taramasayısı için bildirilen Mux tarama sonuçlarını gözden geçirin.
Not: Bu numarayı çalıştırmak QC gözenekleri sayısı farklı olabilir ve büyük bir fark bir sorunu belirtiyor olabilir.- Varsa etkin gözenekleri QC Çalıştır önemli bir azalma yazılımını yeniden başlatın ve hala önemli bir fark varsa bilgisayarı yeniden başlatın.
- Sıralama yazılımı komut dosyası tarafından belirlendiği şekilde heatsink sıcaklığı 34 ° C olduğundan emin olun.
Not: sıcaklık 34 ° C'de değil ise o zaman olacak çalıştırmak için yeterli etkin gözenekleri. - Histogramı okuma uzunlukları sırası uzunlukları beklenen ve deneysel tasarım için uygun olup olmadığını belirlemek için denetleyin.
- 'X. Quit sıralama yazılımı web GUI kapatarak Kapat' kullanarak masaüstü aracı kapatın. Aygıtı bilgisayardan sökün.
12. Çalıştır ve sonuçları analiz
Not: masaüstü aracı kullanarak "Raporu GÖRÜNTÜLE" programı kullanarak verileri sıralama çalışma sırasında takibi yapılabilir. Sırasında veya çalışma tamamlandıktan sonra dosyalar dosya biçiminde veri → okur → geçmek klasörü (varsayılan) bir FAST5 kullanılabilir. Sıralama etkin durumdayken bu klasör açıksa bilgisayarı donabilir.
- Görüntülemek ve dizileri işlemek için bir "HDFViewer" indir.
- Sıralama dosyaları görüntülemek için doğrudan açık tüm dosya HDF Görüntüleyicisi → açık veri klasörü → seçin → seçin C: sürücü (varsayılan) → verileri seçme → seçin okuma → seçin pass türleri.
- Bulmak ve FASTQ dosyasını sol açılan menüden seçin; dosya açılacak ve FASTQ biçiminde kaydedilmiş olabilir.
Not: öğrenci deneme amacıyla dizileri 350 nükleotit veya daha uzun daha fazla çözümleme için seçildi. Temel arama çıkış sırası okuma boyutuna göre sıralamak kullanıcı sağlar. FASTQ dosyaları diğer Biyoinformatik program veya patlama (NCBI) yolu ile çözümlenebilir.
Sonuçlar
Deneysel tasarım ve nanopore teknoloji toprak DNA sıralamak öğrenciler için hızlı ve ucuz bir yöntem sağladı. Temsilcisi Çalıştır 800'den fazla gözenekli sıralama için mevcut kalite kontrol parametreleri geçirildi. Çalıştır üzerinde 125.000 okuma için çalışma 5,38 kb medyan sıra uzunluğu ile sonuçlandı. Dizileri bir kalite puanı verilir ve sonra yalnızca bu dizileri kabul edilebilir puanları ile analiz edildi. Nasıl yapılacağını çıktı olarak FAST5 biçiminde bu programlar gibi patlama (NCBI), tarafından diziler dizileri patlama analiz için uyumludur FASTQ biçime dönüştürür HDF Görüntüleyicisi'nde görüntülenen kabul edilmez tepkidir. Gelecekte yazılım yinelemelerini, verileri HDF görüntüleyiciyi ihtiyacını ortadan kaldırarak bir FASTQ biçimi olarak kullanılabilir. Bkz: takıma giren rakamlar 1, 2 ve 3.
Elli uzunluğu 350 nükleotit dizilerinin vardı tabi BLASTN (nukleotid nükleotit veritabanı) veya BLASTX (amino asit dizini tercüme nukleotid) göre analiz (NCBI) toprak örneği organizmalarda tanımlamak için. Zaman verilen kısıtlamaları sınıf ve ders odak laboratuvar yöntemleri ve değil Biyoinformatik olduğunu, dizileri bu parametreler içinde analiz öğrenciler oldu. Biyoinformatik sınıflarımız verileri ardışık düzen analizi araçlarının gelişimi için kullanabilirsiniz. Bu düzey Biyoinformatik analizi dayalı laboratuvar sınıfta kapsamında değildir. Tablo 1 4 e-04 e değerleri ile tespit edilmiştir organizmalar listesidir. Genellikle, 4 e-04'den az olan e-değerlerin Giriş dizisi (sorgu) ve eşleşen dizini arasında güçlü benzerlik gösterir. (Olmayan (nr) veritabanında) BLASTN tarafından tanımlanan organizmalar çeşitli türler arasında olan ve kullanılabilir daha fazla genomik ve protein analizleri. Hangi ne toprak organizmalar olabileceğini belirlemek önceki veri vardı bu yüzden bu örnek bir bahçe Baltimore İlçesi, MD geldi hangi toprak, hiçbir zaman, test. BLASTN (nr veritabanıyla) çözümleme Ayrıca hiçbir benzerlik nr veritabanındaki sıralarıyla olduğunu belirtti. Bunlar gerçekten roman dizileri olup olmadığını belirlemek için daha fazla çalışma gereklidir. BLASTX tarafından çeşitli serileri analizini BLASTN analizde temsil edilmeyen organizmalar üzerinden çeşitli protein saptandı. Tablo 2 endopeptidases ve ATPases gibi proteinler de dahil olmak üzere birkaç olası proteinler listeler. Bu kütüphane dizileri kullanarak, öğrenciler eğitmen tarafından Yönetmen: Eğer daha fazla çözümleme yapmak için daha sofistike Biyoinformatik araçlarını kullanmak için fırsat var.
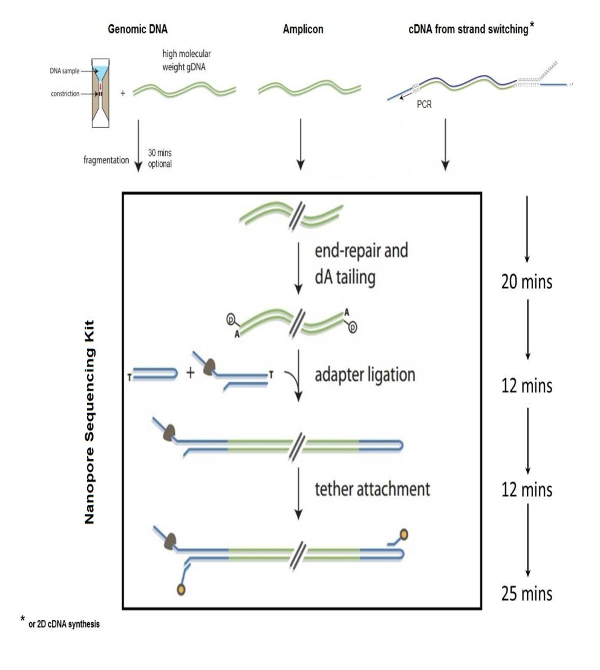
Resim 1 : Genomik Kütüphanesi hazırlık. Nanopore teknolojisini kullanarak sıralama için genomik DNA Kütüphane hazırlanması için adımlar. Bu rakam gerekli izinlere sahip değiştirdi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Resim 2 . Bir nanopore kullanarak DNA sıralama şematik. DNA nanopore membran elektrik sinyali üretimi ve baz kimlik ile geçiyor. Bu rakam gerekli izinlere sahip değiştirdi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
| Organizma | e değeri |
| Streptomyces sp. | 3.00E-06 |
| Nocardoides sp. | 5.00E-04 |
| Gordonia sp. | 5.00E-04 |
| Hyphomicrobium nitrativorens | 1.00E-139 |
| Starkeya novella | 1.00E-31 |
| Hyphomicrobium denitrificans | 1.00E-30 |
| Fibomicrobium sp. | 5.00E-17 |
| Pseudomonas sp. | 2.00E-15 |
| Rhodothemus marinus | 1.00E-17 |
| Turneiella parva | 2.00E-24 |
| E. coli | 0.00E + 00 |
| Bradyrhizobium sp. | 3.00E-30 |
| Gemmatirosa kalamazoonesis | 1.00E-20 |
| Burkholderia sp. | 8.00E-10 |
| Sphingomonas sp. | 8.00E-10 |
| Cellumonas sp. | 6.00E-11 |
Tablo 1: BLASTN incelemenin sonuçlarını seçili. Toprak metagenomics DNA dizileri BLASTN benzerlik arama NCBI olmayan nükleotit veritabanıyla tabi. Rapor verileri e-değerleri 4 x e-4 veya daha az olan.
| Protein | Organizma |
| Örnek protein | Acidobacter bakteri |
| SAM bağımlı metiltransferaz | H. denitrificans |
| ATPaz | Jannaschia sp. |
| Endopeptidase | Shigella sonnei |
| Lizis protein | E. coli |
Tablo 2: BLASTX incelemenin sonuçlarını seçili. Toprak metagenomics DNA dizileri BLASTX benzerlik arama NCBI yedek protein veritabanıyla tabi.
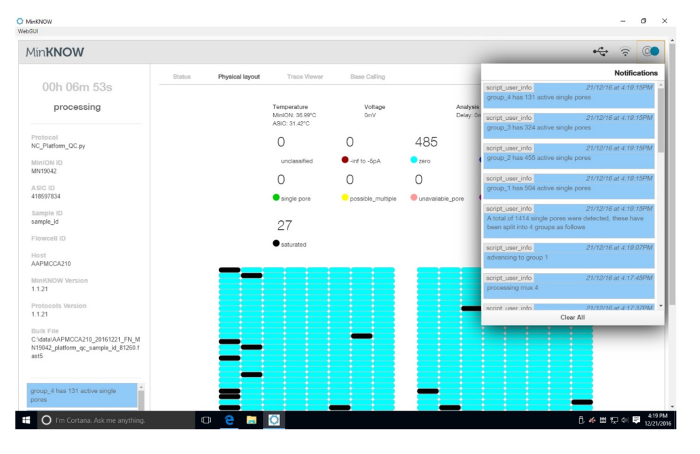
Tamamlayıcı şekil 1: Platform QC sonuç. QC platformun sonuçları sunulmuştur. Sağ üst köşedeki QC etkin gözenekleri için sonuçları vardır. Akış hücrenin her çeyrek için etkin gözenekleri mercek altına alındı. Ana sayfa dinamik ve her çeyrekte mercek altına alındı olarak değişir. Bu vadede 1.414 tek gözenekleri tespit edildi.Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
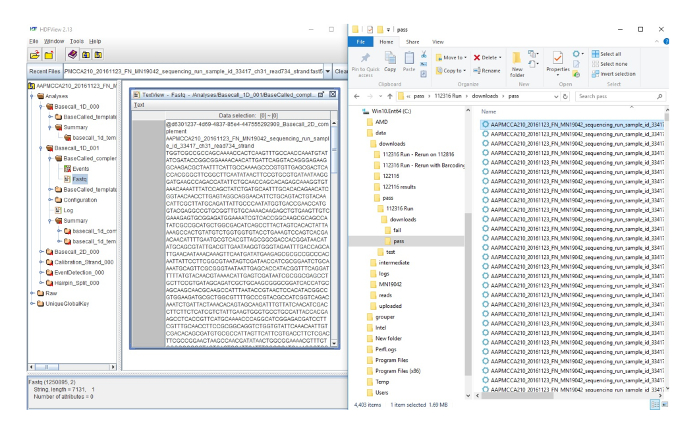
Tamamlayıcı Resim 2: FAST5 dönüştürme dosyaları FASTQ dosyaları için. Burada sağ tarafta veri üzerinden sıralama çalışma çıktı. Her satır bir bireysel sıra temsil eder. Rakam sol tarafında daha fazla çözümleme için kullanılacak bir FASTQ dosyasının sıra dönüştürür HDF Görüntüleyici sağ taraftan vurgulanan sırası gösterilmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
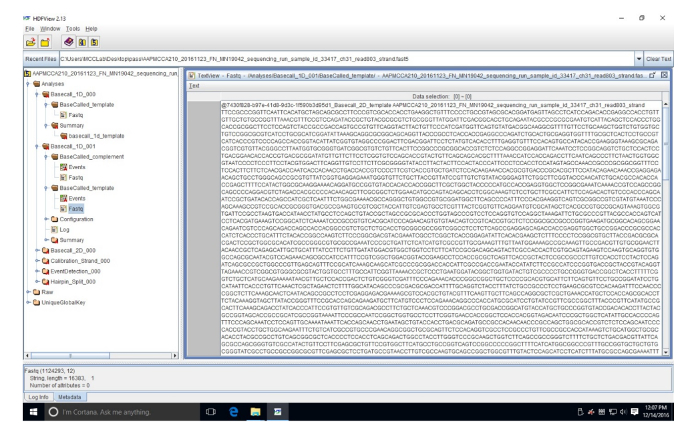
Tamamlayıcı şekil 3: örnek FASTQ sıra HDF Görüntüleyicisi'nde. Bu (okuma 803), FAST5 veri nükleotid dönüştürür bir FASTQ dosyasında sırasıdır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tartışmalar
Birçok sonraki nesil sıralama yöntemleri oluşturulmuştur ve her sentezi tarafından sıralaması üzerinde bağlıdır ancak farklı nükleotit tanımlamak için algılama platformlar. NS, belgili tanımlık pazar yeri yanı sıra en son zamanlarda farklı bir yöntem tamamen, hangi does değil istemek bir sıralama tepki veya nükleotid etiketli kullanır. Onlar bir elektrikli gözenek geçerken bu yöntem zaten dahil nükleotit ücret diferansiyel yararlanır. Onlar nanopore geçerken her nükleotit tanımlaması farklı üsleri tarafından elektrik akımı modülasyon tarafından oluşur. Çoğaltılmış bu sistem birçok parçaları teker teker sıralamak kullanıcı sağlar. Her iki ipliklerini DNA Sekanslama tarafından sıra doğruluğunu önemli ölçüde artar ve bir dizüstü bilgisayara yüklenebilir, sıralama yazılımı sinyalleri işleme ve analiz edilebilir sırası bilgi sağlar.
Pyrosequencing, sentez (SBS) yöntemi, bir sıralama içinde şablonu içine dahil belirli Bankası tespiti luciferase tahlil ve chemiluminescent sinyalleri8nesil bağlıdır. İyon yarı iletken sıralama, pH azalır yayımlanan hidrojen iyon bir iyon sensörü9tarafından algılanır. Hangi algılama için anonim nükleotit öğesini bir floresan molekül aydınlatan sıfır modu dalga kılavuzu (ZMW)10, tek molekül gerçek zamanlı sıralama bağlıdır. SBS oluşturulan13benzersiz sıraların kümeleri vardır öyle ki hedef DNA yükseltmek için benzersiz bir yöntem kullanır. Tagged nükleotit Floresans kaydedildiğinde eklenen nükleotit algılanması sağlanır. Bu sınırlı teknik kaynaklar gerektirir, taşınabilir, uzun sıralama okuma üretir, önceden hiçbir DNA amplifikasyon için ve düşük bir maliyetle diğer yöntemlerine göre işletilen NS diğer yandan eşsiz avantaja diğer yöntemler üzerinde sahiptir. Öğrencilerimiz olmak basit ve mükellef bir üç saat laboratuvar sınıf Kütüphane daha yeni, hızlı hazırlık Protokolü bulundu. Bazı karşılaştığımız sorunları kaldırmak zor olan akış hücre kabarcıkları, önemli bilgisayar güç (depolama bir terabayt) gerekli, alınan geçerli veri FAST5 dosyasında çıktı, ve sıralama akışı hücre daha önce sınırlı bir raf ömrü vardır bozulur. Ayrıca, NS ligasyonu sıralama Protokolü (uzun protokol) diğer dezavantajları olduğunu o, birkaç Kütüphane hazırlık adımları gerektirir moleküler biyoloji teknikleri uzmanlık gerektirir ve ne zaman bazı göre azalan sıralama sadakat oluşturur sıralama yöntemleri3. Ancak, son gelişmeler yeni hızlı Kütüphane hazırlık kiti ile sadece 10 dakika için kütüphane hazırlık gerektirir ve azalan sıralama hata oranı göstermiştir. Yeni kitaplık hazırlama yöntemi bir laboratuar sınıfı kullanmak için çok müsait.
Akış hücre QC özellikle iletişim kuralında, çeşitli kritik adımlar vardır. Bu ilk bir QC akışı hücre makbuzu beş gün içinde yapmak ve bunları 8 hafta içinde kullanarak içerir. Her ne kadar biz-si olmak kullanılmış 8 hafta olduğunu akışı hücreleri, aç/etkin gözenekleri sayısı önemli ölçüde azalır. Bu deneyler nerede en fazla kullanımı akış hücrelerinin elde bir zaman çizelgesi sığacak şekilde planlanmaktadır önemlidir. Biz temizlik protokolü kullanılan ve başarı ile akış hücreleri yeniden.
Toprak temsil mikrobiyal çeşitlilik kullanılmayan bir genetik rezervuarı metagenomics araştırıyor. Örneğin, toprak bir gram arasında 10 içeren yaklaşık7 - 109 prokaryotik hücreler14. Ayrıca, toprak organizmalar roman doğal ürünler, enzimler ve antibiyotik ana kaynağı vardır. Böylece, toprak metagenomics DNA dizi analizi eğitimin her seviyedeki öğrencilere değerli bir öğretim araç temsil eder. NS teknolojinin kolaylığı kullanım ve düşük maliyetli olun bu sistemi çok etkili öğretim aracı. Öğrenciler çevre örnekleri sıra ve sırası tamamlandıktan sonra tanımlamak ve test örnekleri mikroplar ve metagenomics serilerinde karakterize için kullanılabilir Biyoinformatik araçlarını kullanın. NS teknolojisini kullanarak, öğrenciler kadar dışarı-in şimdi laboratuar derslerinde kullanım için gelişmiş teknik uzmanlık ve diğer sıralama platformlarda yüksek reaktif, ekipman ve bakım maliyetleri nedeniyle ulaşmak vardır gerçek eller deneyimi var. Son zamanlarda, biri (J. Harrison, kişisel iletişim) çiftlik toprak, çevre izleme projesinde bu teknolojinin kullanımı bildirdi. Biz çok daha fazla uygulama için bu teknoloji eğitim alan içinde olacak bekliyoruz.
Açıklamalar
Yazarlar hiçbir açıklamalar var.
Teşekkürler
Bu proje destek kısmen Johns Hopkins Üniversitesi, Provost Office tarafından yapıldı Ağ Geçidi bilim girişimi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Thermal Cycler | LifeECO | BTC42096 | |
| Covaris g-TUBE | Covaris | 520079 | |
| NEBNext End Repair Module | New England BioLab | E7546 | |
| Eppendorf LoBind Centrifuge Tubes | Sigma-Aldrich | Z666505 | |
| NEB Blunt/TA Ligase Master Mix | New England BioLab | MO367 | |
| MyOne C1 Strepavidin Beads | Thermo Fisher | 65001 | |
| Eppendorf microcentrifuge | Eppendorf | Model 5420 | |
| Nanodrop UV spectrophotometer | Thermo Fisher | ND-2000 | Model 2000 |
| Belly Dancer Orbital Shaker | Sigma-Aldrich | Z768499 | |
| Power Soil DNA Isolation Kit | MO BIO | 12888-50 | |
| Ligation Sequencing Kit 2D | Oxford Nanopore | SQK-LSK208 | |
| Rapid Sequencing Kit for Genomic DNA | Oxford Nanopore | SQK-RAD002 | |
| End-Prep Reaction Buffer and enzyme mix (NEB Blunt/TA Ligase Naster Mix) | New England BioLab | MO367 | |
| AmPure XP Magnetic Beads | Beckman Coulter | A63880 | |
| Basic Starter Pack | Oxford Nanopore | Includes MinION Sequencing Device and Flow Cell | |
| MinKNOW software | Oxford Nanopore | ||
| Rapid Sequencing Kit for Genomic DNA | Oxford Nanopore | SQK-RAD002 | Includes Running Buffer with Fuel (RBF), Fragmentation Mix (FRM) Rapid Adapter (RAD) Library Lodaing Beads (LLB) |
Referanslar
- Quick, J., et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
- Greninger, A. L., et al. Rapid metagenomics identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome Med. 7, 99 (2015).
- Quick, J., et al. Rapid draft sequencing and real-time nanopore sequencing in a hospital outbreak of Salmonella. Genome Biology. 16, 114 (2015).
- Ashton, P. M., et al. MinION nanopore sequencing identifies the position and structure of a bacterial antibiotic resistance island. Nat Biotechnol. 33, 296-300 (2015).
- Loman, N. J., Quick, J., Simpson, J. T. A complete bacterial genome assembled de novo. using only nanopore sequencing data. Nat Method. 12, 733-735 (2015).
- Hoenen, T., et al. Nanopore Sequencing as a Rapidly Deployable Outbreak Tool. Emerg Infect Dis. 22 (2), 331-334 (2016).
- Petrosino, J. F., Highlander, S., Luna, R. A., Gibbs, R. A., Versalovic, J. Metagenomic pyrosequencing and microbial identification. Clin Chem. 55 (5), 856-866 (2009).
- Cummings, P. J., Ahmed, R., Durocher, J. A., Jessen, A., Vardi, T., Obom, K. M. Pyrosequencing for Microbial Identification and Characterization. J. Vis. Exp. (78), e50405 (2013).
- Rothberg, J. W., et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., et al. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Stoddart, D., Heron, A. J., Mikhailova, E., Maglia, G., Bayley, H. Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. PNAS. 106, 7702-7707 (2009).
- Shendure, J., Hanlee, J. Next generation DNA sequencing. Nat Biotechnol. 26, 1135-1145 (2008).
- Daniel, R. The Metagenomics of Soil. Nat Rev Microbiol. 3, 470-478 (2005).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır