Method Article
Nanopore DNA-Sequenzierung für metagenomische Bodenanalyse
In diesem Artikel
Zusammenfassung
Nanopore Technologie für Sequenzierung Biomoleküle hat breite Anwendungen in den Lebenswissenschaften einschließlich der Identifizierung von Krankheitserregern, Lebensmittelüberwachung Sicherheit, Genomanalyse, metagenomic Umweltüberwachung und Charakterisierung von bakteriellen Antibiotika-Resistenz. In diesem Artikel wird das Verfahren für die metagenomische Boden DNA-Sequenzierung zur Speziesidentifizierung mit Nanopore Sequenzierung Technik demonstriert.
Zusammenfassung
Dieser Artikel beschreibt die Schritte zum Aufbau einer DNA-Bibliothek von Boden, Zubereitung und Verwendung der Durchflusszelle Nanopore und Analyse von DNA-Sequenzen identifiziert mittels Computersoftware. Nanopore DNA-Sequenzierung ist eine flexible Technik, die ermöglicht schnelle mikrobielle Genomsequenzierung, bakterieller und viraler Spezies zu identifizieren um Bakterienstämme zu charakterisieren und zu genetische Mutationen zu erkennen, die Resistenz gegenüber Antibiotika verleihen. Nanopore (NS) für Life Sciences-Sequenzierung Vorteile seine geringe Komplexität, reduzierte Kosten und schnelle Echtzeit-Sequenzierung des gereinigten genomischer DNA, PCR-Amplifikate, cDNA Proben oder RNA. NS ist ein Beispiel für "Strang Sequenzierung" einhergehende DNA Sequenzierung durch die Führung von stranded DNA Moleküls durch eine Nanopore, die in eine synthetische Polymermembran eingesetzt wird. Die Membran hat einen elektrischen Strom angewendet über es, so wie die einzelnen Basen durch die Nanopore den elektrischen Strom in unterschiedlichem Maße durch die vier Nukleotidbasen gestört wird. Die Identifizierung der einzelnen Nukleotid erfolgt durch den Nachweis der charakteristischen Modulation des elektrischen Stroms durch die verschiedenen Basen als Durchgang durch die Nanopore. Das NS-System besteht aus einem handheld, Stromversorgung über USB portable Gerät und ein Einweg-Messzelle, die ein Nanopore Array enthält. Das tragbare Gerät steckt in einem standard-Notebook-Computer, der liest und zeichnet die DNA-Sequenz mit Computersoftware.
Einleitung
Das Ziel dieses Verfahrens ist für die Vorbereitung einer ökologischen DNA-Bibliothek für die Sequenzierung, Nutzung eines Nanopore Fluß Zelle Sequenzierung Geräts erforderlichen Schritte demonstrieren und die generierte DNA-Sequenzen mit Systemsoftware analysieren und das National Center for Biotechnology Information (NCBI) Bioinformatik, mikrobielle Spezies im Boden zu identifizieren. Derzeit verlangen die meisten DNA-Sequenzierung-Plattformen eine große Investition in technische Schulungen und komplexe Instrumentierungen, was nicht machbar in Ressource armen Umgebungen oder im Feldeinsatz ist. Die Nanopore-Sequenzierung (NS)-Plattform beseitigt diese Probleme mit einer kostengünstigen, einfach zu bedienen, Protokoll Bibliothek Vorbereitung und einem tragbaren Gerät, zu sequenzieren und analysieren eine Vielzahl von verschiedenen Arten von Nukleinsäuren1,3. Wir haben mehrere Labor-Klassen für Master-Studierende die NS-Plattform integriert.
Nanopore Technologie für Sequenzierung Biomoleküle hat breite Anwendungen in den Lebenswissenschaften, einschließlich Ermittlung der bakterielle und virale Erreger1,2,6, ökologischen Biodiversität gezeigt. Studien, Lebensmittel-Sicherheit monitoring, Genomanalyse3,5und Charakterisierung von bakteriellen Resistenz gegen Antibiotika4. NS ist eine schnelle und genaue Methode zur Sequenz Nukleinsäuren, die auf dem Prinzip der basiert "Sequenzierung Strang" durch das Erkennen von elektrischen Störungen durch individuelle Nukleotidbasen wenn einzelne stranded DNA eine Nanopore in einem elektrifizierten eingefügt durchläuft synthetische Polymermembran. Die Schritte bei der Vorbereitung von DNA für NS 3' dA-Tailing genomische Fragmente, Adapter und Haltegurt Glühen an die DNA, Genom Fragmentierung und Ende Reparatur enthalten DNA Bibliothek Reinigung und die Bibliothek in der Nanopore-Flow-Zelle-Gerät laden. Zersplitterung des Genoms in ~ 8 kb Größe wird durch Zentrifugieren 1-2 µg genomische DNA durch ein g-Tube Fragmentierung Rohr erreicht. Die fragmentierten genomischen Enden sind dann repariert und angebundene mit Poly-dA mit einem kommerziell erhältlichen Kit. Einzelnen gestrandeten Adapter Sequenzen, die kompatibel mit dem Nanopore motor Protein sind, werden hinzugefügt, DNA-enden, die verwendet werden, um die DNA-Sequenz durch die Nanopore (Abbildung 1) zu führen. Der Haltegurt-Sequenzen sind für DNA-Reinigung und zur Lokalisierung der DNA-Moleküle an die Pore Membran erforderlich. Die Haarnadel entsteht durch Ligation einen Haarnadel-Adapter an einem Ende der dA tailed Bibliothek. Die Haarnadel-Strukturen an den Enden der DNA erlaubt das Auslesen der Sense und Antisense Stränge wie die DNA durch die Nanopore (Abbildung 2). Die vorbereitete genomische Bibliothek wird dann aus der Reaktion gereinigt, mit Streptavidin-Perlen mit einem magnetischen Feld, gefolgt von der Probe in die Nanopore Messzelle für die Analyse zu laden.
Die sequenzierte DNA bemisst sich für Qualität und Sequenzierung liest, die akzeptabel für die Analyse sind unterliegen dann mehrere Bioinformatik, Mikroben zu identifizieren. Die Sequenzen sind in einer FASTQ aus einem FAST5 Format "übersetzt". Im FASTQ-Format können die Sequenzen dann BLAST Analyse genutzt werden.
Protokoll
Hinweis: Metagenomische DNA wird aus dem Boden (Baltimore County, Maryland) mit einer im Handel erhältlichen Boden genomische Isolation gereinigt kit (siehe Tabelle der Materialien). Mit einer UV-Spektralphotometer (siehe Tabelle der Materialien), die gereinigte genomische DNA sollte haben ein 260/280 (nm) Verhältnis > 1,8 und 260/230 Verhältnis zwischen 2.0-2.2 um sicherzustellen, dass die Probe ist frei von Verunreinigungen. Die Höhe der genomischen DNA erforderlich für NS reicht von 200 ng zu 2 µg.
1. schnelle Bibliothek Zubereitungsart (kurzes Protokoll)
Hinweis: Dies ist ein kurzes Protokoll. Sehen Sie Tabelle der Materialien für das schnelle Sequencing Kit.
- In einem 0,2 mL dünnwandigen Rohr (siehe Tabelle der Materialien) addieren Sie 200 ng des hohen Molekulargewichts DNA in einem Finale des Volumens von 7,5 µL destilliertes Wasser.
- Fügen Sie 2,5 µL Fragmentierung Mix (FM) aus dem schnellen Bibliothek Vorbereitungssatz und Mischung sanft durch Umkehrung. Puls-Zentrifuge nach unten drehen (1.000 x g für 5 s).
- Legen Sie die Probe in einem Thermocycler (siehe Tabelle der Materialien) für eine Runde bei 30 ° C für 1 min, gefolgt von 75 ° C für 1 min. entfernen die Rohr- und Puls Zentrifuge, die Probe zu spinnen.
- Fügen Sie 1 µL schnelle Adapter und 0,2 µL Blunt/TA-Ligase-master-Mix, und speichern Sie die angepasste und gefesselte Bibliothek auf Eis.
Hinweis: Wenn mit dem kurzen Protokoll mit Schritt 8 fortfahren.
(2) Ligation Sequenzierung Protokoll (lang): Metagenomische DNA-Fragmentierung
Hinweis: Siehe Tabelle der Materialien für die Ligatur Sequencing Kit.
- Verdünnen Sie 1 µg gereinigten genomic DNA zu einem Endvolumen von 46 µL in entionisiertem Wasser.
- Übertragung die Probe zu einer DNA-Fragmentierung Rohr (siehe Tabelle der Materialien) und für 1 min bei Raumtemperatur bei 8.000 x g mit einem Microcentrifuge Zentrifugieren (siehe Tabelle der Materialien), ca. 8 kb genomische Fragmente zu produzieren. Stellen Sie sicher, dass die Flüssigkeit in dem Sammelrohr bestanden hat.
- Invertieren Sie die Fragmentierung Röhre, Rückkehr in die Zentrifuge und Zentrifuge für 1 min die fragmentierten genomischen DNA in die untere Kammer zu sammeln.
- Die fragmentierten genomischen DNA an eine sterile Low-DNA-Bindung-1,5 mL-Tube übertragen, und einen Teil auf einem niedrigen Prozentsatz Agarose-Gel (0,6 %) um Fragmentierung zu bestätigen zu analysieren > 30 kb.
(3) fragmentiert genomische DNA-Ende-Vorbereitung
- Mit 800 ng fragmentierten genomic DNA in 45 µL deionisiertes Wasser hinzufügen 7 µL-Ende-Prep-Reaktion (siehe Tabelle der Materialien), 3 µL Enzym-Mix-Puffer (siehe Tabelle der Materialien), und 5 µL Nuklease Wasser frei.
- Mix von Inversion und Puls Zentrifuge (1.000 x g für 5 s).
- Probe auf einem 0,2 mL dünnwandigen Rohr übertragen und Probe für 5 min bei 20 ° C, gefolgt von Inkubation bei 65 ° C für 5 min inkubieren.
- Puls-Zentrifuge, Inhalt auf den Boden des Rohres zu bringen.
- Übertragen Sie Probe auf einem 1,5 mL niedrig-DNA-Bindung Microcentrifuge Schlauch.
- Bereiten Sie magnetische Beads (siehe Tabelle der Materialien). Fügen Sie 60 µL resuspendierte Perlen der Ende Prep Reaktion aus Schritt 3.5 und Mix durch pipettieren.
- Auf einem Rotator-Mixer bei 100 u/min (siehe Tabelle der Materialien) für 5 min bei Raumtemperatur inkubieren.
- Pulse Zentrifugieren die Probe zu pellet-Perlen, dann legen Sie die Probe neben Magnet.
- Sobald die Perlen der Magnet eingehalten werden, pipette überstand und verwerfen.
- Entfernen Sie das Rohr aus Magnet und waschen Perlen mit 200 µL von frisch zubereiteten 70 % Ethanol durch Pipettieren sanft auf die Probe.
- Puls-Zentrifuge, pellet-die Perlen und der Magnet wieder.
- Den Überstand entfernen und entsorgen. Wiederholen Sie den Waschschritt erneut mit 200 µL 70 % Ethanol.
- Puls die Röhre, Rückkehr zu den Magneten Zentrifugieren und alle 70 % Ethanol waschen zu entfernen.
- Öffnen Sie mit dem Microcentrifuge Schlauch Deckel, lassen Sie die Perlen an der Luft trocknen für 5 min bei Raumtemperatur.
- Entfernen Sie das Rohr aus Magnet und auszusetzen Sie das Pellet in 31 µL steriler, Nuklease-freie Wasser. 2 min bei Raumtemperatur inkubieren.
- Zurückkehren Sie das Rohr zu den Magneten, bis die Perlen oelletiert haben und das Eluat klar ist.
- Übertragen Sie das Eluat auf einem 1,5 mL Microcentrifuge Schlauch. Mit 1 µL der Probe, quantifizieren die Ende vorbereitet DNA mit einem UV-Spektralphotometer bei 260 nm (siehe Tabelle der Materialien).
Hinweis: Sollte die prozentuale Erholung ~ 70 % (700 ng) aus einer Start Konzentration von 1 µg genomische DNA.
(4) Adapter und Tether Ergänzung zu Ende vorbereitet genomische DNA-Fragmente
- Mischen der Blunt/TA-Ligase Röhren master-Mix durch Umkehrung, und dann Puls Zentrifuge Inhalt auf den Boden zu bringen.
- Fügen Sie mithilfe 30 µL DNA Ende vorbereitet-20 µL des Adapter-Mix und 50 µL Blunt/TA-Ligatur-master-Mix.
- Durch Umkehrung, Puls-Zentrifuge, mischen und 10 min bei Raumtemperatur inkubieren.
(5) magnetische BEad Vorbereitung
- Vortex tube Perlen und dann Transfer 50 µL des suspendierten Perlen zu einer 1,5 mL Microfuge.
- Platzieren Sie das Rohr auf den Magneten zu die Perlen Pellets bis das Eluat genehmigt.
- Den Überstand entfernen und entsorgen.
- 100 µL der Wulst Bindung Puffer hinzufügen (siehe Tabelle der Materialien für Bead-Kit), die Perlen, Wirbel, Perlen, Aufschwemmen pellet die Perlen auf den Magneten, den Überstand entfernen und entsorgen. Wiederholen Sie das Waschen mit 100 µL Bindung Puffer.
- Die gewaschenen Perlen 100 µL der Wulst Bindung Puffer hinzufügen. Wirbel um die Perlen aufzuwirbeln.
6. Bibliothek Reinigung
- Fügen Sie mithilfe einer Mikropipette P200 40 µL Perlen aus dem vorherigen Schritt zur genomischen DNA angepasst/angebunden. Mischen Sie die Probe durch Pipettieren sorgfältig.
- Inkubieren Sie die Probe auf einem Rotator-Mixer bei 100 u/min für 5 min bei Raumtemperatur.
- Legen Sie die Probe auf den Magneten und lassen Sie die Perlen zu begleichen. Pipette aus der Überstand und entsorgen.
- Die Perlen in 140 µL Wulst Bindung Puffer durch Pipettieren aufzuwirbeln. Legen Sie die Probe gegen den Magneten, den Überstand verwerfen und waschen wieder mit einer anderen 140 µL Wulst Bindung Puffer.
- Puls Zentrifugieren Sie das Rohr, das Rohr auf den Magneten zu platzieren, für 2 min. entfernen den restlichen Wulst Bindung Puffer aus dem Pellet.
7. die Elution der Bibliothek von Magnet-Perlen
- Aufschwemmen der magnetischen Beads in 15 µL Elution Puffer durch pipettieren. Inkubieren Sie die Probe für 10 min bei Raumtemperatur.
- Legen Sie das Rohr gegen den Magneten zu die Perlen Pellets.
Hinweis: Sollte die prozentuale Erholung ~ 25 % (250 ng) aus einer Start Konzentration von 1 µg genomische DNA.
8. starten eine Run/Qualitätskontrolle
- Messzelle aus der Verpackung nehmen und portable, Echtzeit-Sequenzer die Durchflusszelle zuordnen (siehe Tabelle der Materialien).
- Sequencer über USB-Kabel an einen Computer anschließen und die Sequenzer-Software zu starten (siehe Tabelle der Materialien).
- Klicken Sie auf "Connect" Gerät durch die Sequenzer-Software, wählen Sie "NC_Platform_QC.py", und klicken Sie auf "Start".
- Die Qualitätskontrolle (QC) dauert ca. 6-7 min. in Anspruch; Suchen Sie nach einem "Sea of Green" (große Flächen des Ausgangs) auf die QC-Anzeige zu bestätigen, dass es genügend aktive Poren (> 800) für die DNA-Sequenzierung.
9. starten einen Sequenzierung Run
- Öffnen Sie die Sequenzierung Programm; ein Dialogfeld wird angezeigt. Proben-ID im Feld name der Probenmaterials.
- Klicken Sie auf das Feld "Flow Cell-ID" und geben Sie den Code auf dem Aufkleber auf der Oberseite der Durchflusszelle gefunden.
- Den Fluss Zelle Deckel öffnen und dann schieben Sie die Probe-Port-Abdeckung nach rechts (im Uhrzeigersinn) so dass die Probenöffnung sichtbar ist. Wenn der Port geöffnet, überprüfen Sie für eine kleine Blase ist. Wenige Mikroliter der Puffer einschließlich der Blase zu entfernen.
- Vergewissern Sie sich, dass über das Sensor-Array gibt es Puffer. Machen den Priming-Puffer durch das Mischen von 480 µL Puffer mit Kraftstoff-Gemisch ausgeführt (RBF-1, siehe die Tabelle der Materialien) (Achten Sie darauf, mischen Sie gründlich erste) mit 520 µL Nuklease-freies Wasser.
- Die Grundierung Hafen 800 µL des Puffers Priming hinzufügen. Heben Sie vorsichtig den Deckel "SpotOn" zugänglich zu machen.
- Fügen Sie nach 5 min eine zusätzliche 200 µL Puffer Grundierung zu Priming-Port hinzu.
10. Laden der Bibliothek
- Vor der Vorbereitung des Bibliothek Ortes der folgenden Reagenzien auf Eis: RBF-1 (aus dem Sequencing Kit), angepasst und angebunden, und Bibliothek laden Perlen (LLB; aus dem Sequencing Kit).
- Eine 0,2 mL Zentrifugenröhrchen fügen Sie hinzu, 25,5 µL RBF und 12 µL Nuklease-freies Wasser bei Raumtemperatur aufbewahrt.
- Mischen Sie die LLB durch Pipettieren wechselvollen. Das 0,2 µL Rohr 26,5 µL der LLB hinzufügen. Mischen Sie die Reagenzien durch Pipettieren wechselvollen.
Hinweis: Die LLB neigt dazu, aus der Mischung zu begleichen. - Das 0,2 µL Rohr 11 µL der angepasst und gefesselte Bibliothek hinzufügen. Abmischen von Inversion und Spin durch Zentrifugation Puls.
- Fügen Sie 75 µL Probe aus Schritt 9.4 tropfenweise an den "SpotOn"-Port. Stellen Sie sicher, dass jeder Tropfen in den Hafen fließt, bevor Sie den nächsten hinzufügen.
- Sorgfältig ersetzen Sie die Probe-Port-Abdeckung zu, so dass der Spund der Hafen einläuft, und setzen Sie den Deckel des Gerätes.
11. eine Sequenzierung Software Protokoll Skript gestartet wird
- Öffnen Sie das Dropdown-Menü unter "Select-Programm" und wählen Sie "48 Stunden Sequenzierung."
- Klicken Sie auf die Schaltfläche "Ausführen" um das Skript zu starten.
- Überprüfen Sie die Ergebnisse des scan-Mux in der Benachrichtigung-Stream für die Anzahl der aktiven Poren in der scan-MUXberichtet.
Hinweis: Diese Zahl kann abweichen von der Anzahl der Poren in den QC laufen und ein großen Unterschied kann auf ein Problem hinweist.- Wenn es eine signifikante Reduktion der aktiven Poren aus dem QC-Lauf starten Sie die Software, und wenn es noch ein signifikanter Unterschied, den Computer neu starten.
- Überprüfen Sie, dass die Kühlkörper-Temperatur 34 ° C ist, wie die Sequenzierung Software Skript berichtet.
Hinweis: Wenn die Temperatur nicht bei 34 ° C ist dann es nicht genügend aktive Poren für den Lauf. - Überprüfen Sie das Histogramm lesen Sie Längen zu bestimmen, ob die Sequenz Längen erwarteten und für den experimentellen Entwurf geeignet sind.
- Schließen Sie die Desktop-Agent mithilfe von "x. beenden die Sequenzer-Software in der Nähe" schließen Sie die Web-GUI. Trennen Sie das Gerät vom Computer.
12. Analyse ausführen und Ergebnisse
Hinweis: Während der Sequenzierung Ausführung, können die Daten überwacht werden mit dem "Bericht anzeigen"-Programm, mit dem Desktop-Agent. Während oder nach Abschluss des Laufs Dateien stehen in einem FAST5 Dateiformat, in dem Daten → liest → pass Ordner (Standard). Wenn dieser Ordner geöffnet ist, während Sequenzierung aktiv ist, kann der Computer fixieren.
- Zum ansehen und bearbeiten Sequenzen, ein "HDFViewer" herunterladen.
- Um die Sequenzierung Dateien anzuzeigen, direkt zu öffnen die HDF Viewer → offene Daten Ordner → wählen Sie, die alle Datei-Typen → wählen Sie C: Laufwerk (Standard) → Daten auswählen → Wählen Sie liest → select Pass.
- Finden Sie und wählen Sie die FASTQ-Datei im linken Drop-Down-Menü; Dateien werden geöffnet und können im FASTQ-Format gespeichert werden.
Hinweis: Für die Zwecke der Student Experiment wurden Sequenzen von 350 Nukleotide oder länger zur weiteren Analyse ausgewählt. Ausgang aus der Basis Berufung ermöglicht dem Benutzer, Sequenz liest basierend auf Größe zu sortieren. FASTQ-Dateien können über BLAST (NCBI) oder anderen Bioinformatik-Programme analysiert werden.
Ergebnisse
Die experimentelle Design und Nanopore Technologie zur Verfügung gestellt, einer schnelle und kostengünstigen Methode für Studierende zu Boden DNA-Sequenz. Die repräsentative Ausführung übergeben der Qualitätskontrolle Parameter mit mehr als 800 Poren zur Sequenzierung. Der Lauf führte über 125.000 liest, die für das Studium mit einer medianen Sequenz Länge von 5,38 kb. Sequenzen sind ein Qualitätsfaktor und nur diese Sequenzen mit akzeptablen Resultate wurden dann analysiert. Als Ausgang für die Sequenzierung ist die Reaktion in FAST5 Format, die nicht von Programmen wie BLAST (NCBI), akzeptiert die Sequenzen im HDF Viewer angesehen wurden, die die Sequenzen in FASTQ Format wandelt, die kompatibel zur Explosion Analyse ist. In Zukunft werden Iterationen der Software, die Daten als FASTQ Format, wodurch die Notwendigkeit für die HDF-Viewer zur Verfügung. Siehe ergänzende Abbildungen 1, 2 und 3.
Fünfzig Sequenzen von über 350 Nukleotide in der Länge waren Gegenstand der Analyse von BLASTN (Nukleotide Nukleotid-Datenbank) oder SEQUENZIERT (Nukleotide in Aminosäuresequenz übersetzt) (NCBI), Organismen in der Bodenprobe zu identifizieren. In Anbetracht der Zeit Zwänge der Klasse und, dass der Schwerpunkt des Kurses auf Labormethoden und nicht Bioinformatik, wir haben die Studenten Sequenzen innerhalb dieser Parameter zu analysieren. Unsere Bioinformatik-Klassen können die Daten für die Entwicklung des Pipeline-Analyse-Tools verwenden. Dieser Analyseebene Bioinformatik fällt nicht in die Labor-basierte Klasse. Tabelle 1 finden Sie eine Liste der Organismen, die mit e-Werte von weniger als 4 e-04 identifiziert wurden. Allgemein, e-Werte von weniger als 4 e-04 zeigen starke Ähnlichkeit zwischen der Eingabesequenz (Abfrage) und die aufeinander abgestimmten Reihenfolge. Die Organismen gekennzeichnet durch BLASTN (gegen die nicht-redundanten (nr)-Datenbank) sind aus einer Vielzahl von Arten und stehen für weitere genomische und Protein-Analysen. In diesem Beispiel des Bodens, die aus einem Garten in Baltimore County, MD kam war nie getestet worden, so gab es keine vorherigen Daten aus, um festzustellen, was Organismen im Boden sein könnte. BLASTN Analyse (anhand der nr-Datenbank) die Analyse ergab auch, gab es Sequenzen keine Ähnlichkeiten in der nr-Datenbank. Um festzustellen, ob dies wirklich neuartige Sequenzen sind, ist weitere Studien erforderlich. Analyse der mehrere Sequenzen von SEQUENZIERT ergab mehrere Proteine von Organismen, die nicht in der BLASTN Analyse dargestellt. Tabelle 2 listet einige mögliche Proteine einschließlich Endopeptidasen und ATPasen-ähnliche Proteine. Mit dieser Bibliothek von Sequenzen, haben Studenten die Möglichkeit, komplexere Bioinformatik Tools verwenden, um weitere Analyse durchführen, wenn der Kursleiter die Regie.
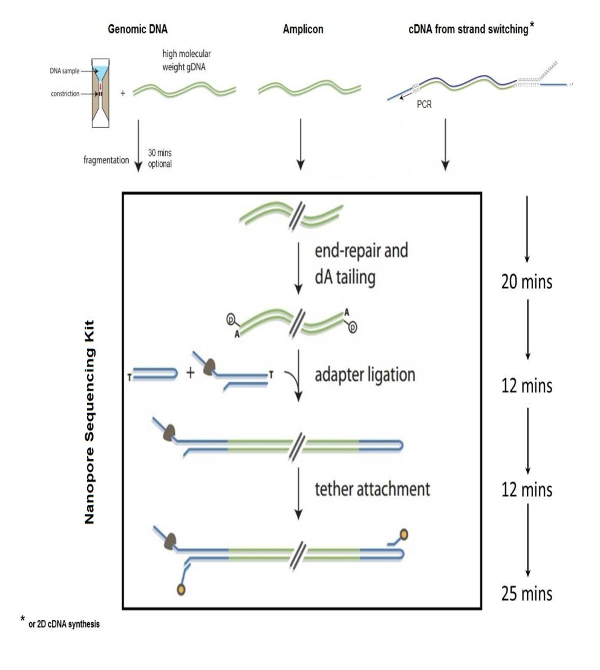
Abbildung 1 : Genomic Bibliothek Vorbereitung. Schritte zur Vorbereitung der genomischen DNA-Bibliothek für die Sequenzierung mit Nanopore Technologie. Diese Zahl wurde mit den erforderlichen Berechtigungen geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2 . Schaltplan der DNA-Sequenzierung mit einer Nanopore. DNA, die durch eine Nanopore Membran mit elektrisches Signal Erzeugung und Basis-Identifikation. Diese Zahl wurde mit den erforderlichen Berechtigungen geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Organismus | e-Wert |
| Streptomyces-sp. | 3.00E-06 |
| Nocardoides sp. | 5.00E-04 |
| Gordonia sp. | 5.00E-04 |
| Hyphomicrobium nitrativorens | 1.00E-139 |
| Starkeya-Novelle | 1.00E-31 |
| Hyphomicrobium denitrificans | 1.00E-30 |
| Fibomicrobium sp. | 5.00E-17 |
| Pseudomonas sp. | 2.00E-15 |
| Rhodothemus marinus | 1.00E-17 |
| Turneiella parva | 2.00E-24 |
| E. coli | 0.00E + 00 |
| Bradyrhizobium sp. | 3.00E-30 |
| Gemmatirosa kalamazoonesis | 1.00E-20 |
| Burkholderia sp. | 8.00E-10 |
| Sphingomonas sp. | 8.00E-10 |
| Cellumonas sp. | 6.00E-11 |
Tabelle 1: ausgewählte Ergebnisse der BLASTN Analyse. Boden Metagenomik DNA-Sequenzen BLASTN Ähnlichkeitssuche anhand der NCBI-nicht-redundanten Nukleotid-Datenbank unterzogen. Die gemeldeten Daten haben e-Werte von 4 X e-4 oder weniger.
| Protein | Organismus |
| Hypothetische protein | Acidobacter Bakterium |
| SAM-abhängige-methyltransferase | H. denitrificans |
| ATPase | Jannaschia sp. |
| Endopeptidase | Shigella sonnei |
| Lyse protein | E. coli |
Tabelle 2: ausgewählte Ergebnisse der Analyse SEQUENZIERT. Boden Metagenomik DNA-Sequenzen SEQUENZIERT Ähnlichkeitssuche anhand der NCBI-nicht-redundanten Protein-Datenbank unterzogen.
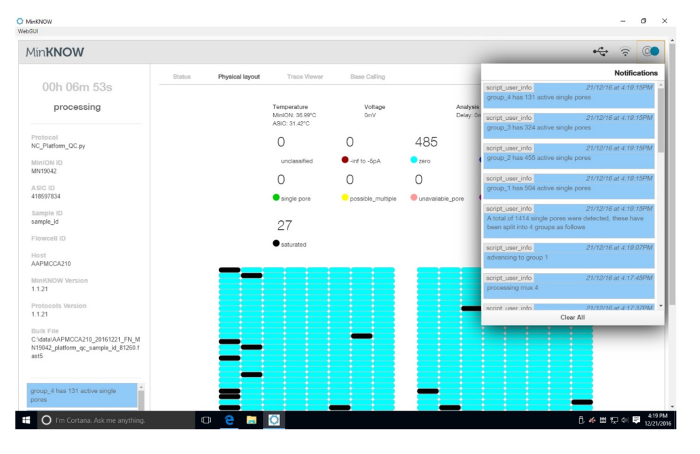
Ergänzende Abbildung1: Plattform QC Ergebnisse. Die Ergebnisse der QC-Plattform. In der oberen rechten Ecke sind die Ergebnisse des QZ für aktive Poren. Jeder Quadrant der Durchflusszelle ist für aktive Poren getestet. Die Hauptseite ist dynamisch und ändert sich wie jeder Quadrant getestet wird. In diesem Lauf waren 1.414 einzelne Poren erkannt.Bitte klicken Sie hier, um eine größere Version dieser Figur.
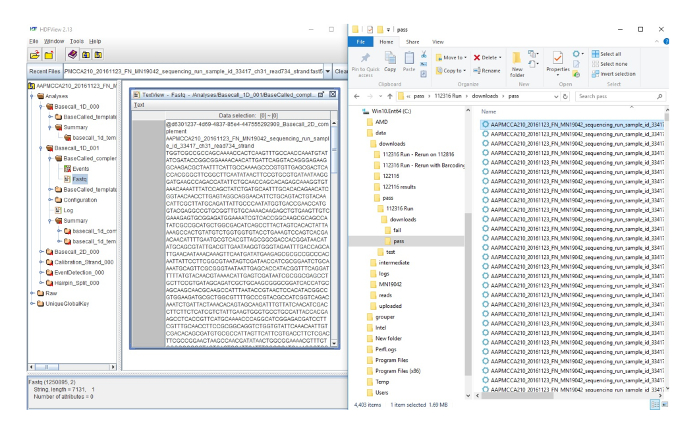
Ergänzende Abbildung2: zu FASTQ Dateien Dateien konvertieren FAST5. Hier ist auf der rechten Seite die Ausgabe der Daten aus der Sequenzierung Flucht. Jede Zeile repräsentiert eine einzelne Sequenz. Die linke Seite der Abbildung zeigt die markierte Sequenz von der rechten Seite im HDF Viewer, der die Sequenz in eine FASTQ-Datei umwandelt, die zur weiteren Analyse verwendet werden können. Bitte klicken Sie hier für eine größere Version dieser Figur.
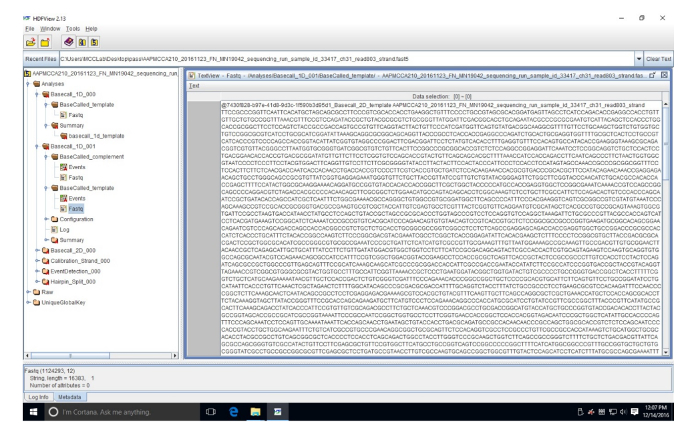
Ergänzende Abbildung 3: Beispiel FASTQ Sequenz im HDF Viewer. Diese Sequenz (gelesen 803) ist in eine FASTQ-Datei, die Nukleotide FAST5 Daten umwandelt. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Viele nächste Generation Sequenzierung Methoden erzeugt wurden und jedes hängt von Sequenzierung durch Synthese aber Erkennung Plattformen zur Identifizierung von Nukleotide unterscheiden. NS, die jüngste Ergänzung zum Marktplatz, verwendet eine andere Methode völlig, die erfordert keiner Sequenzierung Reaktion oder Nukleotide bezeichnet. Diese Methode nutzt kostenlos Differential bereits eingebaute Nukleotide eine elektrifizierte Pore durchschreiten. Die Identifizierung der einzelnen Nukleotid erfolgt durch Modulation des elektrischen Stroms durch die verschiedenen Basen als Durchgang durch die Nanopore. Dieses gebündelte System ermöglicht dem Benutzer viele Fragmente zu einem Zeitpunkt zu sequenzieren. Durch die beiden Stränge der DNA-Sequenzierung, die Genauigkeit der Sequenz wird deutlich erhöht und die Sequenzer-Software, die auf einem Laptopcomputer geladen werden kann, verarbeitet die Signale und die Sequenz informiert, die analysiert werden können.
Detektion der spezifischen Basis in die Vorlage aufgenommen hängt Pyrosequenzierung, eine Abfolge von Synthese (SBS)-Methode, die Luciferase Assay und der Erzeugung von Chemolumineszenz Signale8. In Ion Halbleiter Sequenzierung wird die freigesetzte Wasserstoff-Ionen, die den pH-Wert sinkt durch eine Ionen-Sensor9erkannt. Echtzeit-Sequenzierung Einzelmolekül richtet sich nach der Null Modus Welle Führer (ZMW)10, die für die Erkennung ein fluoreszierendes Molekül markiert, die eingebaute Nukleotid beleuchtet. SBS verwendet eine einzigartige Methode, um die Ziel DNA zu verstärken, so dass einzigartige Sequenzen generierten13sind. Erkennung des zusätzlichen Nukleotids wird erreicht, wenn die Fluoreszenz des tagged Nukleotids aufgezeichnet wird. NS hat auf der anderen Seite einzigartige Vorteil gegenüber anderen Methoden, dass es begrenzte technischen Ressourcen erfordert, tragbar ist, lange Sequenzierung liest produziert, keine vorherige DNA Verstärkung erfordert und zu einem reduzierten Preis im Vergleich zu anderen betrieben werden kann. Unsere Schüler fanden das neuere, schnelle Bibliothek Prep Protokoll, unkompliziert und offen für eine drei-Stunden-Labor-Klasse sein. Einige der Fragen, denen, die wir begegnet, sind Luftblasen in der Messzelle, die schwer zu entfernen waren, es erforderte erhebliche Rechenleistung (ein Terabyte Speicherkapazität) ist die aktuelle Ausgabe von Daten in einer Datei FAST5 und die Sequenzierung Durchflusszelle hat eine begrenzte Haltbarkeit davor verschlechtert sich. Darüber hinaus weitere Nachteile der NS-Ligation Sequenzierung Protokoll (lange) ist, dass es erfordert mehrere Bibliothek Vorbereitungsschritte, erfordert Erfahrung in molekularbiologischen Techniken und reduzierte Sequenzierung Treue im Vergleich zu einigen erzeugt Sequenzierung Methoden3. Jedoch die jüngsten Fortschritte mit der neuen schnellen Bibliothek Vorbereitungssatz erfordert nur 10 min für Bibliothek Vorbereitung und hat eine reduzierte Sequenzierung Fehlerquote unter Beweis gestellt. Die neue Bibliothek Vorbereitung Methode war sehr zugänglich für den Einsatz in einem Labor-Klasse.
Besonders in den QC der Durchflusszelle gibt es mehrere wichtige Schritte in das Protokoll. Dazu gehören Durchführung einer ersten QC innerhalb von fünf Tagen nach Erhalt der Messzelle und mit ihnen innerhalb von 8 Wochen. Obwohl wir Durchflusszellen, die über 8 Wochen waren verwendet haben, ist die Anzahl der Poren öffnen/aktive stark reduziert. Es ist wichtig, dass Experimente geplant sind, um eine Zeitleiste zu passen, wo maximale Nutzung der die Durchflusszellen erreicht. Wir haben die Reinigung Protokoll verwendet und wiederverwendet die Durchflusszellen mit Erfolg.
Untersuchen die Metagenomik der Erde repräsentieren eine ungenutzte genetisches Reservoir der mikrobiellen Diversität. Z. B. ein Gramm Boden wird voraussichtlich zwischen 10 enthalten7 - 109 prokaryotische Zellen14. Bodenorganismen sind darüber hinaus eine Hauptquelle von neuartigen Naturprodukte, Enzyme und Antibiotika. Daher stellt Boden Metagenomik DNA-Sequenzanalyse ein wertvolles Lehrvideos Instrument für Studierende auf allen Ebenen der Bildung. Der NS-Technologie Benutzerfreundlichkeit und niedrige Kosten machen dieses System sehr effektiv Lehrmittel. Studenten können Sequenz Umweltproben und nach Abschluss der Sequenz zur Verfügung Bioinformatik-Werkzeuge verwenden, um zu identifizieren und zu charakterisieren, Mikroben und Metagenomik-Sequenzen in Proben. Mit der NS-Technologie, haben Studenten echte praktische Erfahrung, die bis jetzt von wegen der fortgeschrittenen technischen Know-how und hohe Reagenz, Ausrüstung und Wartung Kosten in anderen Sequenzierung Plattformen für den Einsatz in Laborübungen erreichen hat. Einer unserer Schüler (J. Harrison, persönliche Mitteilung) berichtete kürzlich, den Einsatz dieser Technologie in ein Umweltprojekt Überwachung der landwirtschaftlichen Böden. Wir rechnen damit, dass viele weitere Anwendungen für diese Technologie im Bereich Bildung.
Offenlegungen
Die Autoren haben keine Angaben.
Danksagungen
Dieses Projekt war teilweise Unterstützung von Johns Hopkins Universität, Büro des Propstes durch die Gateway Science Initiative.
Materialien
| Name | Company | Catalog Number | Comments |
| Thermal Cycler | LifeECO | BTC42096 | |
| Covaris g-TUBE | Covaris | 520079 | |
| NEBNext End Repair Module | New England BioLab | E7546 | |
| Eppendorf LoBind Centrifuge Tubes | Sigma-Aldrich | Z666505 | |
| NEB Blunt/TA Ligase Master Mix | New England BioLab | MO367 | |
| MyOne C1 Strepavidin Beads | Thermo Fisher | 65001 | |
| Eppendorf microcentrifuge | Eppendorf | Model 5420 | |
| Nanodrop UV spectrophotometer | Thermo Fisher | ND-2000 | Model 2000 |
| Belly Dancer Orbital Shaker | Sigma-Aldrich | Z768499 | |
| Power Soil DNA Isolation Kit | MO BIO | 12888-50 | |
| Ligation Sequencing Kit 2D | Oxford Nanopore | SQK-LSK208 | |
| Rapid Sequencing Kit for Genomic DNA | Oxford Nanopore | SQK-RAD002 | |
| End-Prep Reaction Buffer and enzyme mix (NEB Blunt/TA Ligase Naster Mix) | New England BioLab | MO367 | |
| AmPure XP Magnetic Beads | Beckman Coulter | A63880 | |
| Basic Starter Pack | Oxford Nanopore | Includes MinION Sequencing Device and Flow Cell | |
| MinKNOW software | Oxford Nanopore | ||
| Rapid Sequencing Kit for Genomic DNA | Oxford Nanopore | SQK-RAD002 | Includes Running Buffer with Fuel (RBF), Fragmentation Mix (FRM) Rapid Adapter (RAD) Library Lodaing Beads (LLB) |
Referenzen
- Quick, J., et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
- Greninger, A. L., et al. Rapid metagenomics identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome Med. 7, 99 (2015).
- Quick, J., et al. Rapid draft sequencing and real-time nanopore sequencing in a hospital outbreak of Salmonella. Genome Biology. 16, 114 (2015).
- Ashton, P. M., et al. MinION nanopore sequencing identifies the position and structure of a bacterial antibiotic resistance island. Nat Biotechnol. 33, 296-300 (2015).
- Loman, N. J., Quick, J., Simpson, J. T. A complete bacterial genome assembled de novo. using only nanopore sequencing data. Nat Method. 12, 733-735 (2015).
- Hoenen, T., et al. Nanopore Sequencing as a Rapidly Deployable Outbreak Tool. Emerg Infect Dis. 22 (2), 331-334 (2016).
- Petrosino, J. F., Highlander, S., Luna, R. A., Gibbs, R. A., Versalovic, J. Metagenomic pyrosequencing and microbial identification. Clin Chem. 55 (5), 856-866 (2009).
- Cummings, P. J., Ahmed, R., Durocher, J. A., Jessen, A., Vardi, T., Obom, K. M. Pyrosequencing for Microbial Identification and Characterization. J. Vis. Exp. (78), e50405 (2013).
- Rothberg, J. W., et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., et al. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Stoddart, D., Heron, A. J., Mikhailova, E., Maglia, G., Bayley, H. Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. PNAS. 106, 7702-7707 (2009).
- Shendure, J., Hanlee, J. Next generation DNA sequencing. Nat Biotechnol. 26, 1135-1145 (2008).
- Daniel, R. The Metagenomics of Soil. Nat Rev Microbiol. 3, 470-478 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten