È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
In tempo reale immagini di leucotriene B 4 Migrazione cellulare mediata e BLT1 Interazioni con β-arrestina
In questo articolo
Riepilogo
Questo documento descrive la metodologia per determinare la risposta chemiotattica dei leucociti ai ligandi specifici e identificare le interazioni tra recettori di superficie della cellula e le proteine citosoliche utilizzando vivere tecniche di imaging cellulare.
Abstract
Recettori accoppiati alla proteina G (GPCR) appartengono alla famiglia delle sette proteine transmembrana e mediare la trasduzione di segnali extracellulari alle risposte intracellulari. GPCR controllo diverse funzioni biologiche come la chemiotassi, rilascio del calcio intracellulare, regolazione genica in modo ligando dipendente tramite eterotrimeriche proteine G 1-2. Di legame induce una serie di cambiamenti conformazionali che portano all'attivazione di eterotrimeriche G-proteine che modulano i livelli di secondi messaggeri come adenosina monofosfato ciclico (cAMP), inositolo trifosfato (IP3) e glicerolo diacyl (DG). In concomitanza con l'attivazione del legame con il ligando del recettore avvia anche una serie di eventi per attenuare il recettore di segnalazione tramite desensibilizzazione, sequestro e / o internalizzazione. Il processo di desensibilizzazione dei GPCR avviene attraverso la fosforilazione del recettore per G-proteina chinasi del recettore (GRKs) e vincolante successiva β-arrestine 3. β-arrestine sono proteine citosoliche e traslocare a membrana su attivazione GPCR, legandosi ai recettori fosforilati (la maggior parte dei casi) ci internalizzazione del recettore, facilitando 4-6.
Leucotriene B 4 (LTB 4) è un pro-infiammatorie molecola lipidica derivati dal percorso di acido arachidonico e media tramite le sue azioni GPCR, LTB 4 recettore 1 (BLT1, un recettore ad alta affinità) e LTB 4 recettore 2 (BLT2, un recettore a bassa affinità ) 7-9. Il 4-BLT1 LTB percorso ha dimostrato di essere critici in diverse malattie infiammatorie tra cui, l'asma, l'artrite e l'aterosclerosi 10-17. Nel documento descrive le metodologie sviluppate per il monitoraggio LTB4-indotto migrazione dei leucociti e le interazioni di BLT1 con β-arrestina e traslocazione dei recettori in cellule vive utilizzando tecniche di imaging microscopia 18-19.
Il midollo osseo le cellule dendritiche derivate da C57BL / 6 topi sono state isolate e coltivate come descritto in precedenza 20-21. Queste cellule sono state testate dal vivo nei metodi di imaging cellulare per dimostrare LTB 4 migrazione cellulare indotta. Il BLT1 umano è stato taggati con rosso proteina fluorescente (BLT1-RFP) a C-terminale e β-arrestin1 taggati con proteina fluorescente verde (β-arr-GFP) e transfettate entrambi i plasmidi in Rat basofili Leukomia (RBL-2H3) linee cellulari 18-19. La cinetica di interazione tra queste proteine e localizzazione sono stati monitorati utilizzando vivere videomicroscopia cellulare. Le metodologie nel documento corrente descrivono l'uso di tecniche microscopiche per studiare le risposte funzionali di G recettori accoppiati alle proteine in cellule vive. Nel documento descrive inoltre l'utilizzo di software Metamorph per quantificare l'intensità di fluorescenza per determinare la cinetica dei recettori e le interazioni proteina citoplasmatica.
Protocollo
Metodologia
Descrizione del microscopio
Dal vivo gli esperimenti di imaging cellulare eseguita utilizzando TE-FM Epi-Fluorescenza sistema collegato al microscopio invertito Nikon Eclipse TE300. Il microscopio dotato fase di riscaldamento. Un fresco scatto HQ digitale B / N CCD (Roper scientifico) della fotocamera e LAMDA 10-2 filtro ottico changer (società strumento Sutter) è collegata al microscopio. Lunghezze d'onda di eccitazione ed emissione sono controllati con ruote filtro e controllato da Lamba 10-2 regolatore ruota portafiltri, Sutter Instruments Co. 500 ms Tempo di esposizione dovrebbe essere sufficiente per visualizzare RFP o GFP in cellule vive. Controllo hardware e acquisizione delle immagini sono controllati da software Metamorph. La scelta dei filtri per il presente studio sono, set di filtri S480/20x, S525/40m e S565/25x, S620/60m per la GFP e RFP, rispettivamente; EGFP / DsRed dual dicroiche, Tecnologia Chroma. Questa ruota filtro in grado di ospitare fino a sei set di filtri. Microscopio è collegato con regolatore Prima tappa Proscan con bastone Gioia. Tutti questi allegati hardware può essere controllato da un software Metamorph. Il microscopio è inoltre collegato con Micromanipolatori per tenere la micropipette.
A. Misurazione leucotrienici B 4 migrazione Regia cellule dendritiche
Utilizzando l'impostazione sopra descritta microscopio, abbiamo sviluppato metodi per seguire ligando la migrazione delle cellule dendritiche diretto in tempo reale. Due micromanipolatori (Narishige) sono collegati al microscopio. La chemiotassi di midollo osseo dei topi cellule dendritiche derivate (BMDCs) verso LTB 4 gradiente è stato registrato. Qui, descriviamo i metodi per preparare il micro-pipette con micro-pipetta estrattore, il caricamento del ligando in micro-pipetta e di iniziare l'esperimento chemiotassi sul palco riscaldato del microscopio di monitorare la migrazione direzionale delle cellule. Questo metodo può essere applicato a testare l'efficacia di diversi fattori chemiotattici nell'indurre la migrazione così come effetto degli inibitori della chemiotassi in cellule vive. L'isolamento di BMDCs 21 da topo è stato descritto in diverse pubblicazioni tra cui JOVE 22.
1. Preparazione del BMDCs
- Targhetta del BMDCs (dopo coltura per 10 giorni) al piatto copertura Fluoro fondo antiscivolo e permettere loro di crescere per 16 ore prima dell'esperimento.
- Lavare le cellule con 3 ml di PBS 1X almeno 2 volte (con l'aggiunta di 3 ml di PBS 1X alla piastra ed aspirando il fuori buffer).
- Aggiungere 2 ml di 1XPBS al piatto. Le cellule sono ora pronti per l'esperimento. Mantenere le cellule in incubatore a 37 ° C prima di sperimentare.
Preparazione del ligando caricata micro pipetta:
- Fissare i tubi di vetro capillari (vetro di serie, 0,75 mm x 0,4 mm, 6 ", Cat # 625500, AM Systems, INC) sulla verticale micropipetta estrattore (Narishige International USA, Inc).
- Mantenere la temperatura a 52 ° C e permettono di fondere il vetro e tirare fuori per formare punte spigolo vivo.
- Fissare la pipetta sulla micro-pipetta titolare.
- Smussare i bordi grezzi di micro pipetta con Micro forgiare M-900 (Narishige International USA, Inc), osservando al microscopio.
- Preparare almeno il 10-20 pipette per ciascun esperimento. Nota: Il micropipette possono anche essere acquistati da Strumenti di precisione mondiale (TIP μ, T1801TW1F).
- Prendere concentrazione desiderata (500 l di 100 nM LTB 4 in l'esperimento in corso) di legante in 1 ml di tubo effendorf e mantenere la punta micro pipetta nella soluzione e lasciare che la soluzione per entrare nel micro pipetta (il riempimento).
- Prendere 1 ml di legante e nella siringa (1 cc siringa da tubercolina) e lentamente riempire il ligando tramite Micro-Fill ago (28G MF-5, strumenti di precisione mondiale) nella pipetta micro dall'alto.
- Collegare il tubo, che si adatta perfettamente alla pipetta pipette e tubo per l'ago (21 G 11 / 2) collegato al siringa da 1 ml, che è pieno di legante.
- Ora, muovere con cautela il ligando riempito micro-pipetta alla fase microscopio e fissare su micro-manipolatori.
- Applicare una pressione poco da siringa per fare ligando che si sta lentamente liberando dalla punta.
- Portare il BMDCs placcato immaturo al microscopio e tenerli sul piatto riscaldato (37 ° C) palco.
- Togliere il coperchio del piatto fluoro.
- Concentrare le cellule utilizzando immersione 60X olio lente dell'obiettivo.
- Attenzione, far cadere il ligando caricata pipetta usando 'grossolana Manuale Manipolatore MN-188NE' per la vicinanza delle cellule.
- Messa a fuoco con la pipetta 'Micromanipolatore idraulico Belle MN-188 NE'.
- Applicare una pressione poco da siringa per assicurarsi che ligando sta rilasciando in piatto.
- Impostare i parametri di acquisizione di immagini per l'acquisizione in brillanti depositato (10 esposizione ms) in ogni 15 secondi, per 2 ore utilizzando il software Metamorph.
- Continuare a monitorare ogni 30 min per progression di migrazione verso ligando.
- Se le cellule sono affollate di punta della pipetta, modificare la posizione della punta della piastra per continuare la migrazione di esperimento.
B. Misurazione GPCR (BLT1) e proteina citoplasmatica (β-arrestina) Interazioni, traslocazione in cellule vive
1. Preparazione di costrutti di DNA plasmidi
I dettagli dei costrutti di DNA plasmidico sono state descritte in Jala et al. (2005) 18.
2. Trasfezione di Human BLT1-RFP e β-arrestina-GFP in cellule RBL-2H3
- Mantenere la Rat basofili Leukomia (RBL-2H3) Cells (60 al 70% confluenza) a 37 ° C in atmosfera umidificata del 95% di aria, 5% di CO 2 come colture monostrato in terreni di coltura (DMEM integrato con 15% FBS, 2 mM L-glutammina, 100 U / ml penicillina e 100 mg / ml streptomicina) nel T75 fiaschi.
- Staccare le cellule dal piatto / fiaschi utilizzando 6 mL di tripsina-EDTA (0,05% tripsina, 0,53 mM EDTA) ed incubare per 5 minuti a 37 ° C in atmosfera umidificata del 95% di aria, 5% di CO 2. Agitare delicatamente i flaconi di monitorare l'entità del distacco delle cellule.
- Aggiungere 6 ml di terreni di crescita a pallone contenente 6 ml di tripsina-EDTA e raccogliere le cellule mescolandoli pipettando su e giù più volte. Trasferire le cellule in 15 ml tubi falco.
- Contare le cellule utilizzando un emocitometro e prendere 4 x 10 6 cellule in provette 15 ml centrifuga e centrifugare a 480g per 3 giri min e risospendere in 200 ml di mezzi di trasfezione (DMEM, 20% FBS, 50 HEPES mM). Se si prevede di effettuare 3 trasfezioni, preparare le cellule e dei media tranfection di conseguenza dal numero di cellulare e aumentando il volume del mezzo di trasfezione.
- Preparare i plasmidi da transfettate alle concentrazioni di almeno 1,5 mg / mL. Prepariamo il DNA plasmidico utilizzando DNA Maxi QIAGEN kit plasmide. Aggiungere l'individuo o sia la codifica plasmide DNA umano per BLT1-RFP (25 mcg) e β-arrestin1-GFP (15 mcg) per elettroforesi cuvette sterili, (Bio-Rad # 16552088), che ha distanza tra gli elettrodi 0,4 cm.
- Prendete 200 ml di cellule sopra risospeso e metterli in provette contenenti sia hBLT1-RFP o β-arrestin1-GFP o entrambe insieme e mescolare delicatamente con pipetta da 1 ml sterile.
- Lasciare la cuvette sopra a temperatura ambiente per 10 min.
- Electroporate le cellule in Gene Pulser II con tensione a 250 V e capacità di 500 uF.
- Dopo l'elettroporazione permettere alle cellule rimanere a temperatura ambiente per 10 min.
- Prendere 1 ml di mezzo di crescita regolare e mescolarlo con le cellule elettroporate nella cuvette.
- Distribuire 300 ml di questa miscela in 35 millimetri piatti fondo fibra di vetro cultura (piatto Fluoro, strumenti di precisione Mondiale) contenente 2 ml di terreni di crescita regolare e incubare per 1 ora a 37 ° C in atmosfera umidificata del 95% di aria, 5% CO 2. Permettono alle cellule di aderire al fondo del piatto.
- Sostituire i media dopo 1 ora di incubazione con i media una crescita regolare e permettere loro crescere a 37 ° C in atmosfera umidificata al 95% di aria, 5% CO 2 incubatore per 18-24 ore.
3. Cellulare Imaging in diretta
Acquisizione di immagini
- Dopo 18-24 ore, lavare le cellule 2 o 3 volte con 2 ml di caldo rosso fenolo libero RPMI contenente 10 HEPES mM, pH 7,55 (Aggiungere 2 ml di mezzi di comunicazione calda direttamente alla piastra e media aspirare. Ripetere 2 o 3 volte). Aggiungere 1,8 ml di caldo rosso fenolo libero RPMI contenente 10 mM HEPES.
- Posto 30 millimetri di vetro coprioggetto fondo piatti di coltura contenente RBL-2H3 cellule, transfettate con hBLT1-RFP e β-arrestin1-GFP sul palco microscopio riscaldata (37 ° C).
- Raccogliere le immagini delle cellule con ingrandimento 600x con 60 X obiettivo Nikon Piano Apo 60X/1.4 numerica olio apertura dell'obiettivo immersione (disponibile nel nostro sistema microscopio).
- Mettere una goccia di olio sulla lente 60 x obiettivo e mettere a fuoco le cellule con regolare campo chiaro luce trasmessa toccando il fondo della piastra.
- Per monitorare la fluorescenza delle proteine transfettate, passare da campo luminoso (luce trasmessa) a fluorescenza filtro (RFP filtro, se volete vedere dei recettori o GFP, se volete vedere β-arrestin1).
- Scegliere la cella luminoso e sano, che esprime hBLT1-RFP sulla superficie cellulare utilizzando RFP filtro e catturare l'immagine.
- Poi passare alla GFP filtro e fare in modo che β-arrestina-GFP espresso nel citoplasma e catturare l'immagine. Poi il colore pseudo entrambe le immagini, prima che il tempo trascorso catturare immagini cellule vive per assicurarsi che sanguinano attraverso di fluorescenza non sta che si verificano tra GFP e RFP.
- Una volta che la scelta del vostro cellulare fatto, impostare i parametri in base alle proprie finalità utilizzando il software (software Metamorph in questo caso).
- Nella dimostrazione corrente, sedici bit sono le immagini acquisite con il sira impostata binning a 1 X 1 combinato con 60 Piano di X obiettivo Nikon Apo 60X/1.4 numerica olio apertura dell'obiettivo immersione.
- Raccogliere le immagini a fluorescenza per la richiesta di offerta e fluorocromi GFP simultaneamente a 30 sec intervallo di tempo (se le interazioni traslocazione / sono troppo veloce, si possono ridurre gli intervalli di tempo) di queste ruote con filtro controllato dal software Metamorph.
- I tempi di esposizione della fotocamera impostata a 1000 msec per la GFP e RFP. (Impostare i tempi di esposizione specifici a seconda dell'intensità di fluorescenza GFP e RFP).
- In primo luogo il ritiro delle immagini per 1 minuto, senza l'aggiunta di ligando, che serve come punto 0 tempo.
- Aggiungere i 200 ml di legante (dal magazzino di 10 micron ad una concentrazione finale di 1 mM) dopo 1 min senza disturbare la piastra / o la posizione della cella.
- Le immagini a fluorescenza sono raccolti per 60 minuti dopo l'aggiunta del legante e memorizzati come immagini TIFF con ordine crescente dei nomi dei file.
- Tutti questi file possono essere effettuate come file stack con immagini di fluorescenza singoli / combinati con data e ora utilizzando il software.
Processo di immagini e quantificazione della fluorescenza di proteine e localizzazione
- Acquisire le immagini con i media pianura (senza cellule) con filtri RFP e fluorescente GFP e salvarli come immagini di sfondo. Usare queste immagini per sottrarre le immagini reali dati ottenuti in precedenza.
- Preparare la pila di immagini singolarmente per le immagini GFP e RFP.
- Selezionare / definire le diverse regioni (superficie vs citosolico) per misurare l'intensità di fluorescenza di GFP e RFP (BLT1 e β-arrestina traslocazione)
- Tracciare la fluorescenza importo come la funzione del tempo. Questo grafico fornirà le informazioni relative cinetica di traslocazione di data molecola.
Rappresentante Risultati
A. Misurazione leucotrienici B 4 migrazione Regia cellule dendritiche
Il metodo descritto in questo protocollo è stato utilizzato per determinare la migrazione delle cellule dendritiche verso leucotriene B 4 (Figura 1 e video allegato 1) 21. Siamo in grado di applicare la metodologia simile a determinare la capacità chemiotattica di cellule di un ligando particolare in cellule vive.
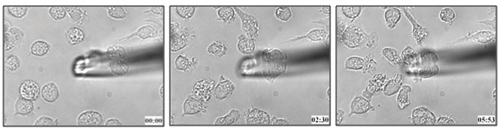
Figura 1. Midollo osseo del mouse delle cellule dendritiche derivate migrazione verso il 100 nM LTB 4.
Video 1. Dal vivo la migrazione delle cellule di BMDCs verso LTB 4. Clicca qui per vedere il video .
B. Misurazione GPCR (BLT1) e proteina citoplasmatica (β-arrestina) Interazioni, traslocazione in cellule vive
Al termine di questa procedura, si possono ottenere le seguenti informazioni in eventi GPCR segnalazione.
- La localizzazione dei GPCR e proteine citosoliche e nucleo (Figura 2).
- Interazione ligando indotta di superficie GPCR (BLT1 in questo caso) con la proteina citoplasmatica (β-arrestina in questo caso) (Fig. 3).
- Cinetica di internalizzazione recettoriale e β-arrestina traslocazione alla membrana seguita da internalizzazione insieme con i recettori (Figura 4) (Jala et al. 2005) 18.
- Si può determinare la dipendenza ligando stato di attivazione dei recettori in cellule vive (in allegato il video 2) e determinare motivi critici o processi coinvolti nella attivazione del recettore (Jala et al. 2005) 18.
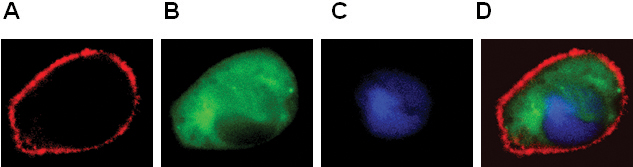
Figura 2. Espressione di BLT1-RFP (A), β-arrestina-GFP (B), pNuc-CFP (C) in cellule RBL-2H3. Colore immagine combinata mostrato nel pannello D.
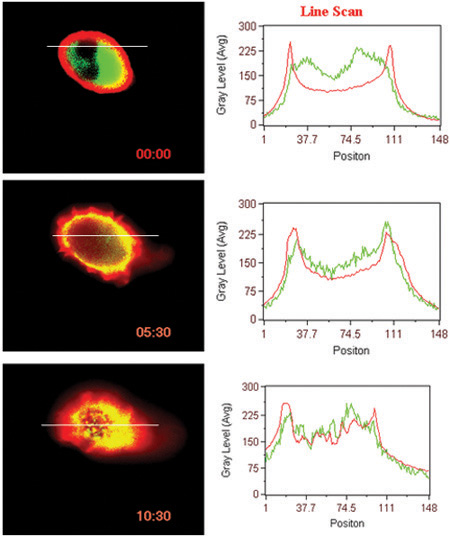
Figura 3. Traslocazione ligando del recettore indotta e β-arrestina. Scansione linea di intensità fluorescente nelle cellule sono mostrati.
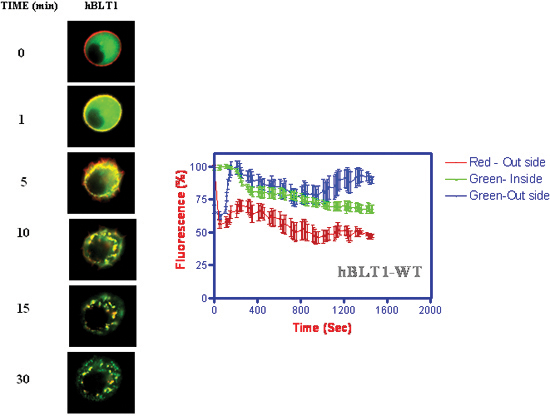
Figura 4. Cinetica di internalizzazione recettoriale e β-arrestina traslocazione dopo l'aggiunta di 1 mM LTB 4. L'intensità di fluorescenza è stata misurata in funzione del tempo a membrana e luoghi citosolico della cellula.
Video 2 immagini dal vivo di cellule che esprimono BLT1-RFP e β-arrestina-GFP sul aggiunta di 1 mM LTB 4. Clicca qui per guardare il video
Discussione
L'imaging cellulare Live è un potente strumento per dimostrare la funzione e le interazioni delle proteine specifiche che si verificano in tempo reale. I metodi descritti in questo manoscritto mostrano chiaramente che LTB 4 può provocare una rapida migrazione delle cellule dendritiche. Questi metodi non solo ampliare gli aspetti della funzione di LTB 4 tipi di cellule diverse, consentono metodi simili da applicare ad una varietà di altre chemochine e prova la loro efficacia come agenti...
Divulgazioni
Riconoscimenti
La ricerca è sostenuta dal National Institutes of concede Salute AI-52381, CA138623 e Kentucky consiglio Lung Cancer Research e il sostegno istituzionale di James Graham Brown Cancer Center.
Materiali
| Name | Company | Catalog Number | Comments |
| Rat Basophilic Leukomia Cell line (RBL-2H3) or HEK293 cells. | ATCC | CRL-2256 | |
| Delbecco’s modified Eagle’s Medium (DMEM) | Invitrogen | 11995 | |
| Phenol red free RPMI or DMEM | Invitrogen | 11835-030 | |
| Fetal Bovine Serum | Invitrogen | 16000-044 | |
| L-Glutamine (200 mM) | Invitrogen | 25030 | |
| Penicillin-streptomycin (10000 U/mL) | Invitrogen | 15140 | |
| Trypsin, 0.05% (1X) with EDTA 4Na, liquid | Invitrogen | 25300 | |
| HEPES (1M) | Invitrogen | 15630 | |
| 35 mm sterile glass coverslip-bottomed Fluoro dishes (0.17 mm thick) (WillCo-dish) | World Precision Instruments, Inc. | FD35-100 | |
| Sterile Gene Pulser Cuvette (0.4 cm electrode gap) (Bio-Rad) | Bio-Rad | 16552088 | |
| Gene Pulser II electroporater | Bio-Rad | ||
| TE-FM Epi-Fluorescence system attached to Nikon Inverted Microscope Eclipse TE300 | Nikon Instruments | ||
| Metamorph Software | Universal Imaging | ||
| Vertical Micro-pipette puller | Narishige International | ||
| Micro-Forge M-900 | Narishige International | ||
| Hadraulic Micromanipulator MO-188NE | Narishige International | ||
| Coarse Manual Manipulator, MN-188NE | Narishige International | ||
| cDNA constructs: | |||
| cDNA of G-Protein coupled receptor tagged with red fluorescence protein at C-terminus (hBLT1-RFP) | Jala et al 2005 | ||
| cDNA of cytosolic protein tagged with GFP (β-arrestin1-GFP in present study). | Jala et al 2005 |
Riferimenti
- Wess, J. G-protein-coupled receptors: molecular mechanisms involved in receptor activation and selectivity of G-protein recognition. FASEB J. 11, 346-354 (1997).
- Gether, U. Uncovering molecular mechanisms involved in activation of G protein-coupled receptors. Endocr Rev. 21, 90-113 (2000).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Lefkowitz, R. J. G. protein-coupled receptors. III. New roles for receptor kinases and beta-arrestins in receptor signaling and desensitization. J Biol Chem. 273, 18677-18680 (1998).
- Shenoy, S. K., Lefkowitz, R. J. Multifaceted roles of beta-arrestins in the regulation of seven-membrane-spanning receptor trafficking and signalling. Biochem J. 375, 503-515 (2003).
- . Beta-arrest or. Nature. 383, 447-450 (1996).
- Serhan, C. N., Haeggstrom, J. Z., Leslie, C. C. Lipid mediator networks in cell signaling: update and impact of cytokines. Faseb J. 10, 1147-1158 (1996).
- Tager, A. M., Luster, A. D. BLT1 and BLT2: the leukotriene B(4) receptors. Prostaglandins Leukot Essent Fatty Acids. 69, 123-134 (2003).
- Toda, A., Yokomizo, T., Shimizu, T. Leukotriene B4 receptors. Prostaglandins Other Lipid Mediat. 68-69, 575-585 (2002).
- Haribabu, B. Targeted disruption of the leukotriene B(4) receptor in mice reveals its role in inflammation and platelet-activating factor-induced anaphylaxis. J Exp Med. 192, 433-438 (2000).
- Subbarao, K. Role of leukotriene B4 receptors in the development of atherosclerosis: potential mechanisms. Arterioscler Thromb Vasc Biol. 24, 369-375 (2004).
- Jala, V. R., Haribabu, B. Leukotrienes and atherosclerosis: new roles for old mediators. Trends Immunol. 25, 315-322 (2004).
- Heller, E. A. Inhibition of atherogenesis in BLT1-deficient mice reveals a role for LTB4 and BLT1 in smooth muscle cell recruitment. Circulation. 112, 578-586 (2005).
- Miyahara, N. Requirement for leukotriene B4 receptor 1 in allergen-induced airway hyperresponsiveness. Am J Respir Crit Care Med. 172, 161-167 (2005).
- Terawaki, K. Absence of leukotriene B4 receptor 1 confers resistance to airway hyperresponsiveness and Th2-type immune responses. J Immunol. 175, 4217-4225 (2005).
- Shao, W. H., Del Prete, A., Bock, C. B., Haribabu, B. Targeted disruption of leukotriene B4 receptors BLT1 and BLT2: a critical role for BLT1 in collagen-induced arthritis in mice. J Immunol. 176, 6254-6261 (2006).
- Kim, N. D., Chou, R. C., Seung, E., Tager, A. M., Luster, A. D. A unique requirement for the leukotriene B4 receptor BLT1 for neutrophil recruitment in inflammatory arthritis. J Exp Med. 203, 829-835 (2006).
- Jala, V. R., Shao, W. H., Haribabu, B. Phosphorylation-independent beta-arrestin translocation and internalization of leukotriene B4 receptors. J Biol Chem. 280, 4880-4887 (2005).
- Jala, V. R., Haribabu, B. Real-time analysis of G protein-coupled receptor signaling in live cells. Methods Mol Biol. 332, 159-165 (2006).
- Del Prete, A., A, . Regulation of dendritic cell migration and adaptive immune response by leukotriene B4 receptors: a role for LTB4 in up-regulation of CCR7 expression and function. Blood. 109, 626-631 (2007).
- Salogni, L. Activin A induces dendritic cell migration through the polarized release of CXC chemokine ligands 12 and 14. Blood. 113, 5848-5856 (2009).
- Boudreau, J., Koshy, S., Cummings, D., Wan, Y. Culture of myeloid dendritic cells from bone marrow precursors. J Vis Exp. , (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon