Method Article
Isolamento dei ribosomi Tradurre contenenti peptidil-tRNA per le analisi funzionali e strutturali
In questo articolo
Riepilogo
Un ostacolo importante per le analisi biochimiche dei ribosomi contenente nascente peptidil-tRNA è stata la presenza di ribosomi altri nei medesimi campioni, i ribosomi non coinvolti nella traduzione della sequenza di mRNA specifico oggetto di analisi. Abbiamo sviluppato una metodologia semplice per purificare, esclusivamente, i ribosomi contenente il nascente peptidil-tRNA di interesse.
Abstract
Recentemente, studi strutturali e biochimici hanno dettagliato molti degli eventi molecolari che si verificano nel ribosoma durante l'inibizione della sintesi delle proteine dagli antibiotici e durante la sintesi polipeptidica nascente. Alcuni di questi antibiotici, e di regolamentazione polipeptidi nascenti per lo più in forma di peptidil-tRNA, inibire sia la formazione di legame peptidico o cessazione traduzione 1-7. Questi eventi inibitori può fermare il movimento del ribosoma, un fenomeno definito "arresto traslazionale". Arresto traduzione sia indotta da un antibiotico o di un polipeptide nascente ha dimostrato di regolare l'espressione di geni coinvolti in diverse funzioni cellulari quali la crescita cellulare, la resistenza agli antibiotici, traslocazione metabolismo delle proteine e delle cellule 8-13. Conoscenza di come antibiotici e regolamentari polipeptidi nascenti alterano la funzione dei ribosomi è essenziale se si vuole comprendere il ruolo completo del ribosoma in traduzione, in ogni organismo.
Qui, descriviamo una metodologia semplice che può essere usato per purificare, esclusivamente, per l'analisi, i ribosomi traduzione di un mRNA specifico e contenente uno specifico peptidil-tRNA 14. Questa procedura è basata su isolamento selettivo dei ribosomi tradurre associato a un marcato con biotina mRNA. Questi complessi traslazionale sono separati da altri ribosomi nella miscela stessa, utilizzando perline paramagnetici streptavidina (SMB) e un campo magnetico (MF). MRNA marcato con biotina sono sintetizzati dal run-off test trascrizione utilizzando come modelli PCR generati frammenti di DNA che contengono promotori T7 trascrizionale. T7 RNA polimerasi incorpora biotina-16-UMP dalla biotina-UTP; nelle nostre condizioni circa dieci biotina-16-UMP molecole sono incorporate in un mRNA 600 nt con un contenuto del 25% dell'Ump. Questi biotina marcata con mRNA vengono poi isolate, e utilizzati in test in vitro traduzione eseguita con fattore di rilascio 2 (RF2)-impoverito cell-free estratti ottenuti da ceppi di Escherichia coli contenenti wild-type o mutante ribosomi. I ribosomi tradurre la biotina marcata con sequenze di mRNA sono in stallo presso la regione codone di stop, per l'assenza della proteina RF2, che compie normalmente terminazione traduzione. Ribosomi in fase di stallo che contiene il nuova sintesi peptidil-tRNA sono isolati e rimossi dalle reazioni traduzione usando SMB e un MF. Queste perle solo legano biotina contenenti messaggi.
L'isolato, complessi traslazionale, può essere utilizzato per analizzare le caratteristiche strutturali e funzionali di tipo selvatico o mutanti componenti ribosomiale, o peptidil-tRNA sequenze, oltre a determinare l'interazione ribosoma con antibiotici o altri fattori molecolari 1,14-16. Per esaminare la funzione di questi complessi isolati ribosoma, peptidil-transferasi analisi può essere eseguita in presenza dell'antibiotico puromicina 1. Per studiare i cambiamenti strutturali in complessi traslazionale, procedure consolidate possono essere utilizzate, come me) reticolazione a specifici amminoacidi 14 e / o ii) test di protezione alchilazione 1,14,17.
Protocollo
1. Preparazione cellulare gratis Estrai.
- Due grammi di batteri Escherichia coli secco pellet ottenuti a partire da metà-log culture fase vengono lavati, due volte, con mezzo litro di tampone A contenente 10 mM Tris-acetato, pH 8.0, 14 mM acetato di magnesio, 60 mM acetato di potassio, e 50 mcg / phenylmethanesulphonylfluoride mL (PMSF) per centrifugazione a 5000 X g per 5 minuti a 4 ° C. Dopo il lavaggio, il pellet batterico viene risospeso in 40 ml di tampone A.
- Le cellule batteriche vengono interrotte con un stampa francese, l'applicazione di pressione 6000 psi. Questa pressione corrisponde a 600 unità con un pollice pistone. La sospensione interrotto viene raccolto in un tubo di vetro pulito (50 ml).
- La sospensione interrotto viene trattata con 1 mM dithiotreitol (DTT) e viene centrifugato a 30.000 xg per 30 min. La procedura di centrifugazione si ripete, usando il surnatante risultante. Il supernatante finale è erogato (1 ml) in microtubi. Infine, le aliquote sono congelati con un secco ghiaccio miscela etanolo o azoto liquido e conservati a -60 ° C o -80 ° C.
2. Preparazione di estratti cellulari gratis impoverito di RF2.
- 4,5 ml di proteina A perline 4B sefarosio liquami vengono miscelati con 5 ml di un anti-RF2 anti-siero con una ruota in movimento a temperatura ambiente, per un'ora. La miscela viene centrifugata a 2500 xg per separare le perle dal antisiero. Su scartando il supernatante, queste perle contenenti anti-RF2 anticorpi (anti-RF2 perle) sono poi lavati dalla risospensione in 1 ml di tampone B contenente 35 mM Tris-acetato pH 7,8, 10 mM acetato di magnesio, 30 mM ammonio acetato, 60 mM glutammato di potassio e 5 mg / ml di inibitore della proteinasi leupeptina. L'anti-RF2 perline vengono separati dai buffer di B per centrifugazione utilizzando le stesse condizioni sopra indicate. La procedura di lavaggio è ripetuta due volte.
- Un ml della cellula senza estratto è mescolato con 150 ml di anti-RF2 perline con una ruota in movimento, a 4 ° C, per due ore. La miscela è centrifugato a 10.000 xg per separare la cellula senza estratto le perline. Il surnatante viene rimosso e nuovamente trattati con un altro 150 microlitri di anti-RF2 perline. Questa procedura viene ripetuta ancora una volta con la conseguente cellula senza soluzione di estratto. La finale cell-free soluzione estratto è dispensato (100 mL) in microtubi. Le aliquote sono congelati con un secco ghiaccio miscela etanolo o azoto liquido e conservati a -60 ° C o -80 ° C.
3. Preparazione di un modello di DNA.
- Preparare un 1 ml di reazione PCR mescolando 600 femtomoles del modello plasmide contenente le sequenze da tradurre (Fig. 1), con 0,4 nanomoli di ciascuno dei oligodeossinucleotidi indicato in Fig. 1, 0,2 micromoli di ciascun dNTP e 50 U di Taq- DNA polimerasi nel buffer fornito da Stratagene Co. Eseguire la reazione di amplificazione alle condizioni seguenti: 94 ° C per 2 minuti, (94 ° C per 30 s, 55 ° C per 30 s, 72 ° C per 1 min, per 30 cicli) e 72 ° C per 12 min.
- Purificare la PCR-prodotti facendo precipitare il DNA con l'aggiunta di volumi 1 / 10 di acetato di sodio 3 M, pH 5,2, e 2 volumi di etanolo ghiacciato. Ripetere la procedura di precipitazioni ancora una volta. Risospendere il DNA in 100 ml di dietil-pirocarbonato (DEPC) trattati con acqua.
In genere, questa procedura produce 100 mg di un prodotto di 600 bp del DNA. - Verificare l'integrità dei prodotti di PCR mediante elettroforesi su gel di agarosio.
4. Preparazione di mRNA marcate con biotina.
- Preparare un 100 ul nella reazione di trascrizione in vitro DEPC acqua trattata mescolando 5 mcg della PCR generati frammento di DNA con 0,5 micromoli di ATP, CTP, GTP, 0,3 micromoli di UTP, 50 nanomoli di biotina-16-UTP, e 10 L del mix enzima T7 nel buffer fornito da Promega Co. Incubare la miscela di reazione a 37 ° C per 3 ore.
Per quantificare la quantità di mRNA ottenuto, il modello del DNA dovrebbe essere eliminato con l'aggiunta di DNasi RNasi-free. Incubare la reazione a 37 ° C per 10 min. - Purificare l'mRNA prodotti facendo precipitare l'RNA aggiungendo volume di 1 / 10 di sodio acetato 3 M, pH 5,2, e 2 volumi di etanolo freddo. Ripetere la procedura di precipitazioni ancora una volta. Risospendere il mRNA in 100 ml di acqua DEPC trattati.
Di solito, questa procedura rendimenti tra 1-2 mg di biotina 600 nt etichettato prodotto mRNA. - Verificare l'integrità dei prodotti contrassegnati mRNA biotina mediante elettroforesi su gel di agarosio.
5. Isolamento dei ribosomi Tradurre contenente un peptidil-tRNA.
- Preparare 500 microlitri di una traduzione nella miscela di reazione in vitro DEPC acqua trattata mescolando 10-15 mg di biotina-mRNA marcato con 75 nanomoli di ciascuno degli anni aminoacidi-tutti, tranne l'aminoacido che sarà sostituito da un acido radioattivo amino , 50 μCi di un acido radioattivo aminoacidi (37MBq) e 20-5 0 l di RF2-impoverito cell-free estratto, in una miscela di reazione tamponata contenente 40 mM Tris-acetato, pH 8.0, 10 mM acetato di magnesio, 175 mM acetato di potassio, acetato di ammonio 10 mM, DTT 2 mM, 2 mM ATP, 0,5 mM GTP, 30 mM fosfoenolpiruvato (PEP), 0,3 U / mL piruvato chinasi (PK), 3,5% polietilenglicole 8000, 1 spermidina mm, 20 mg / ml acido folinico, e 250 mg / ml tRNA da E. coli. Incubare la miscela di reazione a 37 ° C per 10 minuti.
L'ordine di aggiunta dei componenti della reazione è molto importante. Il cell-free estratto deve essere miscelato prima con la soluzione tampone contenente gli aminoacidi e la miscela finale incubata per 5 minuti a temperatura ambiente per permettere l'attivazione dei ribosomi. Più tardi, la biotina marcata con mRNA sono aggiunti. Per le analisi strutturali mediante procedure modificazione chimica, l'aggiunta di un acido radioattivo amino non è necessaria. - Aggiungere alla miscela di reazione traduzione - 3 ml di perline paramagnetici streptavidina (SMB) sospese in tampone C contenente 35 mM Tris-acetato, pH 8.0, 10 mM acetato di magnesio, 175 mM acetato di potassio, acetato di ammonio 10 mM e 1 mM DTT. Incubare la nuova sospensione a temperatura ambiente per 10 min.
- Separare le PMI dalla miscela applicando un campo magnetico (MF) sta usando separazione magnetica.
- Risospendere le PMI nel buffer C e separare le sfere di nuovo con il MF. Ripetere questa procedura di lavaggio due volte. Risospendere le sfere in 500 ml di tampone C, conservare la sospensione su ghiaccio, ed eseguire la procedura successiva immediatamente.
6. L'analisi dei ribosomi isolati contenenti le peptidil-tRNA.
Per peptidil-tRNA
- Miscelare 10 ml di SMB (sospeso in tampone C) con 10 ml di un buffer di carico contenente 50 mM Tris-HCl, pH 6,8, 2,5% (v / v) glicerolo, solfato di sodio al 4% dodecil, 2 mM DTT e 0,2 mg / mL bromofenolo blu. Risolvere i componenti attaccati ai talloni esecuzione dei campioni nel 10% Tris-tricine gel di poliacrilammide.
- Asciugare il gel con un aspirapolvere gel-asciugatrice.
- Verificare l'integrità e la purificazione del peptidil-tRNA esponendo il gel essiccato di un X-ray film.
Per l'RNA ribosomiale
- Aggiungere 190 ml di 2 etilendiamminatetraacetato mM (EDTA) soluzione preparata con DEPC trattati con acqua e 200 ml di fenolo equilibrato con acqua, a 10 ml di una sospensione tallone. Mescolare la sospensione vortexando vigoroso e separare la fase inorganico dalla fase organica per centrifugazione a 10.000 xg per 3 minuti a temperatura ambiente. Raccogliere lo strato superiore dell'acqua in un microtubo nuovo.
- Precipitare l'RNA dallo strato d'acqua con l'aggiunta di volume di 1 / 10 di sodio acetato 3 M, pH 5.2, 1 ml di 20 mg / mL di soluzione di glicogeno, e 2 volumi di etanolo ghiacciato. Risospendere l'RNA in 10 ml di DEPC trattati con acqua.
- Verificare l'integrità del RNA ribosomiale mediante elettroforesi su gel di agarosio.
Di solito, 50 ml di sospensione perline rese 1μg di RNA ribosomiale.
7. Rappresentante dei risultati:
La figura 2 presenta i risultati di una serie di analisi valutare la qualità e la funzionalità dei ribosomi tradurre isolato utilizzando la procedura descritta. L'osservazione di una band unica risolto in gel di poliacrilammide indica la presenza di polipeptidi legati alla biotina marcata con mRNA attaccato al SMB (Figura 2A). La purificazione di RNA ribosomiale utilizzando questa procedura prevede la presenza di ribosomi legato a questi biotina-etichettati mRNA, come pure (Figura 2B). L'aggiunta di puromicina, un antibiotico che induce la peptidil-transferasi attività del ribosoma, provoca la rottura della nascente peptidil-tRNA 1. Questo è osservato come un cambiamento nel modello migratorio del polipeptide isolato in gel di poliacrilammide (Fig. 2C). Insieme, questi dati indicano che la biotina marcata con mRNA attaccato al SMB contengono ribosomi funzionale con peptidil-tRNA.
L'isolamento dei ribosomi tradurre contenenti specifiche peptidil-tRNA permette lo studio degli effetti della peptidil-tRNA, antibiotici e altre molecole, sulla funzione dei ribosomi (Figura 3A), e sulla struttura dei ribosomi (Figura 3B). Negli esempi illustrati di seguito il sparsomycin antibiotico e l'aminoacido triptofano (Trp) inibiscono la scissione idrolitica del nascente tnaC-tRNA peptidil-tRNA indotta da RF2 nei ribosomi isolati (Figura 3A). L'interazione di sparsomycin o Trp con il ribosoma induce anche cambiamenti strutturali, in alcuni nucleotidi che costituiscono l'rRNA 23S del ribosoma tradurre (Fig. 3B). In sintesi, il materiale biologico ottenuto mediante la procedura descritta qui sono utili per ottenere ulteriori comprensione dei contatti strutturali coinvolti nella inibizione della funzione dei ribosomi sulla sua interazione con i vari fattori molecolari.
ove_content ">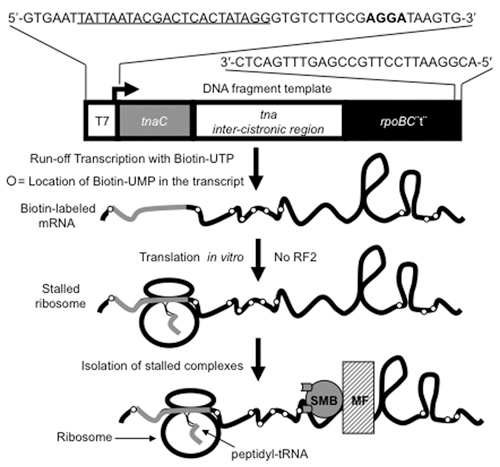
Figura 1. Procedura utilizzata per produrre e purificare ribosomi tradurre contenente peptidil-tRNA. Frammenti di DNA ca. 650 bp di lunghezza, contenente il gene tnaC e la sua regione adiacente, come mostrato in figura, sono utilizzati come modelli per la preparazione mRNA contenenti biotina, [(bio) UMP] mRNA. Questi frammenti di DNA sono prodotte da procedure di PCR utilizzando i oligodeossinucleotidi indicato nella parte superiore della figura. Lettere sottolineate contrassegnare la posizione del promotore T7 codificati nel fondo nucleotide. Grassetto indicano la Shine-Dalgarno sequenza che viene utilizzato nella traduzione delle sequenze di mRNA tnaC. Il promotore T7 nel frammento di DNA è riconosciuta dalla RNA polimerasi di T7 che viene utilizzato per trascrivere (freccia) la sequenza tnaC e non codificanti valle sequenza. La trascrizione è terminato quando la RNA polimerasi raggiunge il 3'-end del frammento di DNA, dopo le sequenze di terminazione rpoBC (rpoBC "t"). La stem-loop struttura del terminatore rpoBC protegge l'mRNA dal 3'-5 'presente attività exoribonuclease nella cella senza estratti per l'nelle reazioni di traduzione in vitro. L'isolato [(bio) UMP] mRNA sono usati per generare in fase di stallo, i ribosomi tradurre, mediante prove in traduzione in vitro. La sequenza tnaC nel [(bio) UMP] mRNA è tradotto da un ribosoma che si blocca quando l'acido ultima aminoacido è incorporato, per l'assenza del fattore di rilascio (RF2) dalla miscela di reazione. Il ribosoma-[(bio) UMP] complessi mRNA sono isolati dalla miscela di reazione totale utilizzando streptavidina-magnetico-perline (SMB) che legano i nucleotidi marcati con biotina incorporato nel mRNA. Le piccole e medie imprese legate al ribosoma-[(bio) UMP] complessi mRNA vengono estratti dalla miscela di reazione totale utilizzando un campo magnetico (MF).
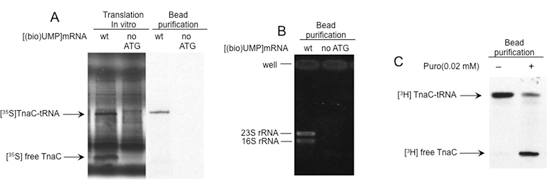
Figura 2. Analisi strutturale e funzionale dei complessi isolati. A) Nelle reazioni traduzione in vitro sono stati effettuati con [(bio) UMP] mRNA in cui è stato sostituito il codone di inizio del gene tnaC da un codone di stop. I prodotti finali sono stati isolati con perline streptavidina, come è indicato nella figura 1. Prodotti di reazione e le molecole isolate sono stati poi analizzati mediante elettroforesi su gel di poliacrilammide. Il tnaC-tRNA e tnaC bande peptide sono contrassegnati. B) RNA totale è stato isolato dai complessi stallo utilizzando procedure di estrazione fenolo. Ogni RNA isolato è stato risolto mediante elettroforesi in gel di agarosio. RNA ribosomiale (rRNA) sono indicati. C) complessi isolati contenenti tnaC-tRNA sono stati incubati con (+) o senza (-) 0,02 mM puromicina (Puro) a temperatura ambiente per 10 min. Il peptide tnaC sono stati etichettati durante la traduzione con [35S]-metionina (A) o [3H]-prolina (B).
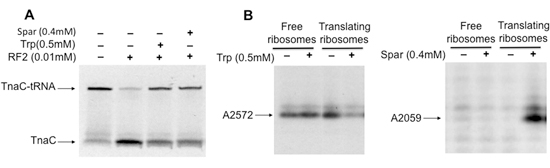
Figura 3. Analisi strutturale e funzionale dei ribosomi contenente tnaC-tRNA che sono legati al sparsomycin antibiotico, o l'aminoacido triptofano. A) i complessi isolati contenenti tnaC-tRNA sono stati incubati con (+) o senza (-) sparsomycin (Spar) o triptofano (Trp) a temperatura ambiente per 5 min. Più tardi, le miscele sono state mescolate con RF2 e incubate a 37 ° C per 20 min. B) Analisi dei cambiamenti nella metilazione del rRNA 23S indotto da molecole vincolante all'interno del ribosoma. Complessi isolati sono stati pre-incubati con (+) o senza (-) 2 mM Trp o Spar per 5 min a 37 ° C. Poi, un agente di alchilazione, solfato di dimetile, è stato aggiunto e le miscele di incubazione a temperatura ambiente per altri 20 min. RNA totale è stato estratto e saggi di primer extension sono stati eseguiti con una [32P]-marcato oligodeoxynucleotide complementare al nucleotidi di rRNA 23S che sono distanziati 100 nucleotidi a valle del nucleotide modificato. I prodotti finali del test estensione sono stati risolti mediante elettroforesi in gel di poliacrilammide. I nucleotidi rRNA 23S influenzata dalla presenza di Trp o Spar sono indicati.
Discussione
Questa procedura di isolamento descritti in questo rapporto è altamente riproducibile. Purtroppo, la presenza di brevi peptidil-tRNA prodotto da le stesse sequenze tradotti non può essere comodamente evitati; quali peptidil-tRNA può corrispondere al 15-20% del materiale totale isolato (Figura 2C). Questi contaminanti possono aumento della concentrazione quando più alte concentrazioni di mRNA marcati con biotina sono stati utilizzati o quando la cella senza estratti utilizzati sono vecchi o sono stati riutilizzati più volte. Pertanto, è molto importante controllare la qualità e la concentrazione dei componenti della traduzione nelle reazioni in vitro. In alternativa, commerciali ad alta efficienza ricostituito cell-free estratti sono disponibili che possono essere utilizzati per le stesse finalità (sistema PURE) 18.
Utilizzando questo metodo, biotina-16-UMP è incorporato in modo casuale su tutta la lunghezza del mRNA bersaglio. C'è la possibilità che alcuni ORF contenente tratti uridina può avere incorporato questo modificate nella loro sequenza nucleotidica, agendo sulla loro traduzione. Variabile concentrazione relativa di biotin-16-UTP/UTP devono essere testati durante la sintesi del mRNA al fine di ottenere la traduzione più efficiente. Metodi alternativi di etichettatura biotina può essere utilizzato e destinato alle 5'-end, la fine più stabile del mRNA. (I) biotina può essere collegato al 5'-end del mRNA con 3'-NH2ATP (3'-amino, 3'-deossiadenosina trifosfato) e T4 RNA ligasi 19. (Ii) 5'-end biotina marcata oligonucleotide può essere collegato al 5'-end del mRNA tramite ligasi T4 RNA e 20. (Iii) 3'-end biotina-etichettati oligodeossinucleotidi che corrispondono alla sequenza complementare al 5'-end del mRNA potrebbe anche essere utilizzato per isolare i complessi traduzione. Queste oligodeossinucleotidi biotina etichettati potrebbe essere collegato al SMB prima dell'isolamento. In seguito la SMB attaccato al biotina-etichettati oligodeossinucleotidi potrebbe essere miscelato con la miscela di reazione traduzione per consentire l'interazione con le loro sequenze di mRNA complementare. Dopo l'isolamento, l'ibrido RNA-DNA regione - e la regione ibridazione tra il oligodeoxynucleotide biotina-etichettati e la sequenza di mRNA - potrebbe essere degradata mediante RNasi H, che separa i complessi in fase di stallo dal perline streptavidina. Questo metodo alternativo può permettere i complessi da utilizzare in futuro le analisi strutturali mediante metodi di risoluzione più elevata.
Divulgazioni
Riconoscimenti
LRC-V. vuole dedicare questo lavoro alla memoria del Dr. Adriel D. Johnson, un professore la cui meravigliosa priorità numero uno è sempre stata la formazione degli studenti. Ci manchi, Adriel. Siamo grati a Jacqueline Moreno per il suo aiuto nello svolgimento degli esperimenti descritti in questo studio. Questo studio è stato sostenuto da una borsa di CY fornito dalla National Science Foundation, MCB-0615390, e da start-up fondi previsti per LRC-V. presso la University of Alabama a Huntsville.
Materiali
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Tris base | Sigma | 252859 | ||
| Magnesium acetate | Fluka | 63049 | ||
| Potassium glutamate | Sigma | G1501 | ||
| Ammonium acetate | Fluka | 09688 | ||
| PMSF | Sigma | P7626 | ||
| Protein A sepharose 4B slurry beads | Sigma | P9424 | ||
| Leupeptin inhibitor | Sigma | L9783 | ||
| Taq-DNA polymerase | Stratagene | 201223 | ||
| T7 Maxiprep kit | Promega | P1300 | ||
| SMB | Promega | Z5482 | ||
| Biotin-16-UTP | Roche | 1388908 | ||
| ATP | Sigma | A7699 | ||
| GTP | Sigma | G8877 | ||
| PEP | Sigma | P7002 | ||
| PK | Sigma | P7768 | ||
| Polyethylenglycol 8000 | Fluka | 81268 | ||
| Folinic acid | Fluka | 47612 | ||
| Spermidine | Fluka | 85558 | ||
| tRNA from E. coli | Sigma | R1753 | ||
| DEPC | Sigma | D5758 | ||
| Tricine | Sigma | T5816 | ||
| L-amino acids | Sigma | LAA21 | ||
| DTT | Fluka | 43815 | ||
| magnetic separation stands | Promega | Z5342 |
Riferimenti
- Cruz-Vera, L. R., Gong, M., Yanofsky, C. Changes produced by bound tryptophan in the ribosome peptidyl transferase center in response to tnaC, a nascent leader peptide. Proc. Natl. Acad. Sci. U. S. A. 103, 3598-3603 (2006).
- Garza-Ramos, G., Xiong, L., Zhong, P., Mankin, A. Binding site of macrolide antibiotics on the ribosome: new resistance mutation identifies a specific interaction of ketolides with rRNA. J. Bacteriol. 183, 6898-6907 (2001).
- Gaynor, M., Mankin, A. S. Macrolide antibiotics: binding site, mechanism of action, resistance. Curr. Top. Med. Chem. 3, 949-961 (2003).
- Polacek, N., Mankin, A. S. The ribosomal peptidyl transferase center: structure, function, evolution, inhibition. Crit. Rev. Biochem. Mol. Biol. 40, 285-311 (2005).
- Steitz, T. A. Structural insights into the functions of the large ribosomal subunit, a major antibiotic target. Keio J. Med. 57, 1-14 (2008).
- Tenson, T., Mankin, A. Antibiotics and the ribosome. Mol. Microbiol. 59, 1664-1677 (2006).
- Xiong, L., Korkhin, Y., Mankin, A. S. Binding site of the bridged macrolides in the Escherichia coli ribosome. Antimicrob Agents Chemother. 49, 281-288 (2005).
- Child, S. J., Miller, M. K., Geballe, A. P. Translational control by an upstream open reading frame in the HER-2/neu transcript. J. Biol. Chem. 274, 24335-24441 (1999).
- Fang, P., Spevak, C. C., Wu, C., Sachs, M. S. A nascent polypeptide domain that can regulate translation elongation. Proc. Natl. Acad. Sci. U. S. A. 101, 4059-4064 (2004).
- Janzen, D. M., Frolova, L., Geballe, A. P. Inhibition of translation termination mediated by an interaction of eukaryotic release factor 1 with a nascent peptidyl-tRNA. Mol. Cell. Biol. 22, 8562-8570 (2002).
- Nakatogawa, H., Ito, K. The ribosomal exit tunnel functions as a discriminating gate. Cell. 108, 629-636 (2002).
- Onouchi, H. Nascent peptide-mediated translation elongation arrest coupled with mRNA degradation in the CGS1 gene of Arabidopsis. Genes Dev. 19, 1799-1810 (2005).
- Raney, A., Law, G. L., Mize, G. J., Morris, D. R. Regulated translation termination at the upstream open reading frame in s-adenosylmethionine decarboxylase mRNA. J. Biol. Chem. 277, 5988-5994 (2002).
- Cruz-Vera, L. R., Rajagopal, S., Squires, C., Yanofsky, C. Features of ribosome-peptidyl-tRNA interactions essential for tryptophan induction of tna operon expression. Mol. Cell. 19, 333-343 (2005).
- Cruz-Vera, L. R., Yanofsky, C. Conserved Residues Asp16 and Pro24 of tnaC-tRNAPro Participate in Tryptophan Induction of tna Operon Expression. J. Bacteriol. 190, 4791-4797 (2008).
- Cruz-Vera, L. R., Yang, R., Yanofsky, C. Tryptophan inhibits Proteus vulgaris tnaC leader peptide elongation, activating tna operon expression. J Bacteriol. 191, 7001-7006 (2009).
- Cruz-Vera, L. R., New, A., Squires, C., Yanofsky, C. Ribosomal features essential for tna operon induction: tryptophan binding at the peptidyl transferase center. J. Bacteriol. 189, 3140-3146 (2007).
- Shimizu, Y. Cell-free translation reconstituted with purified components. Nat. Biotechnol. 19, 751-755 (2001).
- Kinoshita, Y., Nishigaki, K., Husimi, Y. F. l. u. o. r. e. s. c. e. n. c. e. -. isotope- or biotin-labeling of the 5 '-end of single-stranded DNA/RNA using T4 RNA ligase. Nucleic Acids Res. 25, 3747-3748 (1997).
- Nishigaki, K., Taguchi, K., Kinoshita, Y., Aita, T., Husimi, Y. Y-ligation: an efficient method for ligating single-stranded DNAs and RNAs with T4 RNA ligase. Mol Divers. 4, 187-190 (1998).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon