Method Article
Isolamento de Ribossomos Traduzindo Contendo peptidil-tRNAs para análises funcionais e estruturais
Neste Artigo
Resumo
Um grande impedimento para análises bioquímicas dos ribossomos contendo nascente peptidil-tRNAs foi a presença de ribossomos outros nas mesmas amostras, os ribossomos não estão envolvidos na tradução da seqüência de mRNA específico que está sendo analisado. Nós desenvolvemos uma metodologia simples para purificar, exclusivamente, os ribossomos que contém a nascente peptidil-tRNA de interesse.
Resumo
Recentemente, estudos estruturais e bioquímicos têm detalhada muitos dos eventos moleculares que ocorrem no ribossomo durante a inibição da síntese proteica por antibióticos e, durante a síntese de polipeptídeos nascentes. Alguns destes antibióticos, e regulamentar polipeptídeos nascentes principalmente na forma de peptidil-tRNAs, inibem ou formação da ligação peptídica ou término de tradução 1-7. Estes eventos inibitória pode parar o movimento do ribossomo, um fenômeno denominado "prisão de translação". Prisão tradução induzida por qualquer um antibiótico ou um polipeptídeo nascente foi mostrado para regular a expressão de genes envolvidos em diversas funções celulares, como crescimento celular, a resistência aos antibióticos, a translocação de proteínas e metabolismo celular 8-13. Conhecimento de como antibióticos e regulamentares polipeptídeos nascentes alterar a função do ribossomo é essencial se quisermos compreender o papel completo do ribossoma na tradução, em cada organismo.
Aqui, descrevemos uma metodologia simples que podem ser usados para purificar, exclusivamente, para análise, os ribossomos traduzindo um mRNA específico e contendo um determinado peptidil-tRNA 14. Este procedimento é baseado no isolamento seletivo de ribossomos traduzindo vinculado a um mRNA biotina-rotulados. Estes complexos de translação são separados dos ribossomos outros na mesma mistura, usando pérolas paramagnética estreptavidina (SMB) e um campo magnético (MF). MRNAs biotina-rotulados são sintetizados por ensaios de run-off de transcrição usando como modelos de fragmentos de PCR gerado DNA que contêm promotores T7 transcricional. T7 RNA polimerase incorpora biotina-16-UMP de biotina-UTP; nas nossas condições aproximadamente dez biotina-16-UMP moléculas são incorporadas em um mRNA nt 600 com um teor UMP 25%. Estes biotina-rotulados mRNAs são então isolados, e utilizado em ensaios in vitro realizados com tradução fator de liberação de 2 (RF2) estão esgotados livre de células extratos obtidos a partir de cepas de Escherichia coli contendo o tipo selvagem ou mutante ribossomos. Ribossomos traduzindo a biotina marcada com seqüências de mRNA estão paralisadas na região códon de parada, devido à ausência da proteína RF2, que normalmente realiza encerramento de tradução. Ribossomos parado contendo o recém-sintetizado peptidil-tRNA são isolados e removidos a partir de reações de tradução usando SMB e um MF. Estas contas só ligar biotina contendo mensagens.
Os isolados, complexos de translação, pode ser usado para analisar as características estruturais e funcionais do tipo selvagem ou mutante componentes ribossomal, ou peptidil-tRNA seqüências, bem como determinar a interação ribossomo com antibióticos ou outros fatores moleculares 1,14-16. Para examinar a função desses complexos isolados ribossomo, peptidil-transferase ensaios podem ser realizados na presença do antibiótico puromicina 1. Para estudar as mudanças estruturais em complexos de translação, procedimentos bem estabelecidos podem ser utilizados, tais como i) crosslinking a 14 aminoácidos específicos e / ou ii) ensaios alquilação proteção 1,14,17.
Protocolo
1. Preparação de células Extrato grátis.
- Dois gramas de uma coli Escherichia seca bacteriana pellet obtido a partir de meados log-culturas fase são lavados, por duas vezes, com meio litro de tampão contendo 10 mM A Tris-acetato, pH 8,0, 14 mM de acetato de magnésio, 60 mM acetato de potássio e 50 mg / phenylmethanesulphonylfluoride mL (PMSF) por centrifugação a 5000 X g por 5 min a 4 ° C. Após a lavagem, o pellet bacteriano é ressuspenso em 40 mL de tampão A.
- As células bacterianas são interrompidos usando uma imprensa francesa, a aplicação de pressão 6000 psi. Esta pressão corresponde a 600 unidades com um pistão de uma polegada. A suspensão interrompido é coletado em um tubo de vidro limpo (50 mL).
- A suspensão interrompido é tratada com 1 mM ditiotreitol (DTT) e é centrifugado a 30.000 xg por 30 min. O procedimento é repetido centrifugação, utilizando o sobrenadante resultante. O sobrenadante final é dispensado (1 mL) em microtubos. Finalmente, as alíquotas são congeladas usando uma mistura de gelo etanol seco ou nitrogênio líquido, e são armazenadas a -60 ° C ou -80 ° C.
2. Preparação de Extratos celular grátis empobrecido de RF2.
- 4,5 mL de proteína A Sepharose 4B contas de chorume são misturados com 5 mL de um anti-soro anti-RF2 usando uma roda girando em temperatura ambiente, durante uma hora. A mistura é centrifugada a 2500 xg para separar as contas do anti-soro. Após descartar o sobrenadante, estes grânulos contendo os anticorpos anti-RF2 (anti-RF2 esferas) são posteriormente lavadas por ressuspensão em 1 mL de tampão B contendo 35 mM Tris-acetato pH 7,8, 10 mM de acetato de magnésio, 30 mM acetato de amónio, 60 glutamato de potássio mM e 5 mg / mL de leupeptin inibidor de protease. As esferas anti-RF2 são então separados do buffer B por centrifugação utilizando as mesmas condições acima indicadas. O procedimento de lavagem é repetida duas vezes.
- Um mL do extrato livre de células é misturado com 150 mL de anti-RF2 contas usando uma roda giratória, a 4 ° C, por duas horas. A mistura é centrifugada a 10.000 xg para separar o extrato livre de células dos grânulos. O sobrenadante é removido e tratado novamente com outro mL 150 de anti-RF2 contas. Este procedimento é repetido mais uma vez com a solução de extrato livre de células resultantes. A solução extrato final livre de células é dispensado (100 mL) em microtubos. As alíquotas são congeladas usando uma mistura de gelo etanol seco ou nitrogênio líquido, e são armazenadas a -60 ° C ou -80 ° C.
3. Elaboração de um modelo de DNA.
- Prepare a 1 mL de reacção da PCR através da mistura de 600 femtomoles do modelo plasmídeo contendo a seqüência a ser traduzido (Fig. 1), com 0,4 nanomoles de cada um dos oligodeoxynucleotides indicado na Fig. 1, 0,2 micromoles de cada dNTP e 50 U de Taq- DNA polimerase no buffer fornecido pela Stratagene Co. Executar a reação de amplificação nas condições a seguir: 94 ° C por 2 min, (94 ° C por 30 s, 55 ° C por 30 s, 72 ° C por 1 min; por 30 ciclos) e 72 ° C por 12 min.
- Purificar o PCR-produtos por precipitar o DNA pela adição de volume de 1 / 10 de 3 M acetato de sódio, pH 5,2 e 2 volumes de etanol gelado. Repita o procedimento de precipitação mais uma vez. Ressuspender o DNA em 100 mL de dietil-pyrocarbonate (DEPC) de água tratada.
Normalmente, este procedimento produz 100 mg de um produto DNA 600 pb. - Verificar a integridade dos produtos de PCR por eletroforese em gel de agarose.
4. Preparação de Biotina mRNA rotuladas.
- Prepare uma mL 100 in vitro a reação de transcrição na água tratada DEPC através da mistura de 5 mg do fragmento de DNA PCR gerados com 0,5 micromoles de ATP, CTP, GTP, 0,3 micromoles de UTP, 50 nanomoles de biotina-16-UTP, e 10 mL de T7 mix enzima no buffer fornecido pela Promega Co. Incubar a mistura de reação a 37 ° C por 3 horas.
Para quantificar a quantidade de mRNA obtidos, o modelo de DNA deve ser eliminada pela adição de RNase DNase. Incubar a reação a 37 ° C por 10 min. - Purificar os produtos mRNA por precipitar o RNA, adicionando volume 1 / 10 de acetato de sódio 3 M, pH 5,2 e 2 volumes de etanol frio. Repita o procedimento de precipitação mais uma vez. Ressuspender o mRNA em 100 mL de água tratada DEPC.
Normalmente, este procedimento produz entre 02/01 mg de biotina a 600 nt rotulados produto mRNA. - Verificar a integridade dos produtos mRNA biotina marcada por eletroforese em gel de agarose.
5. Isolamento de Ribossomos Traduzindo Contendo um peptidil-tRNA.
- Prepare 500 mL de uma mistura de reação in vitro de tradução em água tratada DEPC pela mistura de 10-15 mg de biotina marcado mRNA com 75 nanomoles de cada um dos dezenove aminoácidos-todos, exceto o aminoácido que será substituído por um ácido amino radioativo , 50 μCi de um ácido amino radioativo (37MBq) e 20-5 0 mL de RF2 depleção extrato livre de células, em uma mistura de reação contendo tampão 40 mM Tris-acetato, pH 8,0, 10 mM de acetato de magnésio, 175 mM de acetato de potássio, acetato de amônio 10 mM, 2 mM DTT, 2 mM ATP, 0,5 GTP mM, 30 mM fosfoenolpiruvato (PEP), 0,3 piruvato quinase U / mL (PK), 3,5% de polietileno glicol 8000, um espermidina mM, 20 mcg / mL de ácido folínico, e 250 mcg / mL tRNA de E. coli. Incubar a mistura de reação a 37 ° C por 10 minutos.
A ordem de adição dos componentes de reação é muito importante. O extrato livre de células deve ser misturado primeiro com a solução tampão contendo os aminoácidos e da mistura final incubadas por 5 min à temperatura ambiente para permitir a ativação do ribossomos. Mais tarde, a biotina marcada com mRNAs são adicionados. Para as análises estruturais utilizando procedimentos de modificação química, a adição de um ácido amino radioativo não é necessária. - Adicione à mistura de reação de tradução - 3 mL de contas paramagnética estreptavidina (SMB) suspenso em tampão C contendo 35 mM Tris-acetato, pH 8,0, 10 mM de acetato de magnésio, 175 mM de acetato de potássio, 10 mM acetato de amônio e 1 mM DTT. Incubar a nova suspensão em temperatura ambiente por 10 min.
- Separar o SMB a partir da mistura pela aplicação de um campo magnético (MF), utilizando se de separação magnética.
- Ressuspender o SMB em tampão C e separar as contas novamente usando o MF. Repita este procedimento de lavagem duas vezes. Volte a suspender as contas em 500 mL de tampão C, loja da suspensão sobre o gelo e realizar o próximo procedimento imediatamente.
6. Análise dos ribossomos isolados Contendo o peptidil-tRNA.
Para peptidil-tRNA
- Misturar 10 mL do SMB (suspenso em tampão C) com 10 mL de um tampão de carregamento contendo 50 mM Tris-HCl, pH 6,8, 2,5% (v / v) de glicerol, sulfato de sódio 4% dodecil, 2 mM DTT e 0,2 mg / mL de azul de bromofenol. Resolver os componentes ligados às esferas executando as amostras em 10% géis de poliacrilamida Tris-tricine.
- Secar o gel usando um secador de vácuo gel.
- Verificar a integridade e purificação do peptidil-tRNA pela exposição do gel seco a um filme de raios-X.
Para RNA ribossomal
- Adicionar 190 mL de solução de 2 mM etilenodiaminotetracetato (EDTA) preparado com água tratada DEPC-, e 200 mL de fenol equilibrado com a água, a 10 mL de uma suspensão de esferas. Misture a suspensão, por vórtex vigoroso e separar a fase inorgânica da fase orgânica por centrifugação a 10.000 xg por 3 min à temperatura ambiente. Coletar a camada de água superior em um microtubo novo.
- Precipitar o RNA a partir da camada de água, adicionando volume 1 / 10 de acetato de sódio 3 M, pH 5.2, 1 mL de 20 mg / mL de solução de glicogênio, e 2 volumes de etanol gelado. Ressuspender o RNA em 10 mL de água tratada DEPC-.
- Verificar a integridade dos RNAs ribossomais por eletroforese em gel de agarose.
Normalmente, 50 mL de suspensão contas rendimentos 1μg de RNA ribossômico.
7. Resultados representativos:
A Figura 2 apresenta os resultados de uma série de análises a avaliação da qualidade e funcionalidade dos ribossomos traduzindo isolado usando o procedimento descrito. A observação de uma banda única resolvidos em géis de poliacrilamida indica a presença de polipeptídeos ligados ao mRNAs biotina marcado ligado ao SMB (Figura 2A). A purificação de RNAs ribossomais usando este procedimento estabelece a presença de ribossomos ligados a estes mRNAs biotina-rotulados, também (Figura 2B). Além de puromicina, um antibiótico que induz a atividade de peptidil-transferase do ribossomo, resulta na clivagem da nascente peptidil-tRNAs 1. Isto é observado como uma mudança no padrão de migração do polipeptídeo isolado em gel de poliacrilamida (Fig. 2C). Juntos, esses dados indicam que a biotina marcada com mRNAs anexado ao SMB contêm ribossomos funcionais com peptidil-tRNAs.
O isolamento dos ribossomos traduzindo contendo específicas peptidil-tRNAs permite o estudo dos efeitos da peptidil-tRNAs, antibióticos e outras moléculas, em função do ribossomo (Figura 3A), e sobre a estrutura do ribossomo (Figura 3B). Nos exemplos aqui apresentados sparsomycin o antibiótico eo aminoácido triptofano (Trp) inibir a clivagem hidrolítica do nascente tnaC-tRNA peptidil-tRNA induzida por RF2 nos ribossomos isolados (Figura 3A). A interação de sparsomycin ou Trp com o ribossoma também induz mudanças estruturais em alguns nucleotídeos que constituem o rRNA 23S do ribossomo traduz (Fig 3B). Em resumo, o material biológico obtido utilizando o procedimento descrito aqui são úteis na obtenção de entendimento adicional dos contatos estruturais envolvidos na inibição da função do ribossomo em cima de sua interação com vários fatores molecular.
ove_content ">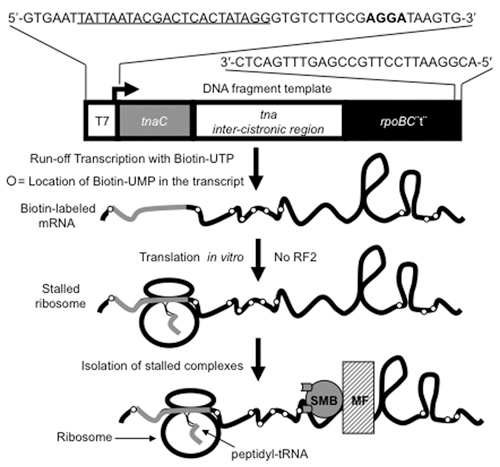
Figura 1. Procedimento usado para produzir e purificar ribossomos traduzindo contendo peptidil-tRNAs. DNA fragmentos ca. 650 bp de comprimento, contendo o gene tnaC e sua região adjacente, mostrado na figura, são usados como modelos na preparação de mRNAs que contém biotina, [(bio) UMP] mRNA. Estes fragmentos de DNA são produzidos por PCR usando os procedimentos oligodeoxynucleotides indicado na parte superior da figura. Letras sublinhadas marcar a localização do promotor T7 codificada na cartilha de nucleotídeos. Letras em negrito indicam a seqüência Equipamento para engraxar os Dalgarno que é usado na tradução das seqüências de mRNA tnaC. O promotor T7 no fragmento de DNA é reconhecido por T7 RNA polimerase, que é usado para transcrever (seta) a seqüência tnaC ea jusante não-codificantes seqüência. Transcrição termina quando a RNA polimerase chega ao fim 3'-do fragmento de DNA, depois de as seqüências rpoBC terminator (rpoBC "t"). A estrutura do haste-laço do terminator rpoBC protege o mRNA da 3'-5 apresentam atividade "exoribonuclease nos extratos livres de células utilizadas na tradução em reações in vitro. O isolado [(bio) UMP] mRNAs são usados para gerar parado, ribossomos traduzindo, usando tradução em ensaios in vitro. A seqüência tnaC no [(bio) UMP] mRNA é traduzida por um ribossomo que barracas, quando o último aminoácido é incorporada, devido à ausência do Fator de Liberação (RF2) a partir da mistura de reação. O ribossomo-[(bio) UMP] complexos mRNA são isolados a partir da mistura de reação total com estreptavidina-magnético-beads (SMB) que se ligam a biotina marcada com nucleotídeos incorporados no mRNA. O SMBs ligado ao ribossomo-[(bio) UMP] complexos mRNA são extraídos da mistura de reação total usando um campo magnético (MF).
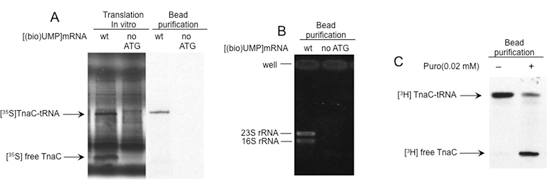
Figura 2. Análises estruturais e funcionais dos complexos isolados. A) Nas reações de tradução in vitro foram realizados com [(bio) UMP] mRNAs em que o códon de iniciação do gene tnaC foi substituído por um códon de parada. Os produtos finais foram isolados usando pérolas de estreptavidina, como é indicado na Figura 1. Produtos de reacção e moléculas isoladas foram então analisadas por eletroforese em gel de poliacrilamida. O tnaC-tRNA e bandas tnaC peptídeo são marcados. B) Total RNA foi isolado a partir dos complexos parado utilizando procedimentos de extração fenol. Cada RNA isolado foi resolvido por eletroforese em gel de agarose. RNAs ribossomais (rRNAs) são indicados. C) complexos isolados contendo tnaC-tRNAs foram incubadas com (+) ou sem (-) 0,02 mM puromicina (Puro) em temperatura ambiente por 10 min. O peptídeo tnaC foram marcados durante a tradução usando [35S]-metionina (A) ou [3H]-prolina (B).
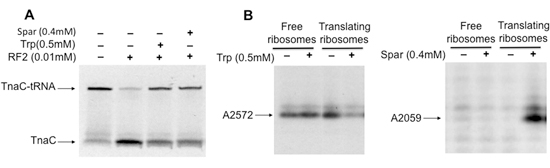
Figura 3. Análises estrutural e funcional dos ribossomos contendo tnaC-tRNA que estão vinculados ao sparsomycin antibiótico, ou do aminoácido triptofano. A) complexos isolados contendo tnaC-tRNAs foram incubadas com (+) ou sem (-) sparsomycin (Spar) ou triptofano (Trp) em temperatura ambiente por 5 min. Mais tarde, as misturas foram misturados com RF2 e incubados a 37 ° C por 20 min. B) Análise das mudanças de metilação no 23S rRNA induzida por moléculas de adesão dentro do ribossomo. Complexos isolados foram pré-incubadas com (+) ou sem (-) 2 mM Trp ou Spar por 5 min a 37 ° C. Então, um agente de alquilação, sulfato de dimetilo, e foi adicionado a misturas incubadas em temperatura ambiente por um adicional de 20 min. RNA total foi extraído e ensaios de extensão de primer foram realizadas com [32P]-rotulados oligodeoxynucleotide complementares para os nucleotídeos do rRNA 23S, que são espaçados 100 nucleotídeos a jusante do nucleotídeo modificado. Os produtos finais dos ensaios de extensão foram resolvidos por eletroforese em gel de poliacrilamida. Os nucleotídeos 23S rRNA afetada pela presença de Trp ou Spar são indicados.
Discussão
Este procedimento de isolamento descritas neste relatório é altamente reprodutível. Infelizmente, a presença de curto peptidil-tRNAs produzido a partir de seqüências de mesmo traduzido não pode ser convenientemente evitada; tais peptidil-tRNAs podem corresponder a 15-20% do material total isolada (Figura 2C). Estes contaminantes podem aumentar em concentração quando maiores concentrações de mRNA biotina-rotulados foram usadas ou quando os extratos de células livres usados são antigos ou que tenham sido re-utilizado várias vezes. Portanto, é muito importante para controlar a qualidade e concentração dos componentes da tradução em reações in vitro. Alternativamente, alta eficiência comercial reconstituído livre de células extratos estão disponíveis, que podem ser utilizados para os mesmos fins (sistema PURE) 18.
Usando este método, biotina-16-UMP é incorporado aleatoriamente ao longo do comprimento do mRNA alvo. Há uma possibilidade de que alguns ORFs contendo uridina tratos podem ter incorporado este nucleotídeo modificado em suas seqüências, afetando a sua tradução. Variável concentrações relativas de biotin-16-UTP/UTP têm de ser testados durante a síntese do mRNA, a fim de obter a tradução mais eficiente. Métodos alternativos de rotulagem biotina pode ser usado, visando o 5'-final, final mais estável do mRNAs. (I) A biotina pode ser ligado à extremidade 5'-do mRNA usando 3'-NH2ATP (3'-amino, trifosfato de 3'-desoxiadenosina) e T4 RNA ligase 19. (Ii) 5'-end biotina marcada com oligonucleotídeo pode ser ligado à extremidade 5'-do mRNA usando RNA ligase T4 bem 20. (Iii) 3'-end biotina-rotulados oligodeoxynucleotides que correspondem à seqüência complementar no final 5'-do mRNA também poderia ser usado para isolar os complexos de tradução. Estes oligodeoxynucleotides biotina marcada poderia ser anexado ao SMB antes de isolamento. Mais tarde, o SMB ligado à biotina-rotulados oligodeoxynucleotides poderia ser misturado com a mistura de reação de tradução para permitir a interação com suas seqüências de mRNA complementar. Após o isolamento, a região RNA-DNA híbrido - e na região hibridação entre o oligodeoxynucleotide biotina marcado ea seqüência de mRNA - poderiam ser degradadas com RNAse H, separando os complexos parado dos grânulos estreptavidina. Este método alternativo pode permitir que os complexos a ser empregada, no futuro, análises estruturais usando métodos de resolução mais alta.
Divulgações
Agradecimentos
LRC-V. desejos de dedicar este artigo à memória do Dr. Adriel D. Johnson, um professor maravilhoso, cuja prioridade número um era sempre a educação dos estudantes. Nós sentimos sua falta, Adriel. Somos gratos a Jacqueline Moreno por sua ajuda na realização das experiências descritas neste estudo. Este estudo foi suportado por uma concessão fornecida para CY da National Science Foundation, MCB-0615390, e por fundos start-up desde a LRC-V. da Universidade do Alabama, em Huntsville.
Materiais
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Tris base | Sigma | 252859 | ||
| Magnesium acetate | Fluka | 63049 | ||
| Potassium glutamate | Sigma | G1501 | ||
| Ammonium acetate | Fluka | 09688 | ||
| PMSF | Sigma | P7626 | ||
| Protein A sepharose 4B slurry beads | Sigma | P9424 | ||
| Leupeptin inhibitor | Sigma | L9783 | ||
| Taq-DNA polymerase | Stratagene | 201223 | ||
| T7 Maxiprep kit | Promega | P1300 | ||
| SMB | Promega | Z5482 | ||
| Biotin-16-UTP | Roche | 1388908 | ||
| ATP | Sigma | A7699 | ||
| GTP | Sigma | G8877 | ||
| PEP | Sigma | P7002 | ||
| PK | Sigma | P7768 | ||
| Polyethylenglycol 8000 | Fluka | 81268 | ||
| Folinic acid | Fluka | 47612 | ||
| Spermidine | Fluka | 85558 | ||
| tRNA from E. coli | Sigma | R1753 | ||
| DEPC | Sigma | D5758 | ||
| Tricine | Sigma | T5816 | ||
| L-amino acids | Sigma | LAA21 | ||
| DTT | Fluka | 43815 | ||
| magnetic separation stands | Promega | Z5342 |
Referências
- Cruz-Vera, L. R., Gong, M., Yanofsky, C. Changes produced by bound tryptophan in the ribosome peptidyl transferase center in response to tnaC, a nascent leader peptide. Proc. Natl. Acad. Sci. U. S. A. 103, 3598-3603 (2006).
- Garza-Ramos, G., Xiong, L., Zhong, P., Mankin, A. Binding site of macrolide antibiotics on the ribosome: new resistance mutation identifies a specific interaction of ketolides with rRNA. J. Bacteriol. 183, 6898-6907 (2001).
- Gaynor, M., Mankin, A. S. Macrolide antibiotics: binding site, mechanism of action, resistance. Curr. Top. Med. Chem. 3, 949-961 (2003).
- Polacek, N., Mankin, A. S. The ribosomal peptidyl transferase center: structure, function, evolution, inhibition. Crit. Rev. Biochem. Mol. Biol. 40, 285-311 (2005).
- Steitz, T. A. Structural insights into the functions of the large ribosomal subunit, a major antibiotic target. Keio J. Med. 57, 1-14 (2008).
- Tenson, T., Mankin, A. Antibiotics and the ribosome. Mol. Microbiol. 59, 1664-1677 (2006).
- Xiong, L., Korkhin, Y., Mankin, A. S. Binding site of the bridged macrolides in the Escherichia coli ribosome. Antimicrob Agents Chemother. 49, 281-288 (2005).
- Child, S. J., Miller, M. K., Geballe, A. P. Translational control by an upstream open reading frame in the HER-2/neu transcript. J. Biol. Chem. 274, 24335-24441 (1999).
- Fang, P., Spevak, C. C., Wu, C., Sachs, M. S. A nascent polypeptide domain that can regulate translation elongation. Proc. Natl. Acad. Sci. U. S. A. 101, 4059-4064 (2004).
- Janzen, D. M., Frolova, L., Geballe, A. P. Inhibition of translation termination mediated by an interaction of eukaryotic release factor 1 with a nascent peptidyl-tRNA. Mol. Cell. Biol. 22, 8562-8570 (2002).
- Nakatogawa, H., Ito, K. The ribosomal exit tunnel functions as a discriminating gate. Cell. 108, 629-636 (2002).
- Onouchi, H. Nascent peptide-mediated translation elongation arrest coupled with mRNA degradation in the CGS1 gene of Arabidopsis. Genes Dev. 19, 1799-1810 (2005).
- Raney, A., Law, G. L., Mize, G. J., Morris, D. R. Regulated translation termination at the upstream open reading frame in s-adenosylmethionine decarboxylase mRNA. J. Biol. Chem. 277, 5988-5994 (2002).
- Cruz-Vera, L. R., Rajagopal, S., Squires, C., Yanofsky, C. Features of ribosome-peptidyl-tRNA interactions essential for tryptophan induction of tna operon expression. Mol. Cell. 19, 333-343 (2005).
- Cruz-Vera, L. R., Yanofsky, C. Conserved Residues Asp16 and Pro24 of tnaC-tRNAPro Participate in Tryptophan Induction of tna Operon Expression. J. Bacteriol. 190, 4791-4797 (2008).
- Cruz-Vera, L. R., Yang, R., Yanofsky, C. Tryptophan inhibits Proteus vulgaris tnaC leader peptide elongation, activating tna operon expression. J Bacteriol. 191, 7001-7006 (2009).
- Cruz-Vera, L. R., New, A., Squires, C., Yanofsky, C. Ribosomal features essential for tna operon induction: tryptophan binding at the peptidyl transferase center. J. Bacteriol. 189, 3140-3146 (2007).
- Shimizu, Y. Cell-free translation reconstituted with purified components. Nat. Biotechnol. 19, 751-755 (2001).
- Kinoshita, Y., Nishigaki, K., Husimi, Y. F. l. u. o. r. e. s. c. e. n. c. e. -. isotope- or biotin-labeling of the 5 '-end of single-stranded DNA/RNA using T4 RNA ligase. Nucleic Acids Res. 25, 3747-3748 (1997).
- Nishigaki, K., Taguchi, K., Kinoshita, Y., Aita, T., Husimi, Y. Y-ligation: an efficient method for ligating single-stranded DNAs and RNAs with T4 RNA ligase. Mol Divers. 4, 187-190 (1998).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados