È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia intravitale della Milza: analisi quantitativa della mobilità Parasite e del flusso sanguigno
In questo articolo
Riepilogo
Mostriamo il metodo per l'esecuzione di microscopia intravitale della milza con GFP parassiti della malaria transgenici e la quantificazione della mobilità parassita e del flusso sanguigno all'interno di questo organo.
Abstract
L'avvento della microscopia intravitale in modelli sperimentali malaria roditore ha permesso importanti progressi alla conoscenza del parassita-ospite 1,2 interazioni. Così, in imaging in vivo di parassiti della malaria durante la pre-eritrocitaria fasi hanno portato alla luce l'ingresso attivo di parassiti nei linfonodi pelle 3, il completo sviluppo del parassita nella pelle 4, e la formazione di un derivato degli epatociti merosome per assicurare la migrazione e rilascio di merozoiti nel flusso sanguigno 5. Inoltre, lo sviluppo di parassiti singoli negli eritrociti è stata recentemente documentata mediante imaging 4D e ha sfidato la nostra visione attuale in materia di esportazione delle proteine della malaria 6. Così, l'imaging intravitale ha radicalmente cambiato la nostra visione sugli eventi chiave nello sviluppo Plasmodium. Purtroppo, gli studi del passaggio dinamico di parassiti della malaria attraverso la milza, un organo importante linfoide squisitamente adattate per cancellare infetti rosso blood cellule sono carenti a causa di vincoli tecnici.
Utilizzando il modello murino della malaria da Plasmodium yoelii in topi BALB / c, abbiamo implementato l'imaging intravitale della milza e ha riportato un rimodellamento differenziale di essa e l'aderenza dei globuli rossi parassitati (pRBCs) alle cellule barriera di origine fibroblastica nella polpa rosso durante infezione con il non-letale parassita linea P.yoelii 17X rispetto alle infezioni con la linea 17XL P.yoelii letale parassita 7. Per arrivare a queste conclusioni, una metodologia specifica con ImageJ software libero è stato sviluppato per consentire la caratterizzazione del veloce movimento tridimensionale del singolo pRBCs. I risultati ottenuti con questo protocollo consentono di determinare il tempo della velocità, direzionalità e di soggiorno dei parassiti nella milza, tutti i parametri di indirizzamento aderenza in vivo. Inoltre, riportiamo la metodologia per quantificare il flusso di sangue al microscopio intravitale e l'uso di DIFferent coloranti al fine di conoscere la struttura complessa del microcircolo della milza.
Dichiarazione etica
Tutti gli studi su animali sono stati effettuati presso le strutture animale dell'Università di Barcellona, in conformità alle linee guida e protocolli approvati dal Comitato Etico per la Sperimentazione Animale dell'Università di Barcellona CEEA-UB (protocollo n. DMAH: 5429). Femmina Balb / c topi di 6-8 settimane di età sono stati ottenuti da Charles River Laboratories.
Protocollo
Questo metodo è stato utilizzato nella ricerca riportata in 7.
1. Infezione animale con Green Fluorescent Protein (GFP) parassiti transgenici
- P. yoelii-GFP linee transgeniche di 17XL e 17X sono stati generati utilizzando gli stessi vettori, il targeting strategia e protocolli descritti altrove per P. berghei 8. Essi esprimono il mutante 3 variante di GFP 9 anni sotto il promotore onnipresente di P. berghei allungamento fattore 1 (Pbeef1a), che dirige l'espressione costitutiva di GFP di parassita citosol durante l'intero intra-eritrocitaria ciclo di sviluppo.
- Iniettare per via intraperitoneale con animali parassitati globuli rossi (pRBCs) di P. yoelii-GFP linee transgeniche 17XL e 17X ottenute dal sangue coda di topo donatore parassitemia al 5-10% e diluito in PBS. Utilizzare una dose di 5x10 5 pRBCs / mouse per raggiungere una parassitemia periferica dell '1% al giorno 3 post-infectisu (pi).
- Al 3 ° giorno pi, controllare che parassitemia di topi infettati con entrambe le linee parassita è lo stesso facendo uno striscio di sangue con una goccia di sangue coda seguita dalla colorazione Giemsa e osservazione al microscopio ottico con un obiettivo di olio 100x. Parassitemia è stimata calcolando la percentuale di globuli rossi pRBCs più totale in tre settori ottica di circa 300 GR.
- Animali di controllo iniettati con FITC globuli rossi può essere utilizzato per caratterizzare il movimento di queste cellule nella milza normale.
2. L'etichettatura dei globuli rossi con FITC e iniezione agli animali di controllo
- Raccogliere 1 ml di sangue totale attraverso la puntura cardiaca di un Balb / c del mouse in 200 ml di PBS contenente etilendiamminotetraacetico (EDTA) (100 g / L, pH 7,4) e lavare il pellet di RBC in PBS / EDTA (0,1 g / L, pH 7,4) attraverso la centrifugazione a 300 xg per 5 minuti (min) a temperatura ambiente (RT).
- Risospendere 200 ul del pellet RBC in 300 μ l di PBS / EDTA (0,1 g / L, pH 8) contenente FITC (10 g / L) e incubare per 2 ore a temperatura ambiente al buio scuotendo con cautela. Trascorso tale termine, il surnatante viene rimosso e le cellule lavate cinque volte (300 xg, 5 min, RT) in PBS / EDTA (0,1 g / L, pH 7,4).
- Per esperimenti in vivo, diluire 10 ml di marcata con FITC RBC pellet in 200 ml di PBS e iniettare per via endovenosa ad un Balb / c del mouse al fine di raggiungere l'1% FITC-GR in circolazione.
3. Le procedure chirurgiche
- Preparare anestetici iniettabili composto da 100 mg / kg di ketamina e 5 mg / kg di Midazolam dose al secondo il peso dell'animale. Iniettare il topo per via intraperitoneale con una dose di anestetico. Readminister metà della dose ogni 30 minuti per mantenere il mouse a tempo pieno anestetizzati.
- Tenere il mouse caldo e verificare che il mouse è completamente anestetizzato (di solito dopo 5-20 min) pizzicando le zampe prima di procedere.
- Allo scopo difacilitare la somministrazione endovenosa di sostanze nel corso dell'esperimento, cannulate la vena della coda del mouse utilizzando una cannula 27G. Controllare che l'ago sia ben posizionato all'interno della vena, iniettando 20-50 ml di soluzione salina tampone e sigillare con nastro adesivo. Se si ostruisce, ripetere la cannulazione a monte della vena. Fare attenzione a non introdurre bolle d'aria.
- Esporre la parte inferiore della milza attraverso una piccola incisione nella pelle e muscolatura sul lato sinistro dorsale dell'animale. Posizionare la milza, dove si osserva il movimento respiro meno PBS e applicare sulla superficie esposta per mantenerla pulita dei capelli del mouse e idratata.
- Tenuta una copertura antiscivolo di 60x24mm con adesivo cianoacrilato (Super Glue Loctite-3) per la cute circostante la milza per permettere la visualizzazione.
4. Imaging di parassiti che vivono nella milza
- Esperimenti di microscopia intravitale sono stati effettuati in un TCS-SP5 microscopio confocale Leica (LeicaMicrosistemi, Heidelberg, Germania) dotato di un sistema di incubazione con controllo di temperatura, un APO 63x ad immersione lente obiettiva glicerolo (NA 1.3), scanner di risonanza a 8000 linee / s ed un Argon (488 nm) e HeNe (594 nm, 633 nm) laser. Laser addizionali, come diodo blu (405 nm) e diodo-pompa-a stato solido (561 nm), può essere necessaria per l'eccitazione delle sonde elencate nella Tabella 1.
- Posto l'animale sul palco del microscopio con la cover-scivolato milza rivolto verso il basso l'obiettivo. Una visione generale della struttura del microcircolo della milza può essere opzionalmente visualizzato utilizzando un obiettivo 20x. Contrasto riflessione RBC sarà utile per selezionare le diverse regioni di interesse per l'immagine a maggiore ingrandimento dopo.
- Attenzione le regioni di interesse selezionati con lenti dell'obiettivo 63x ad immersione glicerolo con autofluorescenza dei tessuti. Parassiti GFP sono osservate passando attraverso diverse aree della milza.
- La fluorescenza è registrato su due differenti channels (eccitazione / d'onda di emissione 488/505-580 nm per FITC / GFP e 488/570-630 nm per autofluorescenza del tessuto) con il set pinhole a 3,0 unità di Airy. RBC riflessione (488/480-495 nm), insieme con le tinture fluorescenti per etichettare i vasi del sangue (vedi Tabella 1), sono utilizzati per ottenere informazioni aggiuntive sulla zona viene esposta e negli esperimenti di flusso di sangue descritta di seguito.
- Catturare immagini attraverso cinque Z-stack che coprono una profondità di 8 micron per la tridimensionalità di un organo, ad una velocità di 8 kHz per generare video di 1.5 min.
- Registrare video di diverse zone della milza per l'analisi quantitativa.
5. Microscopia intravitale del microcircolo della milza e di acquisizione delle immagini per la misurazione del flusso sanguigno
- Vital sonde fluorescenti disciolti in soluzione salina isotonica può essere iniettato per vena della coda durante l'esperimento di immagine l'intuizione vascolarizzazione e ottenere nella struttura della milza. Un elencodi sonde e la loro applicazione è presentato nella tabella 1 10.
- Per etichettare il sistema vascolare con destrano fluorescente, preparare 1 mg di 70 kDa destrano marcati con Texas Red in 100 ml di soluzione salina tampone.
- Utilizzare la vena cannulata coda per iniettare il destrano fluorescente per l'animale sta creando l'immagine.
- Impostare i vasi in orizzontale, in direzione di scansione laser, con rotazione di campo ottico (che non influenzano la velocità). Usa xy e xt linea di scansione modalità nel lume centrale della nave. Utilizzare la scansione bidirezionale con una media di 32 linee a una velocità di 8 kHz per ottenere un'immagine di 512x512 pixel.
- Acquisire immagini di vasi su tre canali diversi (eccitazione / emissione nm di lunghezza d'onda 488/505-580, 594/605-660 nm, 488/480-495 nm per FITC / GFP, destrano-Texas Red e riflessione degli eritrociti, rispettivamente).
- Prendere immagini di vasi con diversi diametri e in diverse fasi del ciclo cardiaco per compensare le fluttuazioni. In queste immagini, le striature derivanti dalle cellule in movimento verranno utilizzati per quantificare il flusso di sangue 11.
6. Elaborazione delle immagini e l'analisi quantitativa della mobilità parassita utilizzando il software ImageJ
- Creare un video in tempo reale dalla sequenza di immagini generati usando il software ImageJ (versione 1.39o, Wayne Rasband, NIH, www.macbiophotonics.ca ).
- Aprire il file ". LIF" file in ImageJ tenere sequenza "xyzct" e canali separati.
- Registrati alcune informazioni utili dal file di metadati: dblvoxelX-voxel-width, dblvoxelY-voxel-altezza, dblvoxelZ-voxel-profondità e l'intervallo tra fotogrammi consecutivi Z-frame e tra le pile. Queste informazioni saranno utilizzate per la calibrazione.
- Sottrarre autofluorescenza (canale 2) alla GFP-parassita immagini (canale 1). Filtrare le immagini con Controllo sfocatura = 1. Si prega di ricordare che con filtro di sfocatura gaussiana delle immagini deve essere dichiarato per pubbns. Salva il file "animal1_m1_substract.seq".
- A Z-coded video a colori è nato come materiale di supporto per l'analisi quantitativa della mobilità parassita, in quanto faciliterà la singola particella di identificazione delle particelle in rapido movimento e Z-movimento caratterizzazione.
- Convertire la pila di immagini 5D, con la terza dimensione è la dimensione Z e la quarta è il tempo. Dare un colore diverso per ogni Z e sovrapposizione.
- Progetto tutte le Z con massima intensità su tutti i tempi per creare un Z-coded video a colori. Salva con nome "animal1_m1_Z_color.avi".
- Classificare ed etichettare tutte le particelle che compaiono nei primi 10 tempi del video in base al numero di fotogrammi di residenza (da 1 a 10). In ogni video, 20 particelle saranno monitorati seguendo le proporzioni ottenute. In totale, 120 i parassiti saranno quantificati da 3 animali, con sei video / animale che rappresentano diverse zone della milza.
- Segnala i fotogrammi di soggiorno per ogni Ze sopra l'intero film per tutte le particelle da quantificare.
- Eseguire 4D (x, y, z, t) di tracciamento manuale di particelle usando il plugin MTrackJ (scritto da E. Meijering). Aprire il file "animal1_m1_substract.seq" come immagine 5D e impostare le proprietà di un'immagine utilizzando le informazioni registrate prima da pixel di larghezza, altezza, profondità (in micron) e l'intervallo stack (in sec). Configurare le impostazioni della traccia come segue: "passare alla prossima volta" e "applicare locale cursore luminoso centroid/25x25pixel". Configurare la visualizzazione di: "mostrare origine", "immagine spettacolo", "spettacolo traccia attiva", "mostrano solo le tracce presenti nei canali di corrente", "mostrare solo il punto di traccia al momento attuale".
- Aggiungere una traccia per ogni particella. Considerare il movimento in Z solo se lo spostamento è superiore a 6 micron (diametro medio di un pRBC). Seguite la particella su un massimo di 100 fotogrammi.
- Save the x, y, z e t coordinate misurate dalla pista come "animal1_m1_p1". Xls.
- Misure di spostamento (D = SQRT ((x finaleInital-x) 2 + (y-y finale inital) 2 + (z-z finale inital) 2); lunghezza del percorso (P = Σ n = 0 SQRT → finale ((x n +1-x n) 2 + ( y n +1-y n) 2 + (z n +1 n-z) 2) con n indicando ogni posizione cingolati; velocità media e tempo di residenza può essere calcolato utilizzando i valori x, y, z, t coordinate tracciati calibrati secondo i dati registrati. Direzionalità delle particelle è definito come il quoziente di spostamento vs lunghezza del percorso, con valori prossimi a 1 indicano il movimento diretto e valori vicini a 0 indicano movimento trattenuto 12. Un modello per il calcolo è facilitata chiuso.
7. Calcolo del flusso di sangue volumetrico
- Il flusso di sangue volumetrico è stimato come Q = V * π * D v 2 / 4, con V, la velocità degli eritrociti sulla sezione attraversare un° D v, lume del vaso di diametro 11.
- Per calcolare V, misurare gli angoli (θ) di cinque strisce di particelle che mostra la riflessione luminosa (RBC) e quattro strisce di particelle mostrando fluorescenza verde (pRBC-GFP) in ogni immagine xt utilizzando il software ImageJ. Misura vaso diametro del lume sull'immagine xy.
- La velocità è quindi espressa in V = 1/tan (θ) * D e / D v per normalizzare per eritrociti (D e = 6 micron) e diametri nave lumen.
- Quantificare un minimo di tre navi con diversi diametri e cinque immagini xt per ciascuna nave.
8. L'analisi statistica
- Per analisi statistica, direzionalità trama, velocità media e tempo di permanenza, distribuzioni di densità e utilizzare l'uguaglianza d'mediani test STATA (IC10) per valutare le differenze tra le due linee parassita.
9. Rappresentante Risultati
Intravitale imaging di parassiti GFP nella milza ha rivelato differenze di mobilità tra i due ceppi di parassiti. Analisi quantitativa dei parametri di mobilità dei singoli parassiti indicato ridotto la velocità, la mancanza di direzionalità e tempo di residenza aumentata di parassiti di topi infettati con il ceppo 17X. Inoltre, il flusso di sangue nei vasi volumetrico non è stato alterato tra ceppi 7. La procedura tecnica è presentata nella Figura 1A. Figura 1B mostra una visione generale di una milza normale di un mouse iniettati con FITC globuli rossi, con uno zoom in nella polpa rossa e un altro ad una nave (Figura 1B, zoom 1 e 2, rispettivamente). Vascolarizzazione è stato evidenziato da iniettando 70 kDa Destrano-Texas Red insieme a contrasto riflessione degli eritrociti. Altri coloranti fluorescenti riassunti nella Tabella 1 può essere utilizzato per ottenere informazioni sull'organo di essere ripreso, come la Hoechst (Figura 1C, 1D).
Imaging in tempo reale dei parassiti del ceppo 17XL e 17X è presentato in Film 1 e 2,con alcuni 17X-pRBC (Movie 2) mostra una rolling-cerchio comportamento. Analisi quantitativa dei parametri di mobilità è stato realizzato attraverso il monitoraggio dei parassiti individuale con l'aiuto di Z-coded immagini a colori. Figura 2A mostra un Z-proiezione di un Z-coded stack dei colori, dove la particella circondato appare muoversi su piani diversi. Figura 2B e 2C rappresentano le tracce per diversi parassiti nelle infezioni 17X e 17XL, rispettivamente. I risultati di direzionalità e tempo di permanenza di tutte le particelle quantificati sono presentati come una mappa di distribuzione della densità di popolazione di parassiti nella figura 2D e 2E, rispettivamente. Per monitorare il flusso di sangue nella milza usando la microscopia intravitale, le striature ottenute nelle immagini xt del lume dei vasi centrale risultanti dal movimento degli eritrociti sono stati misurati per calcolare la velocità 11. Le immagini mostrano una scansione xy di una nave (Figura 2F) con il corrispondente xt linea di scansione (Figura 2G).
| Prob fluorescentee | Localizzazione | 1 fotone di eccitazione (nm) | 2 eccitazione fotone (nm) | Emissione rilevati (nm) | Quantità / peso del mouse |
| Hoechst 33342 | Membrana permeanti che legano il DNA sonda. E le etichette nuclei di tutte le cellule (vivi e morti) dopo l'iniezione endovenosa. | 405 | 800 | 410-480 | 12,5 g / Kg |
| Propidio ioduro | Membrana impermeant che legano il DNA sonda. E le etichette nuclei delle cellule con membrana compromessa (cellule apoptotiche e necrotiche). | 561 | 800 | 570-650 | 250 mg / Kg |
| 70.000 mol-wt destrano fluorescente (FITC, Texas Red) | Fase fluida marcatore che migliora il contrasto di plasma. | FITC 488 Texas Red 594 | 800 | 500-540 600-650 | 50 mg / Kg |
| Sodio fluoresceina | Bulk fase fluida marcatore albumina che migliora il contrasto di plasma. | 488 | 800 | 500-540 | 2 mmol / Kg |
| Evans Blue | Bulk fase fluida marcatore albumina che migliora il contrasto di plasma. | 633 | ° | 645-700 | 20 mg / Kg |
| Rhodamine R6 | Sonda di vitale importanza che si accumula nei mitocondri attivi. E le etichette cellule endoteli e bianchi circolanti dopo l'iniezione endovenosa. | 561 | 800 | 570-650 | 25 mg / Kg |
| Fluospheres-1micron diametro | Perle che sono uptaken da cellule ad attività fagocitaria. | 488 | 800 | 500-540 | ° |
| Alexa488 marcato fibrina IIβ catena specifici anticorpi | Sonda che le etichette fibrina IIβ catena | 488 | 800 | 500-540 | 0,3 mg / Kg |
Tabella 1. Sonde fluorescenti per la microscopia intravitale. Vital coloranti fluorescenti con localizzazioni diverse che possono essere utilizzati per etichettare la milza in vivo. Eccitazione / emissione (Ecc / em) varia da utilizzare con un fotone (o microscopia a due fotoni) sono forniti. La dose indicata è sciolto in 0,1-0,2 ml di tampone soluzione fisiologica e iniettata nella vena della coda del mouse. [Nd: non determinato in questo studio.
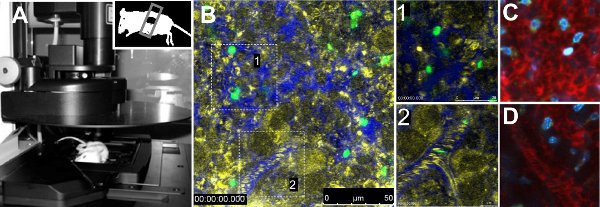
Figura 1. Microscopia intravitale della milza. A. Leica TCS-SP5 microscopio confocale con un mouse posizionato sul palcoscenico del microscopio. Il mouse ha la parte inferiore della milza esposti e sigillato con una copertura antiscivolo. B. Immagine di una zona di rappresentante della milza di un non-infetti animali iniettati con marcato con FITC globuli rossi e70 kDa Destrano-Texas Red per visualizzare la vascolarizzazione. Riflessione (giallo), destrano (blu) e FITC-GR (verde) vengono mostrati. Gigantografie in scatole bianche rappresentano aperta circolazione (1) e chiudere-circolazione (2) le aree. Open-circolazione (C) e da vicino la circolazione (D) colorati con kDa destrano 70 (rosso) e Hoechst 33342 (blu).
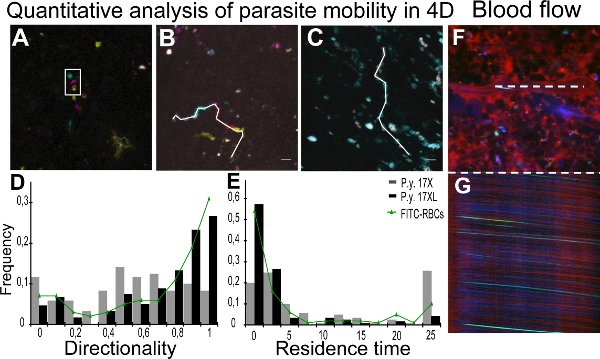
Figura 2. Quantificazione della mobilità parassita e il flusso di sangue. AC. L'analisi quantitativa del movimento delle particelle nelle quattro dimensioni (4D) è facilitata dall'uso di colori diversi di elaborazione delle immagini. Tracking A. è stata effettuata con i dati di profondità da Z-coded immagini a colori, rappresentato utilizzando la proiezione massima intensità di cinque diverse profondità. Rettangolo bianco rappresenta la particella stessa in diverse Z in un punto del tempo. Diverse posizioni sono dovute a cadute di tempo tra l'acquisizionedi diverse immagini Z. Codice Profondità:. Gialla (0 micron), arancione (2 micron), rosa (4 micron), blu (6 micron), verde (8 micron) B, proiezioni C. Tempo di movimento delle particelle con ogni intervallo di tempo colorata come: grigio (0-2,4 sec), ciano (2,4-4,8 sec), magenta (4,8-7,0 sec), rosso (7,0-9,4 sec) e giallo (9,4-11,8 sec). Linea bianca rappresenta 4D tracciamento manuale di particelle di 17X (11,8 s) (B) e 17XL (4,8 sec) (C) parassiti GFP utilizzando MTrackJ. D, E. La distribuzione della densità di particelle GFP da valori di direzionalità (D) e tempo di permanenza (E). I dati corrispondono a 120 particelle di ogni linea di parassiti e 100 GR FITC-marcato da tre esperimenti indipendenti analizzati con l'uguaglianza d'mediani di prova. Mediane 17X/17XL/FITC-RBCs sono 0.53/0.75/0.85 (D) e 4.61/0.67/0.9 sec (E). Differenze tra le due linee in rong> (D) e (E) sono statisticamente significativi (P <0,001). Differenze tra coniugati con fluoresceina globuli rossi e parassiti 17XL non sono statisticamente significativi (p> 0,05). F, G. Milza misure di flusso di sangue. Rappresentazione di xy immagine (F) e xt immagine (G) da una linea di scansione del lume centrale della stessa nave (linea bianca). Milza nave mostrando al plasma con kDa destrano 70 (rosso), pRBC (verde) e la riflessione degli eritrociti (blu).
Film 1 e 2. Time-lapse immagini di microscopia intravitale della milza murini infettati con 17XL (1) o 17X (2) GFP-transgenici parassiti al 10% parassitemia (Z-max di proiezione). Parassita e autofluorescenza dei tessuti sono mostrati in verde e rosso rispettivamente. Barre di scala rappresentano 10 micron e l'intervallo di tempo è in secondi.
v "> Clicca qui per vedere Movie 1.
Clicca qui per vedere Movie 2.
Discussione
L'attuazione di microscopia intravitale della milza in questo modello la malaria roditore ha aperto la possibilità di indagare il passaggio dinamica dei parassiti attraverso questo organo che fino ad ora è stato considerato un "black-box" a causa di considerazioni tecniche. Qui, un grande sforzo è stato messo per adattare un metodo quantitativo che permette l'analisi comparativa delle diverse linee di parassiti a livello singolo e della popolazione. A differenza di altri tessuti e cellule, che era s...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Siamo particolarmente grati a S. Graewe e V. Heussler per la formazione iniziale e continua di ingresso in microscopia intravitale di parassiti della malaria, a J. Burns per la donazione GFP parassiti transgenici, ad A. Bosch (Unità confocale, CCIT-UB, IDIBAPS) per l'assistenza in analisi di immagini e quantificazione e di P. Astola per assistenza tecnica. Ringraziamo Tous R. e Caralt I. per la produzione video. MF è un destinatario di una borsa di studio laureati dal Generalità della Catalogna. HAP è un professore di ricerca ICREA. Il lavoro nel laboratorio di HAP è finanziato dal Settimo programma quadro della Comunità europea (FP7/2007-2013) sotto della convenzione di sovvenzione n. 242095, dalla Fondazione Privata CELLEX (Catalogna, Spagna), e dal Ministero spagnolo della Scienza e dell'Innovazione ( SAF2009-07760).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
| Leica TCS-SP5 microscopio confocale | Leica Microsystems, Heidelberg, Germania | TCS-SP5 non seriale. 5100000419 | |
| Ketamina (Ketolar 50 mg / ml) | Pfizer | 631028 | |
| Midazolam 15 mg / 3 ml | Normon | 838193 | |
| 70.000 MW Destrano, coniugato con Texas Red | Molecular Probes | D1830 | |
| Fluoresceina isotiocianato, isomero I (CIC) | Sigma | F7250 | |
| Hoechst 33342 | Sigma | H1399 | |
| Giemsa | Sigma | GS1 | Soluzione di lavoro è al 10% in acqua distillata |
| Super Glue Loctite-3 | Loctite | 9975-0880 |
Riferimenti
- Amino, R., Menard, R., Frischknecht, F. In vivo imaging of malaria parasites--recent advances and future directions. Curr. Opin. Microbiol. 8, 407-414 (2005).
- Heussler, V., Doerig, C. In vivo imaging enters parasitology. Trends. Parasitol. 22, 192-195 (2006).
- Amino, R., Thiberge, S., Blazquez, S., Baldacci, P., Renaud, O., Shorte, S. Imaging malaria sporozoites. in the dermis of the mammalian. 2, 1705-1712 (2007).
- Gueirard, P., Tavares, J., Thiberge, S., Bernex, F., Ishino, T., Milon, G. Development of the malaria parasite in the skin of the mammalian host. Proc. Natl. Acad. Sci. U. S. A. 107, 18640-18645 (2010).
- Sturm, A., Amino, R., van de Sand, C., Regen, T., Retzlaff, S., Rennenberg, A. Manipulation of host hepatocytes by the malaria parasite for delivery into liver sinusoids. Science. 313, 1287-1290 (2006).
- Gruring, C., Heiber, A., Kruse, F., Ungefehr, J., Gilberger, T. W., Spielmann, T. Development and host cell modifications of Plasmodium falciparum blood stages in four dimensions. Nat. Commun. 2, 165-165 (2011).
- Martin-Jaular, L., Ferrer, M., Calvo, M., Rosanas-Urgell, A., Kalko, S., Graewe, S. Strain-specific spleen remodelling in Plasmodium yoelii infections in Balb/c mice facilitates adherence and spleen macrophage-clearance escape. Cell. Microbiol. 13, 109-122 (2011).
- Linden, M. v. a. n. d. e. r., R, . A Plasmodium berghei reference line that constitutively expresses GFP at a high level throughout the complete life cycle. Mol. Biochem. Parasitol. 137, 23-33 (2004).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein. 173, 33-38 (1996).
- Dunn, K. W., Sandoval, R. M., Kelly, K. J., Dagher, P. C., Tanner, G. A., Atkinson, S. J. Functional studies of the kidney of living animals using multicolor two-photon microscopy. Am. J. Physiol. Cell. Physiol. 283, C905-C916 (2002).
- Zhong, Z., Petrig, B. L., Qi, X., Burns, S. A. In vivo measurement of erythrocyte velocity and retinal blood flow using adaptive optics scanning laser ophthalmoscopy. Opt. Express. 16, 12746-12756 (2008).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Bowdler, A. J. . The complete spleen. , (2002).
- Grayson, M. H., Hotchkiss, R. S., Karl, I. E., Holtzman, M. J., Chaplin, D. D. Intravital microscopy comparing T lymphocyte trafficking to the spleen and the mesenteric lymph node. Am. J. Physiol. Heart. Circ. Physiol. 284, H2213-H2226 (2003).
- Khandoga, A. G., Khandoga, A., Reichel, C. A., Bihari, P., Rehberg, M., Krombach, F. In vivo imaging and quantitative analysis of leukocyte directional migration and polarization in inflamed tissue. PLoS. One. 4, 4693-4693 (2009).
- Weiss, L., Geduldig, U., Weidanz, W. Mechanisms of splenic control of murine malaria: reticular cell activation and the development of a blood-spleen barrier. Am. J. Anat. 176, 251-285 (1986).
- Swirski, F. K., Nahrendorf, M., Etzrodt, M., Wildgruber, M., Cortez-Retamozo, V., Panizzi, P. Identification of splenic reservoir monocytes and their deployment to inflammatory sites. Science. 325, 612-616 (2009).
- Grayson, M. H., Chaplin, D. D., Karl, I. E., Hotchkiss, R. S. Confocal fluorescent intravital microscopy of the murine spleen. J. Immunol. Methods. 256, 55-63 (2001).
- Bajenoff, M., Glaichenhaus, N., Germain, R. N. Fibroblastic reticular cells guide T lymphocyte entry into and migration within the splenic T cell zone. J. Immunol. 181, 3947-3954 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon