È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
In elettroporazione ovo di miRNA-based plasmidi nel tubo neurale sviluppo e valutazione di fenotipi da Injection DiI in Open-book Preparazioni
In questo articolo
Riepilogo
Un metodo di espressione genica nel tubo neurale può essere downregulated in una cella di tipo-specifico, tracciabile modo descritto. Noi dimostrare come In ovo di microRNA basati plasmidi che suscitano spatiotemporally controllato interferenza RNA può essere usato per studiare commissurali guida assonale nel tubo sviluppo neurale.
Abstract
Commissurali DI1 neuroni sono stati ampiamente studiati per chiarire i meccanismi alla base di orientamento degli assoni durante 1,2 sviluppo. Questi neuroni sono situati nel midollo spinale dorsale e inviare loro assoni lungo traiettorie stereotipati. Assoni commissurali inizialmente proiettare ventralmente verso e poi in tutto il fondello. Dopo aver attraversato la linea mediana, questi assoni compiere una brusca virata rostrale e progetto longitudinalmente verso il cervello. Ciascuna di queste fasi è regolata dalle attività coordinate di spunti di orientamento attrattive e repulsive. La corretta interpretazione di questi segnali è fondamentale per la guida degli assoni lungo il loro percorso delimitata. Pertanto, il contributo fisiologica di una particolare molecola di guida assonale commissurali è idealmente esaminati nel contesto dell'embrione vivente. Pertanto, knockdown gene in vivo devono essere controllati con precisione per distinguere attentamente attività di orientamento assone di geni che possono svolgere moltepliciruoli durante lo sviluppo.
Qui, si descrive un metodo per l'espressione del gene knockdown nel tubo neurale pollo in una cella di tipo-specifico, modo tracciabile. Usiamo nuovi vettori plasmidici 3 harboring cellule tipo-specifici promotori / enhancer che guidano l'espressione di una proteina marker fluorescente, seguito direttamente dal miR30-RNAi trascrizione 4 (situato all'interno del 3'-UTR del cDNA che codifica la proteina fluorescente) ( la figura 1). Quando elettroporati nel tubo neurale in via di sviluppo, questi vettori suscitare sottoregolazione efficace di espressione genica e di esprimere proteine marker luminosi fluorescenti per attivare l'analisi diretta delle cellule vivendo knockdown 3. Mescolando diversi vettori RNAi prima elettroporazione permette knockdown simultanea di due o più geni indipendenti in regioni del midollo spinale. Questo consente complesse interazioni cellulari e molecolari da esaminare durante lo sviluppo, in un modo che è veloce, sattua, preciso ed economico. In combinazione con DiI tracciamento delle traiettorie assoni commissurali in open-book preparati 5, questo metodo è un utile strumento per studi in vivo dei meccanismi cellulari e molecolari della crescita degli assoni commissurali e di orientamento. In linea di principio, qualsiasi promotore / enhancer potrebbe essere utilizzato, rendendo potenzialmente la tecnica più ampiamente applicabile per studi in vivo della funzione del gene durante lo sviluppo 6.
Questo video mostra prima come gestire e uova finestra, l'iniezione di plasmidi di DNA nel tubo neurale e la procedura di elettroporazione. Per studiare la guida commissurali dell'assone, il midollo spinale viene rimosso dall'embrione come un libro aperto di preparazione, fissato, e iniettato con DiI per consentire percorsi assoni da tracciare. Il midollo spinale è montata tra coprioggetto e visualizzati usando la microscopia confocale.
Protocollo
1. Preparazione di DNA plasmidico RNAi per Cell Type-specifico silenziamento genico
Plasmidi (Figura 1) sono sintetizzati usando tecniche standard di clonazione molecolare, come precedentemente descritto in dettaglio 3,4.
1,1 clonazione nei vettori: design oligonucelotide
- Usiamo gli stessi oligonucleotidi universali e protocolli clonazione che sono descritti nelle informazioni sul prodotto fornite con il vettore pRFPRNAiC 4 (ARK-Genomics).
- Per clonazione nel sito tornante prima:
Primer 5 'HP1:
5'-GGCGGGGCTAGCTGGAGAAGATGCCTTCCGGAGAGGTGCTGCTGAGCG
Innesco 3 'HP1:
5'-GGGTGGACGCGTAAGAGGGGAAGAAAGCTTCTAACCCCGCTATTCACCACCACTAGGCA - Per clonazione nel sito tornante secondo:
Innesco 5 'HP2:
5'-GGCGGGACGCGTGCTGTGAAGATCCGAAGATGCCTTGCGCTGGTTCCTCCGTGAGCG
Innesco 3 'HP2: - 5'-CGCCGCGCATGCACCAAGCAGAGCAGCCTGAAGACCAGTAGGCA
- Per clonazione nel sito tornante prima:
- Usiamo target GenScript Finder di siRNA per selezionare gene-specifiche sequenze target: https://www.genscript.com/ssl-bin/app/rnai .
- Fondi per la clonazione di un gene specifico miRNA nel sito primo tornante:
Un esempio per silenziamento GFP è mostrato sotto.
Obiettivo sequenza (22nt): 5'-GGCACAAGCTGGAGTACAACTA
GFP avanti HP1 = 59mer 
GFP Reverse HP1 = 58mer 
Ci sono sequenze comuni in questi oligo che fanno parte delle sequenze di miRNA di accompagnamento (pollo-specifico), e comuni loop / stelo sequenze (da umano miRNA30). Il gene-specifiche sequenze bersaglio sono sottolineate. Si noti che non è amIsMatch all'estremità 5 'base del filamento in avanti (mostrato in grassetto; G → A in questo esempio) per imitare il disallineamento naturale in miRNA30 in questa posizione.
- Fondi per la clonazione di un gene specifico miRNA nel sito tornante secondo:
Un esempio per silenziamento LacZ è mostrato sotto.
Obiettivo sequenza (22nt): 5'-CGCGCTGTATCGCTGGATCAAA
LacZ avanti HP2: 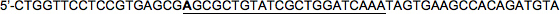
LacZ Reverse HP2: 
Notare che nuovamente, il 5 'base della sequenza bersaglio nel filamento in avanti è stato cambiato (mostrato in grassetto; C → A in questo esempio) in modo che la sequenza antisenso disallineamenti, mimano miRNA30.
1,2 PCR Reazione e subcloning
- Il gene-specifici oligonucleotidi noied insieme con gli oligonucleotidi universali in una reazione di PCR per generare la miRNA30 tipo forcina con pollo miRNA sequenze fiancheggianti.
| La clonazione nel sito primo tornante: 1 pl - 10 ng GFP Forward fondo HP1 1 ml - 100 ng innesco 5 'HP1 Primer 100 ng 3 'HP1 - 1 ml 1 pl dNTP (10 mM) 5 microlitri tampone di reazione 10x Pfu 1 microlitri Pfu DNA polimerasi (Promega) 39 microlitri PCR-Grade acqua | O | La clonazione nel sito tornante secondo: 1 pl - 10 ng LacZ Forward fondo HP2 1 ml - 100 ng innesco 5 'HP2 1 ml - 100 ng innesco 3 'HP2 1 pl dNTP (10 mM) 5 microlitri tampone di reazione 10x Pfu 1 microlitri Pfu DNA polimerasi (Promega) 39 microlitri PCR-Grade acqua |
Cicli:
| 94 ° C | 94 ° C | 55 ° C | 72 ° C | 72 ° C | 4 ° C |
| 1 min | 30 sec | 30 sec | 1 min | 9 min | tenere |
| 30 cicli | |||||
- Purificare il prodotto di PCR, digestione con enzimi di restrizione e subclone nel vettore:
- Primo tornante sito: uso NheI e MluI
- Secondo tornante sito: uso MluI e SphI
- Seguire le tecniche standard per la trasformazione di cellule batteriche competenti. Celle a piastre di agar LB (contenente ampicillina) e DNA raccolto da midipreparation plasmide (ad esempio NucleoBond Xtra Midi kit, Machery-Nagel).
- Sospendere concentrato DNA plasmidicoin sterili DDH 2 0, misurare la concentrazione per spettrofotometria e conservare a -20 ° C.
1,3 sequenziamento miRNA plasmidi
In condizioni normali la reazione di sequenziamento non riesce spesso a causa di una forte struttura secondaria delle forcine. Per migliorare questo 7:
- Effettuare reazione di sequenziamento in 10 mM Tris-Cl con 0,01 mM EDTA (pH 8,0) al posto dell'acqua. Questo aumenta la conversione di DNA superavvolto a ssDNA, che è più sensibile per il sequenziamento.
- Aggiungere una fase di denaturazione termica (98 ° C, 5 min) prima del sequenziamento. Questo converte superavvolto DNA plasmidico di ssDNA.
2. Elettroporazione
2,1 Egg gestione
- Mettere le uova in un incubatore a 38,5 ° C e umidità ~ 45%.
- Incubare le uova fino a quando gli embrioni hanno raggiunto lo stadio di sviluppo desiderato. Per studiare la guida degli assoni dei neuroni commissurali, abbiamo typically electroporate embrioni quando hanno raggiunto Hamburger & Hamilton (HH) fase 17-18 (dopo circa 3 giorni di incubazione) 8. Tuttavia, l'iniezione ed elettroporazione deve essere effettuata prima che la proteina di interesse è accumulato, per fare knockdown efficiente.
- Rimuovere le uova dal termostato e metterli in posizione orizzontale stabile per 20 min, per riposizionare l'embrione in cima al tuorlo sul lato superiore dell'uovo. Riportare le uova nell'incubatrice durante questo periodo.
2.2 Preparazione dei reagenti e apparecchiature
- Preparare tampone fosfato (PBS) e sterilizzare in autoclave o passare la soluzione attraverso un filtro 0,2 micron.
- Rendere micropipette di vetro tirando capillari (World Precision Instruments 1B120F-4, 1,2 / 0,68 OD / ID (mm)) in un dispositivo adatto trazione (es. Narishige PC-10). Rompere la punta di una micropipetta per ottenere un diametro di punta di ~ 5 micron e collegarlo ad un pezzo di tubo conil diametro appropriato.
- Montare gli elettrodi di platino (0,5 cm di lunghezza), saldamente in mano telaio, con un inter-elettrodo distanza di 0,5 cm. Collegare gli elettrodi ad un generatore di impulsi ad onda quadra (BTX ECM 830).
- Impostare il generatore di impulsi con i seguenti parametri:
- Per elettroporazione unilaterale: Tensione: 25 V, numero di impulsi: 5, lunghezza di impulso: 50 msec, dell'impulso intervallo: 1 sec
- Per bilaterale elettroporazione: Tensione: 18 V, numero di impulsi: lunghezza 5, di impulso: 50 msec, dell'impulso intervallo: 1 sec
- Preparare una siringa con ago da 18 G, un bisturi, una bottiglia spritz di etanolo 70% e nastro.
- Melt paraffina in un becher su una piastra riscaldante a 80 ° C.
2,3 Windowing
- Pulire le uova con un tessuto imbevuto di etanolo al 70%.
- Una striscia di nastro lungo l'asse dell'uovo.
- Fare due piccoli fori nel guscio d'uovo con un bisturi, uno al'estremità piatta del l'uovo e l'altro in un angolo dell'area da finestre.
- Usando una siringa, rimuovere ca. 3 ml di albumina di ogni uovo inserendo l'ago con un angolo di 45 ° nel foro all'estremità smussata dell'uovo. Accuratamente evitare di danneggiare il tuorlo.
- Tagliare una finestra nel guscio d'uovo con piccole forbici, tenuto in orizzontale per evitare di danneggiare l'embrione. Se necessario, la posizione del l'embrione può essere verificato e contrassegnato con una matita tenendo una luce forte contro l'estremità smussata dell'uovo. Una finestra di 1,5 a 2 cm quadrati è utile per la maggior parte delle applicazioni.
- Sigillare il foro all'estremità smussata dell'uovo e eventuali crepe nel guscio con paraffina fusa, applicato con un pennello.
- Sigillare la finestra con del nastro trasparente (es. Scotch Magic).
- Ritorna l'uovo alla incubatrice.
2,4 elettroporazione
- Preparare gli strumenti sterili e pulire l'area di lavoro con il 70% di etanolo.
- Preparare il injection soluzione. La concentrazione di DNA appropriata deve essere determinata dall'utente e varierà a seconda del promotore / enhancer utilizzato per guidare l'espressione. A titolo indicativo, si utilizzano in genere 0,2-2,0 mg / pl (vedi Discussione). In un totale di 20 pl, la miscela di iniezione dovrebbe contenere:
| RNAi DNA plasmidico (in H 2 0) | X pl |
| 20x PBS | 1 ml |
| 0,4% trypan blue | 2 microlitri |
| sterile DDH 2 0, per un volume finale di | 20 pl |
Utilizzare aspirazione delicata per caricare la miscela DNA nel bicchiere microcapillare attaccato al tubo.
- Rimuovere il nastro dalla finestra e l'uovo in scena l'embrione secondo Hamburger e Hamilton 8.
- Utilizzare pinze e forbici a molla per rimuovere con attenzione la extraemembrane mbryonic dalla metà caudale dell'embrione. Le membrane possono essere facilmente sollevato dalla embrione nella regione in cui destra e sinistra vene principali vitellina immettere il tronco. Delicatamente strappare o tagliare le membrane e tirare verso la coda. Se necessario, l'embrione può essere visualizzato meglio iniettando una soluzione di inchiostro di china oVerde tra disco embrionale e tuorlo d'uovo, come mostrato in un video Boulland e colleghi 9.
- Iniettare la soluzione di DNA nel canale centrale del tubo neurale, appena sopra le zampe posteriori. Controllare il volume di iniezione per via orale. Il colorante blu deve diffondersi dalla punta della coda fino al ventricolo del cervello sviluppo.
- Aggiungere qualche goccia di PBS sterile sulla parte superiore di un embrione.
- Posizionare gli elettrodi paralleli al asse anteriore-posteriore dell'embrione. Evitare di toccare l'embrione o vasi sanguigni.
- Tenere gli elettrodi costante e electroporate.
- Rimuovere con attenzione gli elettrodi e risciacquare conacqua sterile per rimuovere le proteine denaturate dal bianco d'uovo.
- Per elettroporazione bilaterale, cambiare la polarità degli elettrodi, riposizionare gli elettrodi paralleli l'embrione e ripetere l'elettroporazione. Sciacquare gli elettrodi in acqua sterile.
- Cadere ancora un po 'sterile PBS sul embrione. Richiudere l'uovo con del nastro e tornare al incubatrice fino allo stadio di sviluppo desiderato è raggiunto. Tenuta corretta è fondamentale per evitare la disidratazione dell'embrione.
3. Preparazione del midollo spinale
3,1 Dissezione di embrioni
- A HH25-26 (giorno 5 di incubazione), rimuovere l'embrione dall'uovo utilizzando pinze o un cucchiaino. Collocarlo in PBS in una piastra di Petri rivestita con silicone (elastomero Sylgard).
- Rimuovere le membrane extraembrionali e laici l'embrione sul dorso. Stabilizzarlo sulla piastra da pinning attraverso il collo e la coda (utilizzando sugli insetti 0,20 mm), con s dolcetretching.
- Embrioni interi (per il sezionamento in seguito) può essere fissato qui, sostituendo il PBS con 4% paraformaldeide (PFA) e incubando per 1 ora a temperatura ambiente. Per rendere open-book preparati, continuare il protocollo con tessuto non fissato come descritto di seguito.
3,2 Isolamento del midollo spinale da embrioni
- Pin arti, assicurando che i perni sono inseriti in un angolo lontano dal embrione in modo che non interferiscano con la dissezione. L'embrione deve essere illuminato dal basso per consentire densità tissutale essere percepito nei passi seguenti.
- Rimuovere il cuore e gli organi interni con le forbici a molla e delicatamente raschiare con una pinza. Le vertebre segmentato e il midollo spinale deve essere visibile se tutti gli organi sono stati rimossi completamente.
- Con le forbici a molla, fare un taglio poco profondo attraverso le vertebre sovrastanti il midollo spinale a livello del collo. Ruotare di 180 ° embrione e fare due brevitagli longitudinali attraverso le vertebre su entrambi i lati del midollo spinale, dal collo verso la coda. Utilizzare pinze per sollevare il lembo di vertebre dal midollo spinale e buccia del tessuto (contenente tutte le vertebre) in una singola striscia verso la coda.
- Delicatamente allungare e ri-pin l'embrione attraverso la coda e gli arti.
- Utilizzare un bel bisturi microchirurgico (ad esempio Grieshaber 68101) o un ago di tungsteno per tagliare via le meningi che ricoprono il midollo spinale. Cercare una scura, linea denso di tessuto tra il tubo neurale e gangli delle radici dorsali. Regolare l'angolo di illuminazione, se necessario. Delicatamente tagliare longitudinalmente lungo questa linea su ciascun lato del midollo spinale, dal collo alla coda. Le meningi dovrebbe separare dal midollo spinale dovuta alla stretching dell'embrione appuntato.
- Tagliare il midollo spinale a livello del germoglio ala e caudale al germoglio arto, e sollevare il midollo spinale di tutta l'embrione in una rostrale liscia caudale movimento, l'uso di pinze. Il midollo spinale isolato dovrebbe essere mantenuto immerso in PBS durante questa fase. Non sollevarlo del piatto.
3,3 fissazione di open-libri
- Diffondere il midollo spinale isolato su una spatola e trasferirlo in un nuovo silicone rivestito piastra Petri contenente il 4% di paraformaldeide in PBS.
- Produrre un piatto preparato con cura montaggio pinning del midollo spinale in sei posizioni (rostrale, medialmente e caudalmente su ogni lato, con 0,10 perni insetti mm). Ci etichettare ogni preparazione da un po 'bandiera' che non solo identifica l'embrione, ma indica anche la fine anteriore del libro aperto preparazione.
- Incubare a temperatura ambiente per 30 minuti a 1 ora. Open-libri non dovrebbero essere troppo fissato come questo riduce l'efficienza di diffusione DiI 10 e aumenta di sfondo.
- Versare con cautela fuori dal 4% PFA e sostituirlo con PBS. Tenere i piatti a 4 ° C fino al momento di iniettare con DiI o mount.
- Osservare le open-libri sotto miscroscopy fluorescenza e selezionare il lato appropriato del libro aperto per iniettare con DII.
- Preparare veloce DiI (5 mg / ml in etanolo) e disegnare la soluzione in una micropipetta di vetro attaccato al tubo di plastica. Rompere la fine della micropipetta più finemente possibile ottenere una punta di diametro molto piccolo. Inserire l'ago in un piatto di PBS e controllare che il DiI non fuoriuscire dal ago. In caso di perdite Dii, il diametro dell'ago è troppo grande. In tal caso, preparare un nuovo ago.
- Illuminare gli spazi-book preparati dal basso. Cerca una striscia longitudinale di tessuto denso, situato a circa 1/5 della larghezza di un hemibook dal bordo laterale della preparazione. Ciò corrisponde ai corpi cellulari dei neuroni commissurali, che si trovano appena ventralmente alla piastra del tetto. Partendo da un estremo del libro aperto, inserire l'ago nel tessuto di vetro e, come l'ago is ritirata, sfoglia in una piccola quantità di DiI usando una pipetta a bocca.
- Lavorare rapidamente, rendendo diverse iniezioni lungo la lunghezza del libro aperto ad intervalli regolari di circa 0,5 mm. Se l'ago si intasa, con cura in chiaro con una pinza. Se la punta diventa troppo grande (e perdite DII), sostituire l'ago.
- Al termine di ogni libro aperto, utilizzare una pipetta di trasferimento per aspirare ed eliminare ogni eccesso, trapelato DII. In caso contrario, questo si tradurrà in fondo elevato.
- Lasciare i preparativi per circa tre giorni a 4 ° C per consentire l'DiI a diffondersi lungo gli assoni.
3.5 Montaggio per l'imaging
- Utilizzare una siringa con un ago da 18 G a diffondere un sottile bordo ininterrotta di grasso per vuoto (ad esempio Dow Corning # 976V) lungo i bordi di mm 24 x coprioggetto da 24 mm. Aggiungere qualche goccia di PBS sterile al pozzo. Si noti che un mezzo di montaggio contenente glicerolo con n-propil gallato non può essere compatible con DiI 11. Analogamente, grasso per vuoto di bassa viscosità possono mescolare con PBS e il risultato in fondo elevato.
- Rimuovere i perni dal libro aperto e le trasferisce in la goccia PBS. Immergere il libro aperto e posizionarla al centro del pozzo.
- Con cautela, inserire un altro 24 mm x 24 mm coprioggetti in alto, assicurandosi che il libro aperto rimane aperto. Se necessario, il coprioggetto può essere rimosso, e il libro aperto riposizionato. Premere delicatamente intorno ai bordi della preparazione per creare una tenuta completa di grasso. PBS in eccesso sarà spremuto durante questa fase. Evitare bolle d'aria.
- Conservare i preparati al buio a 4 ° C fino al momento ispezione e documentazione mediante microscopia a fluorescenza. Questo dovrebbe essere fatto rapidamente (entro una settimana), per garantire che i preparativi non si seccano.
4. Risultati rappresentativi
Elettroporazione ed espressione di plasmidi
Sotto l'condizioni sopra descritte, proteina fluorescente deve essere chiaramente rilevabile nel tipo cellulare appropriata senza necessità di ulteriore amplificazione del segnale mediante l'etichettatura anticorpo. La proteina fluorescente deve essere rilevabile nel tipo cellulare desiderato / s. Esempi rappresentativi di open-book preparati e sezioni di embrioni elettroporate con i plasmidi diverse sono mostrati in Figura 2.
Efficienza dei miRNA artificiali
MiRNA artificiali nei confronti di un nuovo gene di interesse deve prima essere sottoposti a screening per l'efficienza e la specificità dei loro effetti smontabili. Troviamo che β-actina promotore-driven costrutti, elettroporate a 0,25 mg / mL, sono appropriati per questo 3. Knockdown in vivo può essere testato mediante immunoistochimica e ibridazione in situ.
DiI etichettatura
Altamente mirato iniezioni DII in embrioni di tipo selvaticodovrebbe produrre più del 80% dei siti di iniezione con traiettorie ideali, archetipiche 3, come mostrato in Figura 3. Animale alla variabilità degli animali dovrebbe essere bassa.
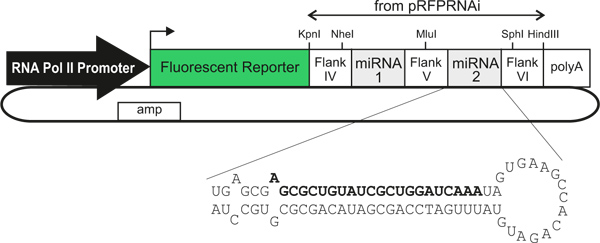
Figura 1. Schema generalizzata dei miRNA che esprimono vettori plasmidici. L'uso di diversi promotori RNA polimerasi II / esaltatori consente cellulare specifico del tipo di espressione. Le cellule trasfettate sono identificabili mediante l'espressione di un reporter fluorescente che è direttamente collegata (all'interno di un singolo trascritto) per uno o due miRNA artificiali, che abbattono espressione genica. Il testo in grassetto indica il filamento senso di un miRNA artificiale contro LacZ, come descritto nel testo.
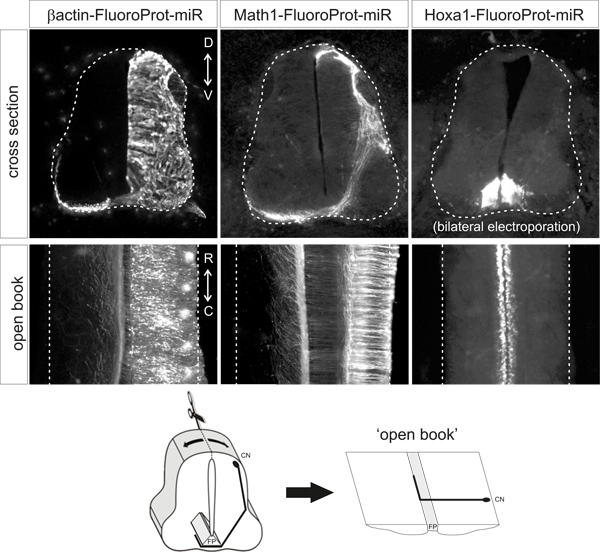
Figura 2. Rappresentativa esempi of fluorescenti pattern di espressione proteica ottenuto a seguito elettroporazione dei vettori indicati plasmidici. Sezioni trasversali e libri aperti sono da HH25-26 embrioni di pollo che sono stati elettroporate a HH18. β-actina promotore pilota l'espressione ubiquitaria, Math1 enhancer pilota l'espressione in DI1 neuroni e HOXA1 enhancer pilota l'espressione particolare nella lamiera di fondo. CN, commissurali neurone, FP, lastra di fondo.
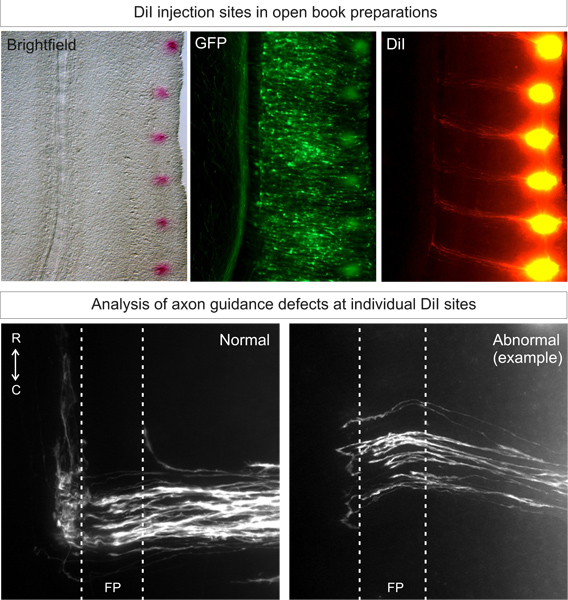
Figura 3. Applicazione ed analisi dei siti di iniezione Dii in preparati libro aperto. DiI deve essere iniettato in un pattern puntiforme, vicino al margine laterale del libro aperto, sul lato elettroporata (identificato da espressione della proteina fluorescente). Dopo 3 giorni di diffusione, commissurali traiettorie assoni devono poter essere visualizzati al microscopio a fluorescenza. Traiettorie assoni normali crescerà verso il pavimento pfine, attraversare la lastra di fondo e poi girare e crescere rostralmente. Fenotipi anomali derivanti da colpo gene verso il basso può essere paragonato a questa traiettoria archetipo. Nell'esempio, alcune stallo assoni nella lamiera di fondo o fare erronee decisioni tornitura sul lato controlaterale.
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo semplice, vettoriale artificiale strategia miRNA può essere utilizzato per l'espressione del gene endogeno knockdown nel tubo neurale pollo. Questi strumenti funzionali offrono silenziamento genico multipla, il controllo temporale del tipo cellulare e specificità, per facilitare la spiegazione dei complessi percorsi di sviluppo. In particolare, abbiamo dimostrato l'utilità di questi plasmidi nell'orientamento dell'assone commissurali, poiché i plasmidi possono essere utilizzati per atterrament...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Il lavoro nel laboratorio di ES è supportato dal Fondo nazionale svizzero della scienza. Vorremmo ringraziare il Dr. Beat Kunz per l'assistenza con le riprese.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
| 0,5 millimetri capillari di vetro | Mondo Strumenti di precisione | 1B120F-4 | |
| Vetro ago estrattore | Narishige | PC-10 | |
| Elettroporatore | BTX | ECM 830 | |
| Sylgard silicone elastomero | Mondo Strumenti di precisione | SYLG184 | |
| Tungsteno filo, 0,075 millimetri | Mondo Strumenti di precisione | TGW0325 | |
| Perni insetti, 0,20 millimetri | Strumenti Scienza Belle | 26002-20 | |
| Perni insetti, 0,10 millimetri | Strumenti Scienza Belle | 26002-10 | |
| Primavera forbici | Scienza Belle Perols | 15003-08 | |
| Dumont # 5 forcipe | Strumenti Scienza Belle | 11252-20 | |
| Dumont N ° 55 pinze | Strumenti Scienza Belle | 11255-20 | |
| Veloce DiI | Molecular Probes | D-7756 | |
| Microscopi a fluorescenza | Olimpo | SZX12, BX51 |
Riferimenti
- Chédotal, A. Further tales of the midline. Current Opinion in Neurobiology. 21, 68-75 (2011).
- Avraham, O., Hadas, Y., Vald, L., Zisman, S., Schejter, A., Visel, A., Klar, A. Transcriptional control of axonal guidance and sorting in dorsal interneurons by the Lim-HD proteins Lhx9 and Lhx1. Neural Development. 4, 21(2009).
- Wilson, N. H., Stoeckli, E. T. Cell type specific, traceable gene silencing for functional gene analysis during vertebrate neural development. Nucleic Acids Research. 39, e133(2011).
- Das, R. M., van Hateren, N. J., Howell, G. R., Farrell, E. R., Bangs, F. K., Porteous, V. C. A robust system for RNA interference in the chicken using a modified microRNA operon. Developmental Biology. 294, 554-563 (2006).
- Perrin, F. E., Stoeckli, E. T. Use of lipophilic dyes in studies of axonal pathfinding in vivo. Microscopy Research and Technique. 48, 25-31 (2000).
- Timmer, J., Johnson, J., Niswander, L. The use of in ovo electroporation for the rapid analysis of neural-specific murine enhancers. Genesis. 29, 123-132 (2001).
- Kieleczawa, J. Simple modifications of the standard DNA sequencing protocol allow for sequencing through siRNA hairpins and other repeats. Journal of Biomolecular Techniques. 16, 220-223 (2005).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. Journal of Morphology. 88, 49-92 (1951).
- Boulland, J., Halasi, G., Kasumacic, N., Glover, J. C. Xenotransplantation of Human Stem Cells into the Chicken Embryo. J. Vis. Exp. (41), e2071(2010).
- Kim, B. G., Dai, H. -N., McAtee, M., Vicini, S., Bregman, B. S. Labeling of dendritic spines with the carbocyanine dye DiI for confocal microscopic imaging in lightly fixed cortical slices. Journal of Neuroscience Methods. 162, 237-243 (2007).
- Murphy, M. C., Fox, E. A. Anterograde tracing method using DiI to label vagal innervation of the embryonic and early postnatal mouse gastrointestinal tract. Journal of Neuroscience Methods. 163, 213-225 (2007).
- Helms, A. W., Abney, A. L., Ben-Arie, N., Zoghbi, H. Y., Johnson, J. E. Autoregulation and multiple enhancers control Math1 expression in the developing nervous system. Development. 127, 1185-1196 (2000).
- Li, X., Lufkin, T. Cre recombinase expression in the floorplate, notochord and gut epithelium in transgenic embryos driven by the Hoxa-1 enhancer III. Genesis. 26, 121-122 (2000).
- Mauti, O., Baeriswyl, T., Stoeckli, E. T. G. ene Silencing by Injection and Electroporation of dsRNA in Avian Embryos. Cold Spring Harbor Protocols. , (2008).
- Krull, C. E. A primer on using in ovo electroporation to analyze gene function. Developmental Dynamics. 229, 433-439 (2004).
- Cullen, B. R. Enhancing and confirming the specificity of RNAi experiments. Nature Methods. 3, 677-681 (2006).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon