Method Article
Il Hindbrain Mouse come modello di studio della neurogenesi embrionale
In questo articolo
Riepilogo
Questo articolo viene illustrato come del hindbrain embrionali del mouse può essere utilizzato come modello per lo studio della neurogenesi inerente allo sviluppo sia dell'intero organo e tessuto sezione preparati.
Abstract
Il proencefalo di embrione di topo è il sistema più comunemente impiegato per lo studio della neurogenesi dei mammiferi durante lo sviluppo. Tuttavia, il neuroepithelium altamente piegato del forebrain non è assoggettabile a wholemount analisi per esaminare i modelli di organo-wide neurogenesi. Inoltre, i meccanismi della neurogenesi del forebrain la definizione non sono necessariamente predittivi della neurogenesi in altre parti del cervello; ad esempio, a causa della presenza dei sottotipi specifici del forebrain progenitrici. Il hindbrain del mouse fornisce un modello alternativo per lo studio della neurogenesi embrionale che è favorevole alla wholemount analisi, così come sezioni di tessuto per osservare la distribuzione spazio-temporale e il comportamento dei progenitori neurali. Inoltre, che facilmente viene sezionato per altre applicazioni a valle, quali analisi di isolamento o biologia molecolare delle cellule. Come il hindbrain del mouse può essere analizzato facilmente nel vasto numero di cella lignaggio reporter e ceppi di topi mutanti che si sono resi disponibili, offre un potente modello per lo studio dei meccanismi cellulari e molecolari della neurogenesi inerente allo sviluppo in un mammifero organismo. Qui, presentiamo un metodo semplice e rapido per utilizzare il hindbrain di embrione di topo per analizzare il comportamento delle cellule (NPC) progenitrici neurali dei mammiferi in preparazioni wholemount e sezioni di tessuto.
Introduzione
Durante lo sviluppo embrionale dei mammiferi, NPC dividono nelle subventricolare (SVZ) aree di espansione neuroepithelium e ventricolare (VZ) per generare nuovi neuroni in un processo chiamato 'neurogenesi'. La generazione di nuovi neuroni e loro precursori NPC è stata studiata estesamente nel proencefalo1, mentre di meno è conosciuto circa questo processo in altre regioni.
Proencefalo è una struttura complessa e intricata piegata che è in gran parte ha studiata con i metodi istologici dopo il taglio del tessuto, che rende i modelli di neurogenesi comprensione attraverso l'intero organo impegnativo. Inoltre, studi di neurogenesi del forebrain non sono necessariamente predittivi del comportamento neurogeno in altre regioni del cervello o del midollo spinale. Ad esempio, segnalazione cues può suscitare le risposte diverse in regioni distinte del CNS, come osservato nel caso di fattore neurotrofico ciliare e fattore inibitorio di leucemia, che promuovono auto-rinnovamento di NPC nel laterale gangliare Eminenza, ma in auto differenziazione dei progenitori di midollo spinale2. Inoltre, una sottoclasse di NPC che popolano il proencefalo in via di sviluppo e contribuire significativamente all'espansione della corteccia cerebrale1 sono assenti del hindbrain e del midollo spinale3. Inversamente, è concepibile che il midollo spinale e hindbrain contengano alternativi sottotipi NPC non presenti nella corteccia.
Il hindbrain di embrione di topo è la regione più antica evolutiva del cervello dei mammiferi e genera il cervelletto e il tronco cerebrale. Nonostante la sua conservazione in specie, relativamente piccolo è conosciuto circa neurogenesi hindbrain, tra cui sottotipi NPC o loro regolazione. La maggior parte della ricerca del hindbrain nel topo si è concentrata sul processo di segmentazione del tessuto, guidata da geni di Hox4e la campitura di neuroni post-mitotici5. Inoltre, il hindbrain è stato utilizzato come modello per studiare i meccanismi di angiogenesi inerente allo sviluppo6.
In contrasto con il hindbrain del mouse, del hindbrain di zebrafish è stato usato estesamente per seguire NPC differenziazione e progressione di lignaggio in un organismo vertebrato modello (ad esempio,7,8). Il hindbrain pulcino è stato impiegato anche per studiare la neurogenesi durante lo sviluppo dei vertebrati (per esempio,9,10). Simile al zebrafish hindbrain11, il hindbrain pulcino può essere dal vivo imaged per studiare il comportamento NPC e regolamento nel tempo12. Analoga osservazione longitudinale da formazione immagine dal vivo non è attualmente possibile negli organismi dei mammiferi, perché si sviluppano nell'utero. Inoltre, mirata manipolazione attraverso tecniche come l'elettroporazione può essere facilmente applicato a embrioni di zebrafish dissipati o pulcino embrioni in ovo (ad es.,13), ma tali tecniche sono anche più impegnativo in utero.
Tuttavia, il hindbrain di embrione di topo è squisitamente adatto per definire i meccanismi molecolari e cellulari che regolano la neurogenesi. In primo luogo, analisi del hindbrain mouse fornirà, in molti casi, informazioni più pertinenti agli sviluppi di esseri umani di quella ottenuta attraverso lo studio di vertebrati inferiori. Mette inoltre a disposizione un vasto numero di ceppi di topi geneticamente modificati che può essere usato per destino Mappatura murino NPC o alterare meccanismi regolatori con alleli del mutante condizionali o costitutivi dei geni rilevanti. Infine, recentemente è stato dimostrato che microinjection del hindbrain ventricolare progenitori ex vivo permette almeno un breve esame del hindbrain NPC cinetica14. Ancora, allo stato attuale, pochissimo è conosciuto circa l'organizzazione spazio-temporale e il comportamento del hindbrain NPC in un contesto di organo intero.
Qui, dimostriamo un metodo semplice e rapido per utilizzare il hindbrain come un potente modello per analizzare il comportamento dei mammiferi di NPC in preparazioni wholemount e sezioni di tessuto. Forniamo ulteriori protocolli per utilizzare immunolabeling per lo studio di parametri differenti neurogenesi e processo hindbrain campioni ulteriormente per applicazioni molecolari a valle come trascrittasi inversa quantitativa (qRT)-PCR.
Protocollo
Tutto il lavoro animale è stato effettuato secondo UK Home Office e orientamenti etici locali.
1. Riepilogo dei passaggi e la tempistica
- Eseguire accoppiamenti temporizzati di topi adulti da un ceppo adatto a rispondere alla domanda biologica sotto inchiesta per ottenere gravidanze di 9,5-e13.5 giorno embrionale (e); richiede 12-15 giorni.
- Facoltativamente, preparare 5-bromo-2'-deoxyuridine (BrdU) / soluzione 5-Etinil-2'-deoxyuridine (EdU) ed eseguire l'iniezione (protocollo sezione 2); richiede ~ 1 h il giorno prima o il giorno di isolamento dell'embrione, a seconda della lunghezza desiderata della etichettatura di EdU/BrdU.
- Eseguire la dissezione di isolamento e hindbrain embrione (protocollo sezione 3); richiede ~ 10 min/embrione.
- Eseguire wholemount immunofluorescenza etichettatura (protocollo sezione 4); richiede 3 giorni.
- Sezione utilizzando un vibratomo ed eseguire immunofluorescenza di sezione galleggiante etichettatura (protocollo sezione 4): richiede 2 giorni.
- Sezione utilizzando un criostato ed eseguire immunofluorescenza etichettatura di cryosections (protocollo sezione 5); richiede 2 giorni.
2. iniettare Mouse femmina incinto con BrdU o EdU (facoltativo)
- Sciogliere BrdU o EdU in soluzione salina sterile tamponata al fosfato (PBS) a concentrazioni di 10 mg/mL e 1 mg/mL, rispettivamente.
Attenzione: BrdU ed EdU sono tossici; indossare una protezione appropriata. - Pesare il mouse incinto e calcolare il volume di soluzione di BrdU o EdU che deve essere somministrato per raggiungere 100 mg/kg BrdU o 5 mg/kg EdU.
- Iniettare la soluzione di BrdU o EdU per via intraperitoneale o 1 h o 1 giorno prima della raccolta degli embrioni, a seconda della lunghezza necessaria etichettatura.
Nota: Etichettatura per 1 h Visualizza cellule hindbrain in fase S. Etichettatura per 1 giorno Visualizza la progenie del hindbrain NPC.
3. dissezione di Hindbrains da e9.5 - embrioni del Mouse e13.5
- Eutanasia di un topo femmina temporizzato-incinta utilizzando una procedura eticamente approvata nella fase gestazionale richiesto (ad es., la dislocazione cervicale). Utilizzando delle forbici affilate, tagliare aperto della cavità peritoneale e attentamente asportare l'utero. Posizionare l'utero asportato contenente gli embrioni in un piatto di plastica 60 mm contenente 20 mL di PBS ghiacciata.
Nota: La tecnica asettica non è richiesta. - Eseguire tutte le ulteriore dissezione utilizzando un microscopio per dissezione. Utilizzando orologiaio numero forcipe 5, rottura della parete muscolare uterina per esporre gli embrioni, rilasciare ogni embrione da recidere il cordone ombelicale e rimuovere il sacco vitellino.
- Utilizzando una pipetta Pasteur sterile con apertura larga-foro, trasferire ogni embrione in un piatto di plastica pulito con PBS ghiacciata.
- Utilizzando orologiaio forcipe numero 55, recidere la testa (Figura 1E).
- Facoltativamente, conservano un piccolo pezzo di tessuto (ad es., ¼ di un vitellino o la punta della coda circa 2 mm) per isolamento di DNA genomic e genotipizzazione successiva.
- Utilizzando orologiaio forcipe numero 55, asportare il tessuto che contiene il hindbrain parallelo e immediatamente sotto il neuroepithelium hindbrain separato dal tessuto facciale e del forebrain (Figura 1F).
- Posizione del hindbrain e lato dorsale del tessuto testa caudale e identificare ventricoloth 4, che è coperto da un sottile strato di tessuto (piatto di tetto). Attentamente forare la lamiera del tetto utilizzando orologiaio forcipe numero 55 (Figura 1) e la buccia assente tessuto in eccesso con il forcipe, rostralmente in movimento lungo la linea mediana sopra il mesencefalo e poi caudalmente sopra il posteriore hindbrain e del midollo spinale (Figura 1 ); il hindbrain ora deve essere esposta in preparazione un libro aperto (Figura 1 H).
- Preparare hindbrains per wholemount immunolabeling (opzione 1).
- Utilizzando orologiaio numero forcipe 55, attentamente tease distanza rimanente testa mesenchima e qualsiasi meningi attaccate al lato pial del hindbrain usando il forcipe (Figura 1I). Rimuovere il mesencefalo e il tessuto del midollo spinale (Figura 1J) per lasciare solo il tessuto del hindbrain (Figura 1 K).
Nota: Questo passaggio della procedura non deve essere seguito a e9.5 o e 10.5, perché tenta di farlo probabilmente danneggerebbe il neuroepithelium hindbrain; Inoltre, rimuovendo le meningi non è essenziale perché il neuroepithelium hindbrain è sufficientemente sottile in questa fase per consentire la penetrazione efficiente degli anticorpi per immunolabeling. Questo passaggio dovrebbe, tuttavia, essere seguita da e11.5 in poi, quando il tessuto meningeo consolida, per migliorare la penetrazione dell'anticorpo neuroepithelium hindbrain. La dissezione è più semplice quando eseguita in PBS ghiacciata (uso pulito PBS per ogni hindbrain).
- Utilizzando orologiaio numero forcipe 55, attentamente tease distanza rimanente testa mesenchima e qualsiasi meningi attaccate al lato pial del hindbrain usando il forcipe (Figura 1I). Rimuovere il mesencefalo e il tessuto del midollo spinale (Figura 1J) per lasciare solo il tessuto del hindbrain (Figura 1 K).
- Preparare la hindbrains per Vibratomo e cryosectioning (opzione 2)
- Utilizzando orologiaio forcipe numero 55, rimuovere il mesencefalo ed il tessuto del midollo spinale (Figura 1J).
Nota: Non è necessario rimuovere il mesenchima e nelle meningi di hindbrains destinati al sezionamento (come mostrato in Figura 1I).
- Utilizzando orologiaio forcipe numero 55, rimuovere il mesencefalo ed il tessuto del midollo spinale (Figura 1J).
- Trasferimento di hindbrains dal piatto di plastica per provette a fondo tondo 2 mL utilizzando una pipetta Pasteur, aspirare tutte PBS e difficoltà per 2 h a 4° C con brezza-agitazione in appena scongelati formaldeide di 4% w/v disciolto in PBS.
Attenzione: La formaldeide è tossica; indossare una protezione appropriata. - Sciacquare hindbrains x 3 volte con PBS. Conservare a 4 ° C in PBS se immunolabeling sarà iniziare entro 2-3 giorni o sostituire PBS con 50% methanol/50% PBS per 5 min prima del trasferimento al 100% di metanolo per conservarli più a lungo a-20° C.
4. Wholemount immunofluorescenza
- Se necessario, reidratare hindbrains in serie decrescente diluizioni di metanolo in PBS (ad es., 75% metanolo, metanolo al 50%, 25% metanolo) a temperatura ambiente (TA) per 5 minuti ciascuno e poi trasferire su PBS.
Nota: Una serie graduata di metanolo è necessaria reidratare hindbrains e garantire la conservazione corretta dei tessuti delicatamente. - Permeabilize hindbrains per 30 min a 4 ° C in PBS contenente 0,1% Triton X-100 (PBT) con agitazione delicata.
- Incubare hindbrains per 1 h a 4 ° C in PBT contenente 10% siero di capra inattivati con agitazione delicata.
Nota: Usare il siero da specie ospiti che gli anticorpi secondari sono stati sollevati in. Per gli anticorpi primari generati in capra, Incubare in privo di siero proteine bloccare, ad esempio 5% albumina di siero bovino in PBS o un'alternativa commerciale adatta (Vedi Tabella materiali). Questo ridurrà la colorazione osservata frequentemente quando usando gli anticorpi primari generati in capra aspecifiche. - Incubare hindbrains durante la notte a 4 ° C in PBT contenente 1% di siero inattivato con calore e gli anticorpi primari con agitazione delicata (ad es., istone di coniglio anti-fosfo H3 [pHH3] diluito 1: 400).
Nota: Per gli anticorpi primari generati in capra, utilizzare PBT senza siero. - Lavare la hindbrains a 4 ° C 5 x con PBT per 1h ciascuna.
- Incubare hindbrains durante la notte a 4 ° C in PBT contenente appropriati fluoroforo secondari anticorpi coniugati (ad es., capra anti-coniglio Alexa Fluor 488) a 1: 200 in PBT mirate contro gli anticorpi primari utilizzati. Tenere hindbrains nel buio da qui per proteggere i fluorophores da photobleaching.
Nota: Per gli anticorpi primari generati in capra, utilizzare anti-capra frammenti Fab di anticorpi secondari per ridurre la colorazione aspecifica. - Lavare la hindbrains a 4 ° C 5 x con PBT per 1h ciascuna.
- Postfix hindbrains in formaldeide 4% in PBS per 15 min a RT per conservazione a lungo termine di grippaggio dell'anticorpo. Risciacquare brevemente due volte in PBS.
- Coprire un vetrino per microscopio con due strati di nastro isolante nero e asportare un piccolo quadrato dal nastro a più livelli per creare una tasca sufficientemente grande da contenere un hindbrain.
- Trasferire ogni hindbrain in una tasca con una pipetta Pasteur, rimuovere il liquido in eccesso e aggiungere un reagente antifade appropriato alla tasca prima di coprirla lentamente con un vetrino coprioggetti per evitare bolle d'aria di intrappolamento sotto il vetrino coprioggetti. Sigillare il vetrino coprioggetto e fissarlo alla diapositiva con un sottile strato di smalto. Conservare il vetrino a 4 ° C al buio fino alla acquisizione immagine (protocollo sezione 7).
5. Vibratome sezionamento e sezione immunofluorescenza di galleggiamento
- Se necessario, reidratare hindbrains da metanolo come descritto al punto 4.1.
- Incorporare hindbrains nel fuso 3% p/v agarosio preparato in acqua distillata.
Nota: Consentire agarosio fuso raffreddare a circa 55° C brevemente prima di incorporare per evitare danni dovuti al calore hindbrain. - Tagliare sezioni trasversali hindbrain ad uno spessore di 70 µm utilizzando un vibratomo. Trasferire ogni sezione appena tagliata con un pennello in un pozzetto di una piastra a 24 pozzetti contenenti PBS ghiacciata.
- Etichettare le sezioni galleggiante come descritto ai punti 4.2-4.7, ma modificare la procedura come segue: lavare il galleggiante sezioni a 4 ° C 3x con PBT per 15 min ciascuno, diminuire il tempo di incubazione per anticorpi secondari a 2h e incubare in anticorpo secondario a TA.
- Facoltativamente, dopo l'etichettatura con gli anticorpi per altri epitopi come descritto al punto 5.4, rilevare i nuclei di EdU+ utilizzando un EdU etichettatura kit secondo le istruzioni del produttore. Incubare le sezioni galleggiante nella reazione cocktail per 30 min a 37 ° C al buio. Lavare hindbrain sezioni a 4 ° C x 3 con PBT per 15 minuti ciascuno.
- Facoltativamente, dopo l'etichettatura per altri epitopi come descritto al punto 5.4, rilevare i nuclei BrdU+ come segue.
- Incubare il galleggiante sezioni in acido cloridrico N 2 a 37 ° C per 30 min al buio.
- Incubare il galleggiante sezioni in tampone borato di sodio 0,1 M a pH 8,5 due volte per 5 min a temperatura ambiente al buio per neutralizzare l'acido cloridrico.
- Lavare sezioni galleggiante 3 x brevemente in PBS a 4 ° C.
- Eseguire immunofluorescenza di etichettatura per BrdU come descritto al punto 5.4.
- Incubare le sezioni galleggiante per 2 min a RT a 10 µ g/mL 4', 6-diamidino-2-phenylindole dicloridrato (DAPI) in PBS per colorante di contrasto nuclei delle cellule.
- Postfix le sezioni galleggiante in formaldeide 4% per 15 minuti a TA.
- Lavare galleggiante sezioni brevemente in PBS. Trasferire con cautela sezioni galleggiante per un vetrino per microscopio utilizzando un pennello. Assorbire l'eccesso di PBS intorno alla sezione utilizzando la carta assorbente o tessuto.
- Montare le sezioni utilizzando lentamente 80% glicerolo in PBS e coprire con un vetrino coprioggetti per evitare bolle d'aria di intrappolamento. Conservare il vetrino a 4 ° C al buio fino alla acquisizione immagine (passaggio 7).
6. criostato sezionamento e donatrici immunofluorescenza etichettatura
- Se necessario, reidratare hindbrains da metanolo come descritto al punto 4.1.
- Incubare hindbrains in saccarosio 30% w/v in PBS per cryoprotect hindbrains prima del congelamento.
Nota: Hindbrains sono pronti per il congelamento quando hanno affondato alla parte inferiore del tubo, che in genere dura ≤ 2h per hindbrains e9.5-10.5 e 3-6 h per hindbrains e11.5-13.5. - Immergere hindbrains in temperatura di taglio ottico composto (OCT) e congelare rapidamente trasferendo stampi contenenti hindbrains in ottobre a isopentano raffreddato a tra-40 ° C e -50 ° C il ghiaccio secco.
Nota: Hindbrains congelati, incorporato può essere conservata a-20 ° C a breve e a lungo termine-80 ° C fino al sezionamento. - Tagliare sezioni trasversali hindbrain ad uno spessore di 10 µm utilizzando un criostato e trasferimento a vetrini da microscopio elettrostaticamente adesivo.
Nota: Hindbrain cryosections possono essere conservati a-20 ° C a breve e a lungo termine-80 ° C fino a etichettatura. - Incubare i vetrini a temperatura ambiente per 15 minuti ad asciugare le sezioni per le diapositive. Lavare cryosections con PBS per sciogliere OCT e mark una barriera idrofoba intorno cryosections utilizzando una penna PAP. Ripetere i passaggi da 5.4-5.8.
- Ripetere il passaggio 5.10 usando un mezzo di montaggio a base alcool polivinilico al posto di glicerolo.
7. acquisizione immagini
- Esempi di immagine utilizzando un epifluorescente o un microscopio confocale laser-scanner dotati di lenti adatte per diapositive di supporto montato acquose e filtri ottici adatti per fluorofori utilizzati per immunolabeling.
- Per realizzare l'immagine del hindbrain intera o una sezione del hindbrain, usare una lente di ingrandimento 10x (ad es., fig. 2B); per visualizzare le aree di hindbrains per la quantificazione, è necessario utilizzare un 40 X ingrandimento (ad es., Figura 2D) e per visualizzare le singole celle, utilizzare una lente con un ingrandimento di 63 x (ad esempio, Figura 2).
8. metodi alternativi
- Seguito passo 3.8, omogeneizzare unfixed hindbrains per produrre una sospensione singola cella per citometria a flusso applicazioni15.
- Seguito passo 3.8, omogeneizzare unfixed hindbrains per estrarre RNA da sospensioni di singola cellula per RT-qPCR (ad es., 16).
Nota: Assicurarsi che tutti i reagenti e le attrezzature sono tenute sterile e RNAsi-libera in tutto per evitare la degradazione del RNA. - Seguito passo 3.8, omogeneizzare unfixed hindbrains per isolare gli NPC e propagarle in vitro come neurosfere per analisi del hindbrain NPC comportamento17.
Nota: Assicurarsi che tutti i reagenti e le attrezzature sono tenute sterile per prevenire la contaminazione batterica/fungina delle culture neurosphere.
Risultati
In questa sezione sono riportati esempi di risultati che possono essere ottenuti quando si studia la neurogenesi del hindbrain embrionali del mouse attraverso l'analisi di sezione wholemount e tessuto.
Indichiamo che immunolabeling wholemount del hindbrain microdissected con un anticorpo per il pHH3 di mitotic marcatore Visualizza dividendo i PNG di VZ (Figura 2B – D). Vi mostriamo pHH3+ NPC a un forte ingrandimento per evidenziare le diverse fasi della mitosi (Figura 2). Abbiamo illustrato che questo metodo di etichettatura è adatto per essere eseguita attraverso diverse tappe consecutive dello sviluppo del hindbrain di osservare il corso di tempo della mitosi NPC in questo organo (Figura 2D).
Indichiamo che l'imaging trasversale immunolabeled vibratome sezioni del hindbrain 1 h dopo l'iniezione di EdU, Visualizza l'orientamento di clivaggio degli NPC mitotico (Figura 3B), il pattern di migrazione nucleare pseudostratified, intercinetica del ciclismo progenitori18 (Figura 3B, D) e la struttura complessiva di VZ (Figura 3B – D). Si noti che pHH3 mitotica+ NPC sono presenti solo alla superficie ventricolare e non più basally (Figura 3), che contrasta il modello di divisione basale dei PNG più impegnati nel proencefalo19.
Illustriamo anche come ciclismo NPC e la loro progenie differenziata possa essere etichettato con BrdU o EdU per valutare la progressione di lignaggio NPC (Figura 4). Il immunolabeling di cryosections trasversale del hindbrain mouse 1 giorno dopo l'iniezione di BrdU per BrdU e Ki67viene illustrato il numero e il posizionamento del ciclismo NPC in neuroepithelium (Figura 4A, B), per cui il numero di autorinnovanti NPC può essere definito calcolando la percentuale di Ki67+ + di BrdU cellule tra tutte le celle di BrdU+ (Figura 4A, C).
Infine, vi mostriamo un esempio di immunolabeling di sezioni vibratome hindbrain per RC2, un antigene nel filamento intermedio neurali specifici nestina, per visualizzare NPC endfeet (figura 5B) e processi (Figura 5). Sezioni del VIBRATOME, anziché cryosections sottili, permettono una migliore osservazione degli NPC altamente ramificati e anche della struttura complessiva di neuroepithelial.

Figura 1 : Passi cruciali nella dissezione di un hindbrain da un embrione di topo e11.5. (A – D) Raffigurazione schematica del hindbrain microdissection. (A) il tessuto testa caudale viene rimosso da recidere lungo le linee tratteggiate rosse. (B) lamiera del tetto è trafitto e poi strappato via in direzione rostrale e caudale lungo la linea mediana (linee verticali rosse) e poi lateralmente per rivelare il neuroepithelium hindbrain. (C) il neuroepithelium è curiosato lontano le meningi dopo aver inserito il forcipe tra entrambe le strutture e i tessuti del midbrain e del midollo spinale vengono rimossi da recidere il tessuto con il forcipe lungo le linee tratteggiate rosse. (D) questa procedura produce un hindbrain appiattito. (E–K) Acquisizione di immagini delle fasi principali della procedura di microdissection del hindbrain. (E), l'embrione viene decapitato da recidere lungo la linea tratteggiata. (F) del hindbrain e 4th ventricolo, racchiuso da lamiera del tetto, vengono asportati e separati dalla testa da recidere lungo le linee tratteggiate. (G) il ventricoloth 4 è orientato verso l'alto prima un piccolo foro è perforato in lamiera del tetto (asterisco). Il foro è allargato dal peeling il tessuto con cura sia rostralmente caudalmente lungo la linea mediana (frecce) per esporre il hindbrain. Il hindbrain è posizionato pial lato verso il basso e a sinistra per piegare fuori in preparazione un libro aperto (H; nero Sagittaria indica tessuto neurale hindbrain). Se il hindbrain è necessaria per wholemount immunolabeling, la membrana pial viene rimosso facendo leva delicatamente il neuroepithelium hindbrain dalle membrane meningee circostante usando il forcipe (io). L'eccesso di mesencefalo (mb) e del midollo spinale (sc) il tessuto è rimosse (linee tratteggiate in J) per isolare il hindbrain (K). Barra della scala: 500 µm per (E), 300 µm per (F – K). Clicca qui per visualizzare una versione più grande di questa figura.
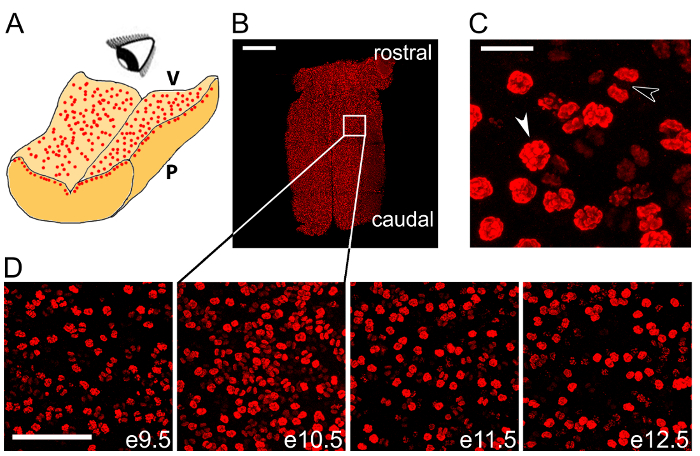
Figura 2 : Wholemount immunolabeling può essere utilizzato per quantificare i mitoses NPC attraverso il hindbrain. Schema (A) illustrando en affrontare imaging di NPCs mitotica nello strato ventricolare hindbrain. V, lato ventricolare hindbrain; P, lato pial hindbrain. (B) mattonelle confocale scansione stack z del hindbrain e 10.5 seguendo wholemount immunolabeling per la pHH3 di mitotic indicatore (rosso). La scatola bianca indica l'area mostrata a maggiore ingrandimento nel secondo pannello (D). (C) pre-anafase (freccia bianca) e le figure mitotic anafase (freccia nera) sono distinguibili all'interno della coorte totale di mitotic NPC. (D) corso di tempo wholemount pHH3 etichettatura da e9.5-13,5. Scala bar: 500 µm (B); 20 µm (C); 100 µm (D). Clicca qui per visualizzare una versione più grande di questa figura.
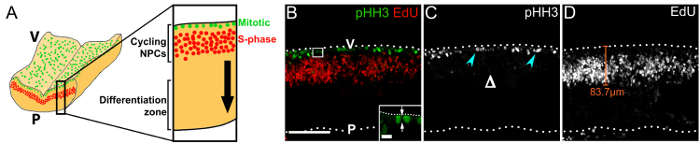
Figura 3 : I PNG in bicicletta all'interno della loro zona germinale del hindbrain rostrale. Schema (A) raffigurante la posizione di mitotica (verde) e NPC S-fase (rosso) alla superficie ventricolare del hindbrain e nell'area periventricolare, rispettivamente. Una sezione trasversale virtuale attraverso il hindbrain viene visualizzata a maggiore ingrandimento sulla destra. La freccia nera indica la direzione della migrazione della progenie NPC che hanno terminato il ciclo cellulare verso la zona di differenziazione. Stack di Confocal z (B) di una sezione del hindbrain e11.0 galleggiante da un embrione che ha ricevuto un 1h EdU impulso; immunolabeling per pHH3 ed EdU è stato usato per prevedere mitotica (verde) e gli NPC S-fase (rosso), rispettivamente. Superfici del hindbrain sono indicate da linee tratteggiate; V, lato ventricolare hindbrain; P, lato pial hindbrain. L'area indicata da una scatola bianca è mostrato ad più alto ingrandimento nella rientranza; le frecce indicano l'aereo di fenditura di un NPC in anafase, riguardante la superficie ventricolare. (C) singolo canale visualizzazione pHH3 immunolabeling solo. NPC mitotic sono indicati da frecce ciano; la mancanza di divisioni basale è indicata da Δ. (D) visualizzati da EdU etichettatura solo monocanale. EdU+ NPC adottare una distribuzione pseudostratified a causa della loro migrazione nucleare intercinetica. La distanza che i PNG migrano lontano dalla superficie ventricolare può essere misurata, come indicato con una linea arancione rappresentativa. Scala bar: 100 µm (B -D); 10 µm in inserto in (B). Clicca qui per visualizzare una versione più grande di questa figura.
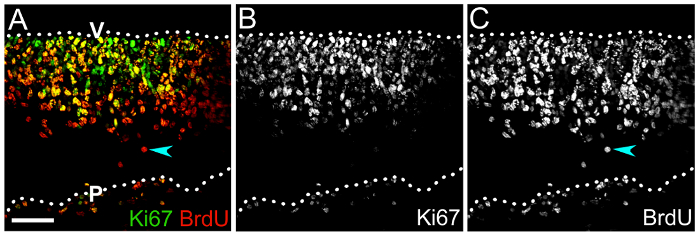
Figura 4 : Combinando BrdU con etichettatura Ki67 per determinare la capacità di auto-rinnovamento NPC hindbrain. (A) dello stack z Confocal di un donatrici del hindbrain e 10.5 dopo l'etichettatura per Ki67 (verde) e impulso di BrdU (rosso) seguendo un 1 giorno BrdU. Le cellule doppio-positivi nella zona ventricolare sono gli NPC che hanno incorporato BrdU e sono ancora in movimento attraverso il ciclo cellulare, come indicato dall'etichettatura Ki67. La proporzione di cellule doppio-identificato tra la popolazione totale di BrdU+ rappresenta la percentuale di auto-propagarsi NPC. (B, C) singoli canali visualizzati Ki67 (B) e immunolabeling di BrdU (C) solo. Una cella di BrdU+ che manca di Ki67 e quindi probabilmente è stato terminato il ciclo cellulare è indicata da una freccia ciano (A,C). Superfici del hindbrain sono indicate da linee tratteggiate; V, lato ventricolare hindbrain; P, lato pial hindbrain. Barra della scala: 50 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5 : Immunolabeling nestina Visualizza processi NPC ed endfeet. (A – C) Immunofluorescenza per RC2, che riconosce un epitopo sul filamento intermedio neurale nestina, illustra la morfologia NPC all'interno neuroepithelium hindbrain. Stack di Confocal z (A) di una sezione galleggiante da un hindbrain e11.5 seguito RC2 immunolabeling; le zone "in box" vengono mostrate a maggiore ingrandimento in (B, B'; regione apicale/ventricolare) e (C, C'; basale regione). Superfici del hindbrain sono indicate da linee tratteggiate; V, lato ventricolare hindbrain; P, lato pial hindbrain. (B, C) Pile di Confocal z di zone "in box" in (A); singole sezioni ottiche sono mostrate in ('B, C'), rispettivamente. Un NPC apicale endfoot ancorato alla superficie ventricolare è indicata da una freccia (B'). Singoli e scanditi NPC processi sono indicati da frecce bianche e nere (C'), rispettivamente. Scala bar: 100 µm, (A); 25 µm per (B, C). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Questo protocollo viene descritto come utilizzare il hindbrain embrionali del mouse come un modello per studiare i meccanismi della neurogenesi inerente allo sviluppo. Utilizzando una varietà di metodi diversi immunolabeling, possono essere fruiti hindbrain NPC e il loro numero quantificato in sezioni di tessuto o organo wholemounts. La facilità di dissezione e anatomia piatto assicura che il hindbrain può essere imaged in una preparazione di 'libro aperto' per raccogliere informazioni sui modelli di livello di organo neurogenesi.
Più ulteriormente indichiamo che NPC morfologia e ciclo cellulare relativi NPC posizionamento possono essere facilmente visualizzati in galleggiante- o cryosections del hindbrain. Entrambi i comportamenti possono essere sfruttati per definire nuove popolazioni di cellule progenitrici, come in precedenza è stata eseguita nel telencefalo dei mammiferi20. Ad esempio, primi formata Sox2+ neuroepithelia e glia radiale apicale di Pax6+ sono presenti nel hindbrain21,22, ma manca il hindbrain Tbr2+ basali progenitori3 .
Il protocollo descritto qui può anche essere adattato per osservare il comportamento di specifiche sottopopolazioni di NPC di etichettatura fluorescente per formazione immagine dal vivo e/o l'analisi di lignaggio in tessuti fissi. Ciò può essere ottenuto, ad esempio, studiando hindbrains da topi portatori di tamoxifen-inducible transgene Sox1-iCreERT2 e il reporter Rosa26tdTomato 23.
Oltre a migliorare la conoscenza della neurogenesi murina, studiando il hindbrain può delucidare largamente pertinenti meccanismi neurogeni che sono condivise tra specie, perché il hindbrain è una regione altamente conservata del cervello che dovrebbe essere più simile tra le specie di vertebrati rispetto del forebrain.
Come nella neurogenesi hindbrain avviene sopra una finestra di tempo comparativamente più breve rispetto del forebrain neurogenesi23, è importante considerare la necessità di confrontare adeguatamente in scena gli embrioni. Di conseguenza, bias sperimentale è evitato da conteggio e registrazione del numero di coppie di somite in un embrione prima del suo hindbrain di isolamento. Il tessuto del hindbrain stesso è fragile e forcipe dovrebbero pertanto essere gestito con attenzione quando separando il tessuto del hindbrain dal mesenchima testa e meningi; un paio di 'pratica corre' quindi potrebbe essere consigliabile prima tentativo di dissezione di embrioni preziosi. Inoltre, hindbrains dovrebbe essere trasferito da una provetta a altra utilizzando una pipetta di Pasteur wide-bore, piuttosto che con il forcipe per evitare danni (apertura della pipetta può essere ampliata con il taglio della punta con forbici pulite). Infine, anche se raro, nella misura di EdU/BrdU incorporazione in fase S NPC può essere variabile, in particolare durante gli impulsi brevi (cioè, 1 h). Per migliorare l'etichettatura, garantire che EdU/BrdU è dissolto correttamente prima di caricare la soluzione nella siringa e iniettare con cura nella cavità peritoneale. Iniezioni di povere, come quelli realizzati per via sottocutanea da incidente, verranno causare perdere o intrappolando la soluzione EdU/BrdU e impedire di entrare la circolazione.
Divulgazioni
Nessuno degli autori hanno interessi concorrenti o conflitto di interessi.
Riconoscimenti
Ringraziamo Vasiliki Chantzara per lo svolgimento di accoppiamenti temporizzati e il personale dell'unità risorse biologiche presso la UCL Institute of Ophthalmology per allevamento del mouse. Questo studio è stato supportato da un Wellcome Trust Investigator Award 095623/Z/11/Z a CR.
Materiali
| Name | Company | Catalog Number | Comments |
| Round-bottomed reagent tube, 2.0 ml (Safe-Lock) | VWR | 211-2120 | Also available from other commercial suppliers |
| Plastic cell culture dish, 60 mm | Thermo Fisher | 150288 | Also available from other commercial suppliers |
| Cell culture plates, 12-well | Thermo Fisher | 150628 | Also available from other commercial suppliers |
| Pasteur pipettes | Copan | 200C | Also available from other commercial suppliers |
| Dumont Watchmaker forceps, no. 5 | FST | 91150-20 | |
| Dumont Watchmaker forceps, no. 55 | FST | 11295-51 | |
| 29G needle/syringe | BD | BD Micro-Fine +1ml | |
| Anti-phospho-histone H3 primary antibody | Millipore | 06-570 | Goat, dilution 1:400 |
| Anti-BrdU primary antibody | Abcam | ab6326 | Rat, dilution 1:400 |
| Anti-Ki67 primary antibody | BD Biosciences | 550609 | Mouse, dilution 1:400 |
| Anti-RC2 primary antibody | Developmental Studies Hybridoma Bank | RC2 | Mouse (IgM), dilution 1:400 |
| Alexa Fluor 488-conjugated goat anti rabbit secondary antibody | Thermo Fisher | A11029 | Dilution 1:200 |
| Alexa Fluor 594-conjugated goat anti rabbit secondary antibody | Thermo Fisher | A11037 | Dilution 1:200 |
| Alexa Fluor 488-conjugated goat anti mouse IgM secondary antibody | Thermo Fisher | A21042 | Dilution 1:200 |
| Alexa Fluor 488-conjugated goat anti mouse secondary antibody | Thermo Fisher | A11001 | Dilution 1:200 |
| Alexa Fluor 594-conjugated goat anti rat secondary antibody | Thermo Fisher | A11007 | Dilution 1:200 |
| 4',6-Diamidino-2-Phenylindole, Dihydrochloride (DAPI) | Sigma | D9542 | Use at 10 μg per mL |
| 5-ethynyl-2’-deoxyuridine (EdU) | Sigma | 900584 | |
| 5-bromo-2´-deoxyuridine (BrdU) | Sigma | B5002 | |
| Click-iT EdU Alexa Fluor 594 Imaging Kit | Thermo Fisher | C10086 | |
| Heat-inactivated goat serum | Sigma | G9023 | |
| Bovine serum albumin | Sigma | A7906 | |
| DAKO protein-block serum free | Agilent | X0909 | |
| Triton X-100 | Sigma | T8787 | Also available from other commercial suppliers |
| Phosphate buffer saline | Sigma | P4417 | Also available from other commercial suppliers |
| Paraformaldehyde | Sigma | P6148 | Also available from other commercial suppliers |
| Sodium tetraborate | Sigma | B9876 | Also available from other commercial suppliers |
| Sucrose | Sigma | S0389 | Also available from other commercial suppliers |
| Agarose | Sigma | A9539 | Also available from other commercial suppliers |
| Super PAP pen liquid blocker | Ted Pella, Inc. | 22309 | |
| SlowFade Antifade Kit | Thermo Fisher | S-2828 | |
| Glycerol | Fisher Scientific | 10337700 | |
| Mowiol | Millipore | 475904 | |
| OCT | Scigen | 4583 | Also available from other commercial suppliers |
| Isopentane | Sigma | M32631 | Also available from other commercial suppliers |
| Methanol | Thermo Fisher | 10675112 | Also available from other commercial suppliers |
| Hydrochloric acid | Thermo Fisher | 10316380 | Also available from other commercial suppliers |
| Microscope slides | VWR | 631-0912 | |
| Superfrost microscope slides | VWR | 631-0108 | |
| Thermometer | VWR | 620-0858 | |
| Cover glass | VWR | 631-0137 | |
| Stereo Microscope, Leica MZ16 | Leica | not applicable | |
| Confocal laser scanning microscope LSM710 | Zeiss | not applicable |
Riferimenti
- Wilsch-Brauninger, M., Florio, M., Huttner, W. B. Neocortex expansion in development and evolution - from cell biology to single genes. Curr Opin Neurobiol. 39, 122-132 (2016).
- Gregg, C., Weiss, S. CNTF/LIF/gp130 receptor complex signaling maintains a VZ precursor differentiation gradient in the developing ventral forebrain. Development. 132 (3), 565-578 (2005).
- Kwon, G. S., Hadjantonakis, A. K. Eomes::GFP-a tool for live imaging cells of the trophoblast, primitive streak, and telencephalon in the mouse embryo. Genesis. 45 (4), 208-217 (2007).
- Krumlauf, R. Hox Genes and the Hindbrain: A Study in Segments. Curr Top Dev Biol. 116, 581-596 (2016).
- Guthrie, S. Patterning and axon guidance of cranial motor neurons. Nat Rev Neurosci. 8 (11), 859-871 (2007).
- Fantin, A., Vieira, J. M., Plein, A., Maden, C. H., Ruhrberg, C. The embryonic mouse hindbrain as a qualitative and quantitative model for studying the molecular and cellular mechanisms of angiogenesis. Nat Protoc. 8 (2), 418-429 (2013).
- Terriente, J., Gerety, S. S., Watanabe-Asaka, T., Gonzalez-Quevedo, R., Wilkinson, D. G. Signalling from hindbrain boundaries regulates neuronal clustering that patterns neurogenesis. Development. 139 (16), 2978-2987 (2012).
- Coolen, M., Thieffry, D., Drivenes, O., Becker, T. S., Bally-Cuif, L. miR-9 controls the timing of neurogenesis through the direct inhibition of antagonistic factors. Dev Cell. 22 (5), 1052-1064 (2012).
- Dias, J. M., Alekseenko, Z., Applequist, J. M., Ericson, J. Tgfbeta signaling regulates temporal neurogenesis and potency of neural stem cells in the CNS. Neuron. 84 (5), 927-939 (2014).
- Jacob, J., et al. Retinoid acid specifies neuronal identity through graded expression of Ascl1. Curr Biol. 23 (5), 412-418 (2013).
- Alexandre, P., Reugels, A. M., Barker, D., Blanc, E., Clarke, J. D. Neurons derive from the more apical daughter in asymmetric divisions in the zebrafish neural tube. Nat Neurosci. 13 (6), 673-679 (2010).
- Peretz, Y., et al. A new role of hindbrain boundaries as pools of neural stem/progenitor cells regulated by Sox2. BMC Biol. 14, 57 (2016).
- Graeden, E., Sive, H. Live imaging of the zebrafish embryonic brain by confocal microscopy. J Vis Exp. (26), (2009).
- Taverna, E., Haffner, C., Pepperkok, R., Huttner, W. B. A new approach to manipulate the fate of single neural stem cells in tissue. Nat Neurosci. 15 (2), 329-337 (2011).
- Menon, V., Thomas, R., Ghale, A. R., Reinhard, C., Pruszak, J. Flow cytometry protocols for surface and intracellular antigen analyses of neural cell types. J Vis Exp. (94), (2014).
- Hutton, S. R., Pevny, L. H. SOX2 expression levels distinguish between neural progenitor populations of the developing dorsal telencephalon. Dev Biol. 352 (1), 40-47 (2011).
- Azari, H., Sharififar, S., Rahman, M., Ansari, S., Reynolds, B. A. Establishing embryonic mouse neural stem cell culture using the neurosphere assay. J Vis Exp. (47), (2011).
- Sauer, F. Mitosis in the neural tube. The Journal of Comparative Neurology. 62 (2), 377-405 (1935).
- Sessa, A., Mao, C. A., Hadjantonakis, A. K., Klein, W. H., Broccoli, V. Tbr2 directs conversion of radial glia into basal precursors and guides neuronal amplification by indirect neurogenesis in the developing neocortex. Neuron. 60 (1), 56-69 (2008).
- Pilz, G. A., et al. Amplification of progenitors in the mammalian telencephalon includes a new radial glial cell type. Nat Commun. 4, 2125 (2013).
- Fantin, A., et al. Tissue macrophages act as cellular chaperones for vascular anastomosis downstream of VEGF-mediated endothelial tip cell induction. Blood. 116 (5), 829-840 (2010).
- Kayam, G., et al. A novel role for Pax6 in the segmental organization of the hindbrain. Development. 140 (10), 2190-2202 (2013).
- Tata, M., et al. Regulation of embryonic neurogenesis by germinal zone vasculature. Proc Natl Acad Sci U S A. 113 (47), 13414-13419 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon