Method Article
Perlina che carica proteine e acidi nucleici in cellule umane aderenti
In questo articolo
Riepilogo
Il carico di perle introduce proteine, plasmidi e particelle nelle cellule di mammifero aderenti. Questa tecnica di caricamento cellulare è economica, rapida e non influisce sostanzialmente sulla salute delle cellule. È più adatto per l'imaging di cellule vive.
Abstract
Molti esperimenti di imaging di cellule vive utilizzano particelle esogene (ad esempio, peptidi, anticorpi, perle) per etichettare o funzionare all'interno delle cellule. Tuttavia, introdurre proteine in una cellula attraverso la sua membrana è difficile. La selezione limitata dei metodi attuali lotta con bassa efficienza, richiede attrezzature costose e tecnicamente impegnative o funzioni all'interno di parametri ristretti. Qui, descriviamo una tecnica relativamente semplice ed economica per caricare DNA, RNA e proteine in cellule umane vive. Il carico di perle induce una temporanea interruzione meccanica della membrana cellulare, consentendo alle macromolecole di entrare nelle cellule di mammifero vive aderenti. A meno di 0,01 USD per esperimento, il caricamento delle perle è il metodo di caricamento delle celle meno costoso disponibile. Inoltre, il caricamento delle perline non stressa sostanzialmente le cellule o influisce sulla loro vitalità o proliferazione. Questo manoscritto descrive le fasi della procedura di caricamento delle pere, gli adattamenti, le variazioni e le limitazioni tecniche. Questa metodologia è particolarmente adatta per l'imaging di cellule vive, ma fornisce una soluzione pratica per altre applicazioni che richiedono l'introduzione di proteine, perle, RNA o plasmidi in cellule di mammifero viventi e aderenti.
Introduzione
Il caricamento di macromolecole in cellule di mammifero richiede una metodologia che consenta loro di attraversare la membrana plasmatica della cellula1. Diversi metodi possono introdurre plasmidi nelle cellule di mammifero attraverso la trasfezione, tra cui la trasfezione liposomiale2 e la trasfezione dietilamminoetil-destrano3. Tuttavia, i metodi per caricare proteine o particelle impermeabili alla membrana nelle cellule sono più limitati.
Diverse tecniche hanno aggirato questo difficile ostacolo utilizzando varie strategie. In primo luogo, la microiniezione fornisce particelle attraverso una micropipetta in cellule vive al microscopio4. Mentre probabilmente il metodo più controllato e meno invasivo, questa tecnica è relativamente a basso rendimento perché le celle devono essere caricate una per una. Inoltre, la microiniezione richiede attrezzature specializzate ed è tecnicamente impegnativa.
In secondo luogo, l'elettroporazione è un modo per iniettare elettro-proteine nelle cellule tramite la rottura della membrana indotta dalla tensione5,6,7. Tuttavia, questo metodo richiede ancora una volta attrezzature specializzate e costose e lo shock può causare stress e mortalità cellulare. Inoltre, le celle devono essere tripsinizzate prima dell'elettroporazione e successivamente riplazionate, limitando il lasso di tempo in cui le celle possono essere studiate dopo l'elettroporazione.
In terzo luogo, le membrane cellulari possono essere modificate chimicamente per una permeabilizzazione temporanea e reversibile8,9. Il carico di streptolisina-O inserisce un'endotossina nelle membrane cellulari, che forma pori temporanei, consentendo alle particelle esogene impermeabili alla membrana, comprese le proteine e i plasmidi del DNA, di entrare nelle cellule10. Dopo un recupero di 2 ore, circa la metà delle cellule ripara questi pori e ferma le particelle internalizzanti dalla soluzione. Tuttavia, questa tecnica richiede un lungo tempo di recupero ed è incompatibile con i tipi di cellule che non possono tollerare le endotossine.
Quarto, la distruzione meccanica carica le particelle nelle cellule attraverso la perturbazione fisica della membrana cellulare11. Questo può essere fatto in diversi modi, tra cui graffiare, raschiare e rotolare perle in cima alle celle12,13. Già nel 1987, le pere sono state utilizzate per caricare meccanicamente le proteine nelle cellule14. Più recentemente, la tecnica di caricamento delle perle è stata ottimizzata e adattata oltre le proteine per includere il carico di plasmidi e RNA, come descritto qui.
Il caricamento di perle è un metodo facile, economico e veloce per caricare proteine e plasmidi in cellule umane aderenti. Le perle di vetro vengono brevemente arrotolate in cima alle cellule, interrompendo temporaneamente la loro membrana cellulare. Ciò consente alle particelle in soluzione di entrare. Poiché il caricamento delle perle ha una bassa efficienza, è più adatto per esperimenti di microscopia a singola molecola o singola cellula. Il carico di perle può introdurre un'ampia varietà di proteine, tra cui anticorpi frammentati (Fab),15,16 proteine purificate come scFvs,17 intracorpi,18,19o proteine del rivestimento di mRNA, ad esempio, ms2 coat protein (MCP)20,21. I vettori di espressione plasmidicata possono anche essere aggiunti alla soluzione proteica e caricati simultaneamente22,23,24,25.
Oltre alle proteine e ai plasmidi, molecole grandi come 250 nm di polistirene sono state introdotte nelle cellule tramite il caricamento di perline (comunicazione personale). Il caricamento di pere è incredibilmente economico, costa meno di 0,01 USD per esperimento in materiali e non richiede attrezzature aggiuntive costose. Il costo è ulteriormente ridotto riducendo al minimo la quantità di sonde utilizzate per esperimento perché vengono caricate solo le celle nel microwell centrale di 14 mm di diametro di una camera di imaging. Va notato che l'area di carico limitata significa che il carico di pere non è l'ideale per il caricamento di celle alla rinfusa.
Questo manoscritto presenta il processo di caricamento delle perle, incluso come costruire l'apparato di carico delle perle ed eseguire un esperimento. Mostra che proteine, RNA e DNA possono essere caricati in vari tipi di cellule e che due diverse proteine caricate simultaneamente hanno concentrazioni cellulari altamente correlate e varianza relativamente bassa. Sono inoltre discusse le variazioni nel protocollo in base al tipo di cellula e al carico di proteine, plasmidi o RNA. Sebbene si pensi che le perle forino e interrompano la membrana cellulare, se eseguite in modo appropriato, il processo di caricamento delle perle rimuove solo un piccolo numero di cellule dal fondo della camera di imaging. Dopo un breve periodo di recupero, le cellule continuano a crescere e dividersi. Questa metodologia è ideale per esperimenti di microscopia a cellule vive, tra cui il monitoraggio di proteine e RNA a singola molecola, il rilevamento di modificazioni post-traduzionali, l'osservazione di meccanismi cellulari dinamici o il monitoraggio della localizzazione subcellulare15,16,22,26,27.
Protocollo
1. Pulire, sterilizzare e asciugare le perle di vetro per evitare l'aggregazione e garantire che si diffondano uniformemente in cima alle cellule.
- Sterilizzare circa 5 ml di perle di vetro in idrossido di sodio (NaOH). Misurare le perle in un tubo conico da 50 ml. Aggiungere 25 mL di 2 M NaOH e mescolare delicatamente usando uno shaker o un rotatore per 2 ore.
- Decantare il NaOH, conservando il maggior numero possibile di pere. Se le perle sono in sospensione, ruotare brevemente il tubo di perle in una centrifuga (1 minuto a ~ 1000 × g,temperatura ambiente).
- Lavare accuratamente le perline con acqua di coltura cellulare fino a quando il pH non è neutro (utilizzare una striscia reattiva di pH sull'eluente per confermare un pH neutro). Decantare l'acqua di lavaggio ogni volta, come prima.
- Lavare accuratamente le perle con etanolo al 100% 2-3x. Decantare l'etanolo ogni volta, come prima.
- Asciugare le pere. Cospargere le perle per formare uno strato sottile all'interno di un contenitore sterile (come una capsula di Petri di 10 cm). Lasciando il contenitore aperto, lasciare asciugare all'aria le perle in un armadietto di biosicurezza durante la notte. Assicurarsi che le perle siano completamente asciutte picchiettando o scuotendo delicatamente il contenitore e controllando che le perle abbiano una consistenza sabbiosa senza aggregarsi o sfaldarsi.
- Sterilizzare uv le perle secche per 15 minuti.
2. Assemblare l'apparato caricatore di perle.
- Fissare una patch di rete (polipropilene o materiale equivalente, aperture da 105 μm per consentire il passaggio delle perline) per coprire l'intera apertura della camera di tenuta delle perline con nastro adesivo o bloccando la rete tra le estremità maschio e femmina di una camera di imaging riutilizzabile in metallo (Figura 1A).
- Sterilizzare UV l'apparecchio per 15 min. Aggiungere le perle all'apparecchio e sigillarlo saldamente con pellicola cerosa.
NOTA: È essenziale che le pere siano completamente pulite e asciutte in questa fase. Dovrebbero essere sciolti e sembrare sabbiosi senza ciuffi. Se non appaiono così, rilavare e asciugare completamente le pere. - Conservare l'apparecchio in un contenitore asciutto sigillato essiccato con gel di silice o altro mezzo essiccante. Se le perle diventano umide, il che sarà evidente dall'aggregazione delle perle, asciugare e sterilizzare accuratamente il caricatore di perle e sostituirlo con perle fresche.
NOTA: Tutte queste precauzioni impediranno a muffe o batteri di crescere su o intorno alle perle all'interno del caricatore di perle. L'apparato caricatore di perle può essere realizzato in diversi modi. Vedi i dettagli nella discussione.
3. Preparare camere con fondo di vetro di cellule aderenti.
- Semina cellule di mammifero aderenti su una camera di vetro di 35 mm. Assicurarsi che le celle siano confluenti per circa l'80% al momento del caricamento del cordone. (Vedere la Tabella 1 per ulteriori informazioni sui vari tipi di cellule e le note sull'efficacia del caricamento delle perle in diversi tipi di cellule.)
NOTA: le cellule possono essere seminate solo nel microwell al centro della camera per conservare quante cellule vengono utilizzate. - Incubare le cellule in condizioni normali fino a quando non sono completamente aderenti al vetro.
NOTA: è essenziale che la densità cellulare sia sufficientemente elevata e che le cellule siano saldamente aderenti al vetro. Se questi requisiti non sono soddisfatti, le cellule probabilmente si staccano durante il caricamento delle perle. La linea temporale tra la semina cellulare e il caricamento delle pere può essere allungata per garantire la corretta adesione e confluenza cellulare.
4. Celle di carico delle peri
NOTA: Se necessario, lavare brevemente le cellule con soluzione salina tamponata con fosfato (PBS) e quindi aggiungere 2 ml del mezzo ottimale. Incubare per almeno 30 min.
- Fare una soluzione di 3-8 μL contenente i plasmidi, le proteine e/o le particelle desiderate. Utilizzare ~ 1 μg (0,1-1 pmol) di ciascun tipo di plasmide e ~ 0,5 μg (0,01 nmol) di proteine, a seconda delle esigenze sperimentali. Utilizzare un tubo a bassa ritenzione per le proteine in modo che non vengano lasciate sulle pareti del tubo. Portare la soluzione fino ad un minimo di 3 μL con PBS e regolare il volume della soluzione per rivestire l'intera area delle celle da caricare (ad esempio, il microwell della camera, Figura 1B).
- Mescolare accuratamente la soluzione pipettando su e giù e/o facendo scorrere il tubo. Ruotare brevemente la soluzione fino al fondo del tubo in un microfugio da tavolo.
- Trasferire la soluzione di carico del perlina e la camera delle cellule in una cappa di coltura tissutale. Eseguire i passaggi rimanenti nel cappuccio di coltura del tessuto utilizzando la tecnica sterile.
- Rimuovere il mezzo dalle cellule e conservarlo temporaneamente in un tubo sterile. Aspirare delicatamente tutto il mezzo da intorno ai bordi della camera e inclinare la camera con un angolo di circa 45 ° e rimuovere la goccia rimanente di materiale nel microwell centrale. Durante la rimozione del mezzo, assicurarsi di evitare che la punta della pipetta tocchi il vetro, il che potrebbe causare desquamazione e perdita delle cellule. Passare rapidamente al passaggio successivo in modo che le cellule non siano asciutte a lungo.
- Pipettare delicatamente la soluzione di carico del perlo sul microwell di vetro al centro della camera. Opzionale: Incubare con un leggero dondolo per ~ 30 s senza lasciare asciugare completamente la camera.
- Disperdere delicatamente un monostrato di perle di vetro sulla parte superiore delle celle, preferibilmente utilizzando un apparecchio di carico di perle (Figura 1A). Assicurarsi che le perle coprano completamente le celle nel microwell del fondo di vetro.
- Pizzicando la camera con due dita, toccarla contro la superficie del cappuccio sollevandola ~ 2 pollici e portandola verso il basso saldamente. Usa una forza approssimativamente equivalente a far cadere il piatto da quell'altezza. Ripeti per un totale di ~ 10 tocchi.
NOTA: Assicurarsi che i rubinetti non sbuccino sostanzialmente le cellule. Il tocco può essere ottimizzato per il tipo di cella. Se le celle si caricano male, toccare più forte; tuttavia, se molte cellule si staccano, toccare più leggermente. - Aggiungere delicatamente il mezzo nella camera pipettando lentamente sul lato di plastica della camera. Cerca di aspirare eventuali perli galleggianti senza disturbare le cellule. Aggiungere altri supporti preriscaldati in questo passaggio se ne è stato rimosso troppo. Incubare le cellule per 0,5-2 ore nell'incubatrice.
- Sterilizzare UV il caricatore di perle per 15 minuti prima di riportarlo allo stoccaggio in condizioni di essiccazione.
- Aggiungere colorante (ad esempio, DAPI o haloTag ligando la macchia, se richiesto dall'esperimento) alle cellule secondo il protocollo raccomandato dal produttore.
- Lavare le celle 3 volte con il mezzo prima dell'imaging per rimuovere le perle e i componenti di carico in eccesso in soluzione. Evitare il pipettaggio direttamente sulle cellule per evitare che si stacchino.
5. Imaging delle celle caricate con perle
- Immagina le cellule immediatamente o quando richiesto dall'esperimento. Utilizzare un microscopio in grado di catturare la fluorescenza (laser o sorgente luminosa monocromatica). Assicurarsi che le lunghezze d'onda di eccitazione siano appropriate per il fluoroforo o il colorante scelto (ad esempio, luce di lunghezza d'onda di 488 nm per la proteina di fluorescenza verde (GFP)).
NOTA: le proteine caricate con perline possono essere riprese una volta che le cellule si sono riprese (non appena 30 minuti dopo il caricamento per le linee cellulari qui descritte). L'espressione plasmidica richiede ≥2 h a seconda degli elementi vettoriali di espressione(Figura 1Ce ulteriori spiegazioni nella discussione). L'imaging di celle caricate con perle può essere eseguito su qualsiasi microscopio dotato delle sorgenti fluorescenti appropriate associate alle sonde caricate, una telecamera in grado di catturare immagini di fluorescenza, come un dispositivo ad accoppiamento di carica moltiplicatore di elettroni (EMCCD) o una telecamera scientifica complementare a semiconduttore di ossido di metallo (sCMOS) e un incubatore per controllare la temperatura, l'umidità e l'anidride carbonica. Per una guida alla microscopia a fluorescenza, fare riferimento a 27.
Risultati
L'applicazione più comune del carico di pere è quella di introdurre uno o più tipi di proteine nelle cellule umane aderenti. Per illustrare questo, le cellule sono state caricate con una soluzione di una proteina Fab coniugata Cy3 e Alexa488. Sebbene non tutte le cellule del microwell fossero caricate con pere, le cellule che venivano caricate avevano quasi sempre proteine etichettate come Cy3 e Alexa488 insieme (Figura 2A). Secondo una stima precedente, quando 0,5 microgrammi di Fab diluiti in 4 microlitri vengono caricati con perle29, come nella Figura 2A, ogni cella viene caricata con circa 106 molecole Fab.
Anche il DNA plasmidico che codifica per GFP (1 μg di DNA plasmidico, 1,8 μL di una soluzione da 557 ng/μL) e 0,5 μg di Fab con etichetta Cy3 è stato introdotto nelle cellule tramite carico di perle e successivamente espresso e visualizzato (Figura 2B). La fluorescenza GFP ha indicato che il plasmide che codifica GFP non è stato solo caricato nelle cellule, ma anche espresso. Pertanto, nella stessa cellula, il caricamento del perla può introdurre una sonda proteica (ad esempio, Cy3-labeled Fab) e un plasmide reporter (ad esempio, GFP), come eseguito in questo laboratorio in precedenza22,23,24. Abbiamo determinato che il 40% delle cellule erano caricate con proteine Fab e il 21% delle cellule caricate con perle esprimeva il plasmide co-caricato, come mostrato nei campi di vista rappresentativi nella Figura 2B. Tipicamente, ogni camera è caricata con 1-2 μg di plasmide, approssimativamente la stessa quantità della lipofezione.
Le cellule caricate con perle esprimono livelli molto diversi di plasmidi (Figura 2C,D). Per misurare specificamente questo, abbiamo utilizzato il test Fisher Ratio per confrontare le distribuzioni dei dati di intensità delle proteine e dei plasmidi. I risultati hanno mostrato che sebbene le proteine 1 e 2 avessero distribuzioni di intensità simili (p = ~1), ogni proteina aveva una distribuzione significativamente più piccola rispetto al plasmide (p = 3,2e-6 e 1,8e-5). Sebbene ciò possa essere dovuto alla variabilità nel numero di plasmidi caricati per cellula, la maggiore fonte di variabilità può derivare dai molti passaggi richiesti per l'espressione del plasmide che probabilmente variano notevolmente tra le cellule, tra cui l'importazione nel nucleo cellulare, la trascrizione e la traduzione. Al contrario, i livelli di proteine caricate con perle avevano una leggera varianza cellula-cellula e i livelli di due proteine caricate simultaneamente erano altamente correlati tra loro (Figura 2D,E).
L'espressione del plasmide può essere osservata già a 2-4 ore dopo il caricamento del perle, ma può verificarsi in seguito a seconda di quando si ottiene l'espressione ottimale del plasmide. Si consiglia di eseguire un corso temporale per determinare la migliore finestra di espressione per uno specifico plasmide che copre 2-24 ore dopo il caricamento del perlo. Questo può essere fatto in una camera con imaging a lungo termine o caricando perle e scaglionando più camere. Le cellule caricate con pere rimangono aderenti e sono abbastanza sane da crescere e dividersi. Le cellule U2OS umane caricate con pere sono state riprese direttamente prima, subito dopo e 24 ore dopo il caricamento del perno. Il corretto caricamento delle perle non ha avuto quasi alcun effetto evidente sul numero di cellule o sulla loro morfologia, come mostrato nella Figura 3A (a sinistra, al centro).
Al contrario, il carico di pere scadente con troppe pere e l'eccessiva forza di maschiatura sono rappresentati nella Figura 3B. Ciò ha causato molta perdita cellulare (grandi chiazze del vetro di copertura senza cellule e cellule staccate, fluttuanti, fuori fuoco), scarsa morfologia cellulare (cellule che appaiono arrotondate e scarsamente aderenti) e gruppi di perle che rimangono sul vetro di copertura dopo il caricamento delle perle. Sebbene si pensi che le cellule subiscano danni meccanici durante il caricamento delle perle, le cellule sono cresciute e proliferate nella camera caricata correttamente con perle, come evidenziato dall'aumento del numero di celle 24 ore dopo il caricamento delle perle (Figura 3A, a destra). L'effetto sulla vitalità cellulare può essere valutato attraverso una varietà di saggi, come un saggio di bromuro di bromuro di 3-(4,5-dimetiltiazol-2-il)-2,5-difeniltetrazolio (MTT), per confrontare le cellule caricate con perle e le cellule caricate in finta30. Inoltre, questo e il lavoro precedente mostrano che le celle caricate con perle subiscono la divisione cellulare (Figura 3C e Video supplementare 1), e la tempistica della mitosi non è influenzata dal carico di perle31, che serve come ulteriore prova per la salute cellulare sostenuta dopo il carico di perle.
Il caricamento di perline è una tecnica versatile, che ospita diverse linee cellulari aderenti e varie macromolecole. Qui, questa varietà è stata dimostrata caricando le linee cellulari RPE1 e HeLa con Fab (Figura 4A,B). La Tabella 1 fornisce ulteriori esempi di caricamento di perline in diverse linee cellulari, in questo laboratorio e oltre, e sottolinea alcune delle differenze sfumate tra i protocolli di caricamento delle perline di altri laboratori. Da notare, il diametro delle perle di vetro utilizzate per il carico varia notevolmente tra i laboratori, anche se il carico più efficiente è stato trovato per piccole perle di diametro 75 μm in diverse linee cellulari14. Inoltre, questo laboratorio ha iniziato a caricare anche l'RNA (dati non mostrati). La Figura 4C mostra una cellula U2OS rappresentativa caricata con un Cy5-RNA 9mer e Cy3-DNA 28mer insieme.
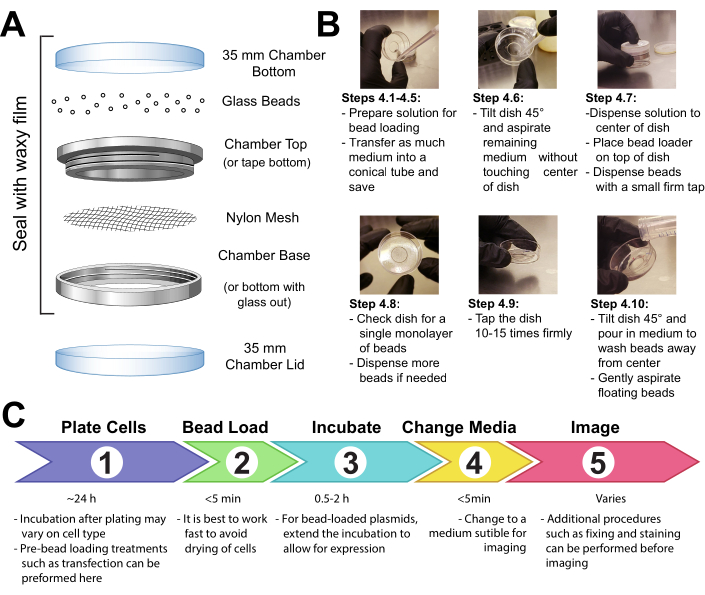
Figura 1: Apparato di caricamento delle perle, tecnica e sequenza temporale Fare clic qui per visualizzare una versione più grande di questa figura.
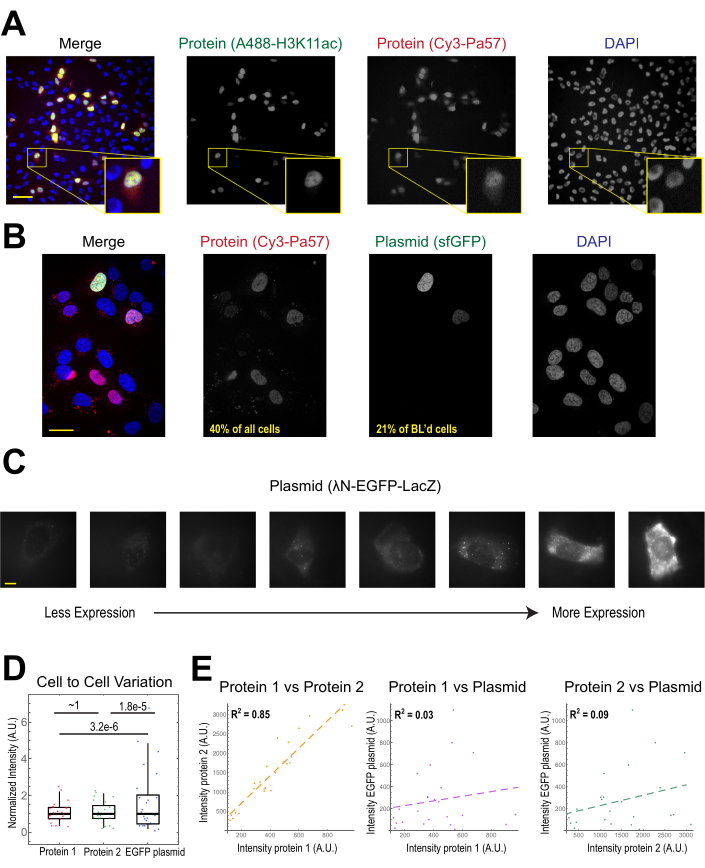
Figura 2. Il carico di perle introduce una bassa variabilità nella concentrazione proteica ma un'elevata variabilità nell'espressione del plasmide. (A) Le cellule sono state caricate con 0,5 μg di ciascuna delle Fab acetilmente coniugate con Alexa488 anti-H3K27 (verde) e Cy3-coniugato anti-RNAPII-Serina 5-fosforilato Fab (rosso) in 4 μL di soluzione di carico del perlato. Le cellule sono state colorate con DAPI (blu) e poi immediatamente con immagini dal vivo. Barre di scala = 20 μm. (B) Le cellule sono state caricate con 0,5 μg di proteina Fab (proteina cy3-coniugata anti-RNAPII-serina 5-fosforilata, rossa) e 1 μg di plasmide che codifica per la supercartella GFP-H2B (verde) in 4 μL di soluzione di carico di perle. Dopo 24 ore, le cellule sono state colorate con DAPI (blu) e sono state riprese dal vivo. Le barre di scala = 30 μm. (C-E) La proteina 1 (JF646-HaloLigand-labeled HaloTag-MCP), la proteina 2 (Cy3-coniugata anti-FLAG Fab) e un plasmide che codifica EGFP (λN-EGFP-LacZ) sono state caricate insieme nelle cellule. L'intensità totale in ciascun canale fluorescente è stata misurata in un cerotto di 1,3 x 1,3 μm nel citoplasma di ciascuna cellula. N = 25 celle. (C) Cellule rappresentative che esprimono il plasmide caricato con perle, λN-EGFP-LacZ. Le stesse condizioni e intensità di imaging sono state utilizzate per tutte le cellule. Le macchie sono aggregati della proteina espressa. Barre di scala = 10 μm. (D) Il grafico mostra l'intensità totale di ogni cellula della proteina 1, della proteina 2 o dell'EGFP espressa dal plasmide. Ogni canale è stato normalizzato alla mediana. I valori P corretti da Bonferroni sono stati calcolati dal test Fisher Ratio per determinare se la distribuzione dei dati di intensità proteica o plasmidica ha la stessa variabilità. Ogni punto rappresenta una cella. (E) Le intensità totali per entrambe le proteine, la proteina 1 e il plasmide, o la proteina 2 e il plasmide, sono tracciate l'una contro l'altra. Vengono visualizzati i valori R2 calcolati. Ogni punto rappresenta una cella. Abbreviazioni: DAPI = 4′,6-diamidino-2-fenilindolo; EGFP = proteina fluorescente verde potenziata; U.A. = unità arbitrarie; MCP = proteina del mantello MS2; RNAPII = RNA polimerasi II. Fare clic qui per visualizzare una versione più grande di questa figura.
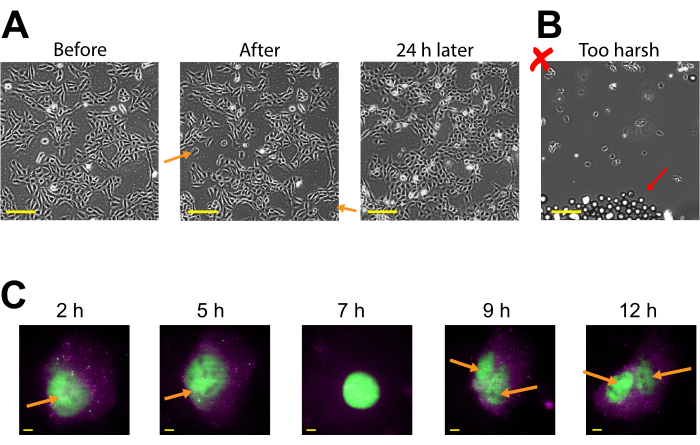
Figura 3: Le celle caricate conperle rimangono aderenti e sono abbastanza sane da crescere e dividersi. (A) Le cellule U2OS sono state caricate con 0,5 μg di Cy3-coniugato anti-FLAG Fab in 4 μL di soluzione di carico di perle. Le celle sono state riprese direttamente prima, direttamente dopo il caricamento delle pere e 24 ore dopo il caricamento delle pere. Le frecce arancioni identificano le aree in cui le cellule si sono staccate durante il caricamento delle perle. Barre di scala = 2 mm. (B) Immagine rappresentativa di celle U2OS caricate con componenti da (A) ma con picchiettature dure e troppe perle. La freccia rossa identifica le perle di vetro extra. Barra di scala = 2 mm. (C) Le celle U2OS sono state caricate con 1,5 μg del plasmide 14,4 kbp smFLAG-KDM5B-15xBoxB-24xMS2, 0,5 μg di Cy3-coniugato anti-FLAG Fab (verde), 130 ng di HaloTag-MCP (magenta) in 8 μL di soluzione di carico di perle. Subito prima dell'imaging, l'HaloTag è stato colorato con JF646-HaloLigand. Gli stem-loop MS2 dell'mRNA trascritto dal plasmide reporter sono etichettati da MCP (macchie magenta) e la proteina reporter tradotta con tag FLAG è etichettata da anti-FLAG Fab (colocalizzazione verde in mRNA). La proteina matura etichettata con Fab si localizza nel nucleo. Questa cella è stata considerata 4-15 ore dopo il caricamento delle pere. Le frecce gialle identificano il nucleo cellulare prima e i nuclei dopo la divisione cellulare. Barre di scala = 5 μm. Abbreviazione: MCP = MS2 coat protein. Fare clic qui per visualizzare una versione più grande di questa figura.
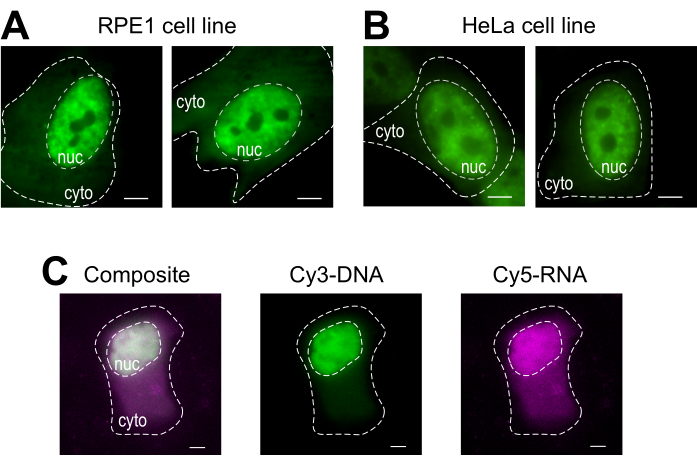
Figura 4: Variazioni nel materiale di carico del tipo di cella del protocollo di caricamento delle perle. (A-B) Le cellule RPE1 (A) e HeLa (B) sono state caricate con 1,5 μg di una proteina fab nucleare (anti-RNAPII-Serina 5-fosforilazione) in 4 μL di soluzione di carico. Il nucleo di ogni cellula (nuc) e il citoplasma (cito) sono marcati. Le cellule sono state riprese 6 ore dopo essere state caricate con pere. Barre di scala = 5 μm. (C) Le cellule U2OS umane sono state caricate con oligos cy5-RNA 9mer (magenta) e Cy3-DNA 28mer (verde), 10 picomoli di ciascuno, in 4 μL di soluzione di carico di perle. Le cellule sono state riprese 4 ore dopo essere state caricate con pere. Tutti i nuclei cellulari sono evidenziati da una linea tratteggiata. Barre di scala = 5 μm. Abbreviazioni: RNAPII = RNA polimerasi II. Fare clic qui per visualizzare una versione più grande di questa figura.
| Linea cellulare | Tipo di cella | Efficacia del caricamento del perno | Note/riferimenti |
| Cellule staminali (umane) | Cellule staminali embrionali | Difficile | * Molte cellule si staccano durante il caricamento delle perle se placcate su piastre rivestite di gelatina |
| HEK 293 · | Cellule renali embrionali umane | Difficile | * È necessario posare una matrice di gel sulla superficie della camera di imaging prima del caricamento del perle. Toccare delicatamente durante il caricamento del perno all'inizio. |
| Neuroni (ratto) | Neuroni embrionali primari (e-18), dissociati | Molto inefficiente | * Il carico efficiente di perle dei neuroni non è stato osservato utilizzando questo protocollo standard di caricamento delle perle. Ciò potrebbe essere dovuto alla natura non aderente dei neuroni o al conseguente danno ai processi neurali. |
| MDCK (cane) | Cellule renali canine di Madin-Darby | Vedi McNeil e Warder (1987)14 | * Caricamento di pere a bassa efficienza14 |
| U2OS (umano) | Osteosarcoma | Protocollo di caricamento delle perle standard | |
| HeLa (umano) | Cancro cervicale | Protocollo di caricamento delle perle standard | |
| RPE1 (umano) | Cellule epiteliali immortalate con hTERT | Protocollo di caricamento delle perle standard | |
| HFF (umano) | Fibroblasti primari del prepuzio | Vedi Besteiro et al. (2009)31 | * Protocollo modificato di inclinazione invece ditoccare 31 |
| BALB/c 3T3, NIH 3T3 e Swiss 3T3 (mouse) | Fibroblasti embrionali | Vedi Gilmore e Romer (1996)32, Emerson et al. (2014)33 e McNeil e Warder (1987)14 | *425–600 μm di perle di vetro riportate32 |
| *Perle di vetro usate da 200–300 μm33 | |||
| *Le perle di vetro da 75 μm hanno dato risultati migliori di 400 μm14 | |||
| DM (muntjac indiano) | Fibroblasti cutanei | Vedi Manders, Kimura e Cook (1999)34 | |
| CHO (criceto) | Cellule ovaie epiteliali | Vedi Memedula e Belmont (2003)35 | *Perle di vetro usate da 425-600 μm35 |
| BAE (bovino) | Cellule endoteliali aortiche bovine (BAEC-11) | Vedi McNeil e Warder (1987)14 | *Le perle di vetro da 75 μm hanno dato risultati migliori di 400 μm14 |
| PtK-2 (Potomus tridaclylis) | Cellule renali epiteliali | Vedi McNeil e Warder (1987)14 | *Le perle di vetro da 75 μm hanno dato risultati migliori di 400 μm14 |
| HUVEC (umano) | Cellule endoteliali della vena ombelicale | Vedi Gilmore e Romer (1996)32 | *Perle di vetro usate da 425-600 μm32 |
| J774 e J774.2 (mouse) | cellule dei macrofagi monociti | Vedi Becker et al. (2005)36 e McNeil e Warder (1987)14 | * Agitazione delicata (invece di picchiettare) e perle di vetro da 425-600μm 36 |
| MS-5 (mouse) | cellule stromali del midollo osseo | Vedi Molenaar et al. (2003)37 | |
| WPE1-NB11 (umano) | cellule epiteliali della prostata | Vedi Gilmore e Romer (1996)32 e | *Perle di vetro usate da 425-600 μm32 |
| Emerson et al. (2014)33 | *Perle di vetro usate da 200–300 μm33 |
Tabella 1: Caricamento di perline in diverse linee cellulari. Per le linee cellulari che non sono ancora state caricate in questo laboratorio, vengono forniti riferimenti e note sulle variazioni del protocollo.
Video supplementare 1: Esempio di una cella caricata con perle in fase di divisione cellulare. Le cellule U2OS sono state caricate con 1,5 μg del plasmide 14,4 kbp smFLAG-KDM5B-15xBoxB-24xMS2, 0,5 μg di Cy3-coniugato anti-FLAG Fab (verde), 130 ng di HaloTag-MCP (magenta) in 8 μL di soluzione di carico di perle. Subito prima dell'imaging, l'HaloTag è stato colorato con JF646-HaloLigand. Gli stem-loop MS2 dell'mRNA trascritto dal plasmide reporter sono etichettati da MCP (macchie magenta) e la proteina reporter tradotta con tag FLAG è etichettata tramite anti-FLAG Fab (colocalizzazione verde in mRNA). La proteina matura etichettata con Fab si localizza nel nucleo. Questa cella è stata considerata 4-15 ore dopo il caricamento delle pere. Barra della scala = 10 μm. Abbreviazione: MCP = MS2 coat protein. Clicca qui per scaricare questo video.
Discussione
La tecnica di caricamento delle perle qui descritta è un metodo economico ed efficiente in termini di tempo per l'introduzione di macromolecole e altre particelle nelle cellule aderenti. Questo processo versatile può caricare proteine (Figura 2A)15,16,26,27, una combinazione di proteine e plasmidi ( Figura2B,C)22,25, RNA ( Figura4C), 100 e 250 nm polistirene perline (corrispondenza personale), coloranti sintetici39 o punti quantici34,40 . Il carico di perle può avere la capacità di caricare anche altri tipi di particelle impermeabili alla membrana. La sua applicazione più frequentemente utilizzata è per caricare anticorpi o Fabs per colpire epitopi endogeni, come le modifiche post-traduzionali (PTM), in cellule vive. I bersagli, come i PTM, sono spesso difficili da etichettare nelle cellule vive senza sonde geneticamente codificate41,42,stabili specifiche per PTM. Al contrario, il caricamento di perle può introdurre più tipi di sonde, reporter o altri strumenti molecolari insieme nella stessa cella per il monitoraggio di più lettura contemporaneamente. Prevediamo che il caricamento delle perle sarà una tecnica utile per caricare una varietà di macromolecole o particelle.
Uno dei principali vantaggi del caricamento delle pere è il basso costo: ogni esperimento costa meno di 0,01 USD. Un apparecchio caricatore di perle può essere realizzato facilmente utilizzando materiali economici che costano in totale ~ $ 150, che è significativamente meno costoso di qualsiasi altro metodo di caricamento delle celle. Il costo di un apparecchio caricatore di perle può essere ulteriormente ridotto a meno di $ 10 sostituendo la camera metallica riutilizzabile con una di plastica. Per questo, praticare un foro in una camera da 35 mm o rimuovere il vetro da una camera con fondo di vetro da 35 mm, quindi fissare saldamente la rete in posizione con del nastro adesivo. Al posto di un apparecchio, il caricamento delle perle può anche essere eseguito utilizzando una punta di pipetta da 1000 μL a foro largo per raccogliere e cospargere le perle sulle celle, sebbene questa variazione renda difficile cospargere un monostrato di perle sulle celle (passaggio 4.6).
Un altro vantaggio del caricamento delle perline è che le cellule possono mantenere la normale morfologia generale, recuperare rapidamente e continuare a crescere e dividersi, almeno per le cellule U2OS, RPE1 e HeLa studiate qui e per le altre linee cellulari studiate altrove (Figura 3; Figura 4A,B; Video supplementare 1; e Tabella 1)31. Durante il carico di perline, le cellule subiscono stress fisico e talvolta si spostano e si staccano (~ 5% delle cellule si stacca in condizioni ottimali, ma una maggiore perdita cellulare può verificarsi se il carico di perline viene eseguito con troppa forza o troppe perle di vetro vengono caricate sopra le cellule, come illustrato nella Figura 3B). Tuttavia, le celle caricate con pere che rimangono attaccate al coperchio di solito appaiono sane e possono essere visualizzate non appena 30 minuti dopo il caricamento del perlo (Figura 3A). Generalmente concediamo alle cellule un periodo di recupero di 30 minuti, ma prevediamo che l'imaging prima dopo il caricamento delle perle sia fattibile.
Uno dei principali svantaggi di questa tecnica è che le cellule devono essere in grado di sopportare piccoli stress fisici durante il carico e rimanere saldamente aderenti al coverslip. Linee cellulari scarsamente / non aderenti o cellule cresciute su piastre rivestite (ad esempio, HEK e cellule staminali) spesso si staccano dopo una delicata picchiettatura durante il caricamento delle perline. Inoltre, l'esperienza ha dimostrato che i neuroni primari sono troppo sensibili per il caricamento delle pere.
Il caricamento del perle è più adatto per esperimenti a singola cellula o singola molecola. Nella nostra esperienza, il carico di perle ha un'efficienza di carico proteico di circa il 20-40% e ~ 20% delle celle caricate con perle ha anche espresso un plasmide co-caricato (Figura 2A, B). Pertanto, i plasmidi di carico di perle possono essere meno efficienti per l'espressione proteica rispetto alle proteine purificate che caricano perle perché i plasmidi non solo devono entrare nelle cellule ma anche essere espressi (il che comporta, tra le altre cose, l'importazione nucleare, la trascrizione e la traduzione, ognuna delle quali può ridurre l'efficienza di espressione). La bassa efficienza dell'espressione del plasmide caricato con perle può essere aggirata utilizzando protocolli di trasfezione alternativi, come la lipofezione, prima di caricare le proteine o le sonde16,27. Inoltre, l'incubazione delle cellule in mezzi ottimali per 30 minuti prima del caricamento delle perle può aiutare l'espressione del plasmide. A causa della bassa espressione del plasmide, il carico di perle non è stato spesso utilizzato come alternativa alla trasfezione basata sulla lipofezione in questo laboratorio. L'unica eccezione è quando una proteina purificata, come Fab, deve essere co-caricata, nel qual caso è abbastanza conveniente caricare la proteina e il plasmide allo stesso tempo. Inoltre, per le cellule che non rispondono o sono intolleranti alla lipofezione, il carico di perle può fornire un metodo alternativo, anche se a bassa efficienza, per l'espressione transitoria del plasmide.
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare.
Riconoscimenti
Siamo grati ai membri del laboratorio Stasevich per le innumerevoli conversazioni che hanno contribuito a migliorare e sviluppare questo protocollo. In particolare, la dott.ssa Linda Forero e il dott. Phil Fox per consigli su come caricare pere diversi tipi di cellule. Vorremmo ringraziare sinceramente la dott.0.00-Takanaka, la dott.0.0.00 e la dott..0.00. Siamo molto grati al Dr. Ashok Prasad e al Dr. Diego Krapf per aver generosamente condiviso i loro protocolli di caricamento delle peri per l'introduzione di particelle inorganiche nelle cellule. Siamo grati al Dr. Travis Sanders, Craig Marshall e al Dr. Thomas Santangelo per aver generosamente condiviso il loro reagente RNA etichettato. ALK, MNS, CAC, GG e TJS sono stati supportati dalla sovvenzione R35GM119728 del National Science Foundation (NSF) e dalla sovvenzione CAREER MCB-1845761 della National Science Foundation (NSF), entrambe a TJS. CAC è stato anche supportato dal premio NSF NRT DGE-1450032.
Materiali
| Name | Company | Catalog Number | Comments |
| 10 cm cell culture dishes | VWR | 82050-916 | Use to culture cells |
| 35 mm cell culture dishes | Falcon | 353001 | Use to construct bead loader |
| Attofluor Cell Chamber | Thermo Fisher Scientific | A7816 | Use to construct the custom bead loader |
| DMEM, high glucose, no glutamine | Thermo Fisher Scientific | 11960069 | Use in general cell culture |
| Drierite Indicating Absorbents | Thermo Fisher Scientific | 07-578-3B | Store the bead loader in a desiccator with these absorbent pellets |
| Fetal Bovine Serum | Atlas Biologics | F-0050-A | Use in general cell culture and as a supplement before bead loading |
| Glass beads, acid washed, ≤106 µm* | Millipore Sigma | G4649 | Sprinkle on cells to bead load plasmid DNA and proteins |
| Glass bottom dishes, 35 mm, #1.5, 14 mm glass | MatTek Corporation | P35G-1.5-14-C | Seed cells onto these chambers for imaging |
| L-Glutamine-200 mM | Thermo Fisher Scientific | 25030081 | Use to make DMEM + media |
| Opti-MEM, Reduced Serum Medium | Thermo Fisher Scientific | 31985070 | Optimal media for incubating cells before bead loading (optional step) |
| Parafilm | VWR | 52858-032 | waxy film used to construct bead loader |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140-122 | Use to make DMEM + media |
| Phenol-free DMEM | Thermo Fisher Scientific | 31053036 | Use on cells before imaging |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | AM9625 | Working stock of sterile 1X PBS |
| Sodium Hydroxide (NaOH) | Thermo Fisher Scientific | S318-500 | Use 2 M solution to wash glass beads |
| Spectra Mesh Woven Filters, Polypropylene, 105 µm opening | Spectrum Labs | 148496 | Use to construct bead loader |
| Trypsin | Thermo Fisher Scientific | 25300062 | Use in general cell culture |
Riferimenti
- Stewart, M. P., et al. In vitro and ex vivo strategies for intracellular delivery. Nature. 538 (7624), 183-192 (2016).
- Felgner, P. L., et al. Lipofection: a highly efficient, lipid-mediated DNA-transfection procedure. Proceedings of the National Academy of Sciences of the United States of America. 84 (21), 7413-7417 (1987).
- Schenborn, E. T., Goiffon, V. DEAE-dextran tansfection of mammalian cultured cells. Methods in Molecular Biology. 130, 147-153 (2000).
- Celis, J. E. Microinjection of somatic cells with micropipettes: comparison with other transfer techniques. Biochemical Journal. 223 (2), 281-291 (1984).
- Chakrabarti, R., Wylie, D. E., Schuster, S. M. Transfer of monoclonal antibodies into mammalian cells by electroporation. Journal of Biological Chemistry. 264 (26), 15494-15500 (1989).
- Wilson, A. K., Horwitz, J., De Lanerolle, P. Evaluation of the electroinjection method for introducing proteins into living cells. American Journal of Physiology. 260 (2), 355-363 (1991).
- Potter, H. Transfection by electroporation. Current Protocols in Molecular Biology. 62 (1), 1-6 (2003).
- Fawell, S., et al. Tat-mediated delivery of heterologous proteins into cells. Proceedings of the National Academy of Sciences of the United States of America. 91 (2), 664-668 (1994).
- Prior, T. I., FitzGerald, D. J., Pastan, I. Translocation mediated by domain II of Pseudomonas exotoxin A: transport of barnase into the cytosol. Biochemistry. 31 (14), 3555-3559 (1992).
- Walev, I., et al. Delivery of proteins into living cells by reversible membrane permeabilization with streptolysin-O. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3185-3190 (2001).
- Pitchiaya, S., Androsavich, J. R., Walter, N. G. Intracellular single molecule microscopy reveals two kinetically distinct pathways for microRNA assembly. EMBO Reports. 13 (8), 709-715 (2012).
- McNeil, P. L., Murphy, R. F., Lanni, F., Taylor, D. L. A method for incorporating macromolecules into adherent cells. The Journal of Cell Biology. 98 (4), 1556-1564 (1984).
- Ortiz, D., Baldwin, M. M., Lucas, J. J. Transient correction of genetic defects in cultured animal cells by introduction of functional proteins. Molecular and Cellular Biology. 7 (8), 3012-3017 (1987).
- McNeil, P. L., Warder, E. Glass beads load macromolecules into living cells. Journal of Cell Science. 88 (5), 669-678 (1987).
- Hayashi-Takanaka, Y., et al. Tracking epigenetic histone modifications in single cells using Fab-based live endogenous modification labeling. Nucleic Acids Research. 39 (15), 6475-6488 (2011).
- Morisaki, T., et al. Real-time quantification of single RNA translation dynamics in living cells. Science. 352 (6292), 1425-1429 (2016).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Zhao, N., et al. A genetically encoded probe for imaging nascent and mature HA-tagged proteins in vivo. Nature Communications. 10 (1), 2947(2019).
- Jedlitzke, B., Mootz, H. D. Photocaged nanobodies delivered into cells for light activation of biological processes. ChemPhotoChem. 5 (1), 22-25 (2021).
- Coulon, A., et al. Kinetic competition during the transcription cycle results in stochastic RNA processing. eLife. 3, 03939(2014).
- Pichon, X., Robert, M. -C., Bertrand, E., Singer, R. H., Tutucci, E. New generations of MS2 variants and MCP fusions to detect single mRNAs in living eukaryotic cells. Methods in Molecular Biology. 2166, 121-144 (2020).
- Koch, A., et al. Quantifying the dynamics of IRES and cap translation with single-molecule resolution in live cells. Nature Structural & Molecular Biology. 27, 1095-1104 (2020).
- Moon, S. L., et al. Multicolor single-molecule tracking of mRNA interactions with RNP granules. Nature cell biology. 21 (2), 162-168 (2019).
- Moon, S. L. Coupling of translation quality control and mRNA targeting to stress granules. Journal of Cell Biology. 219 (8), 202004120(2020).
- Cialek, C. A., et al. Imaging translational control by Argonaute with single-molecule resolution in live cells. bioRxiv. , (2021).
- Forero-Quintero, L. S., et al. Live-cell imaging reveals the spatiotemporal organization of endogenous RNA polymerase II phosphorylation at a single gene. bioRxiv. , (2020).
- Lyon, K., Aguilera, L. U., Morisaki, T., Munsky, B., Stasevich, T. J. Live-cell single RNA imaging reveals bursts of translational frameshifting. Molecular Cell. 75 (1), 172-183 (2019).
- JoVE. Introduction to Fluorescence Microscopy. General Laboratory Techniques. JoVE Science Education Database. , JoVE. Cambridge, MA. (2021).
- Hayashi-Takanaka, Y., Yamagata, K., Nozaki, N., Kimura, H. Visualizing histone modifications in living cells: spatiotemporal dynamics of H3 phosphorylation during interphase. Journal of Cell Biology. 187 (6), 781-790 (2009).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the MTT assay. Cold Spring Harbor Protocols. 2018 (6), (2018).
- Stasevich, T. J., et al. Regulation of RNA polymerase II activation by histone acetylation in single living cells. Nature. 516 (7530), 272-275 (2014).
- Besteiro, S., Michelin, A., Poncet, J., Dubremetz, J. -F., Lebrun, M. Export of a Toxoplasma gondii rhoptry neck protein complex at the host cell membrane to form the moving junction during invasion. PLOS Pathogens. 5 (2), 1000309(2009).
- Gilmore, A. P., Romer, L. H. Inhibition of focal adhesion kinase (FAK) signaling in focal adhesions decreases cell motility and proliferation. Molecular Biology of the Cell. 7 (8), 1209-1224 (1996).
- Emerson, N. T., Hsia, C. -H., Rafalska-Metcalf, I. U., Yang, H. Mechanodelivery of nanoparticles to the cytoplasm of living cells. Nanoscale. 6 (9), 4538-4543 (2014).
- Manders, E. M. M., Kimura, H., Cook, P. R. Direct imaging of DNA in living cells reveals the dynamics of chromosome formation. Journal of Cell Biology. 144 (5), 813-822 (1999).
- Memedula, S., Belmont, A. S. Sequential recruitment of HAT and SWI/SNF components to condensed chromatin by VP16. Current Biology. 13 (3), 241-246 (2003).
- Becker, T., Volchuk, A., Rothman, J. E. Differential use of endoplasmic reticulum membrane for phagocytosis in J774 macrophages. Proceedings of the National Academy of Sciences of the United States of America. 102 (11), 4022-4026 (2005).
- Molenaar, C., et al. Visualizing telomere dynamics in living mammalian cells using PNA probes. The EMBO Journal. 22 (24), 6631-6641 (2003).
- Jones, S. A., Shim, S. -H., He, J., Zhuang, X. Fast, three-dimensional super-resolution imaging of live cells. Nature Methods. 8 (6), 499-505 (2011).
- Sabri, A., Xu, X., Krapf, W. M. Elucidating the origin of heterogeneous anomalous diffusion in the cytoplasm of mammalian cells. Physical Review Letters. 125 (5), 053901(2020).
- Sato, Y., et al. Genetically encoded system to track histone modification in vivo. Scientific Reports. 3, 2436(2013).
- Sato, Y., Stasevich, T. J., Kimura, H. Visualizing the dynamics of inactive X chromosomes in living cells using antibody-based fluorescent probes. X-Chromosome Inactivation. Methods in Molecular Biology. 1861, Humana. New York, NY, USA. 91-102 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon