Method Article
Imaging delle spine dendritiche in Caenorhabditis elegans
In questo articolo
Riepilogo
Le spine dendritiche sono importanti caratteristiche cellulari del sistema nervoso. Qui vengono descritti metodi di imaging dal vivo per valutare la struttura e la funzione delle spine dendritiche in C. elegans. Questi approcci supportano lo sviluppo di schermi mutanti per i geni che definiscono la forma o la funzione della colonna vertebrale dendritica.
Abstract
Le spine dendritiche sono siti specializzati di innervazione sinaptica modulata dall'attività e servono come substrati per l'apprendimento e la memoria. Recentemente, le spine dendritiche sono state descritte per i neuroni DD GABAergici come siti di input dai neuroni colinergici presinaptici nel circuito motorio di Caenorhabditis elegans. Questo circuito sinaptico può ora servire come un nuovo potente modello in vivo della morfogenesi e della funzione della colonna vertebrale che sfrutta la facile genetica e la pronta accessibilità di C. elegans all'imaging di cellule vive.
Questo protocollo descrive le strategie sperimentali per valutare la struttura e la funzione della colonna vertebrale DD. In questo approccio, viene utilizzata una strategia di imaging a super-risoluzione per visualizzare le forme intricate delle spine dendritiche ricche di actina. Per valutare la funzione della colonna vertebrale DD, l'opsina attivata dalla luce, Chrimson, stimola i neuroni colinergici presinaptici e l'indicatore di calcio, GCaMP, riporta i transitori di calcio evocati nelle spine DD postsinaptiche. Insieme, questi metodi comprendono potenti approcci per identificare i determinanti genetici delle spine dendritiche in C. elegans che potrebbero anche dirigere la morfogenesi e la funzione della colonna vertebrale nel cervello.
Introduzione
Le spine dendritiche sono strutture cellulari specializzate che ricevono input dai neuroni vicini per la trasmissione sinaptica. L'attivazione dei recettori dei neurotrasmettitori eleva il calcio intracellulare e le vie di segnalazione a valle in queste caratteristiche protrusioni neuronali1. A causa dell'importanza fondamentale delle spine dendritiche per la neurotrasmissione e la loro errata regolazione nelle malattie dello sviluppo neurologico1, la scoperta di fattori che modulano la morfogenesi e la funzione della colonna vertebrale dendritica è di grande interesse per il campo delle neuroscienze.
Recentemente, sono state identificate spine dendritiche nel sistema nervoso di C. elegans sulla base di caratteristiche chiave condivise con le spine dei mammiferi2. Questa determinazione è cruciale perché apre la possibilità di sfruttare i vantaggi di C. elegans per studiare la biologia della colonna vertebrale. Le spine dendritiche sui motoneuroni dorsali D (DD) ricevono input dai neuroni colinergici (VA e VB) nel cordone nervoso ventrale (Figura 1A)2,3,4. Qui, vengono presentati metodi di imaging per esplorare la struttura delle spine dendritiche DD e la loro funzione in vivo in un sistema nervoso intatto che è facilmente accessibile all'imaging dal vivo e all'analisi genetica. Per il monitoraggio della forma delle spine dendritiche, (1) proteine fluorescenti citosoliche, che riempiono il processo dendritico e le spine; (2) proteine fluorescenti legate alla membrana, che decorano il bordo delle spine dendritiche e dei dendriti; o (3) vengono utilizzati i marcatori di actina, LifeAct5 o Utrophin6, che sono arricchiti in spine dendritiche, rivelando così la loro forma. Per monitorare la funzionalità delle spine DD, la fluorescenza GCaMP viene utilizzata per rilevare i transitori Ca++ evocati dall'attivazione dell'opsina spostata verso il rosso, Chrimson, nei neuroni colinergici presinaptici7. Entrambe le strategie dovrebbero facilitare lo studio delle spine dendritiche DD in animali selvatici e mutanti.
Protocollo
1. Determinazione della struttura delle spine dendritiche DD
- Creare vermi transgenici per etichettare le spine DD
- Utilizzare il promotore flp-13 per costruire un vettore di espressione per l'etichetta di interesse (ad esempio, mCherry citoplasmatico, MYR::mRuby, LifeAct::GFP, GFP::utrophin) (Figura 1). Vedere l'elenco completo dei plasmidi nel file supplementare 1.
- Utilizzare metodi consolidati per generare una linea transgenica che etichetta le spine DD 8,9.
- Preparare il sigillante
- Realizzare una miscela 1:1 di mezzo di incorporamento a base di paraffina10 (vedere Tabella dei materiali).
- Riscaldare il mezzo a 60 °C fino alla fusione, quindi aliquote in provette da microcentrifuga con tappo da 1,5 mL e mantenere un blocco riscaldante a 60-70 °C.
NOTA: Il sigillante può durare per 4 settimane nel blocco di riscaldamento.
- Preparare un anestetico.
- Preparare soluzioni madre in H2O distillato di tricaina all'1% e levamisolo 1 M (vedi tabella dei materiali). Conservare a -20 °C.
- Preparare una soluzione di lavoro allo 0,05% di tricaina e 15 mM di anestetico levamisolo, come descritto nei punti 1.3.3-1.3.511.
- Mescolare 75 μL di 1% di tricaina e 22,5 μL di 1 M levamisolo.
- Aggiungere il buffer M9 a un volume finale di 1,5 ml.
- Aliquote 10 μL di tricaina allo 0,05%, 15 mM di levamisolo in provette da microcentrifuga da 0,5 mL e conservare a -20 °C.
NOTA: La miscela anestetica è sensibile alla temperatura e le singole aliquote della soluzione di lavoro non devono essere ricongelate dopo lo scongelamento per ogni esperimento. Vedere il file supplementare 2 per una ricetta per il buffer M9.
- Acquisizione di immagini ad alta risoluzione
- Preparare il 10% di agarosio e mantenerlo a bagnomaria a 60 °C.
NOTA: vedere il rapporto di Monica Driscoll in WormBook12. - Montare 15-20 giovani adulti su tamponi di agarosio al 10% e aggiungere 3 μL di anestetico (vedi fase 3).
- Applicare il coprislip (i vermi vengono immobilizzati entro 5 minuti).
- Sigillare i bordi del vetrino con una miscela sigillante adesiva fusa (vedi Tabella dei materiali).
- Acquisire immagini
- Acquisizione in super-risoluzione
- Utilizzare un microscopio confocale a scansione laser dotato di microscopia a super-risoluzione con un obiettivo a olio Plan-Apochromat 63x/1.40 per ottenere una dimensione di pixel ridotta (ad esempio, < 50 nm). Acquisire Z-stack utilizzando la dimensione del passo consigliata dal software del produttore (vedere Tabella dei materiali).
- Raccogliere una serie di sezioni ottiche che coprono il volume totale del processo ventrale DD (ad esempio, 15-20 fette a 0,19 μm di dimensione del passo o 2-3μm di spessore). Invia Z-stacks per l'elaborazione delle immagini utilizzando il software del produttore e analizza le immagini con un punteggio superiore a 7 (Figura 1B, Figura 2 e Figura 3).
- Acquisizione di Nyquist
- Utilizzare un microscopio confocale a scansione laser per selezionare la dimensione ottimale dei pixel per la lunghezza d'onda della luce e l'apertura numerica dell'obiettivo in uso (ad esempio, obiettivo 40x/1.4 Plan Fluor oil).
NOTA: La dimensione dei pixel più piccola rivelerà la struttura fine delle spine DD. - Inviare stack per la deconvoluzione 3D utilizzando l'algoritmo automatico (vedere la tabella dei materiali) (Figura 3).
- Utilizzare un microscopio confocale a scansione laser per selezionare la dimensione ottimale dei pixel per la lunghezza d'onda della luce e l'apertura numerica dell'obiettivo in uso (ad esempio, obiettivo 40x/1.4 Plan Fluor oil).
- Utilizzare il più piccolo Z-step possibile (ad esempio, determinato dallo stadio piezoelettrico) perché il sovracampionamento in Z può produrre immagini più nitide dopo la deconvoluzione 3D13.
- Acquisizione in super-risoluzione
- Preparare il 10% di agarosio e mantenerlo a bagnomaria a 60 °C.
- Analisi delle immagini
- Utilizzare un software di elaborazione delle immagini appropriato (vedere Tabella dei materiali) per creare proiezioni di massima intensità degli Z-stacks14.
- Contare manualmente le sporgenze sul dendrite DD.
NOTA: Le sporgenze sono estensioni perpendicolari dall'albero principale (Figura 1B, punte di freccia). - Determinare la lunghezza del dendrite DD segnato per calcolare la densità delle spine per 10 μm di dendrite DD (Figura 1C).
- Classificare le spine come sottili/funghi, filopodiali, tozze o ramificate (Figura 2A).
NOTA: Le spine sottili / fungo mostrano una base stretta (collo) e una punta più ampia (testa). Le spine filopodiali non mostrano una base ristretta (senza collo) ma hanno una larghezza costante. Le spine tozze hanno una base e una punta larghe. Le spine ramificate sono sporgenze con più di una punta.
2. Valutazione dell'attivazione delle spine dendritiche DD mediante segnalazione colinergica presinaptica
- Creare vermi transgenici usando tecniche convenzionali (ad esempio, microiniezione)8,9
- Utilizzare il promotore flp-13 per guidare l'espressione del sensore Ca++ , GCaMP6s, nei neuroni DD e il promotore unc-4 per guidare l'espressione di Chrimson, una channelrhodopsina spostata verso il rosso, nei neuroni VA presinaptici (Figura 4A). Vedere l'elenco dei plasmidi nel file supplementare 1.
- Preparare All-trans Retinal (ATR) e piastre di controllo.
NOTA: ATR è un cofattore necessario affinché Chrimson funzioni come canale ionico optogeneticamente attivato.- Preparare una soluzione madre ATR da 100 mM in etanolo (100%). Conservare a -20 °C in 1 mL di aliquote.
- Sotto una cappa a flusso laminare, aggiungere 300 μL di coltura batterica OP50 durante la notte e 0,25 μL di ATR a ciascuna piastra di agar nutriente NGM (Nematode Growth Medium) da 60 mm e distribuire con una bacchetta di vetro sterile.
- Per i controlli, aggiungere 300 μL di batteri OP50 e 0,25 μL di etanolo (100%) a un gruppo separato di piastre NGM.
- Lasciare riposare le piastre nel cappuccio a temperatura ambiente per 24 ore (al riparo dalla luce ambientale) per consentire la crescita batterica.
NOTA: Le piastre possono essere utilizzate dopo l'incubazione iniziale di 24 ore o mantenute a 4 °C per l'uso entro 5 giorni.
- Impostazione dell'esperimento
- Posizionare cinque larve dello stadio NC3569 L4 su ATR o piastre di controllo con semi OP50 che mancano di ATR e crescono nell'oscurità a 23 ° C.
- Tre giorni dopo, utilizzare un microscopio da dissezione stereoscopico per confermare lo sviluppo della vulva per selezionare la progenie dello stadio L4 da ATR e piastre di controllo per l'imaging come descritto nei passaggi 2.4.1-2.4.3.
- Su un vetrino da microscopio, posizionare 2 μL di poliperline da 0,05 μm (2,5% di solidi p/v) (vedi Tabella dei materiali).
- Utilizzare un filo di platino ("worm pick") per aggiungere un piccolo globulo di super colla alla soluzione e ruotare delicatamente per generare "fili" filamentosi di colla. Quindi aggiungere 3 μL di tampone M9 (Figura 4B).
- Posizionare circa dieci larve di L4 nella soluzione e applicare un vetrino.
NOTA: le fibre di colla contatteranno casualmente i vermi e li immobilizzeranno dopo l'applicazione del vetrino. I vermi che sono incorporati in grandi globuli di colla appaiono essiccati e non dovrebbero essere fotografati. - Sigillare i bordi del foglietto come indicato al punto 1.4.4.
- Registrazione di transitori Ca++ evocati in spine dendritiche.
- Utilizzare un microscopio confocale a disco rotante dotato di una telecamera CCD sensibile, un obiettivo a olio TIRF 100x e linee laser da 488 nm e 561 nm (vedi Tabella dei materiali).
- Regolare lo stadio del microscopio per posizionare le spine DD nel piano focale.
- Impostare l'acquisizione time-lapse per illuminare il campione con la linea laser a 488 nm ogni fotogramma (per rilevare la fluorescenza GCaMP6s) e la linea laser a 561 nm a intervalli periodici (per l'eccitazione Chrimson).
NOTA: ad esempio, utilizzare la luce a 488 nm per acquisire istantanee consecutive (200 ms) del segnale GCaMP6s accoppiato con un impulso di 200 ms di luce di 561 nm ogni 5° fotogramma (Figura 4C-E). Con questa configurazione, i livelli di GCaMP prima e dopo ogni impulso a 561 nm vengono rilevati a ~1 s di distanza (200 ms di laser a 488 nm per rilevare GCaMP prima dell'attivazione di VA, 200 ms di impulso a 561 nm per attivare VA e ~600 ms per passare tra linee laser e filtri di emissione. Con questa configurazione, i neuroni VA vengono attivati ogni 2,5 s.
- Analisi dell'imaging Ca++ in vivo
- Utilizzare la deconvoluzione 2D e l'allineamento dell'immagine per correggere deviazioni minori derivanti dal movimento della vite senza fine durante l'acquisizione (vedere Tabella dei materiali).
- Definire la colonna vertebrale dendritica DD come la regione di interesse (ROI nelle figure 4C-D).
- Duplicare il ROI e spostarlo in una regione vicina all'interno del worm per raccogliere il segnale di fondo (ad esempio, il rumore).
- Utilizzare il software appropriato (vedere Tabella dei materiali) per esportare le intensità GCaMP6s in Excel per ogni punto temporale. Sottrarre la fluorescenza di fondo dalla fluorescenza del ROI della colonna vertebrale.
- Determinare la variazione della fluorescenza sottraendo la fluorescenza GCaMP6s nel fotogramma immediatamente prima dell'eccitazione a 561 nm (F0) da ciascun punto temporale dopo l'eccitazione (ΔF), quindi dividendo per F0 per determinare ΔF/F0 (Figura 4E).
- Rappresentare graficamente le tracce normalizzate (vedere la tabella dei materiali).
- Eseguire un test statistico accoppiato per ogni misurazione della fluorescenza GCaMP6s prima e dopo ogni impulso di luce a 561 nm.
NOTA: Questo approccio esclude efficacemente fluttuazioni casuali nella fluorescenza di GCaMP6s che altrimenti riducono il potere statistico del confronto del segnale medio GCaMP6s da tutte le misurazioni prima e dopo l'eccitazione a 561 nm (Figura 4F). - Per le misurazioni che mostrano una distribuzione normale o gaussiana, utilizzare un test ANOVA parametrico accoppiato e correggere per confronti multipli per ciascuno dei due gruppi (ATR prima vs. dopo, nessun ATR prima vs. dopo). In alternativa, per i dati che non sono normalmente distribuiti, utilizzare un ANOVA non parametrico con correzione posthoc per test multipli.
NOTA: i worm cresciuti su piastre prive di ATR ("no ATR") sono controlli necessari e non dovrebbero mostrare transitori Ca++ attivati a 561 nm perché ATR è richiesto per la funzione Chrimson.
Risultati
Le misurazioni con tre marcatori indipendenti (citosolic mCherry, LifeAct::GFP, MYR::mRuby) hanno prodotto una densità media di 3,4 ± 1,03 spine dendritiche DD per 10 μm di dendrite DD in giovani adulti wild-type (Figura 1B,C). Per questa analisi, le misurazioni ottenute con il marcatore GFP::Utrophin che ha prodotto una densità della colonna vertebrale significativamente inferiore sono state escluse (2,4 ± 0,74, Figura 1) a causa delle interazioni di Utrophin con il citoscheletro di actina6 che potenzialmente guida la morfogenesi della colonna vertebrale15. Le misure della densità della colonna vertebrale al microscopio ottico sono paragonabili al valore di 4,2 spine/10 μm di dendrite ottenuto dalla ricostruzione di 12 spine da micrografie elettroniche del neurone DD12. L'approccio di imaging delle cellule vive ha confermato che la morfologia sottile/a forma di fungo delle spine DD predomina nelle forme della colonna vertebrale adulta rispetto a quelle alternative (ad esempio, filopodiali, tozze, ramificate) (Figura 2B), che è anche tipica per le spine nel sistema nervoso maturo dei mammiferi16.
Una strategia optogenetica è stata utilizzata per chiedere se le presunte spine dendritiche rilevate dalla microscopia ottica ad alta risoluzione (Figura 1 e Figura 2) rispondono al rilascio di neurotrasmettitori dai siti presinaptici, un segno distintivo caratteristico delle spine dendritiche nei neuroni dei mammiferi. La luce verde (561 nm) è stata utilizzata per attivare una variante channelrhodopsin, Chrimson, nei neuroni colinergici presinaptici e la luce blu (488 nm) per rilevare la fluorescenza Ca++-dipendente emessa da una sonda GCaMP citoplasmatica nelle spine dendritiche DD postsinaptiche. Questo esperimento ha rilevato esplosioni transitorie del segnale GCaMP nelle spine DD immediatamente dopo l'attivazione optogenetica di Chrimson nei neuroni VA presinaptici (Figura 3). Il successo di questo esperimento dipende dall'espressione affidabile di Chrimson in tutti i neuroni VA presinaptici. In questo caso, è stato utilizzato un integrante cromosomico17 del marcatore Punc-4::Chrimson per garantire un'espressione VA coerente. Questo esperimento potrebbe anche essere condotto con un array extracromosomico. L'espressione cremisi in uno specifico neurone VA può essere confermata indipendentemente, ad esempio, accoppiando il transgene Chrimson a una sequenza leader transplicata SL2 con una GFP localizzata nucleare a valle come marcatore di co-espressione2. È essenziale eseguire un esperimento di controllo in assenza di ATR per confermare che il segnale GCaMP misurato dipende dall'attivazione optogenetica di Chrimson, che è strettamente ATR-dipendente (Figura 4D). Infine, poiché i segnali Ca++ evocati sono transitori, è fondamentale adottare un protocollo di imaging che consenta la commutazione rapida (<1 s) tra l'eccitazione a 561 nm e l'acquisizione del segnale GCaMP con il laser a 488 nm (Figura 4).

Figura 1: Marcatura delle spine dendritiche DD . (A) (In alto) Sei neuroni dorsali D (DD1-DD6) nel cordone nervoso ventrale di C. elegans. (In basso) Negli adulti, le spine DD dirette ventralmente (punta di freccia) contattano i terminali presinaptici dei motoneuroni Ventrale A (VA) e Ventral B (VB) (magenta) e le commessure DD si estendono al cordone nervoso dorsale per fornire l'output GABAergico ai muscoli del corpo (freccia)18. Questa cifra è stata modificata rispetto al riferimento2. (B) Micrografie fluorescenti (Airyscan) di spine DD marcate con mCherry citosolico, mRuby miristoilato (MYR::mRuby), LifeAct::GFP e GFP::Utrophin in vermi giovani adulti. Le punte di freccia grigie indicano le spine. Barra della scala = 2 μm. (C) Densità (spine/10 μm) delle spine dendritiche del neurone DD marcate con mCherry citosolico (3,77 ± 0,9), MYR::mRuby (3,09 ± 0,8), LifeAct::GFP (3,44 ± 1,1) o GFP::Utrophin (2,41 ± 0,8). Tutti i campioni sono normalmente distribuiti. One-Way ANOVA mostra che le densità della colonna vertebrale per mCherry citosolico, MYR::mRuby e LifeAct::GFP non sono significativamente (NS) diverse, mentre la densità della colonna vertebrale è ridotta per GFP::Utrophin vs. mCherry (p = 0,0016) e LifeAct::GFP (p = 0,0082). La linea rossa tratteggiata rappresenta la densità della colonna vertebrale dei neuroni DD valutati dalla ricostruzione EM 3D (4,2 spine / 10 μm). Questa cifra è stata modificata rispetto al riferimento2. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Imaging delle spine dendritiche DD. (A) (In alto) Schema delle forme della colonna vertebrale. (In basso) Immagini Airyscan di ogni tipo di colonna vertebrale (barra di scala = 500 nm) etichettate con LifeAct::GFP (verde) e ricostruzioni 3D mediante micrografie elettroniche seriali di un adulto congelato ad alta pressione (blu). (B) Frequenza della colonna vertebrale per tipo, visualizzata con LifeAct::GFP: Sottile/Fungo (55,5 ± 14,5%), Filopodiale (10,3 ± 8,70%), Tozzo (18,8 ± 10,7%), Ramificata (15,42 ± 6,01%). Frequenza delle spine per tipo visualizzata con MYR::mRubino: Sottile/Fungo (52,2 ± 16,5%), Filopodiale (5,68 ± 7,0%), Tozzo (33,1 ± 14,8%), Ramificata (9,02 ± 9,6%). Test T spaiato, filopodiale (p = 0,0339); Le spine Stubby (p = 0.0009) e Branched (p = 0.011) etichettate con il marcatore MYR::mRuby sono significativamente diverse da LifeAct::GFP. Questa cifra è stata modificata rispetto al riferimento2. Fare clic qui per visualizzare una versione ingrandita di questa figura.
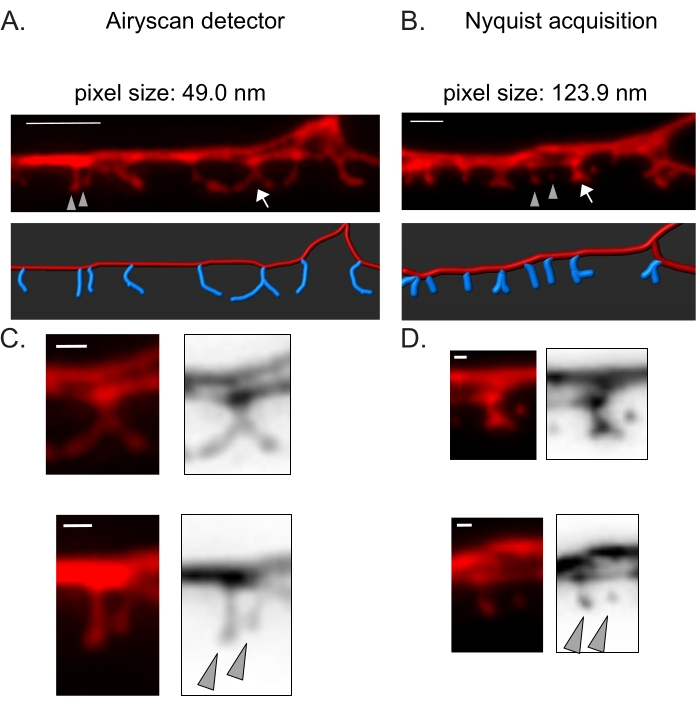
Figura 3: Strategie per l'acquisizione di immagini ad alta risoluzione delle spine DD . (A-B) (Inizio pagina) Immagini fluorescenti di dendriti DD1 marcate con un marcatore citosolico (mCherry) mediante (A) rivelatore Airyscan e (B) acquisizione Nyquist. (In basso) La dendrite DD (rossa) è raffigurata con un software di analisi delle immagini (opzione auto-path del tracciante del filamento) e le spine DD (blu) sono illustrate graficamente utilizzando il modulo di rilevamento delle spine semi-automatico. Le frecce puntano alla spina ramificata allargata in C e D. Le punte di freccia indicano le spine sottili / fungo vicine ingrandite in C e D. Barra di scala = 2 μm. (C-D) Esempi ingranditi di (in alto) spina ramificata (freccia) e (in basso) due spine sottili / a fungo vicine (punte di freccia) ottenute con (C) rivelatore Airyscan o per (D) acquisizione di Nyquist. Barra della scala = 500 nm. Dati riprodotti da2. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: Valutazione della funzione delle spine DD. (A) I motoneuroni DD esprimono l'indicatore Ca++ GCaMP6s (verde) e i motoneuroni VA esprimono la variante channelrhodopsin, Chrimson (magenta)7. (B) Metodo di rappresentazione schematica per il montaggio di worm per misure di Ca++ . (1) Su un vetrino da microscopio pulito, (2) posizionare 2 μL di perline di poli da 0,05 μm, (3) utilizzare un filo di platino ("worm pick") per aggiungere un piccolo globulo di super colla e (4) ruotare nella soluzione per generare fili filamentosi di colla. (5) Aggiungere 3μL di tampone M9. (6) Posizionare circa dieci larve di L4 nella soluzione, (7) applicare il coprislip e sigillare i bordi con vaselina/cera. (C-D) L'attivazione dei neuroni VA è correlata con i transitori Ca++ nelle spine DD1. La fluorescenza GCaMP6s ripresa (a intervalli di 0,5 s) con attivazione periodica della luce di Chrimson (intervalli di 2,5 s) evoca transitori Ca++ con (C) + ATR (n = 12) ma non nei controlli (D) (-ATR, n = 12). I pannelli sono istantanee nel tempo (s), prima e dopo l'impulso di 561 nm di luce (linea rosa verticale). Barre di scala = 2 μm. Il segnale GCaMP6s viene acquisito da un ROI (Regione di interesse) sulla punta di ciascuna spina. (E) Fluorescenza GCaMP6s durante il periodo di registrazione di 10 s tracciato per +ATR (verde) vs -ATR (controllo, grigio) (n = 12 video). Le barre rosa verticali indicano l'illuminazione a 561 nm (ad esempio, l'attivazione Chrimson). Ogni animale è stato stimolato 4 volte con 561 nm di luce. Le misurazioni sono state raccolte prima e dopo ogni impulso di luce a 561 nm. (F) Grafico della fluorescenza GCaMP6s prima e dopo ogni impulso di luce a 561 nm. La fluorescenza GCaMP6s è stata misurata 1 s dopo ogni impulso di luce a 561 nm. Poiché i campioni non sono normalmente distribuiti, è stato applicato un test di Friedman non parametrico accoppiato per correggere i confronti multipli della fluorescenza GCaMP6 prima rispetto a quelli con GCaMP6. dopo stimolazione luminosa a 561 nm per vermi cresciuti con ATR (+ATR, verde) (*** p = 0,0004, n = 48 misurazioni) o in assenza di ATR (-ATR, grigio) (NS, non significativo, p = 0,0962, n = 48 misurazioni). Questa cifra è stata modificata rispetto al riferimento2. Fare clic qui per visualizzare una versione ingrandita di questa figura.
File supplementare 1: Elenco dei plasmidi utilizzati nello studio. Clicca qui per scaricare questo file.
File supplementare 2: Composizione e preparazione del buffer M9. Clicca qui per scaricare questo file.
Discussione
Il rivelatore Airyscan è stato selezionato per acquisire istantanee di spine DD perché offre un rapporto segnale-rumore più elevato e una risoluzione migliore rispetto ai microscopi confocali convenzionali 19,20. L'imaging AiryScan consente anche l'uso di proteine fluorescenti convenzionali (ad esempio, GFP, mCherry, ecc.), ora ampiamente disponibili per C. elegans. Sebbene immagini ad alta risoluzione possano essere ottenute con altri metodi di super-risoluzione (ad esempio, STORM, STED, PALM), questi metodi richiedono proteine fluorescenti foto-attivabili o foto-commutabili21. In alternativa ad Airyscan, si raccomandano microscopi confocali convenzionali. Ad esempio, l'imaging con acquisizione Nyquist (Figura 3) raggiunge la dimensione dei pixel utilizzando un obiettivo 40x/1,3 di 123,9 nm, sufficiente a distinguere i tipi morfologici della colonna vertebrale (Figura 2).
Per determinare la densità della colonna vertebrale, si raccomanda di utilizzare una proteina fluorescente citosolica come (1) mCherry o GFP, (2) LifeAct per marcare il citoscheletro di actina o (3) una proteina fluorescente miristoilata (ad esempio, MYR::mRuby) per marcare la membrana plasmatica (Figura 1B). In confronto, la proteina legante la F-actina Utrophin riduce la densità della colonna vertebrale (Figura 1C), indicando un effetto negativo sulla morfogenesi della colonna vertebrale quando l'Utrophin è sovraespressa.
Gli attuali metodi di imaging dovrebbero aiutare a identificare le varianti genetiche che governano la morfologia della colonna vertebrale 1,16. La morfologia della colonna vertebrale DD (cioè sottile/fungo, filopodiale, tozza, ramificata, vedi Figura 2) può essere valutata da singole proiezioni 2D delle immagini laterali del cordone nervoso ventrale poiché la maggior parte delle spine DD adotta un orientamento tipicamente diretto ventralmente. In questi confronti, è essenziale utilizzare lo stesso marcatore fluorescente per ogni condizione poiché i tipi morfologici apparenti della colonna vertebrale sembrano essere influenzati dal metodo di etichettatura (ad esempio, MYR::mRuby vs. LifeAct::GFP). Inoltre, è stato notato che le forme della colonna vertebrale sono dinamiche e probabilmente cambiano forma in risposta ai segnali esterni 2,16. Pertanto, è anche essenziale confrontare le forme della colonna vertebrale tra genotipi in fasi di sviluppo simili e in condizioni simili.
L'orientamento del cordone ventrale di C. elegans è fondamentale per un'acquisizione accurata delle immagini. Sia le corde ventrali che dorsali sui lati opposti dell'animale dovrebbero essere visibili nello stesso piano Z, indicando che il verme è orientato su un lato (Figura 1B). È meglio non raccogliere immagini di vermi in movimento o in contatto con altri vermi o bolle vicino al cordone ventrale, poiché ciò può degradare le immagini delle spine.
Per l'imaging del calcio in vivo , i vetrini freschi devono essere preparati immediatamente prima di ogni acquisizione. È meglio visualizzare i vermi a contatto solo con fibre di colla sottili vs. "globi" di colla che tendono a seccare i vermi e degradare l'immagine (Figura 4B). Nell'esperimento mostrato in Figura 4, l'impulso della luce a 561 nm attiva l'intero campo visivo. Per aumentare la risoluzione temporale e spaziale per rilevare transitori Ca++ locali, ad esempio, all'interno di singole spine DD, è possibile utilizzare un mini scanner galvo impostato per la linea laser a 561 nm per stimolare una regione di interesse più piccola17.
Divulgazioni
Non dichiariamo conflitti di interesse.
Riconoscimenti
L'imaging e l'analisi su Imaris sono stati eseguiti nella Vanderbilt Cell Imaging Shared Resource (CIRS) supportata da NIH (CA68485, DK20593, DK58404, DK59637 e EY08126). LSM 880 è supportato dalla sovvenzione 1S10OD201630. L'imaging su un disco rotante Nikon è stato eseguito presso il Nikon Center of Excellence. Ringraziamo Jenny Schafer, direttore del CISR, e Bryan Millis per la formazione e le discussioni approfondite e i membri del laboratorio Burnette: Dylan Burnette, Aidan Fenix e Nilay Taneja per i consigli. Questo lavoro è stato supportato da sovvenzioni del National Institutes of Health a DMM (R01NS081259 e R01NS106951) e una sovvenzione dell'American Heart Association ad ACC (18PRE33960581).
Materiali
| Name | Company | Catalog Number | Comments |
| All-trans retinal (ATR) | Sigma-Aldrich | R2500-100MG | Necessary cofactor for neuronal excitation with Chrimson |
| diH2O | MilliQ | To prepare M9 buffer | |
| Ethanol 100% | Sigma | 64-17-5 | To dilute ATR and make control plates for neuronal excitation |
| Ethyl 3-aminobenzoate methanesulfonate salt (tricaine) | To immobilize animals for imaging dendritic spines | ||
| ImageJ | NIH | (Schindelin J et al., 2012) | Open source image processing software |
| KH2PO4 | Fisher Bioreagents | 7758-11-4 | To prepare M9 buffer |
| Levamisole hydrochloride | Sigma | 16595-80-5 | To immobilize animals for imaging dendritic spines |
| MgSO4 | Fisher Chemical | M63-500 | To prepare M9 buffer |
| Microscope cover glass | Fisherbrand | 12542B | To mount animals for microscopy acquisition |
| Na2HPO4 | Fisher Scientific | S369-500 | To prepare M9 buffer |
| NaCl | Fisher Chemical | S671-3 | To prepare M9 buffer |
| NIS Elements version 05.21 | Nikon | To analyze images and movies (e.g., Deconvolution, image alignment) | |
| Polybeads carboxylate 0.05um microspheres | Polysciences, Inc | 15913-10 | To immobilize animals for imaging Ca++ transients |
| Prism | For statistical analysis and graphing normalized Ca++ transients | ||
| SeaKen ME agarose | Lonza | 50014 | To make agarose pads to mount animals for imaging |
| Super Glue | The gorilla company | To immobilize animals for imaging Ca++ transients | |
| Superfrost microscope slides | Fisherbrand | 22-034-980 | To mount animals for microscopy acquisition |
| vaseline | Covidien | 8884430300 | To seal sample for confocal snapshots |
| Wax | Fisherbrand | 23-021-399 | Paraplast tissue embedding medium |
| Microscope for super-resolution imaging | |||
| LSM880 | Zeiss | ||
| AiryScan detector | Zeiss | ||
| Plan Apochromat (oil) 63x/ 1.40 NA, WD = 0.19 mm | |||
| Laser lines | |||
| Stage controller | |||
| Microscope for Nyquist image acquisition | |||
| A1R Confocal | Nikon | ||
| Plan Fluor (oil) 40x/1.3 NA, WD 0.24 mm | |||
| 488 nm, 16mW | |||
| 561 nm, 17mW | |||
| Microscope to monitor evoked Ca++ transients in dendritic spines | |||
| Spinning Disk Confocal | Nikon | ||
| Andor DU-897 EMCCD camera | |||
| Spinning disk Head CSU-X1 | Yokogawa | ||
| Apo TIRF (oil) 100x/1.49 NA ,WD 0.12 mm | |||
| 488 nm, 65mW | |||
| 561 nm, 86mW | |||
| 525 nm (+/- 18 nm) | |||
| 605 nm (+/- 35 nm) |
Riferimenti
- Sala, C., Segal, M. Dendritic Spines: The Locus of Structural and Functional Plasticity. Physiological Reviews. 94 (1), 141-188 (2014).
- Cuentas-Condori, A., et al. C. elegans neurons have functional dendritic spines. Elife. 8, 47918 (2019).
- Philbrook, A., et al. Neurexin directs partner-specific synaptic connectivity in C. Elegans. Elife. 7, 35692 (2018).
- Oliver, D., Alexander, K., Francis, M. M. Molecular Mechanisms Directing Spine Outgrowth and Synaptic Partner Selection in Caenorhabditis elegans. Journal of Experimental Neuroscience. 12, 10-13 (2018).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Ladt, K., Ganguly, A., Roy, S. Axonal actin in action: Imaging actin dynamics in neurons. Methods of Cell Biology. 131, 91-106 (2016).
- Schild, L. C., Glauser, D. A. Dual color neural activation and behavior control with chrimson and CoChR in Caenorhabditis elegans. Genetics. 200 (4), 1029-1034 (2015).
- Mello, C., Kramer, J., Stinchcomb, D., Ambros, V. Efficient gene transfer in C. elegans: extrahcormosomal maintenance and integration of transforming sequences. The EMBO Journal. 10 (12), 3959-3970 (1991).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C . elegans Using Microinjection. Journal of Visualized Experiments. (18), e833 (2008).
- Smith, C. J., et al. Time-lapse imaging and cell-specific expression profiling reveal dynamic branching and molecular determinants of a multi-dendritic nociceptor in C. elegans. Developemntal Biology. 345 (1), 18-33 (2010).
- Mccarter, J., Bartlett, B., Dang, T., Schedl, T. Soma - Germ Cell Interactions in Caenorhabditis elegans : Multiple Events of Hermaphrodite Germline Development Require the Somatic Sheath and Spermathecal Lineages. Developemntal Biology. 181 (2), 121-143 (1997).
- Driscoll, M. Mounting animals for observation with Nomarski DIC optics. WormBook. , (2008).
- North, A. J. Seeing is believing? A beginners' guide to practical pitfalls in image acquisition. Journal of Cell Biology. 172 (1), 9-18 (2006).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9, 676-682 (2012).
- Hotulainen, P., Hoogenraad, C. C. Actin in dendritic spines connecting dynamics to function. Journal of Cell Biology. 189 (4), 619-629 (2010).
- Berry, K. P., Nedivi, E. Spine Dynamics: Are They All the Same. Neuron. 96 (1), 43-55 (2017).
- Miller, D. M., Niemeyer, C. J. Expression of the unc-4 homeoprotein in Caenorhabditis elegans motor neurons specifies presynaptic input. Development. 121 (9), 2877-2886 (1995).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The Structure of the Ventral Nerve Cord of Caenorhadbitis elegans. Philosophical Transactions of the Royal Society. 275 (938), 327-348 (1976).
- Huff, J. The Airyscan detector from ZEISS confocal imaging with improved signal-to-noise ratio and super-resolution. Nature Methods. 12, (2015).
- Huff, J. The Fast mode for ZEISS LSM 880 with Airyscan high-speed confocal imaging with super-resolution and improved signal-to-noise ratio. Nature Methods. 13, (2016).
- Jacquemet, G., Carisey, A. F., Hamidi, H., Henriques, R., Leterrier, C. The cell biologist's guide to super-resolution microscopy. Journal of Cell Science. 133 (11), 240713 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon