È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificazione della citotossicità cellulare anticorpo-dipendente in un modello di sferoide tumorale: applicazione per la scoperta di farmaci
In questo articolo
Riepilogo
Qui, presentiamo un metodo per identificare i composti che modulano il meccanismo ADCC, un importante meccanismo di uccisione delle cellule tumorali degli anticorpi antitumorali. L'effetto citotossico delle cellule NK è misurato negli sferoidi delle cellule del cancro al seno in presenza di Trastuzumab. L'analisi delle immagini identifica le cellule killer vive e morte e le cellule bersaglio negli sferoidi.
Abstract
L'immunoterapia basata su anticorpi monoclonali che ha come bersaglio gli antigeni tumorali è ora un pilastro del trattamento del cancro. Uno dei meccanismi d'azione clinicamente rilevanti degli anticorpi è la citotossicità cellulare anticorpo-dipendente (ADCC), in cui l'anticorpo si lega alle cellule tumorali e impegna la componente cellulare del sistema immunitario, ad esempio le cellule natural killer (NK), per uccidere le cellule tumorali. L'efficacia di queste terapie potrebbe essere migliorata identificando composti adiuvanti che aumentano la sensibilità delle cellule tumorali o la potenza delle cellule immunitarie. Inoltre, interazioni farmacologiche non scoperte in pazienti oncologici co-medicati per condizioni pregresse o sintomi associati al cancro possono determinare il successo della terapia anticorpale; Pertanto, tali interazioni farmacologiche indesiderate devono essere eliminate. Con questi obiettivi in mente, abbiamo creato un modello di ADCC per il cancro e descriviamo qui un semplice protocollo per trovare farmaci modulanti l'ADCC. Poiché i modelli 3D come gli sferoidi delle cellule tumorali sono superiori alle colture 2D nel predire le risposte in vivo dei tumori alle terapie antitumorali, le co-colture sferoidi di cellule di carcinoma mammario HER2+ JIMT-1 che esprimono EGFP e NK92. Le linee cellulari CD16 sono state create e indotte con Trastuzumab, un anticorpo monoclonale clinicamente approvato contro il carcinoma mammario HER2-positivo. Gli sferoidi JIMT-1 sono stati lasciati formare in piastre a 96 pozzetti con fondo a U repellenti per le cellule. Il giorno 3 sono state aggiunte cellule NK e Trastuzumab. Gli sferoidi sono stati poi colorati con Annexin V-Alexa 647 per misurare la morte cellulare apoptotica, che è stata quantificata nella zona periferica degli sferoidi con un microscopio automatizzato. L'applicabilità del nostro test per identificare le molecole modulanti l'ADCC è dimostrata dimostrando che Sunitinib, un inibitore della tirosin-chinasi approvato dalla FDA contro il cancro metastatico, abolisce quasi completamente l'ADCC. La generazione degli sferoidi e le pipeline di acquisizione e analisi delle immagini sono compatibili con lo screening ad alto rendimento per i composti modulanti ADCC negli sferoidi delle cellule tumorali.
Introduzione
Gli sferoidi tumorali multicellulari (MCTS) sono modelli tridimensionali (3D) ampiamente utilizzati che si formano a causa della tendenza delle cellule aderenti ad aggregarsi e rappresentano uno strumento importante per ottenere informazioni meccanicistiche sulla biologia delle cellule tumorali. Possono essere generati da un'ampia gamma di tipi di cellule mediante numerose tecniche, come le colture 3D a base liquida e basata su scaffold1. Il loro principale vantaggio rispetto ai modelli 2D monostrato è che ricapitolano le caratteristiche principali dei tumori in vivo , vale a dire l'organizzazione strutturale e l'ipossia, imitando il comportamento biologico delle cellule tumorali, in particolare i meccanismi che portano alla fuga terapeutica e alla resistenza ai farmaci2. Pertanto, poiché gli MCTS possono migliorare la prevedibilità della tossicità e della sensibilità ai farmaci, sono ampiamente utilizzati per studiare i tumori in 3D e potrebbero migliorare lo sviluppo di farmaci efficaci per diversi tipi di cancro3.
Per studiare qualsiasi malattia, c'è un bisogno critico di modelli pertinenti e convenienti. La creazione di modelli per gli studi di immunologia del cancro è impegnativa perché il sistema immunitario è costituito da più tipi di cellule. Ogni tipo di cellula ha diversi sottotipi e un ampio spettro di stati di attivazione. Questi diversi tipi di cellule immunitarie interagiscono con le cellule tumorali e altri componenti tumorali, influenzando in ultima analisi l'esito della malattia. I metodi di coltura cellulare in vitro 2D non riescono a ricapitolare queste complesse interazioni cellulari, in quanto mancano di traducibilità e non sono in grado di prevedere l'azione di un farmaco a livello di sistema (ad esempio, nei tessuti)4,5. Inoltre, i modelli murini hanno anche gravi limitazioni a causa delle differenze fondamentali tra il sistema immunitario umano e quello murino. I sistemi di coltura 3D possono, quindi, colmare le attuali lacune nei modelli disponibili, fornendo un metodo alternativo e migliorando la nostra comprensione dell'immunologia del cancro6. In particolare, i modelli sferoidi potrebbero essere utilizzati per testare le immunoterapie, principalmente per valutare l'efficienza dello screening farmacologico e degli anticorpi terapeutici per migliorare l'infiltrazione delle cellule immunitarie e gli effetti antitumorali contro i bersagli sferoidi7. Inoltre, è stato ampiamente dimostrato il potenziale degli MCTS composti da cellule in diversi stati metabolici e proliferativi per studiare le interazioni tra cellule dello stroma (ad esempio, linfociti, macrofagi, fibroblasti) e cellule tumorali e per lo sviluppo di nuove strategie antitumorali8. Quindi, c'è una necessità vitale di corroborare piattaforme predittive e accurate al fine di potenziare il processo di test farmacologico, tenendo conto della fisiopatologia del microambiente tumorale.
Il carcinoma mammario (BC) è il tumore più frequente diagnosticato in tutto il mondo nelle donne. La classificazione clinica di questa malattia eterogenea si basa sulla presenza di recettori transmembrana, ad esempio i recettori degli estrogeni (ER) e del progesterone (PR) (collettivamente chiamati recettori ormonali, HR), insieme alla sovraespressione o all'amplificazione della proteina/oncogene del recettore 2 del fattore di crescita epidermico umano (HER2). Sulla base dell'espressione immunoistochimica di questi recettori, sono comunemente riconosciuti quattro sottotipi: carcinoma mammario luminale A (HR+/HER2-), luminale B (HR+/HER2+), HER2-positivo (HR-/HER2+) e carcinoma mammario triplo negativo (HR-/HER2-). Il gruppo HER2+ costituisce il 10-15% dei casi di BC ed è caratterizzato da un'elevata espressione di HER2 con assenza di ER e PR, ha una prognosi peggiore rispetto ai tumori luminali e richiede farmaci specifici diretti contro la proteina HER2/neu9.
Lo sviluppo del BC è un processo in più fasi e una diagnosi precoce è essenziale per un trattamento efficace della malattia10. Tuttavia, nonostante siano emerse di recente opzioni di trattamento personalizzate per la BC (ad esempio, terapie endocrine e anticorpali anti-HER2), la BC continua a rappresentare una sfida per gli oncologi. Proprio come la chirurgia, la chemioterapia e la radioterapia, anche queste terapie personalizzate possono avere gravi effetti avversi e i pazienti possono sviluppare resistenza a questi agenti, rendendo difficile determinare la strategia migliore a lungo termine11,12. Pertanto, una migliore comprensione dell'interazione tra il tumore e il suo microambiente è essenziale e dovrebbe fornire nuove direzioni per lo sviluppo di nuovi trattamenti che tengano conto delle specificità dei diversi sottotipi di BC13. Una nuova ondata di immunoterapie, come i coniugati farmaco-anticorpale, le terapie adottive con cellule T, i vaccini e i nuovi anticorpi monoclonali diretti a HER2 (mAb) sono in fase di studio in un'ampia popolazione di pazienti con tumori che esprimono HER214.
Trastuzumab, ad esempio, rappresenta una modalità di trattamento efficiente per HER2+ BC. Come parte del suo meccanismo d'azione, Trastuzumab media le attività dipendenti dal recettore gamma cristallizzabile (FcγR) del frammento. I FčγR si distinguono per la loro affinità per il frammento Fc e per la risposta immunitaria che avviano. L'attivazione di FcγRIIIa (CD16A) sulle cellule natural killer (NK) è fondamentale per mediare la citotossicità cellulare anticorpo-dipendente (ADCC), mentre l'attivazione di FcγRIIa (CD32A) e FcγRIIIa sui macrofagi induce la fagocitosi cellulare anticorpo-dipendente (ADCP)15. Studi su modelli animali hanno mostrato che i topi privi dei recettori FcγRI (CD64) e FcγRIII (CD16) non erano in grado di avviare risposte immunitarie protettive contro antigeni tumore-specifici, rivelando che l'ADCC è probabilmente un importante meccanismo d'azione per l'anticorpo monoclonale Trastuzumab16.
Poiché le cellule NK ricorrono agli Abs legati alle cellule tumorali per l'uccisione delle cellule tumorali da parte dell'ADCC, l'espressione dei recettori Fc è fondamentale per un trattamento efficace con Trastuzumab17 (Figura 1). Inoltre, la loro azione è efficacemente bilanciata da una stimolazione di recettori attivanti e inibitori, ad esempio i recettori Killer-cell immunoglobulin-like (KIR)18.
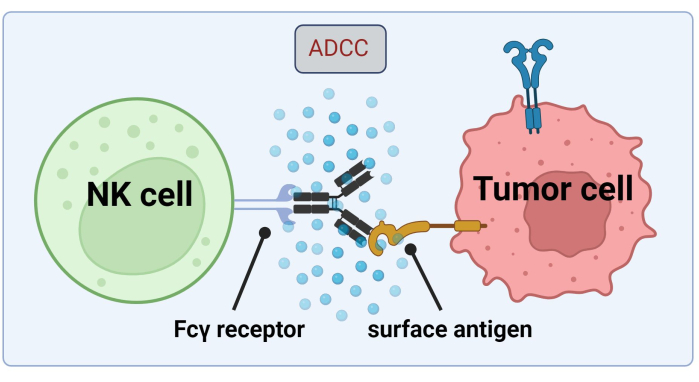
Figura 1. Meccanismo dell'ADCC nel contesto di una risposta antitumorale. Il recettore Fcγ di una cellula natural killer (NK) riconosce la regione Fc di un anticorpo, che in precedenza si era legato a un antigene di superficie su una cellula tumorale. Questa sinapsi immunologica porta alla degranulazione della cellula NK che rilascia mediatori citotossici come i granzimi e la perforina. Queste molecole contribuiscono alla formazione dei pori nella membrana cellulare e attivano le vie apoptotiche causando la morte cellulare programmata della cellula bersaglio (immagine creata con Biorender.com). Fare clic qui per visualizzare una versione più grande di questa figura.
Lo sviluppo dell'immunoterapia per HER2+ BC rappresenta un campo in evoluzione. In questo caso, si dovrebbero considerare le interazioni tra i vari componenti del sistema immunitario. Inoltre, pubblicazioni precedenti hanno ampiamente testato terapie combinate che coinvolgono tutti i tipi di terapie tradizionali, immunitarie o cellulari per identificare combinazioni sinergiche19.
Diversi modelli 3D di HER2+ BC sono stati precedentemente utilizzati per la scoperta di farmaci. Ad esempio, Balalaeva et al. hanno utilizzato sferoidi SKBR-3 che sovraesprimono HER2 per valutare la citotossicità dell'immunotossina mirata a HER2 4D5scFv-PE4020. In un altro studio, è stato creato un sistema di coltura HER2+ BC basato su Matrigel 3D per misurare la crescita cellulare in risposta a Trastuzumab e agenti endocrini21. Questi studi evidenziano l'importanza dei modelli di sferoidi tumorali di cellule tumorali sovraesprimenti HER2 nel rappresentare una strategia efficace per migliorare clinicamente le risposte terapeutiche22.
Il nostro gruppo ha precedentemente identificato Sunitinib, un inibitore multitarget della tirosin-chinasi, come inibitore dell'ADCC dipendente da trastuzumab nelle cellule JIMT-1 HER2+ BC in un test di coltura 2D. Lo studio ha rivelato che Sunitinib induce l'autofagia e compromette la funzione di uccisione delle cellule NK, riducendo l'espressione di HER2 e migliorando l'adesione superficiale delle cellule JIMT-117.
Qui abbiamo stabilito un nuovo modello 3D di ADCC sferoidi (cellule tumorali NK.92.CD16+Trastuzumab+JIMT-1-EGFP) da utilizzare per applicazioni di screening ad alto rendimento e, al fine di convalidare i risultati sopra menzionati, Sunitinib è stato utilizzato come composto modello. In primo luogo, abbiamo generato cellule JIMT-1 esprimenti EGFP17 e abbiamo fatto crescere sferoidi da queste cellule. L'ADCC è stato indotto da cellule NK insieme a Trastuzumab e gli sferoidi sono stati mantenuti in coltura in presenza o assenza di composti in esame per 24 ore (Figura 2). La quantificazione dell'ADCC si basa sulla rilevazione della morte delle cellule tumorali apoptotiche (colorazione con annessina V) utilizzando un sistema di analisi ad alto contenuto.
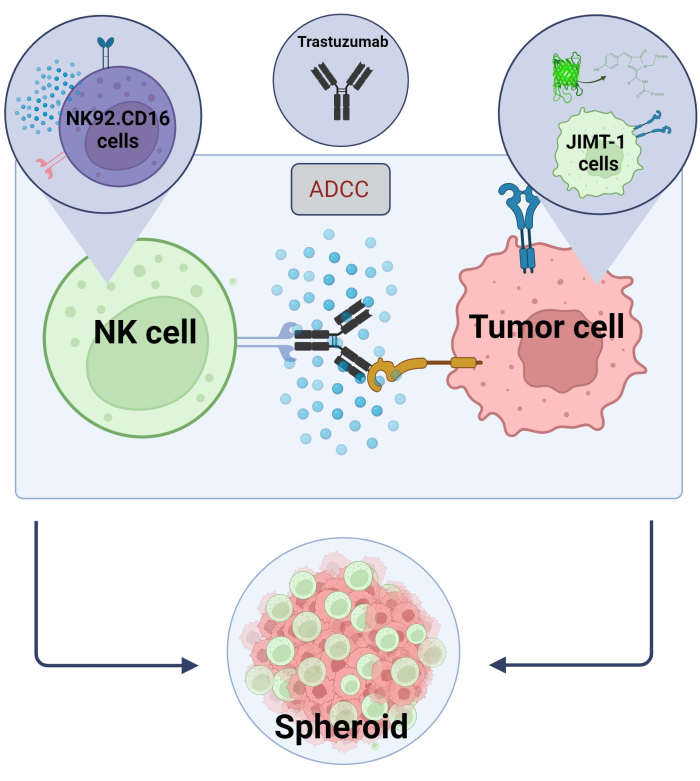
Figura 2. ADCC in un sistema di co-coltura di sferoidi 3D. Le nostre impostazioni sperimentali si basano su un sistema di sferoidi 3D in grado di modellare in modo più accurato il microambiente in vivo rispetto ai modelli 2D. Le cellule di carcinoma mammario JIMT-1 EGFP sono state seminate su un fondo concavo repellente per formare un ammasso cellulare di forma rotonda, chiamato sferoide. L'ADCC è stato quindi avviato con l'aggiunta di NK92. Cellule natural killer CD16 (rapporto E:T = 20:1) e un anticorpo monoclonale anti-HER2, Trastuzumab. Il modello sperimentale si è dimostrato efficiente e facilmente applicabile per l'identificazione di composti modificanti ADCC (immagine creata con Biorender.com). Fare clic qui per visualizzare una versione più grande di questa figura.
Abbiamo dimostrato che l'acquisizione di dati in questo modo può essere effettuata in tempo reale ed è statisticamente robusta per l'uso nello screening ad alto contenuto nella scoperta di farmaci antitumorali. È importante sottolineare che questo modello consente una convalida estesa di un insieme più ampio di composti e può essere applicato a diversi saggi di interesse.
Protocollo
1. Impostazione del modello sferoidale della proteina fluorescente potenziata JIMT-1 (EGFP)
- Per formare un fondo repellente per cellule a forma di U, rivestire la piastra a 96 pozzetti con una soluzione di agarosio-PBS allo 0,5% (30 μL/pozzetto). Incubare la piastra a temperatura ambiente per circa 30-45 min.
- Lavare due volte le cellule JIMT-1-EGFP con 2 mL di PBS sterile (la generazione della linea cellulare JIMT1-EGFP è stata riportata in una precedente pubblicazione17). Utilizzare palloni per coltura tissutale T25 e terreni JIMT-1 (terreno DMEM/F-12 integrato con il 20% di siero fetale bovino (FBS), 0,3 U/mL di insulina (100 UI/mL, Humulin R) e 1% di penicillina-streptomicina) per la coltura cellulare.
- Aggiungere 2 mL di tripsina-EDTA al matraccio e incubare per 10 minuti in un incubatore a CO2 .
- Picchiettare il pallone per verificare se le cellule JIMT-1 si sono staccate dopo il tempo di incubazione.
- Utilizzare 2 mL di terreno JIMT-1 per fermare la digestione e raccogliere la sospensione cellulare in una provetta da 15 mL.
- Contare le cellule in una camera Bürker con lo 0,4% di blu di tripano (80 μL di colorante + 20 μL di sospensione cellulare) e regolare il numero di cellule a 20.000 cellule/mL.
- Pipettare 100 μL della sospensione cellulare (2000 cellule/pozzetto) a ciascun pozzetto della piastra a 96 pozzetti (pre-rivestita con soluzione di agarosio-PBS allo 0,5% come indicato al punto 1.1).
- Lasciare che le cellule si aggreghino durante un periodo di incubazione di 3 giorni a 37 °C in un incubatore a CO2 .
- Controllare regolarmente le dimensioni e la forma degli sferoidi con un microscopio invertito.
2. Rivestimento della piastra HCS
NOTA: Per evitare l'attaccamento degli sferoidi JIMT-1-EGFP alla superficie di vetro della piastra, è fondamentale rivestire la piastra di screening ad alto contenuto (HCS) (altrimenti l'analisi ad alto contenuto non sarebbe possibile).
- Il giorno 3 dopo l'induzione degli sferoidi, rivestire la piastra di screening ad alto contenuto a 96 pozzetti con Pluronic-F127 (0,5% in DMSO, 50 μL/pozzetto) e incubare la piastra per 45 minuti a temperatura ambiente.
- Aspirare la soluzione di rivestimento e lavare i pozzetti due volte con terreno privo di siero DMEM/F-12 (100 μL/pozzetto).
3. Trasferimento degli sferoidi sulla piastra HCS
- Utilizzando una pipetta da 1 mL, trasferire gli sferoidi in triplice copia sulla piastra HCS a 96 pozzetti con fondo in vetro.
4. Pretrattamento degli sferoidi EGFP JIMT-1 con composti di prova
- Aggiungere il composto in esame (ad es. Sunitinib diluito in DMSO a una concentrazione di 40 μM) pipettando 10 μL/pozzetto e aggiungere 10 μL di terreno JIMT-1 fresco ai pozzetti di controllo (CTL).
- Incubare la piastra per 1 ora in un incubatore a CO2 a 37 °C.
5. Induzione dell'ADCC mediante l'aggiunta delle cellule effettrici
NOTA: Le cellule CD16.176V.NK92 (di seguito denominate cellule NK) sono state coltivate in α-MEM integrato con il 20% di FBS, l'1% di MEM-NEAA, l'1% di Na-piruvato, l'1% di glutammina, l'1% di penicillina-streptomicina e 100 UI/mL di IL-2.
- Raccogliere le cellule NK nel matraccio in una provetta da 15 ml. Contare le cellule con blu di tripano (80 μL del colorante + 20 μL della sospensione cellulare) e regolare la densità cellulare al rapporto effettore-bersaglio (E:T) di 20:1 (40.000 cellule NK/pozzetto).
- Colorare le cellule NK con 10 μM Cell Tracker Blue (CTB, 1 μL in 1 mL di terreno NK α-MEM) e metterle in un incubatore di CO2 a 37 °C per 1 ora.
- Centrifugare le cellule NK a 150 x g per 3 minuti a temperatura ambiente due volte per lavare l'eccesso di colorante con 1 mL di terreno α-MEM.
- Risospendere il pellet di cellule NK in 1 mL di terreno JIMT-1 fresco.
- Aggiungere le cellule NK colorate insieme all'anticorpo anti-HER2 (Ab) (Trastuzumab, disciolto in H2O distillato sterile) agli sferoidi JIMT-1 bersaglio pipettando 55 μL/pozzetto di cellule NK colorate con CTB e 55 μL/pozzetto di 10 μg/mL Ab nel terreno JIMT-1 (il volume totale di trattamento aggiunto per l'ADCC è di 110 μL/pozzetto e la concentrazione finale di Sunitinib è di 20 μM).
NOTA: Per la selezione della concentrazione Ab e del rapporto E:T, ci siamo basati sulla nostra precedente pubblicazione17. Sono stati condotti esperimenti preliminari con diversi rapporti E:T e concentrazioni di Trastuzumab per valutare quale fosse il più efficace nell'indurre ADCC negli sferoidi (Figura supplementare S1). - Aggiungere 110 μL/pozzetto di terreno JIMT-1 fresco per controllare (CTL).
- Incubare la piastra per 24 ore in un incubatore CO2 a 37 °C.
NOTA: Poiché è noto che le cellule NK92 esercitano funzioni citotossiche non specifiche, per il controllo utilizzare cellule JIMT-1 co-incubate sia con cellule NK da sole che con un controllo isotipo o un Ab F(ab')2. In questo caso F(ab')2-Trastuzumab (TR-F(ab')2) è stato utilizzato come CTL negativo. Il frammento TR-F(ab')2 è stato preparato come riportato da Tóth et al.19 e aggiunto nello stesso volume di Trastuzumab insieme alle cellule NK come descritto in 5.5. La concentrazione è stata aggiustata a 6,6 μg/mL corrispondente a una concentrazione equimolare con Trastuzumab23.
6. Annessina V-647: colorazione degli sferoidi
- Per misurare la morte cellulare apoptotica, colorare gli sferoidi con coniugato di annessina V-Alexa Fluor 647 per 1 ora in terreno JIMT-1 (1:100) pipettando 50 μL/pozzetto.
7. Imaging
NOTA: La piastra viene ripresa a 24 ore dopo l'aggiunta delle cellule effettrici NK alle cellule bersaglio. Per l'imaging vengono utilizzati un analizzatore ad alto contenuto e un software di analisi delle immagini.
- Selezionare il tipo di micropiastra dall'elenco delle piastre utilizzando l'opzione Tipo di piastra . Utilizzare una piastra di screening ad alto contenuto (HCS) a 96 pozzetti.
- Selezionare l'autofocus a due picchi poiché il test viene eseguito su lastre con obiettivo 10x con apertura numerica di 0,3 (NA), utilizzando rispettivamente le opzioni Messa a fuoco automatica e Obiettivo . Scegliere la modalità confocale con l'opzione Modalità opzionale e applicare il Binning 2 utilizzando l'opzione Binning .
- Per l'imaging degli sferoidi, selezionare i canali appropriati utilizzando l'opzione Selezione canale . Per rilevare le cellule JIMT-1 trasdotte con EGFP scegliere EGFP (tempo di integrazione 200 ms, potenza laser 50%, altezza stack 2,0 μm; es: 488 nm em: 500-550 nm) e per rilevare le cellule apoptotiche utilizzare Alexa 647 (tempo di integrazione 100 ms, potenza laser 50%, altezza stack 10,0 μm; es: 640 nm em: 650-760 nm). Per visualizzare le cellule NK all'interno degli sferoidi, scegliere il seguente canale: DAPI (tempo di integrazione 100 ms, potenza laser 50%, altezza stack 2,0 μm; es: 405 nm em: 435-480 nm).
- Nell'opzione Selezione layout , selezionare le pile Z poiché le cellule negli sferoidi tendono a trovarsi su piani focali diversi del microscopio. 10 piani (con una distanza di 10 μm) sono sufficienti per coprire l'intera regione sferoidale. Impostare i valori del primo piano e dell'ultimo piano rispettivamente a 0 μm e 90 μm.
NOTA: Prima di iniziare la misurazione, è possibile scattare immagini campione con la funzione istantanea per verificare le impostazioni corrette. - Impostare il numero di pozzetti e campi per l'imaging utilizzando l'opzione Definisci layout .
8. Analisi ad alto contenuto (HCA)
NOTA: Analizzare le immagini con il software Harmony o esportare le immagini per l'analisi utilizzando un software di terze parti preferito. Per l'analisi dell'efficienza dell'ADCC, viene misurata l'intensità di fluorescenza dell'annessina V 647. Le cellule bersaglio uccise dall'ADCC appaiono nell'area periferica degli sferoidi. Pertanto, le cellule positive all'annessina V vengono misurate in questo "anello" sferoidale. Al fine di convalidare questo metodo, sono state eseguite analisi con diversi parametri per valutare quale fosse il più affidabile e producesse i migliori risultati (Figura supplementare S2). Nel video viene mostrato un pozzetto ADCC per dimostrare passo dopo passo il processo di analisi delle immagini.
- Identifica gli sferoidi in base alla fluorescenza EGFP delle cellule JIMT-1 utilizzando l'opzione Trova regioni di trama e filtrali per dimensione (> 25.000 μm2).
- Rimuovi gli oggetti bordo tramite l'opzione Seleziona popolamento .
- Poiché le cellule positive all'annessina V (apoptotiche) compaiono alla periferia degli sferoidi, misurare l'intensità dell'annessina in questo "anello" apoptotico, selezionato dall'opzione Seleziona regione (bordo esterno -90%) per determinare la morte cellulare apoptotica.
- Esprimere i valori di intensità dell'annessina V come intensità media.
Risultati
Sono state generate cellule JIMT-1 che esprimono EGFP e da queste cellule sono stati coltivati sferoidi. Sunitinib è stato utilizzato come composto di prova in quanto è stato precedentemente dimostrato che influisce sul decorso dell'ADCC17. Gli sferoidi sono stati lasciati aggregare per 72 ore. Il giorno 3, 10 μg/mL di Trastuzumab (o 6,6 μg/mL TR-F(ab')219) equimolari e cellule NK (20:1) sono stati aggiunti agli sferoidi in presenza o in assenza di 20 μM di ...
Discussione
Nonostante i significativi miglioramenti nel trattamento della BC negli ultimi decenni, i pazienti sviluppano ancora regolarmente resistenza ai farmaci o sperimentano effetti collaterali negativi24. L'elevata morbilità e mortalità legate alla BC richiede una continua indagine sui meccanismi molecolari sottostanti, così come robuste piattaforme di screening per identificare nuove molecole utilizzabili per lo sviluppo terapeutico25. Queste strategie richiedono saggi traduz...
Divulgazioni
Gli autori hanno dichiarato di non avere conflitti di interesse.
Riconoscimenti
LV ha ricevuto finanziamenti dall'Ufficio nazionale per la ricerca, lo sviluppo e l'innovazione sovvenzioni GINOP-2.3.2-15-2016-00010 TUMORDNS", GINOP-2.3.2-15-2016-00048-STAYALIVE, OTKA K132193 e K147482. Questo progetto ha ricevuto finanziamenti dalla rete di ricerca ungherese HUN-REN. Le cellule CD16.176V.NK-92 sono state ottenute dal Dr. Kerry S. Campbell (Fox Chase Center, Philadelphia, PA, per conto di Brink Biologics, lnc. San Diego, CA), sono protetti da brevetti in tutto il mondo e sono stati concessi in licenza da Nantkwest, lnc. (www.nantkwest.com). Gli autori sono grati a György Vereb e Árpád Szöőr per il loro aiuto con l'uso della linea cellulare NK-92 e del TR-F(ab')2, e per la consulenza tecnica.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well glass bottom Cell Carrier Ultra microplates | PerkinElmer, Waltham, MA, USA | LLC 6055302 | for spheroids measurements |
| 96-well tissue culture plates | TPP | 92096 | for cell seeding |
| α-MEM medium | Sigma | M8042 | in NK medium |
| Agarose | Sigma | A9539 | for spheroids seeding |
| Annexin V-Alexa Fluo 647 conjugate | Invitrogen-ThermoFisher Scientific | A23204 | for apoptosis measurement with HCS |
| CD16.176 V.NK-92 cells | Dr. Kerry S. Campbell (the Fox Chase Cancer Center, Philadelphia, PA on behalf of Brink Biologics, Inc. San Diego, CA) | ATCC CRL-2407 | for cell culture |
| Cell Tracker Blue | Invitrogen-Thermo Scientific, Waltham, MA, USA) | C2110 | for staining of NK cells |
| DMEM/F-12 medium | Sigma | D8437 | in JIMT1-EGFP medium |
| Dimethyl sulfoxide (DMSO) | Sigma | D8418 | for coating HCS plate before transfering the spheroids |
| Fetal bovine serum (FBS) | Biosera | FB-1090/500 | JIMT-1-EGFP and NK medium |
| Glutamine | Gibco | 35,050–061 | in NK medium |
| GraphPad Prism 8.0.1 | GraphPad Software Inc., San Diego, CA, USA | for statistical analysis | |
| Harmony software | PerkinElmer, Waltham, MA, USA | for HCA | |
| IL-2 | Proleukin, Novartis Hungária Kft., Budapest, Hungary | PHC0026 | in NK medium |
| Insulin (Humulin R) | Eli Lilly | HI0219 | JIMT-1-EGFP medium |
| JIMT-1 breast cancer cells | for cell culture | ||
| MEM Non-essential Amino Acids (MEM-NEAA) | Gibco | 11,140–050 | in NK medium |
| Na-pyruvate | Lonza | BE13-115E | in NK medium |
| Opera Phenix High-Content Analysis equipment | PerkinElmer, Waltham, MA, USA | HH14001000 | for HCA |
| PBS (Posphate buffered saline) | Lonza | BE17-517Q | for washing the cells |
| Penicillin-Streptomycin | Biosera | LM-A4118 | JIMT-1-EGFP and NK medium |
| pLP-1, pLP-2, pLP-VSV-G, pWOXEGFP | Invitrogen, (Prof. József T zsér, University of Debrecen) zsér, University of Debrecen) | for JIMT-1-EGFP cell line | |
| Pluronic-F127 | Sigma | P2443 | for coating HCS plate before transfering the spheroids |
| Sunitinib malate | SigmaAldrich | PZ0012 | for treatments |
| Trastuzumab Ab (humanized anti-HER2 monoclonal antibody) | Herzuma®, EGIS Pharmaceuticals, Budapest, Hungary | NDC-63459-303-43 | for treatments |
| Trastuzumab-F(ab')2 | Gift from Prof. György Vereb and Árpád Szö r r | Department of Biophysics and Cell Biology, University of Debrecen | for treatments |
| Trypan blue 0.4% solution | Sigma | T8154 | for cell counting |
| Trypsin-EDTA 1X in PBS | Biosera | LM-T1706 | for cells detachment |
Riferimenti
- Jubelin, C., et al. Three-dimensional in vitro culture models in oncology research. Cell Biosci. 12 (1), 155 (2022).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Front Oncol. 7, 293 (2017).
- Badr-Eldin, S. M., Aldawsari, H. M., Kotta, S., Deb, P. K., Venugopala, K. N. Three-dimensional in vitro cell culture models for efficient drug discovery: Progress so far and future prospects. Pharmaceuticals (Basel). 15 (8), 926 (2022).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Pinto, B., Henriques, A. C., Silva, P. M. A., Bousbaa, H. Three-dimensional spheroids as in vitro preclinical models for cancer research. Pharmaceutics. 12 (12), 1186 (2020).
- Fitzgerald, A. A., Li, E., Weiner, L. M. 3D culture systems for exploring cancer immunology. Cancers (Basel). 13 (1), 56 (2020).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: A systematic approach to enhance the biological relevance of data obtained. Sci Rep. 6, 19103 (2016).
- Boucherit, N., Gorvel, L., Olive, D. 3D tumor models and their use for the testing of immunotherapies. Front Immunol. 11, 603640 (2020).
- Li, Y., et al. Recent progress on immunotherapy for breast cancer: Tumor microenvironment, nanotechnology and more. Front Bioeng Biotechnol. 9, 680315 (2021).
- Lo Nigro, C., et al. NK-mediated antibody-dependent cell-mediated cytotoxicity in solid tumors: Biological evidence and clinical perspectives. Ann Transl Med. 7 (5), 105 (2019).
- Sun, Y. S., et al. Risk factors and preventions of breast cancer. Int J Biol Sci. 13 (11), 1387-1397 (2017).
- Liu, P. H., Wei, J. C., Wang, Y. H., Yeh, M. H. Female breast cancer incidence predisposing risk factors identification using nationwide big data: A matched nested case-control study in taiwan. BMC Cancer. 22 (1), 849 (2022).
- Burguin, A., Diorio, C., Durocher, F. Breast cancer treatments: Updates and new challenges. J Pers Med. 11 (8), 808 (2021).
- Costa, R. L. B., Czerniecki, B. J. Clinical development of immunotherapies for HER2(+) breast cancer: A review of HER2-directed monoclonal antibodies and beyond. NPJ Breast Cancer. 6, 10 (2020).
- Musolino, A., et al. Role of fcgamma receptors in HER2-targeted breast cancer therapy. J Immunother Cancer. 10 (1), e003171 (2022).
- Petricevic, B., et al. Trastuzumab mediates antibody-dependent cell-mediated cytotoxicity and phagocytosis to the same extent in both adjuvant and metastatic HER2/NEU breast cancer patients. J Transl Med. 11, 307 (2013).
- Guti, E., et al. The multitargeted receptor tyrosine kinase inhibitor sunitinib induces resistance of HER2 positive breast cancer cells to Trastuzumab-mediated ADCC. Cancer Immunol Immunother. 71 (9), 2151-2168 (2022).
- Barok, M., et al. Trastuzumab decreases the number of circulating and disseminated tumor cells despite Trastuzumab resistance of the primary tumor. Cancer Lett. 260 (1-2), 198-208 (2008).
- Toth, G., et al. The combination of Trastuzumab and Pertuzumab administered at approved doses may delay development of Trastuzumab resistance by additively enhancing antibody-dependent cell-mediated cytotoxicity. MAbs. 8 (7), 1361-1370 (2016).
- Ehlers, F. a. I., et al. ADCC-inducing antibody Trastuzumab and selection of KIR-HLA ligand mismatched donors enhance the NK cell anti-breast cancer response. Cancers (Basel). 13 (13), 3232 (2021).
- Gangadhara, S., Smith, C., Barrett-Lee, P., Hiscox, S. 3d culture of Her2+ breast cancer cells promotes AKT to MAPK switching and a loss of therapeutic response. BMC Cancer. 16, 345 (2016).
- Balalaeva, I. V., Sokolova, E. A., Puzhikhina, A. D., Brilkina, A. A., Deyev, S. M. Spheroids of Her2-positive breast adenocarcinoma for studying anticancer immunotoxins in vitro. Acta Naturae. 9 (1), 38-43 (2017).
- Selis, F., et al. Pegylated Trastuzumab fragments acquire an increased in vivo stability but show a largely reduced affinity for the target antigen. Int J Mol Sci. 17 (4), 491 (2016).
- Johnston, R. L., et al. High content screening application for cell-type specific behaviour in heterogeneous primary breast epithelial subpopulations. Breast Cancer Res. 18 (1), 18 (2016).
- Kandaswamy, C., Silva, L. M., Alexandre, L. A., Santos, J. M. High-content analysis of breast cancer using single-cell deep transfer learning. J Biomol Screen. 21 (3), 252-259 (2016).
- Esquer, H., Zhou, Q., Abraham, A. D., Labarbera, D. V. Advanced high-content-screening applications of clonogenicity in cancer. SLAS Discov. 25 (7), 734-743 (2020).
- Pickl, M., Ries, C. H. Comparison of 3D and 2D tumor models reveals enhanced Her2 activation in 3D associated with an increased response to trastuzumab. Oncogene. 28 (3), 461-468 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon