このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
シングルショウジョウバエ個眼の解剖とイメージング
要約
成人の使用の制限要因ショウジョウバエ目には細胞内プロセスの困難な画像です。我々は薬物療法と高度な画像処理の対象になることができる正真正銘の初代神経細胞培養を、生成する単一のommatidiumの複数形の解剖を説明します。
要約
ショウジョウバエでは神経科学研究に貴重な貢献をしているとので、その強力な遺伝学1の神経変性疾患のモデルとして広く用いられている。特にフライアイは、 ショウジョウバエの神経系の最もアクセスとライフなくても済む部分である、神経変性の研究のための選択の器官となっている。しかし、そのまま目の主な注意点は、神経変性の理解に重要であるようなオートファジーのダイナミクス2のようなイメージング細胞内のイベント、、で、ために色素の強い自家蛍光が、困難です。
我々は最近Dentatorubro核淡蒼球萎縮症(DRPLA)3、4のハエのモデルにおけるオートファジー機能障害の理解に不可欠であった単ommatidiumの複数形3の解剖と文化を使用しています。
我々は現在、劇的に神経変性のためのフライのモデルの可能性を拡大する可能性がある電気生理学的研究5日から適応このテクニックの包括的な説明(図1)、報告する。このメソッドは、画像細胞内イベントを生活し、視細胞(図2)に効果的な薬剤投与を監視するために適応させることができます。モザイクの技法6月8日と組み合わせて使用する場合、遺伝的に異なる細胞の応答は平行(図2)で検定することができる。
プロトコル
1。 ショウジョウバエの網膜の解剖
- リン酸3ウェルガラススライドの1つのウェルを埋めるには、CO 2でハエを麻酔し(PBS 1X)緩衝生理食塩水、と。オスとメスの両方がこの実験に使用することができます。
- ハエが麻酔したら、鉗子を使用して単一のフライをピックアップし、PBSの小さな皿にドロップします。
- 解剖スコープの下では、鉗子で口吻をピンチし、鉗子の2番目のセットを使用して首を切断することによって身体から頭を切り離す。
- 左/右正中脳を明らかにするためにmicroscissorsとハエの頭に沿って半分にカット、テングザルからハエの頭を保持する。
- 網膜を拾うためにカットピペットの先端を使用して、とシュナイダー媒体で満たされた3ウェルガラススライドの第二のウェルに移す。
- 最初に、頭部のクチクラの離れてほとんど剥離し、鉗子で脳を取り除く。粘膜と角膜の間に挟まれた網膜を保つ。
- 次に、スライドガラスを直接配置する新鮮Schniederの培地のドロップ(50μl)の上に網膜を転送する。
2。 ommatidiumの複数形の解剖
- 解剖の最後の目的(直接イメージングや文化かどうか)に、蛍光顕微鏡(直接または逆)の光学系に応じて、このステップでは、顕微鏡スライド上または24ウェルプレートのどちらかの場所を取ることができます。この解剖の最終的な目的のために、ommatidiumの複数形は、スライドガラス上に直接解剖されます。
- どちらの鉗子と網膜の最も背側または腹側の端をつかみます。 microscissorsを使用して、目の真ん中にommatidiumの複数形を公開するために背側/腹側正中線に沿って半分に網膜をカット。
- 角膜がダウンして直面しているとラミナが上を向いているような網膜を把握し続ける。微細なタングステン針を用いて、葉身と角膜からommatidiumの複数形を切り離します。
- シングルommatidiumの複数形とommatidiumの複数形のグループは、ウェルまたはスライドの下部に収集します。先に進む前に、収集した可能性のある大きなごみを取り除きます。
3。治療、染色およびイメージング
- オートファジー分析するために、、シュナイダーのドロップ(シュナイダーの最初の希釈1:10およびその後スライド上のドロップに0.5μLを加える)で1:1000 Lysotrackerを希釈混合し、10秒間待ちます。
- 背後ommatidiumの複数形を残すように注意して、スライドから余分な液体を取り除く。これは、微細なゲルローディングチップで最善を尽くしています。
- ommatidiumの複数形をカバーするためにVectashieldのドロップを追加、nailpolishとカバーとシールを適用します。
4。代表的な結果:
プロトコルの適切な実行は、ラミナの断片によって一緒に保持がうまく角膜側で区切られた単一のommatidiumの複数形のとommatidiumの複数形の小グループの数を残してください。オートファゴソーム、リソソームとautophagolysosomes(図3)を示す、GFPとLysotracker:これらは、Atg8を視覚化するために、この例のように撮像することができます。メソッドは、ライブイメージングに使用することができます、しかし、このケースではシュナイダーのメディアの自己蛍光が困難、低輝度蛍光シグナルを区別するために行うことを留意すべきである。追加の問題は、光受容体に接続されたままに色素顆粒は、特に赤のチャネルで、強い蛍光性であるということです。明視野像は、本物の小器官と区別するために必要となる場合があります。
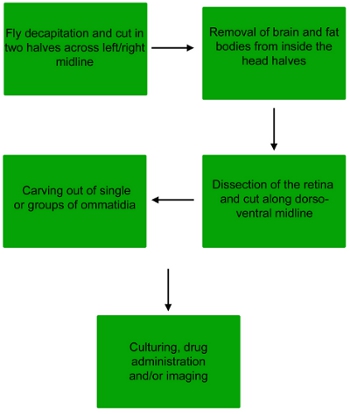
図1解剖の手順のフローチャートは、ハエの網膜から単ommatidiumの複数形やommatidiumの複数形の小さなグループを取得する。
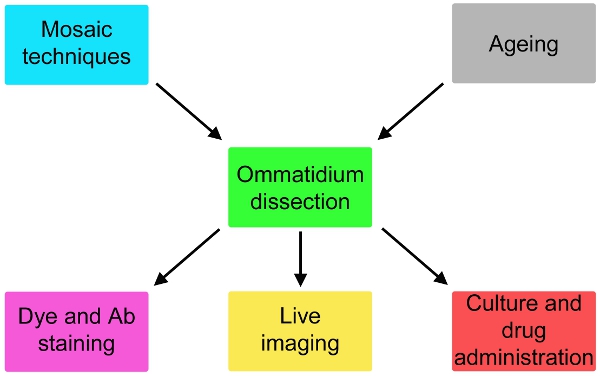
図2。単純なプロトコルの可能なバリエーションがここでは、遺伝学を飛ばすと24時間と解剖の薬剤投与の下流までのために上流の解剖と染色や培養の老化含むものについて説明します。
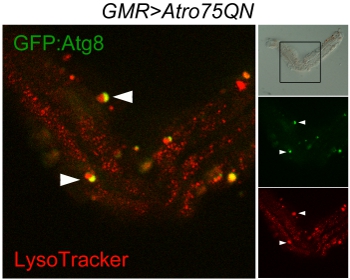
図3 AWから切除した単一の個眼ではオートファゴソームとリソソームの共焦点スキャン、GMR - GAL4、UAS - Atro75QN、UAS - GFP::Atg8 / +フライ、29位のグルタミンAtrophin変異体と歳12日を表す℃に明パネル(右上)は、個眼のほぼそのままの構造を表示し、フレームには、スキャン範囲を示します。赤いマークのリソソーム、オートファゴソーム緑、オートリソソーム、二つの細胞小器官との融合によって、その結果、黄色に表示されます。矢頭は、オートファゴソームとリソソームの間に2つの進行中の融合イベントを示している。
ディスカッション
ここで紹介する単一の個眼の解剖は、 ショウジョウバエの眼でモデル化された神経変性のようなプロセスは、約深い細胞生物学的情報の収集を可能にします。光受容ニューロンは、他のニューロンよりも簡単にアクセスでき、それら細胞質は生体内の神経変性を定量化するための多くのモデルで上手に使用される非常に同じ細胞では、特に大規模、および小胞のダイナミクスを...
開示事項
利害の衝突は宣言されません。
謝辞
我々は議論のためにバーナードシャルーに感謝。この作品は、KCL生物医学学校によって資金を供給された。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント(省略可能) |
|---|---|---|---|
| ポータブルCO2ディスペンサー | Flystuff | 59から150 | また、利用可能なリフィル |
| デュモンピンセットいいえ5 | 寒天サイエンティフィック | T5034 | |
| 3ウェルガラススライド | EMS | 71561〜01 | |
| マイクロはさみ | VWR | HAMMHSB516 - 09 | |
| シュナイダーのミディアム | VWR | 733-1663 | |
| LysoTrackerレッドDND - 99 | インビトロジェン | L7528 | |
| Superfrostスライド | VWR | 631-0108 | |
| DAPIでVectashield | ベクトル研究所 | H - 1200 | |
| カバースリップ | VWR | 631-1336 |
参考文献
- Marsh, J. L., Thompson, L. M. Drosophila in the study of neurodegenerative disease. Neuron. 52, 169-178 (2006).
- Mizushima, N., Levine, B., Cuervo, A. M., Klionsky, D. J. Autophagy fights disease through cellular self-digestion. Nature. 451, 1069-1075 (2008).
- Nisoli, I. Neurodegeneration by polyglutamine Atrophin is not rescued by induction of autophagy. Cell Death Differ. 17, 1577-1587 (2010).
- Charroux, B., Fanto, M. The fine line between waste disposal and recycling: DRPLA fly models illustrate the importance of completing the autophagy cycle for rescuing neurodegeneration. Autophagy. 6, 667-669 (2010).
- Hardie, R. C. Voltage-sensitive potassium channels in Drosophila photoreceptors. J Neurosci. 11, 3079-3095 (1991).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker (MARCM) for Drosophila neural development. Trends in neurosciences. 24, 251-254 (2001).
- Xu, T., Rubin, G. M. Analysis of genetic mosaics in developing and adult Drosophila tissues. Development. 117, 1223-1237 (1993).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. Dev Biol. 351, 128-1234 (2011).
- Napoletano, F. Polyglutamine Atrophin provokes neurodegeneration in Drosophila by repressing fat. The EMBO journal. 30, 945-958 (2011).
- Ravikumar, B. Inhibition of mTOR induces autophagy and reduces toxicity of polyglutamine expansions in fly and mouse models of Huntington disease. Nature. 36, 585-595 (2004).
- Zou, S., Meadows, S., Sharp, L., Jan, L. Y., Jan, Y. N. Genome-wide study of aging and oxidative stress response in Drosophila melanogaster. Proceedings of the National Academy of Sciences of the United States of America. 97, 13726-13731 (2000).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved