È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Singolo Drosophila Ommatidium Dissection e Imaging
In questo articolo
Riepilogo
Il fattore limitante l'uso degli adulti Drosophila per studiare neurodegenerazione e biologia cellulare è l'imaging difficile dei processi intracellulari. Descriviamo la dissezione dei singoli ommatidi per generare una buona fede colture primarie di cellule neuronali, che possono essere soggetti a trattamento farmacologico e di imaging avanzate.
Abstract
La mosca della frutta Drosophila melanogaster ha dato un contributo inestimabile alla ricerca neuroscientifica ed è stato ampiamente utilizzato come modello per le malattie neurodegenerative a causa della sua potente genetica 1. L'occhio vola in particolare è stato l'organo di scelta per la ricerca neurodegenerazione, essendo la parte più accessibile e di vita indispensabili del sistema nervoso Drosophila. Ma l'avvertimento principale degli occhi intatto è la difficoltà, a causa della intensa autofluorescenza del pigmento, in eventi di imaging intracellulari, come autofagia dinamica 2, che sono di primaria importanza per la comprensione della neurodegenerazione.
Recentemente abbiamo usato la dissezione e la cultura dei singoli ommatidi 3 che è stato essenziale per la comprensione delle disfunzioni autofagici in un modello di volo Dentatorubro-Pallidoluysian Atrofia (DRPLA) 3, 4.
Ora segnalare una descrizione completa di questa tecnica (Fig. 1), adattato da studi elettrofisiologici 5, che rischia di ampliare notevolmente la possibilità di modelli di volo per neurodegenerazione. Questo metodo può essere adattato a vivere immagine eventi subcellulare e per monitorare la somministrazione efficace farmaco su cellule fotorecettrici (Fig. 2). Se usato in combinazione con tecniche di mosaico 6-8, le risposte di cellule geneticamente diverse possono essere analizzati in parallelo (Fig. 2).
Protocollo
1. Dissezione della retina Drosophila
- Riempire un pozzo di un 3-Bene vetrino con tampone fosfato (PBS 1X), e poi anestetizzare vola da CO 2. Sia i maschi che le femmine possono essere utilizzati per questo esperimento.
- Una volta che le mosche sono anestetizzati, prendere una mosca con pinze e inseritelo nel piccolo piatto di PBS.
- In un ambito di dissezione, pizzicare la proboscide con una pinza, e poi staccare la testa dal corpo eliminando il collo con una seconda serie di pinze.
- Tenendo la testa vola dalla proboscide, tagliata a metà lungo la linea mediana a sinistra / destra della testa volare con microscissors per rivelare il cervello.
- Utilizzare un puntale tagliato a raccogliere la retina, e trasferirli in un secondo pozzo della 3-Bene vetrino di vetro pieni di medio Schneider.
- In primo luogo, buccia via la maggior parte della cuticola testa e poi rimuovere il cervello con una pinza. Mantenere la retina a sandwich tra la lamina e la cornea.
- Poi, il trasferimento della retina su una goccia (50 ml) di media Schnieder fresca posizionato direttamente del vetrino.
2. Dissezione del ommatidi
- A seconda dello scopo finale della dissezione (sia immagini immediato o la cultura) e l'ottica del microscopio a fluorescenza (diretta o invertita), questo passaggio può avvenire sia su un vetrino o in una 24-pozzetti. Per lo scopo finale di questa dissezione, il ommatidi verrà sezionato direttamente sul vetrino.
- Afferrare una delle estremità più dorsale o ventrale della retina con una pinza. Utilizzando microscissors, tagliare la retina a metà lungo la linea mediana dorsale / ventrale per esporre la ommatidi al centro dell'occhio.
- Continua a cogliere la retina in modo che la cornea è rivolto verso il basso e la lamina è rivolto verso l'alto. Utilizzando un ago di tungsteno fine, staccare il ommatidi dalla lamina e la cornea.
- Singoli ommatidi e gruppi di ommatidi raccoglierà sul fondo del pozzo o diapositiva. Rimuovere eventuali residui che potrebbero essere più grandi raccolte prima di procedere.
3. Trattamento, colorazione e di imaging
- Per analizzare l'autofagia, diluire 1:1000 Lysotracker nel menu Schneider (prima diluita 1:10 in Schneider e poi aggiungere 0,5 microlitri alla goccia sul vetrino), mescolare e attendere 10 secondi.
- Togliere il liquido in eccesso dalla diapositiva, facendo attenzione a lasciarsi alle spalle ommatidi. Questo viene fatto meglio con una punta fine caricamento del gel.
- Aggiungere una goccia di Vectashield per coprire il ommatidi, applicare un vetrino coprioggetti e sigillare con nailpolish.
4. Rappresentante dei risultati:
La buona esecuzione del protocollo dovrebbe lasciare un numero di ommatidi singoli e di piccoli gruppi di ommatidi tenuti insieme da frammenti di lamina ma ben separati dal lato della cornea. Questi possono essere ripreso come in questo esempio per visualizzare Atg8: GFP e Lysotracker, mostrando autophagosomes, lisosomi e autophagolysosomes (Fig. 3). Il metodo può essere utilizzato per l'imaging dal vivo, però in questo caso è da notare che autofluorescenza di media di Schneider renderà difficile distinguere i segnali a bassa intensità fluorescente. Un ulteriore problema è che i granuli di pigmento che rimangono attaccati ai fotorecettori sono intensamente fluorescenti, soprattutto nel canale rosso. Una foto brightfield può essere richiesto per distinguerli dagli organelli genuina.
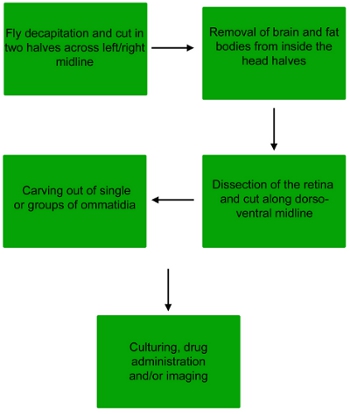
Figura 1. Diagramma di flusso della procedura di dissezione di ottenere ommatidi singoli o piccoli gruppi di ommatidi dalla retina volare.
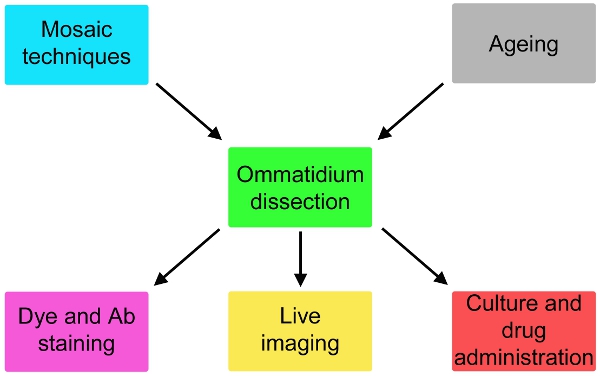
Figura 2. Eventuali variazioni del semplice protocollo qui descritto che coinvolgono la genetica e l'invecchiamento volare a monte della dissezione e colorazione o coltura per un massimo di 24 ore ed a valle della droga amministrazione della dissezione.
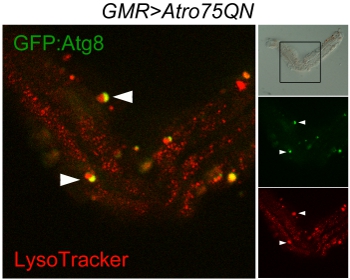
Figura 3 scansione confocale di autophagosomes e lisosomi in un singolo ommatidium sezionato da aw; GMR-GAL4, UAS-Atro75QN; UAS-GFP:.: Atg8 / + volo, esprimendo un poliglutamine Atrophin mutante e dai 12 giorni a 29 ° C. Il pannello di campo chiaro (in alto a destra) mostra la struttura quasi intatta della ommatidium e la cornice indica l'area acquisita. Segni rossi lisosomi, il verde autophagosomes, auto-lisosomi, risultante dalla fusione dei due organelli, di colore giallo. Le frecce indicano due eventi di fusione in corso tra autophagosomes e lisosomi.
Discussione
La dissezione ommatidium singolo presentato qui permette la raccolta di informazioni delle cellule più profonde sui processi biologici come la neurodegenerazione, quando modellato negli occhi Drosophila. I neuroni dei fotorecettori sono più facilmente accessibili di altri neuroni e citoplasma è particolarmente grande, e quindi adatto per studiare la dinamica delle vescicole, nelle cellule stesse utilizzate proficuamente in molti modelli di quantificare neurodegenerazione in vivo. L'aspetto criti...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo Bernard Charroux per le discussioni. Questo lavoro è stato finanziato dalla Scuola LKC biomedica.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
|---|---|---|---|
| Dispenser portatile di CO2 | Flystuff | 59-150 | Ricariche disponibili |
| DUMONT Tweezer n. 5 | AGAR scientifico | T5034 | |
| 3-Bene vetrino | SME | 71561-01 | |
| Micro forbici | VWR | HAMMHSB516-09 | |
| Media Schneider | VWR | 733-1663 | |
| LysoTracker Red DND-99 | Invitrogen | L7528 | |
| Superfrost diapositive | VWR | 631-0108 | |
| Vectashield con DAPI | Vector Labs | H-1200 | |
| Coprioggetto | VWR | 631-1336 |
Riferimenti
- Marsh, J. L., Thompson, L. M. Drosophila in the study of neurodegenerative disease. Neuron. 52, 169-178 (2006).
- Mizushima, N., Levine, B., Cuervo, A. M., Klionsky, D. J. Autophagy fights disease through cellular self-digestion. Nature. 451, 1069-1075 (2008).
- Nisoli, I. Neurodegeneration by polyglutamine Atrophin is not rescued by induction of autophagy. Cell Death Differ. 17, 1577-1587 (2010).
- Charroux, B., Fanto, M. The fine line between waste disposal and recycling: DRPLA fly models illustrate the importance of completing the autophagy cycle for rescuing neurodegeneration. Autophagy. 6, 667-669 (2010).
- Hardie, R. C. Voltage-sensitive potassium channels in Drosophila photoreceptors. J Neurosci. 11, 3079-3095 (1991).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker (MARCM) for Drosophila neural development. Trends in neurosciences. 24, 251-254 (2001).
- Xu, T., Rubin, G. M. Analysis of genetic mosaics in developing and adult Drosophila tissues. Development. 117, 1223-1237 (1993).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. Dev Biol. 351, 128-1234 (2011).
- Napoletano, F. Polyglutamine Atrophin provokes neurodegeneration in Drosophila by repressing fat. The EMBO journal. 30, 945-958 (2011).
- Ravikumar, B. Inhibition of mTOR induces autophagy and reduces toxicity of polyglutamine expansions in fly and mouse models of Huntington disease. Nature. 36, 585-595 (2004).
- Zou, S., Meadows, S., Sharp, L., Jan, L. Y., Jan, Y. N. Genome-wide study of aging and oxidative stress response in Drosophila melanogaster. Proceedings of the National Academy of Sciences of the United States of America. 97, 13726-13731 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon