Method Article
タンデムアフィニティー精製を用いたタンパク質相互作用するパートナーの識別
要約
タンデムアフィニティー精製は、タンパク質の結合パートナーを同定するための堅牢なアプローチである。コンセプトの証明として、この方法論は、翻訳開始に関与する共沈物宿主細胞因子に十分に特徴付けられた翻訳開始因子eIF4Eに適用した。このメソッドは、簡単に任意の細胞またはウイルスのタンパク質に適合されている。
要約
ホストとウイルスタンパク質の機能を理解する上で重要かつしばしば律速段階は、細胞またはウイルスタンパク質パートナー相互作用の識別です。酵母ツーハイブリッドシステムと同様に、組換えタンパク質および質量分析同定続く1内因性タンパク質の免疫沈降を用いたアッセイをプルダウンを含む相互作用パートナーの同定を可能にする多くのアプローチがあります。最近の研究では、相互作用するタンパク質の同定つの特定の溶出ステップで結合し、ダブルアフィニティータグ媒介精製の有用性を強調している。このアプローチでは、と呼ばれるタンデムアフィニティー精製(TAP)は、最初に酵母2,3で使用されていたが、最近哺乳動物細胞で4-8を使用するように適応されています。
概念実証として、私たちは十分に特徴付けられた真核生物の翻訳開始FACを使用してタンデムアフィニティー精製(TAP)法を確立していますTorのeIF4E 9,10。携帯翻訳因子eIF4Eはキャップ依存性翻訳開始10に関与する細胞eIF4F複合体の重要なコンポーネントです。現在の研究で使用されているのTAPタグが2つのプロテインGユニットから構成され、ストレプトアビジン結合ペプチドは、タバコエッチウイルス(TEV)プロテアーゼ切断配列で区切られています。現在の研究で使用されているのTAPタグが2つのプロテインGユニットから構成され、タバコエッチウイルス(TEV)プロテアーゼ切断配列8で区切られたストレプトアビジン結合ペプチドである。クローン細胞株を生成するための必要性をあきらめるために、我々はpMEP4(Invitrogen社)に基づいて、エピソームに保持するプラスミドから、TAPタグ付きベイト蛋白質の発現に依存して迅速なシステムを開発しました。このプラスミドからのタグ付きマウスeIF4Eの発現は、塩化カドミウム誘導性メタロチオネインプロモーターを用いて制御されていました。
Rへの結合を介して発現する細胞とそれに続くアフィニティ精製の溶解abbit IgGアガロース、TEVプロテアーゼ切断は、ストレプトアビジン架橋アガロースに結合して(単独TAPタグを発現する細胞株を制御する場合と比較して)、その後のビオチンの溶出は、プルダウンeIF4Eに明らかに特定の多くのタンパク質を同定した。タンパク質の同定は、1D SDS-PAGEからのバンドの切除とその後のタンデム質量分析により得られた。識別されるコンポーネントは、eIF4Gと4EBP-1の既知のeIF4E結合タンパク質が含まれています。さらに、eIF4EがコンポーネントであるeIF4F複合体の他の成分は、eIF4Aとポリ-結合タンパク質すなわち、同定された。知られている直接の結合パートナーとしてだけでなく、二次相互作用するタンパク質だけでなく、を識別する能力は、さらに、機能未知のタンパク質の特性評価では、このアプローチの有用性を強調しています。
プロトコル
1。細胞株の生成:pMEP4トランスフェクション/式
- pMEP4発現ベクターで細胞をトランスフェクトすると、すべてのモックトランスフェクトした細胞がオフに殺されたまで、ハイグロマイシンB / mlの(Roche)を100μgのを選択します。特定のクローンを単離する必要はありませんので、プラスミドpMEP4はエピソーム維持されます。
- pMEP4ベクターを含む細胞は、16時間、10μMCDCL 2で処理することにより誘導することができる。の発現と誘導性は細胞株の増幅の前に小さな規模で確認されるべきである。プロテインGのドメインは、ほとんどすべての種からの抗体に結合するTAP-tagタンパク質は容易に検出することができます。大規模な精製の ための細胞の一般的に10コンフルエント175cm 2のフラスコが必要となります。これは約2 X10 8を発現する細胞に相当します。
2。細胞ライセートの調製
- PBSに細胞をこすり、単一の50 mlチューブに結合します。 1200倍GFを回転させるまたは5分(これは洗濯後約1.5 mlに減少して起動するにはパックされた細胞は2-3 mlのになる必要があります)。
- 細胞を氷冷PBS(50 MLS各時間)で3回洗浄します。
- 5 mlの溶解バッファーで細胞を溶解液(50mM Tris-HCl(pH7.5)で、125 mM塩化ナトリウム、5%グリセロール、0.2%NP-40、1.5mMのMgCl 2、25mMのNaFを、1mMののNa 3 VO 4及びプロテアーゼ阻害剤。)注:フッ化ナトリウムは、Na 3 VO 4、およびプロテアーゼ阻害剤は、新鮮に追加する必要があります。氷上で5分間出る前に10回ピペッティングします。氷上で5分間氷上で再び出発前に鈍針を用いて注射器を上下に5〜10倍。 syringingを繰り返して、氷上でさらに5分間放置。
- (液体N 2 /またはドライアイスとエタノール)ライセートを二回凍結融解。サンプルは4℃以上に到達することはできません注:処理する時間があるまでは、-80°Cでこのサンプルを保存することができます。
- 1.5 mlチューブに小分けサンプルとunlysed細胞とDを削除遠心分離(4℃、10分°C、16,000 X g)でebris。
- 上清を回収し、15mlファルコンチューブに結合し、(オプションで)タンパク質収量の定量化のために20μlのサンプルを取る前に0.45μmフィルターを通過します。その後の分析(サンプル1)さらに50μlのアリコートを削除します。
3。ウサギIgG-アガロースに結合する
(注:すべてのスピンが特に明記しない限り、1分間、4℃で冷却遠心機で1200倍gで実施する必要があります)
- 静かに瓶を渦巻くことでウサギIgG-アガロース溶液(Sigma-Aldrich社)を懸濁します。 IgGアガロース(カット1ミリリットルピペットチップを使用して)380μlを取り、1分間の遠心分離により出荷/保存液を除去します。明確にするために遠心分離を用いて冷却した溶解バッファーにアガロース三回(4°C)を洗浄します。アガロースの最終収率はパックされたビーズの約250μlのでなければなりません。
- 2.6(15mlファルコン浴槽内から消去細胞ライセートを追加します。e)は4℃で3時間洗浄したウサギ抗IgGアガロースとインキュベート(または一晩)℃まで回転ミキサーを使用します。
- 4°Cで5分間アガロースビーズの回転を停止し、その後の分析(サンプル2)の上清を除去します。注:タンパク質タグ付けされ、TAPは現在、ビーズに関連付ける必要があります。
4。 TEVプロテアーゼ切断
(注:特に断りのない限り、すべてのスピンが、1分間4℃で冷却遠心機で1200倍gで実施する必要があります)
- チルド溶解緩衝液中でウサギIgGアガロースビーズを3回洗浄(4℃)(注:溶解バッファーは、プロテアーゼ阻害剤を含むべきではありません)明確にするために遠心分離を使用します。ケアは洗浄ステップ中にビーズを削除したり、紛失しないように注意する必要があります。
- 明確にするために遠心分離を用いたTEVプロテアーゼ切断緩衝液(10mMトリス-HCl(pH 7.5)を、100mMのNaCl、および0.2%NP-40)でビーズをさらに2回洗浄します。最後の洗浄の後を慎重にすべてのliを削除するビーズから一口分。
- TEVプロテアーゼ切断ミックスを調製する。各サンプルについては、467.5μlのH 2 O、25μlの20倍TEVバッファー(Invitrogen)を、5μlの0.1M DTTおよび2.5μL(25 U)TEVプロテアーゼ(Invitrogen)を含む。パックされたビーズの各サンプルにこのTEV切断ミックス500μlのを追加し、1.7ミリリットルプレ給油管(コスター社)にミックス全体を転送します。インキュベーションの前にその後の分析(サンプル3)30μlのアリコートを削除します。
- 4℃で一晩TEVプロテアーゼ反応をインキュベート°C回転ミキサーを使用します。短いインキュベーション時間は経験的に決定されるべきであるbaitタンパク質とTEVプロテアーゼ切断部位が最小限のインキュベーション時間のアクセシビリティの性質に応じて使用することができないことに注意してください。
5。固定化ストレプトアビジンビーズをプラスULTRALINKに結合
(注:すべてのスピンが特に明記しない限り、1分間、4℃で冷却遠心機で1200倍gで実施する必要があります)
- 1200倍G(4°C)で5分間ウサギIgGアガロースビーズを含むTEV切断反応を遠心します。分析のために明らかにし、上清の20μlのアリコートを取り外します(サンプル4)
- 新鮮1.5ミリリットルプレ給油管(コスター社)に、残りの上清(約480μl)を転送し、氷上でこのままにしておきます。この上清を捨ててはいけない - これはあなたのTEV切断されたbaitタンパク質と関連するすべての結合タンパク質を含んでいます。
- 残りのウサギIgGアガロースビーズを再懸濁してチルドの溶解緩衝液を500μl(2.3節)を追加します。上清を除去し、ステップ5.2からそれと組み合わせる前に、ビーズを明確にするために、この混合物を遠心分離します。 (それは現在のサンプル〜980μlを含む必要があります)氷の上で、このチューブのままにしておきます。
- その後の分析のためにウサギIgGアガロースビーズ(サンプル5)を保持します。注:SDS-PAGEゲル(またはウェスタンブロット)でこのサンプルを実行する場合、それが原因で、重鎖および軽IgGの存在の背景の多くを与えるチェーン。
- 一方、優しくULTRALINKボトルを渦巻くことでストレプトアビジンプラスアガロースビーズ(Pierce)を固定化に再懸濁します。無給油1.5 mlのチューブに、サンプルあたりのストレプトアビジンビーズのアリコートを70μlのエンドカットで200μlのピペットチップを使用して、遠心分離によって出荷/保存液を除去します。注:これらのビーズは非常に小さいので、 "アヒルのくちばし"や平坦である/ナローエンドのヒントは、洗濯中にビーズの損失を最小限にするために使用することができます。
- ストレプトアビジンビーズを明確にするために遠心分離を用いて冷却した溶解用緩衝液で3回(4°C)を洗浄します。これは、チューブあたりパックビーズの約45から50μlを残す必要があります。
- °C回転ミキサーを用いて4℃で3時間(または一晩)洗浄し、ストレプトアビジンビーズとインキュベートを含むチューブにTEVプロテアーゼ切断上清(すなわち、ステップ5.3からのサンプル980μl)を転送します。
- 4℃で5分間ストレプトアビジンビーズの回転を停止し、(その後の分析のために上清を除去サムPLE 6)。ストレプトアビジンビーズを明確にするために遠心分離を用いて冷却した溶解用緩衝液で3回(4°C)を洗浄します。最終洗浄後、慎重に残りのすべての洗浄バッファーを削除します。
6。ストレプトアビジン結合ペプチドとbaitタンパク質のビオチン溶出
(注:すべてのスピンが特に明記しない限り、1分間、4℃で冷却遠心機で1200倍gで実施する必要があります)
- ビオチン(1 mMのD-ビオチン)、回転ミキサーを使用して3時間(または一晩)を4℃でインキュベートした:PBS 500μLを加えることにより、ストレプトアビジンビーズからのHisタグ融合タンパク質を溶出します。
- 4℃で5分間ストレプトアビジンビーズの回転を停止し、1.5ミリリットルプレ給油管(注に上清を除去:これはあなたの最終的なサンプル/溶出(サンプル7)です。
- 残りのストレプトアビジンビーズをPBS、回転ミキサーを使用して2-3時間(または一晩)を4℃でインキュベートする:別の500μlのビオチンを追加します。
- 投稿泥炭は、ステップ6.2および2溶出(サンプル7)を組み合わせる。この最後の溶出は-80℃で保存することができます
- 、ビオチン溶出の効率性を評価し、残りのストレプトアビジンビーズを凍結し、SDSサンプルバッファー(サンプル8)(推奨:フィッシャーからLaneMarker 5倍削減するサンプル緩衝液)の後で沸騰します。
7。タンパク質濃度
- 最後の溶出液(サンプル7)が低いため、タンパク質濃度と相対的に高いボリュームの分析の前に集中する必要があります。これは、低分子量(<5 kDa)のVivaspinスピンカラム(Vivascience)を使用して達成することができます。未満100μlに音量を下げ集中することを目指しています。この最後のサンプルは、煮沸し、タンパク質試料バッファー(推奨:フィッシャーからLaneMarker 5倍削減するサンプル·バッファ)に格納することができます。
8。分析
- サンプル1-8プルダウンの効率を決定するためにウェスタンブロットで分析することができます。注:する最も二次抗体融合タンパク質のプロテインGドメインと交差反応するが、これは、TEVプロテアーゼ切断後に削除されます。
- サンプル7は、1Dまたは2D SDS-PAGEで分析することができます。染色は銀またはクマシー(:SilverQuest銀染色およびInvitrogenからコロイダルブルークマシー染色キット推奨)を使用して実行することができます。タンパク質同定は質量分析を用いて行うことができる。
9。代表的な結果
TAPタグ付けeIF4Eの結合パートナーを識別するには、このプロトコルからの最終的な溶出(サンプル7)の1D SDS-PAGE分析の例を図2に記載されています。この代表的なゲルは、セル内の他のタンパク質とeIF4E相互作用の複雑で豊かな自然を反映しています。また、唯一のTAPタグを発現する細胞株から生成され、図2に示すように、陰性対照との比較は、プルダウン餌このeIF4Eの特異性を示しています。このインスタンス内で濃縮された最終的な溶出の15%(サンプル7)はInvitrogenからSilverquest銀染色キットを用いて染色される前に、市販のプレキャスト勾配ゲルを使用して、1D SDS-PAGEにより分析した。
一般的に残りの濃縮された最終的な溶出の百分の50から85(サンプル7)染色コロイドクマシー(Invitrogen社製)で分析されています。全体の車線からのサンプル(ゲルスライス/バンド)を質量分析によって抽出されたと分析した。
プルダウンeIF4Eに同定されたタンパク質は、非特異的結合パートナーを識別するために、ネガティブコントロールから結合パートナーに対して、ろ過した。 TAPタグ付けeIF4Eのこのプロセスを用いて同定し、最終的なタンパク質は、この手法を用いて同定し、通常のタンパク質の代表である、図2に見ることができます。さらに、eIF3サブユニットの数も(データは示さず)が同定された。
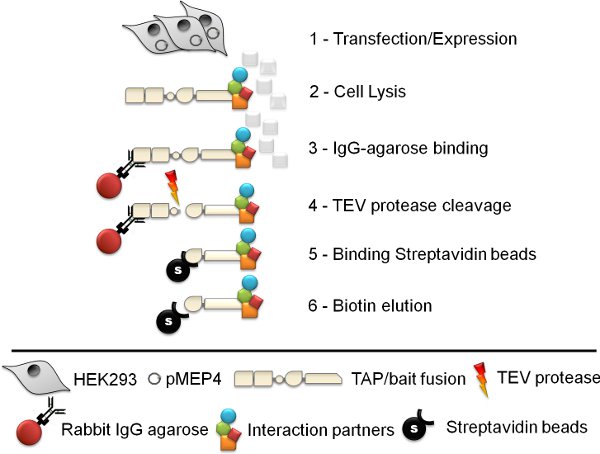
図1。の模式図タンデムアフィニティー精製の 手順は6段階のタンデムアフィニティー精製(TAP)プロトコルは、細胞株の生成、細胞溶解、ウサギIgGアガロース免疫沈降、TEVプロテアーゼ切断、ストレプトアビジンビーズアフィニティ精製し、最後にビオチン溶出が含まれます。
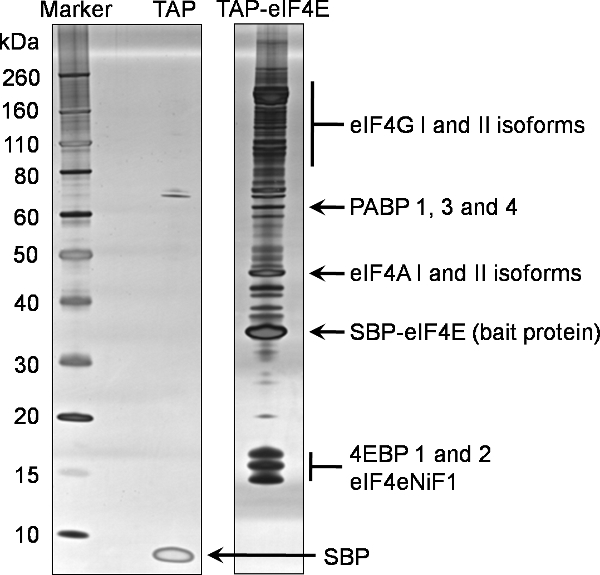
図2。マウスeIF4Eタンパク質のタンデムアフィニティー精製、Nの対話パートナー末端タグ付きのeIF4Eのタップが接続されたプロトコルを使用して、真核HEK293細胞から精製した。最後の溶出(サンプル7)の20%の画分は、プレキャスト4-12%勾配ゲル(TAP-eIF4Eレーン)をSDS-PAGEにより分析した。同等の分析が単独でタグ(TAPレーン)を行った。タンパク質は銀染色を用いて同定した。その後、同じ試料の質量分析法によって同定されたタンパク質は、このゲル上で強調表示されます。
略語:eIF4G、真核生物の翻訳開始F俳優4ガンマ、PABP、ポリA結合タンパク質、eIF4A:真核生物翻訳開始因子4α、SBP-eIF4e、真核生物翻訳開始因子4E結合、真核生物翻訳開始因子4Eに融合し、4EBPsストレプトアビジン結合ペプチドを含む、残りのTAPのbaitタンパク質タンパク質、eIF4eNiF1l真核生物翻訳開始因子4E核インポート因子1、SBP、TEV融合していないTAPのペプチドから残りのストレプトアビジン結合ペプチド。
ディスカッション
ここに示すTAPタギング技術は、真核細胞内でベイトタンパク質の結合パートナーを単離するための高度に特異的かつ厳格な方法を示しています。このアプローチは、細胞とウイルス両方のタンパク質に適用することができます。我々の知る限り、このような手法は翻訳開始因子eIF4Eに適用されたのは今回が初めてです。このテクニックを使用して既知のeIF4E結合タンパク質のeIF4Gと4EBPsの同定は、このようなアプローチの有効性を確認します。さらに、eIF4F複合体の残りのコンポーネントの識別、すなわちeIF4Aと、PABPは、間接的な相互作用および第三複合体が精製プロセス中にそのまま残っていることを確認します。標準的なeIF4e結合タンパク質の複数のアイソフォームの同定にも明らかであった。これらは、図2で詳細に説明されています。
アプローチの限界に関しては、一定の注意が選択肢に関しては注意が必要ですbaitタンパク質およびN末端またはC末端タグtagを配置するか否かを判定する。それは機能的アッセイを実施またはタグ誘導体の機能であることを確認する前に、フルスケールの精製に顕微鏡により融合タンパク質の局在を調べることをお勧めかもしれません。内在性膜や核タンパク質は必ずしも溶解ステップで説明されて相対的に中性洗剤を条件によって解放されない場合があります。すべての免疫沈降と同様のプルダウンアッセイと同様に、変更は溶解の効率を高めるために溶解バッファー中で自然と洗剤の濃度に作ることができる。溶解および洗浄バッファーのイオン濃度はまた、精製のストリンジェンシーを増加または減少するように変更されることがあります。それは、標準的な温和な条件(上記)で初期精製を行うことも可能ですが、その後に続いて増加するイオンの消費で洗浄することができます4月5日のアリコートにストレプトアビジンビーズ(セクション5.8)、分割ditions。これにより、ユーザーは、非特定の相互作用するタンパク質を除去する最適な条件を識別するために有効にすることができます。全体のプロセスは、相互作用パートナーが知られており、それらの有無だけで、その後のウェスタンブロットによって決定される一過性トランスフェクションを使用して実行することができます。このケースでは、発現プラスミド8μgのトランスフェクションした細胞の2 100センチメートル2皿ずつお勧めします。既知の結合パートナーと相互作用するTAP-融合タンパク質の変異体の誘導体の能力を調べるときにこの変更されたプロシージャは、特定の使用である。
テクニックは許容可能な結果を生成するために失敗した銀染色SDS-PAGEゲル(またはウェスタンブロットによる抗体が使用可能な場合)に8を介してサンプル1を分析することで、トラブルシューティングの優れた方法です。それぞれの親和性に基づく降水量と特定の溶出の効率が、この体系的なサンプリング手法を用いて分析することができます。サンプル八から一はoptim中に撮影されるべきそれぞれの新しい餌のためのプロトコルの化。
TAPタグ付け技術は、GSTのような他のアプローチはプルダウンなどの二重アフィニティー精製および特定の溶出ステップ(TEV切断部位とビオチン溶出)で維持する特異性と厳しさを提供して酵母ツーハイブリッドアッセイをに強力かつ堅牢な代替手段を提供精製工程を通して高いレベル。批判的にこの手法は、任意のタンパク質に適用することができるので、目的のタンパク質ターゲットの結合パートナーを同定するための優れた方法を表しています。
開示事項
利害の衝突が宣言されません。
謝辞
本研究では、博士イアングッドフェローに授与ウェルカムトラストシニア·フェローシップによって資金を供給された。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
| ハイグロマイシンB | ロシュ社 | 10843555001 | |
| ウサギIgGアガロース | シグマ | A2909 | |
| AC-TEVプロテアーゼ | インビトロジェン | 12575-015 | |
| プロテアーゼ阻害剤カクテル | カルビオケム | 539134 | |
| UltraLinkは、ストレプトアビジンプラスビーズ固定化 | ピアース | 53116 | |
| Vivaspin 500遠心濃縮機(5KDa) | Vivaspin | V50112 | |
| SilverQuest銀染色キット | インビトロジェン | LC6070 | |
| NOVEXコロイドブルークマシー染色キット | インビトロジェン | LC6025 | |
| 1.7ミリリットル封入管 | 共演者 | 3207 | |
| マイクロキャピラリーピペットチップ | VWR | 37001-150 | |
| NuPage 4から12パーセントビス - トリス勾配ゲル | インビトロジェン | NP0322BOX | |
| CDCL 2 | シグマ | 202908 | |
| 5X SDSサンプルバッファー | フィッシャー | PN39000 |
参考文献
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem. Soc. Trans. 38 (4), 875-878 (2010).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17 (10), 1030-1032 (1999).
- Blackwell, C., Brown, J. D. The application of tandem-affinity purification to Candida albicans. Methods Mol. Biol. 499, 133-148 (2009).
- Cochrane, A. Stable complex formation between HIV Rev and the nucleosome assembly protein, NAP1, affects Rev function. Virology. 388 (1), 103-111 (2009).
- Fernandez, E. Targeted tandem affinity purification of PSD-95 recovers core postsynaptic complexes and schizophrenia susceptibility proteins. Mol. Syst. Biol. 5, 269-269 (2009).
- Gloeckner, C. J. Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag. Methods Mol. Biol. 564, 359-372 (2009).
- Holowaty, M. N. Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7. J. Biol. Chem. 278 (32), 29987-29994 (2003).
- Burckstummer, T. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3 (12), 1013-1019 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4 E. EMBO Rep. 6 (10), 968-972 (2005).
- Goodfellow, I. G., Roberts, L. O. Eukaryotic initiation factor 4E. Int. J. Biochem. Cell Biol. 40 (12), 2675-2680 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved