Method Article
Identificação de parceiros de proteína Interagir utilizando Tandem purificação por afinidade
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Tandem purificação por afinidade é uma abordagem robusta para a identificação de parceiros de ligação às proteínas. Como prova de conceito, este método foi aplicado ao bem caracterizado eIF4E factor de iniciação da tradução para o co-precipitado os factores de células hospedeiras envolvido na iniciação da tradução. Este método é facilmente adaptado a qualquer proteína celular ou virai.
Resumo
Um passo crítico e, muitas vezes limitante na compreensão da função do hospedeiro e proteínas virais é a identificação de interagir parceiros de proteínas celulares ou virais. Existem muitas abordagens que permitem a identificação de parceiros que interagem, incluindo o sistema de levedura duas híbrido, bem como puxar para baixo, utilizando ensaios de proteínas recombinantes e imunoprecipitação de proteínas endógenas seguidos por espectrometria de massa de identificação 1. Estudos recentes revelaram a utilidade de duplo afinidade purificação mediada tag, juntamente com dois passos de eluição específicos na identificação de proteínas que interagem. Esta abordagem, denominado Tandem purificação por afinidade (TAP), foi inicialmente utilizado em levedura 2,3 mas mais recentemente foi adaptado para usar em células de mamíferos 4-8.
Como prova de conceito, criámos uma purificação por afinidade tandem (TAP) usando o método do bem caracterizada eucariótico de iniciação da tradução factor eIF4E 9,10. O eIF4E fator celular de tradução é um componente crítico do complexo eIF4F celular envolvido na cap-dependente de início da tradução 10. A etiqueta de TAP utilizado no presente estudo é composto de duas unidades de proteína G e um péptido de ligação a estreptavidina separados por um vírus de Tabaco Etch (TEV) sequência de clivagem de protease. A etiqueta de TAP utilizado no presente estudo é composto de duas unidades de proteína G e um péptido de ligação a estreptavidina separados por um vírus de Tabaco Etch (TEV) a sequência de clivagem de protease 8. Para renunciar a necessidade para a geração de linhas de células clonais, foi desenvolvido um sistema rápido que depende da expressão da proteína TAP-etiquetados com isco de um plasmídeo episomally mantida com base em pMEP4 (Invitrogen). Expressão de etiquetados eIF4E murino a partir deste plasmídeo foi controlado usando o cádmio cloreto de promotor de metalotioneina indutível.
Lise das células que expressam e subsequente purificação por afinidade através de ligação a rabbit IgG agarose, TEV clivagem protease, a ligação a estreptavidina agarose ligada e eluição biotina subseqüente identificaram proteínas numerosas aparentemente específicos para o eIF4E pull-down (quando comparado ao grupo controle linhas de células que expressam a marca TAP sozinho). As identidades das proteínas foram obtidos por excisão das bandas de espectrometria de massa de SDS-PAGE e subsequente em tandem 1D. Os componentes identificados incluído as proteínas de ligação conhecidos eIF4E eIF4G e 4EBP-1. Além disso, outros componentes do complexo eIF4F, dos quais eIF4E é um componente foram identificados, ou seja, eIF4A e Poly-A proteína de ligação. A capacidade de identificar não apenas conhecidos diretos parceiros de ligação, bem como secundárias proteínas interagem entre si, acentua ainda mais a utilidade desta abordagem para a caracterização de proteínas de função desconhecida.
Protocolo
1. Geração de linhas celulares: pMEP4 transfecção / expressão
- Transfectar células com o vetor de expressão pMEP4 e selecione com 100 mg de higromicina B / ml (Roche), até que todas as células transfectadas simulados foram mortos fora. O plasmídeo pMEP4 é mantida episomally portanto, não há necessidade de isolar clones específicos.
- As células contendo o vector pMEP4 pode ser induzida por tratamento com 10 uM CdCl 2 durante 16 horas. Expressão e indutibilidade deve ser confirmado, em pequena escala antes da amplificação das linhas de células. A proteína TAP-etiquetados pode ser prontamente detectada como os domínios de proteína G se ligam a anticorpos a partir de quase todas as espécies. Para grande escala purificações normalmente 10 confluentes 175 cm 2 frascos de células são necessários. Isso equivale a cerca de 2 X10 8 células que expressam.
2. Preparação lisado celular
- Raspe as células em PBS e combinam em um único tubo ml 50. Spin 1200X gfou 5 minutos (isto deve resultar em 2-3 ml de eritrócitos para começar, o que reduz a cerca de 1,5 ml, após lavagem).
- Lave as células três vezes em PBS gelado (50 ml de cada vez).
- Lisar as células em 5 ml de tampão de lise (50 mM Tris-HCl (pH 7,5), NaCl 125 mM, glicerol 5%, 0,2% de NP-40, 1,5 mM de MgCl2, 25 mM NaF, 1 mM de Na 3 VO 4 e inibidor de protease ). Nota: NaF, Na 3 VO 4 e inibidores da protease devem ser adicionados fresco. Pipetar cima e para baixo 10 vezes antes de sair durante 5 minutos em gelo. Seringas cima e para baixo 5-10 vezes usando uma agulha romba antes de sair novamente em gelo durante 5 minutos em gelo. Repita syringing e deixar durante mais 5 minutos em gelo.
- Freeze-descongelar o lisado duas vezes (gelo N2 líquido / ou seca e etanol). Não permitir amostra para alcançar uma maior do que 4 ° C. Nota: você pode armazenar esta amostra a -80 ° C até que você tenha tempo para processar.
- Alíquota de amostra em tubos de 1,5 ml e remover as células unlysed edebris por centrifugação (10 minutos a 4 ° C, 16.000 X g).
- Recuperar o sobrenadante, combinar num tubo Falcon 15 ml e (opcionalmente) passar através de um filtro de 0,45 antes de tomar uma amostra de 20 uL para a quantificação produção de proteína. Remover uma alíquota de 50 ul adicionais para análise subsequente (Amostra 1).
3. A ligação a IgG de coelho-agarose
(Nota: Todos os spins deve ser realizado a 1200X g numa centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que indicado de outra forma)
- Suavemente ressuspender o IgG de coelho-agarose solução (Sigma-Aldrich) por agitação do frasco. Tome 380 uL de IgG de agarose (usando um corte ponta de pipeta de 1 ml) e remover o transporte / solução conservante por centrifugação durante 1 minuto. Lavar os agarose três vezes em tampão de lise refrigerados (4 ° C) usando centrifugação para clarificar. O rendimento final de agarose deve ser de cerca de 250 uL de pérolas de embalados.
- Adicionar o lisado celular eliminado 2,6 (em uma cuba 15 ml Falcone) para o lavada-coelho IgG de agarose e incubar durante 3 horas (ou durante a noite) a 4 ° C usando um misturador rotativo.
- Girar para baixo as esferas de agarose durante 5 minutos a 4 ° C e remover o sobrenadante para análise subsequente (Amostra 2). Nota: O TAP etiquetados com proteína deve agora ser associada com as esferas.
4. TEV clivagem protease
(Nota: Todos os spins deve ser realizado a 1200X g numa centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que indicado de outra forma)
- Lavar os grânulos de coelho IgG de agarose três vezes em tampão de lise refrigerados (4 ° C) usando centrifugação para clarificar (nota: o tampão de lise não deve conter inibidores de protease). Cuidados devem ser tomados para não remover ou perder todas as pérolas durante as etapas de lavagem.
- Lavar os grânulos mais duas vezes, com TEV protease-tampão de clivagem (10 mM Tris-HCl (pH 7,5), NaCl 100 mM, e 0,2% de NP-40) usando a centrifugação para clarificar. Após a última lavagem cuidadosamente remover todo o liquid a partir dos grânulos.
- Prepara-se uma mistura de clivagem de TEV protease; para cada amostra incluem 467,5 uL de H2O, 25 uL de 20x tampão TEV (Invitrogen), 5 ul 0,1 M DTT e 2,5 ul (25 U) TEV Protease (Invitrogen). Adicionar 500 uL desta mistura de clivagem de TEV para cada amostra de grânulos compactados e transferir toda a mistura para um tubo de 1,7 ml de pré-lubrificado (Costar). Antes da incubação remover uma alíquota 30 ul para análise subsequente (Amostra 3).
- Incubar a reacção protease de TEV durante a noite a 4 ° C utilizando um misturador rotativo. Note-se que tempos mais curtos incubações podem ser utilizadas dependendo da natureza da proteína isco e da acessibilidade do sítio de clivagem da protease de TEV, mas o tempo de incubação mínima deve ser determinado empiricamente.
5. A ligação às ULTRALINK estreptavidina imobilizada Além disso contas
(Nota: Todos os spins deve ser realizado a 1200X g numa centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que indicado de outra forma)
- Centrifugar a reacção de clivagem de TEV contendo os grânulos de coelho IgG de agarose durante 5 minutos a 1200X g (4 ° C). Remover uma alíquota de 20 ul do sobrenadante clarificado para análise (Exemplo 4)
- Transferir o sobrenadante remanescente (cerca de 480 uL) a uma ml 1,5 fresca pré-lubrificado tubo (Costar) e deixar em gelo. Não jogue fora este sobrenadante - contém sua proteína isca TEV clivados e as proteínas associadas vinculativos.
- Adicionar 500 ul de tampão de lise gelada (Seção 2.3) para os restantes coelho IgG contas de agarose e ressuspender. Centrifugar esta mistura a clarificar as esferas antes de remover o sobrenadante e combinando-o com que a partir do passo 5.2. Deixar este tubo em gelo (deve conter agora ~ ul de amostra 980).
- Guarde as contas de IgG de coelho de agarose para análise posterior (amostra 5). Nota: se executar esta amostra num gel de SDS-PAGE (ou Western blot) que irá dar um lote de fundo devido à presença da cadeia pesada de IgG e de luzcadeias.
- Enquanto isso delicadamente ressuspender o Ultralink Imobilizado contas estreptavidina Plus agarose (Pierce) agitando o frasco. Usando uma ponteira 200 ul com o corte final, alíquota de 70 ul de contas estreptavidina por amostra em pré-lubrificadas tubos de 1,5 ml e remover transporte / solução conservante por centrifugação. Nota: estas contas são muito pequenas e por isso "duck-billed" ou apartamento / narrow-ended dicas podem ser usadas para minimizar a perda de talão durante a lavagem.
- Lave as esferas de estreptavidina três vezes em tampão de lise frio (4 ° C) usando centrifugação para clarificar. Isso deve deixar cerca de 45-50 ul de contas embalados por tubo.
- Transferir o sobrenadante de clivagem de protease-TEV (isto é, o uL de amostra 980 a partir do passo 5.3) para o tubo contendo as esferas de estreptavidina lavadas e incubar durante 3 horas (ou durante a noite) a 4 ° C usando um misturador rotativo.
- Girar para baixo as esferas de estreptavidina durante 5 minutos a 4 ° C e remover o sobrenadante para análise subsequente (Samplo 6). Lave as esferas de estreptavidina três vezes em tampão de lise frio (4 ° C) usando centrifugação para clarificar. Após a lavagem final, cuidadosamente remover todo o tampão de lavagem restante.
6. Biotina eluição do péptido de ligação a estreptavidina e proteína isco
(Nota: Todos os spins deve ser realizado a 1200X g numa centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que indicado de outra forma)
- Eluir proteínas marcadas a partir das pérolas de estreptavidina por adição de 500 ul de PBS: Biotina (1 mM D-biotina) e incubando a 4 ° C durante 3 horas (ou durante a noite), utilizando um misturador rotativo.
- Girar para baixo as esferas de estreptavidina durante 5 minutos a 4 ° C e remover o sobrenadante para um tubo de 1,5 ml de pré-lubrificado (Nota: esta é a sua amostra final / eluição (Exemplo 7).
- Adicionar outra Biotina 500 uL: PBS para as esferas de estreptavidina restantes e incubar a 4 ° C durante 2-3 horas (ou durante a noite), utilizando um misturador rotativo.
- Returfa passo 6,2 e combinar as duas eluições (Exemplo 7). Este eluição final pode ser armazenado a -80 ° C.
- Para avaliar a eficiência da eluição biotina, congelar as esferas de estreptavidina restantes e ferver mais tarde, em tampão de amostra SDS (Exemplo 8) (recomendações: LaneMarker tampão de amostra de 5x reduzindo a partir de Fisher).
7. A concentração de proteína
- O eluato final (Amostra 7) tem de ser concentrada antes da análise por causa da concentração de proteína de baixo e de volume relativamente elevado. Isto pode conseguido usando baixo peso molecular (<5 kDa) colunas spin Vivaspin (Vivascience). Destinam-se a concentrar-se o volume até menos de 100 uL. Esta amostra final pode ser fervido e armazenado em tampão de amostra de proteína (recomendações: LaneMarker tampão de amostra de 5x reduzindo a partir de Fisher).
8. Análise
- As amostras 1-8 pode ser analisada por Western blot para determinar a eficiência de puxar para baixo. Nota: A maioria dos anticorpos secundários vontadereagem de forma cruzada com os domínios da proteína G da proteína de fusão, mas este é removido após a clivagem protease de TEV.
- Exemplo 7 pode ser analisada por 1D e 2D SDS-PAGE. Coloração pode ser realizada usando prata ou de Coomassie (recomendações: SilverQuest coloração de prata e coloidais Blue kits coloração com Coomassie de Invitrogen). Identificação de proteínas pode ser realizada utilizando espectrometria de massa.
9. Os resultados representativos
Um exemplo de análise de SDS-PAGE 1D da eluição final (Amostra 7) a partir de este protocolo para identificar parceiros de ligação de eIF4E TAP etiquetados é fornecido na Figura 2. Este gel representante reflecte a natureza complexa e abundante de interacções com outras proteínas eIF4E na célula. A comparação com o controlo negativo também mostrada na Figura 2, gerada a partir de uma linha celular expressando somente a marca TAP, ilustra a especificidade deste eIF4E iscado pull-down. Neste exemplo 15% da eluição concentrado final (Amostra7) foi analisada por 1D SDS-PAGE utilizando disponíveis comercialmente géis pré-moldados de gradiente antes de serem coradas utilizando o kit de coloração de prata Silverquest a partir de Invitrogen.
Tipicamente 50-85% da eluição concentrado restante final (Amostra 7) é analisado com Coomassie coloidal mancha (Invitrogen). As amostras (fatias de gel / bandas) a partir da pista inteira foram então extraídos e analisados por espectrometria de massa.
Proteínas identificadas no eIF4E pull-down foram filtrados contra os parceiros de ligação a partir do controlo negativo para identificar não-específicos parceiros de ligação. As proteínas finais identificados usando este processo de etiquetagem eIF4E TAP pode ser visto na Figura 2, que é representativo das proteínas normais identificados usando esta técnica. Além disso, um certo número de subunidades eIF3 também foram identificados (dados não mostrados).
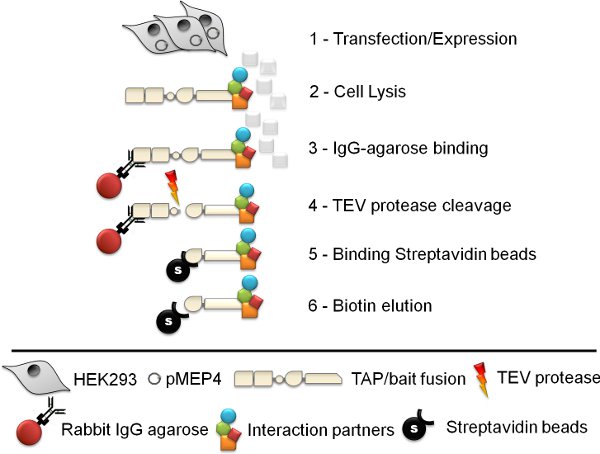
Figura 1. Esquemática doParalelamente processo de purificação de afinidade. O conjunto de seis passos afinidade purificação protocolo (TAP) envolve a geração linha celular, a lise celular, coelho IgG agarose imuno-precipitação, TEV clivagem protease, estreptavidina purificação por afinidade talão e eluição finalmente biotina.
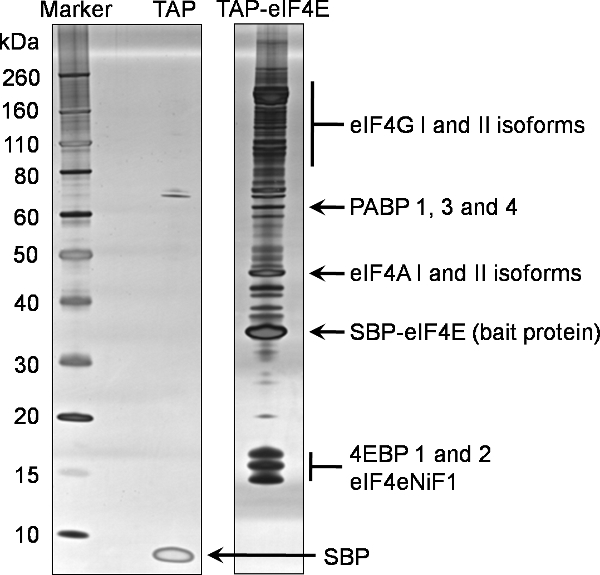
Figura 2. Tandem purificação por afinidade da proteína de murino eIF4E. Interagindo parceiros do N terminal TAP etiquetados eIF4E foram purificados a partir de células HEK293 eucarióticas, utilizando o protocolo em anexo. Uma fracção de 20% da eluição final (Amostra 7) foi analisado por SDS-PAGE num gel de gradiente pré-fundido 4-12% (TAP-eIF4E pista). Uma análise foi realizada equivalente para a marca de sozinho (pista TAP). As proteínas foram identificadas usando coloração com prata. Proteínas subsequentemente identificadas por espectrometria de massa da mesma amostra são destacadas sobre este gel.
Abreviaturas: eIF4G; eucariótico de iniciação da tradução fgama ator 4, PABP; polyA proteína de ligação, eIF4A: fator de iniciação eucariótico tradução 4 alpha, SBP-eIF4E; a proteína isca restante TAP contendo o peptídeo estreptavidina ligação fundida ao eucariótica 4E fator de iniciação da tradução, 4EBPs; tradução eucariótica 4E fator de iniciação de ligação proteínas, eIF4eNiF1l eucariótico de iniciação da tradução fator 4E fator de importação nuclear 1, SBP, o peptídeo estreptavidina restante ligação do peptídeo TAP TEV não fundido.
Discussão
A técnica de marcação TAP ilustrado aqui demonstra um método altamente específico e rigoroso para isolar os parceiros de ligação de proteínas isca em células eucarióticas. Esta abordagem pode ser aplicada a ambas as proteínas celulares e virais. Para o nosso conhecimento, este é a primeira vez que uma tal técnica tem sido aplicada à eIF4E factor de iniciação da tradução. Identificação do conhecido eIF4G proteínas eIF4E vinculativa e os 4EBPs usando esta técnica confirma a validade de tal abordagem. Além disso, a identificação do componente restante do complexo eIF4F, a saber, eIF4A, e PABP confirma que a interacção indirecta e complexos terciárias permanecer intacto durante o processo de purificação. A identificação de múltiplas isoformas dos canónicos eIF4E proteínas de ligação também era evidente. Estes são descritos em mais detalhe na Figura 2.
Com relação às limitações da abordagem, certos cuidados devem ser tomadas medidas em relação à escolhade proteína isco e se ou não a colocar a etiqueta de TAG na extremidade N-ou C-terminal. Pode ser aconselhável realizar um ensaio funcional ou examinar a localização da proteína de fusão por microscopia antes da purificação escala completa para assegurar o derivado marcado é funcional. Membrana Integral ou proteínas nucleares não podem necessariamente ser liberado pelas condições relativamente suaves detergente descritos na etapa de lise. Como com todas as imunoprecipitações e semelhantes pull-down ensaios, as modificações podem ser feitas com a natureza e as concentrações do detergente no tampão de lise para aumentar a eficiência de lise. A concentração iónica dos buffers de lise e de lavagem pode também ser modificado para aumentar ou diminuir o rigor da purificação. É também possível realizar a purificação inicial sob condições padrão suaves (descrito acima), mas posteriormente dividir as esferas de estreptavidina (secção 5.8) em alíquotas 4-5, que podem posteriormente ser lavados sob crescente con iónicoções. Isso pode permitir que o usuário a identificar as condições ideais que removem não-específicas proteínas que interagem. Todo o processo pode também ser realizada usando transfecção transiente em que os parceiros que interagem são conhecidos ea sua presença ou ausência determinada por Western blot subsequente apenas. Neste caso, sugiro dois 100cm 2 pratos de cada células transfectadas com 8 mg de plasmídeo de expressão. Este procedimento modificado é de particular utilidade quando se examina a capacidade de derivados de mutantes da proteína de fusão TAP-para interagir com parceiros de ligação conhecidos.
Análise de amostras de 1 a 8 em corado com prata de SDS-PAGE (gel ou por western blot se um anticorpo está disponível) é um excelente meio de resolução de problemas, se a técnica de falhar para gerar os resultados aceitáveis. A eficiência de cada precipitação afinidade baseada e eluição específico podem ser analisados usando esta abordagem sistemática de amostragem. As amostras de um a oito deve ser tomado durante optimização do protocolo para cada nova isca.
A técnica de marcação TAP fornece uma alternativa poderosa e robusta para outras abordagens, tais como GST pull-baixos, de levedura dois ensaios híbridos etc A purificação por afinidade dupla e os passos de eluição específicos (de clivagem de TEV e eluição biotina) fornecer especificidade e rigor que ser mantido a uma elevado nível durante todo o procedimento de purificação. Criticamente esta técnica pode ser aplicada a qualquer proteína e, portanto, representa um excelente método para identificar parceiros de ligação para um alvo da proteína de interesse.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Esta pesquisa foi financiada por uma bolsa Wellcome Senior confiança atribuído ao Dr. Ian Goodfellow.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| Higromicina B | Roche | 10843555001 | |
| IgG de coelho Agarose | Sigma | A2909 | |
| AC-TEV Protease | Invitrogen | 12575-015 | |
| Cocktail inibidor de protease | Calbiochem | 539134 | |
| Ultralink Imobilizado contas estreptavidina Plus | Perfurar | 53116 | |
| Vivaspin concentrador de 500 centrífugas (5KDa) | Vivaspin | V50112 | |
| SilverQuest kit coloração com prata | Invitrogen | LC6070 | |
| Novex coloidal Azul kit de coloração de Coomassie | Invitrogen | LC6025 | |
| 1.7ml tubos previamente lubrificados | Costar | 3207 | |
| Microcapilar ponteiras | VWR | 37001-150 | |
| NuPage 4-12% gradiente de Bis-Tris géis | Invitrogen | NP0322BOX | |
| CdCl 2 | Sigma | 202908 | |
| Tampão de amostra SDS 5x | Pescador | PN39000 |
Referências
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem. Soc. Trans. 38 (4), 875-878 (2010).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17 (10), 1030-1032 (1999).
- Blackwell, C., Brown, J. D. The application of tandem-affinity purification to Candida albicans. Methods Mol. Biol. 499, 133-148 (2009).
- Cochrane, A. Stable complex formation between HIV Rev and the nucleosome assembly protein, NAP1, affects Rev function. Virology. 388 (1), 103-111 (2009).
- Fernandez, E. Targeted tandem affinity purification of PSD-95 recovers core postsynaptic complexes and schizophrenia susceptibility proteins. Mol. Syst. Biol. 5, 269-269 (2009).
- Gloeckner, C. J. Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag. Methods Mol. Biol. 564, 359-372 (2009).
- Holowaty, M. N. Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7. J. Biol. Chem. 278 (32), 29987-29994 (2003).
- Burckstummer, T. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3 (12), 1013-1019 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4 E. EMBO Rep. 6 (10), 968-972 (2005).
- Goodfellow, I. G., Roberts, L. O. Eukaryotic initiation factor 4E. Int. J. Biochem. Cell Biol. 40 (12), 2675-2680 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados