Method Article
Identification des partenaires protéine interagissant Utilisation Tandem Affinity Purification
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Tandem Affinity Purification est une approche robuste pour l'identification de partenaires de liaison aux protéines. Comme preuve de concept, cette méthodologie a été appliquée à la eIF4E facteur d'initiation de traduction bien caractérisé à la co-précipité les facteurs cellulaires de l'hôte impliqués dans l'initiation de la traduction. Cette méthode est facilement adaptable à n'importe quelle protéine cellulaire ou virale.
Résumé
Une étape cruciale et souvent un facteur limitant dans la compréhension de la fonction de l'hôte et des protéines virales est l'identification des partenaires d'interaction des protéines cellulaires ou virales. Il existe plusieurs approches qui permettent l'identification des partenaires d'interaction, y compris la levure système à deux hybrides, ainsi que déroulez tests utilisant des protéines recombinantes et d'immunoprécipitation de protéines endogènes suivies par spectrométrie de masse d'identification 1. Des études récentes ont mis en évidence l'utilité de la double-purification par affinité médiation tag, couplé avec deux étapes d'élution spécifiques à l'identification des protéines qui interagissent. Cette approche, appelée Tandem Affinity Purification (TAP), a été initialement utilisée dans la levure 2,3, mais, plus récemment, a été adapté pour utiliser des cellules de mammifères 4-8.
Comme preuve de concept que nous avons mis en place un purification par affinité en tandem (TAP) en utilisant la méthode du bien caractérisé eucaryote initiation de la traduction facteur eIF4E 9,10. Le cellulaire de traduction eIF4E facteur est un élément essentiel du complexe eIF4F cellulaire impliquée dans cap-dépendante initiation de la traduction 10. La balise TAP utilisée dans l'étude actuelle est composée de deux unités de protéine G et un peptide de liaison streptavidine séparés par un virus etch du tabac (TEV) séquence de clivage de la protéase. La balise TAP utilisée dans l'étude actuelle est composée de deux unités de protéine G et un peptide de liaison streptavidine séparés par un virus etch du tabac (TEV) séquence de clivage de protéase 8. Pour renoncer à la nécessité pour la génération de lignées cellulaires clonales, nous avons développé un système rapide qui s'appuie sur l'expression de la protéine marquée par TAP-appâts à partir d'un plasmide épisomique maintenue sur la base pMEP4 (Invitrogen). Expression de eIF4E murin étiqueté à partir de ce plasmide a été contrôlé à l'aide du chlorure de cadmium métallothionéine promoteur inductible.
La lyse des cellules exprimant et purification par affinité ultérieure via la liaison à rabbit IgG agarose, clivage de la protéase TEV, la liaison à la streptavidine liée agarose et l'élution ultérieure biotine identifié de nombreuses protéines apparemment spécifiques à l'eIF4E déroulant (par rapport à contrôler des lignées cellulaires exprimant le tag TAP seule). Les identités des protéines ont été obtenues par l'excision des bandes de 1D spectrométrie de masse en tandem SDS-PAGE et les suivantes. Les composants identifiés comprenait les protéines eIF4E connus contraignantes eIF4G et 4EBP-1. En outre, d'autres composants du complexe eIF4F, dont eIF4E est un composant ont été identifiés, à savoir eIF4A et poly-Une protéine de liaison. La capacité à identifier non seulement connus directes des partenaires de liaison ainsi que les protéines en interaction, secondaires souligne en outre l'utilité de cette approche dans la caractérisation des protéines de fonction inconnue.
Protocole
1. Génération de lignées cellulaires: pMEP4 transfection / expression
- Transfecter des cellules avec le vecteur d'expression pMEP4 et sélectionnez avec 100 ug d'hygromycine B / ml (Roche) jusqu'à ce que toutes les cellules transfectées simulées ont été tués hors tension. Le plasmide est maintenu pMEP4 épisomique de sorte qu'il n'est pas nécessaire d'isoler des clones spécifiques.
- Cellules contenant le vecteur pMEP4 peut être induite par un traitement avec 10 pM CdCl 2 pendant 16 heures. Expression et inductibilité doit être confirmée sur une petite échelle avant l'amplification des lignées cellulaires. La protéine TAP-étiqueté peut être facilement détectée que dans les domaines des protéines G se lier à des anticorps de presque toutes les espèces. Pour les grandes purifications typiquement 10 confluentes 175 cm 2 flacons de cellules sont nécessaires. Cela équivaut à environ 2 X10 8 cellules exprimant.
2. Préparation de lysat cellulaire
- Grattez les cellules dans du PBS et de combiner en un seul tube de 50 ml. Spin 1200X gfou 5 minutes (cela devrait aboutir à 2-3 ml de culot globulaire pour commencer, ce qui réduit à environ 1,5 ml après le lavage).
- Laver les cellules trois fois dans PBS glacé (50 ml à chaque fois).
- Lyser les cellules dans 5 ml de tampon de lyse (50 mM de Tris-HCl (pH 7,5), 125 mM de NaCl, de glycérol 5%, 0,2% de NP-40, 1,5 mM MgCl 2, 25 mM de NaF, 1 mM de Na 3 VO 4 et inhibiteur de la protéase ). Note: NaF, Na 3 VO 4 et inhibiteurs de la protéase doit être ajouté frais. Introduire à la pipette de haut en bas 10 fois avant de partir pour 5 minutes sur la glace. Fois seringues haut et en bas 5-10 en utilisant une aiguille émoussée, avant de repartir sur de la glace pendant 5 minutes sur la glace. Répétez seringage et laissez encore 5 minutes sur la glace.
- Gel-dégel du lysat deux fois (la glace N 2 liquide / ou sec et l'éthanol). Ne pas laisser l'échantillon pour atteindre plus de 4 ° C. Remarque: vous pouvez stocker cet échantillon à -80 ° C jusqu'à ce que vous avez le temps de traiter.
- Cellules aliquote de l'échantillon dans des tubes de 1,5 ml et de supprimer non lysées et debris par centrifugation (10 minutes à 4 ° C, 16.000 X g).
- Récupérer le surnageant, mélanger dans un tube Falcon de 15 ml et (éventuellement) passer à travers un filtre de 0,45 um avant de prendre un échantillon de 20 ul pour la quantification rendement en protéines. Suppression d'un aliquote supplémentaire de 50 pi pour une analyse ultérieure (Exemple 1).
3. La liaison aux IgG de lapin-agarose
(Remarque: Toutes les pirouettes doit être effectuée à g 1200X dans une centrifugeuse réfrigérée à 4 ° C pendant 1 minute, sauf indication contraire)
- Resuspendre doucement le lapin IgG-agarose solution (Sigma-Aldrich) en agitant la bouteille. Prenez 380 ul d'IgG d'agarose (en utilisant un embout de pipette 1 ml de coupe) et supprimer l'envoi / solution de conservation par centrifugation pendant 1 minute. Laver les trois agarose fois dans un tampon de lyse froid (4 ° C) en utilisant la centrifugation pour clarifier. Le rendement final d'agarose devrait être d'environ 250 pi de perles emballées.
- Ajouter le lysat cellulaire autorisé de 2,6 (dans une baignoire Falcon de 15 mle) à l'lavé de lapin IgG agarose et incuber pendant 3 heures (ou toute la nuit) à 4 ° C en utilisant un mélangeur rotatif.
- Isoler les billes d'agarose pendant 5 minutes à 4 ° C et retirer le surnageant pour une analyse ultérieure (échantillon 2). Remarque: Le TAP protéine marquée devrait maintenant être associé avec les perles.
4. Clivage de la protéase TEV
(Remarque: Toutes les pirouettes doit être effectuée à 1200X g dans une centrifugeuse réfrigérée à 4 ° C pendant 1 minute, sauf indication contraire)
- Laver les IgG de lapin billes d'agarose à trois reprises en tampon de lyse froid (4 ° C) en utilisant la centrifugation pour clarifier (note: le tampon de lyse ne devrait pas contenir des inhibiteurs de la protéase). Il faut prendre soin de ne pas enlever ou de perdre des perles au cours des étapes de lavage.
- Laver les billes deux autres fois avec un tampon de clivage TEV-protéase (10 mM Tris-HCl (pH 7,5), 100 mM de NaCl, et 0,2% de NP-40) en utilisant la centrifugation pour clarifier. Après le dernier lavage retirez soigneusement tous les liquid des billes.
- Préparer un mélange de protéase TEV clivage; pour chaque échantillon comprennent 467,5 pi H 2 O, 25 pl de tampon 20x TEV (Invitrogen), 5 pl DTT 0,1 M et 2,5 pi (25 U) VET protéase (Invitrogen). Ajouter 500 pl de ce mélange de clivage VET à chaque échantillon de billes emballées et transférer le mélange ensemble d'une 1,7 ml pré-lubrifié tube (Costar). Avant d'incubation supprimer un aliquote de 30 pl pour une analyse ultérieure (Exemple 3).
- Incuber la réaction protéase TEV pendant une nuit à 4 ° C en utilisant un mélangeur rotatif. Notez que des temps plus courts incubations peuvent être utilisés en fonction de la nature de la protéine appât et l'accessibilité du site de clivage de protéase TEV, mais le temps d'incubation minimale devrait être déterminée empiriquement.
5. Liaison à Ultralink streptavidine immobilisée plus perles
(Remarque: Toutes les pirouettes doit être effectuée à g 1200X dans une centrifugeuse réfrigérée à 4 ° C pendant 1 minute, sauf indication contraire)
- Centrifuger la réaction de clivage TEV contenant les IgG de lapin billes d'agarose pendant 5 minutes à 1200X g (4 ° C). Suppression d'un aliquote de 20 ul du surnageant clarifié pour l'analyse (échantillon 4)
- Transférer le surnageant restant (environ 480 pi) dans un nouveau 1,5 ml pré-lubrifié tube (Costar) et le laisser sur la glace. Ne jetez pas ce surnageant - elle contient des protéines de votre TEV appâts clivée et des protéines associées contraignantes.
- Ajouter 500 ul de tampon de lyse froid (section 2.3) pour les perles restantes IgG de lapin d'agarose et remettre en suspension. Centrifugeuse ce mélange pour clarifier les billes avant de retirer le surnageant et en le combinant avec celle de l'étape 5.2. Laissez ce tube sur la glace (il doit maintenant contenir ~ 980 pi d'échantillon).
- Conserver les IgG de lapin billes d'agarose pour une analyse ultérieure (échantillon 5). Remarque: si vous exécutez cet exemple sur un gel SDS-PAGE (ou western blot), il donnera beaucoup de bruit de fond dû à la présence de la lourde IgG et de la lumièreles chaînes.
- Pendant ce temps doucement resuspendre le Ultralink immobilisée streptavidine Plus billes d'agarose (Pierce) en agitant la bouteille. En utilisant une pointe de pipette 200 pi avec la coupe fin, partie aliquote de 70 pl billes de streptavidine par échantillon en pré-lubrifiés tubes de 1,5 ml et supprimer l'envoi / solution de conservation par centrifugation. Remarque: ces perles sont très petites et donc «à bec de canard» ou plat / étroite limitée conseils peuvent être utilisés pour minimiser la perte de bourrelet pendant le lavage.
- Laver les billes de streptavidine trois fois dans un tampon de lyse (réfrigérés à 4 ° C) en utilisant la centrifugation pour clarifier. Cela devrait laisser environ 45-50 ul de billes emballés par tube.
- Transférer le surnageant clivage TEV-protéase (c.-à-ul 980 de l'échantillon de l'étape 5.3) dans le tube contenant les billes de streptavidine lavés et incuber pendant 3 heures (ou toute la nuit) à 4 ° C en utilisant un mélangeur rotatif.
- Isoler les billes de streptavidine pour 5 minutes à 4 ° C et retirer le surnageant pour une analyse ultérieure (Samexemple 6). Laver les billes de streptavidine trois fois dans un tampon de lyse (réfrigérés à 4 ° C) en utilisant la centrifugation pour clarifier. Après le lavage final, retirez soigneusement tous tampon de lavage restant.
6. Élution du peptide biotine streptavidine et protéine appât
(Remarque: Toutes les pirouettes doit être effectuée à g 1200X dans une centrifugeuse réfrigérée à 4 ° C pendant 1 minute, sauf indication contraire)
- Éluer protéines marquées des billes de streptavidine par ajout de 500 ul de PBS: biotine (1 mM D-biotine) et incubation à 4 ° C pendant 3 heures (ou toute la nuit) en utilisant un mélangeur rotatif.
- Isoler les billes de streptavidine pour 5 minutes à 4 ° C et éliminer le surnageant dans un tube de 1,5 ml pré-lubrifié (Note: ceci est votre échantillon final / élution (Exemple 7).
- Ajouter une autre Biotine 500 ul: PBS aux billes de streptavidine restantes et incuber à 4 ° C pendant 2-3 heures (ou toute la nuit) en utilisant un mélangeur rotatif.
- Rétourbe étape 6.2 et de combiner les deux élutions (Exemple 7). Cette élution finale peuvent être stockés à -80 ° C.
- Pour évaluer l'efficacité de l'élution biotine, de geler les billes de streptavidine restants et faire bouillir plus tard dans un tampon d'échantillon SDS (échantillon 8) (recommandations: LaneMarker tampons 5x échantillon réduction de Fisher).
7. La concentration en protéines
- L'éluat final (Exemple 7) doit être concentrée avant l'analyse en raison de la faible concentration de protéines et le volume relativement élevé. Cela peut atteindre à l'aide de bas poids moléculaire (moins de 5 kDa) colonnes de centrifugation Vivaspin (Vivascience). Vise à concentrer le volume à moins de 100 pi. Cette finale de l'échantillon peut être bouilli et stockées dans un tampon échantillon de protéine (recommandations: LaneMarker tampons 5x échantillon réduction de Fisher).
8. Analyse
- Les échantillons 1-8 peuvent être analysés par western blot pour déterminer l'efficacité de la tirer vers le bas. Remarque: La plupart des anticorps secondairesune réaction croisée avec les domaines de protéine G de la protéine de fusion, mais cela est enlevé après clivage de la protéase TEV.
- Exemple 7 peuvent être analysés par 1D ou 2D SDS-PAGE. La coloration peut être effectué en utilisant l'argent ou au bleu de Coomassie (recommandations: coloration à l'argent colloïdal et SilverQuest Blue kits de coloration Coomassie de Invitrogen). Identification de protéines peut être réalisée en utilisant la spectrométrie de masse.
9. Les résultats représentatifs
Un exemple de 1D analyse SDS-PAGE de l'élution finale (Exemple 7) de ce protocole pour identifier les partenaires de liaison de eIF4E TAP marqué est fourni dans la figure 2. Ce gel représentant reflète la nature complexe et abondante des interactions avec d'autres protéines eIF4E dans la cellule. La comparaison avec le témoin négatif a également montré à la figure 2, généré à partir d'une lignée cellulaire exprimant uniquement la balise TAP, illustre la spécificité de cette eIF4E appâté déroulant. Dans ce cas 15% de l'élution concentrée final (échantillon7) a été analysé par SDS-PAGE 1D utilisant des gels de gradient disponibles dans le commerce préfabriqués avant d'être colorées à l'aide du kit de coloration à l'argent Silverquest d'Invitrogen.
Typiquement 50-85% de l'élution restant concentré final (échantillon 7) est analysée avec colloïdal Coomassie tache (Invitrogen). Les échantillons (tranches de gel ou bandes) de la ruelle ont ensuite été extrait et analysé par spectrométrie de masse.
Protéines identifiées dans le eIF4E déroulant ont été filtrés contre les partenaires de liaison de contrôle négatif à identifier les non-partenaires de liaison spécifiques. Les protéines finales identifiés en utilisant ce processus de eIF4E marquage TAP peut être vu dans la figure 2, qui est représentatif des protéines normales identifiés en utilisant cette technique. En outre, un certain nombre de sous-unités des eIF3 ont également été identifiés (données non présentées).
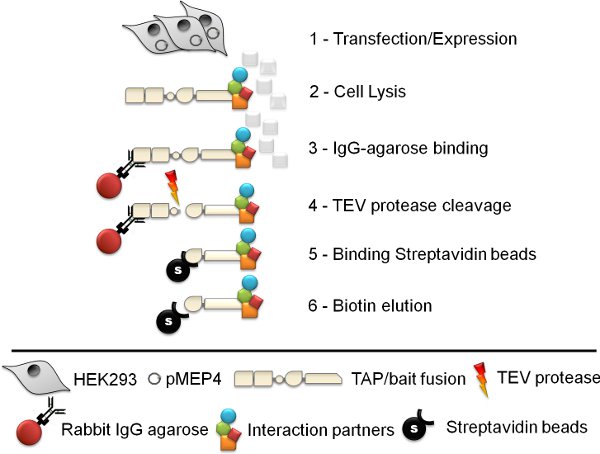
Figure 1. Schéma de l'procédure de purification par affinité en tandem. Le tandem six étape de purification par affinité (TAP) le protocole implique la génération lignée cellulaire, la lyse des cellules, IgG de lapin d'agarose immuno-précipitation, clivage de la protéase TEV, la purification d'affinité streptavidine perles et élution enfin la biotine.
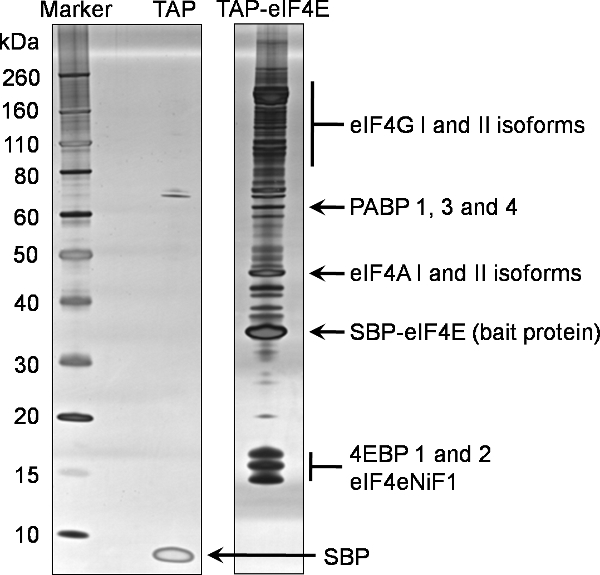
Figure 2. Tandem Affinity Purification de la protéine eIF4E murin. Partenaires d'interaction de la N terminale TAP étiqueté eIF4E ont été purifiés à partir de cellules HEK293 eucaryotes en utilisant le protocole ci-joint. Une fraction de 20% de l'élution finale (Exemple 7) a été analysé par SDS-PAGE sur un gel pré-coulé gradient 4-12% (TAP-eIF4E voies). Une analyse a été effectuée pour l'équivalent de la balise seul (piste TAP). Les protéines ont été identifiées à l'aide coloration à l'argent. Protéines suite identifiés par spectrométrie de masse de l'échantillon même sont mis en évidence sur ce gel.
Abréviations: eIF4G; eucaryote d'initiation de traduction fActeur 4 gamma, PABP; polyA binding protein, eIF4A: eucaryote facteur d'initiation de la traduction 4 alpha, SBP-eIF4E; la protéine TAP restant appâts contenant le peptide streptavidine fusionnée à la 4E facteur d'initiation de traduction eucaryote, 4EBPs; eucaryote 4E facteur d'initiation de traduction de liaison protéines, l'initiation eucaryote eIF4eNiF1l facteur de traduction 4E importation du facteur nucléaire 1, SBP, le peptide de liaison streptavidine reste du peptide TAP infondue TEV.
Discussion
La technique de marquage TAP illustré ici montre une méthode hautement spécifique et rigoureux pour isoler les partenaires de liaison de protéines appâts dans des cellules eucaryotes. Cette approche peut être appliquée à des protéines cellulaires et virales. À notre connaissance, c'est la première fois qu'une telle technique a été appliquée à l'initiation de la traduction eIF4E facteur. Identification de la liaison eIF4E eIF4G connu protéines et les 4EBPs en utilisant cette technique confirme la validité d'une telle approche. En outre, l'identification de la composante restante du complexe eIF4F, nommément eIF4A, et PABP confirme que l'interaction indirecte et des complexes tertiaires restent intactes pendant le procédé de purification. L'identification des isoformes multiples des protéines de liaison eIF4E canoniques était également évident. Ceux-ci sont décrits plus en détail dans la figure 2.
En ce qui concerne les limites de l'approche, certains soins doivent être prises en ce qui concerne le choixde la protéine appât et si oui ou non de placer la balise TAG à l'extrémité N-ou C-terminale. Il pourrait être souhaitable de procéder à un test fonctionnel ou d'examiner la localisation de la protéine de fusion par microscopie avant la purification à grande échelle pour assurer le dérivé marqué est fonctionnel. Membranaire intégrale ou des protéines nucléaires ne sont pas nécessairement être libéré par les conditions relativement douces de détergent décrites dans l'étape de lyse. Comme avec tous les immunoprécipitations et similaires déroulants essais, modifications pourraient être apportées à la nature et les concentrations du détergent dans le tampon de lyse pour accroître l'efficacité de la lyse. La concentration ionique des tampons de lyse et lavage peut également être modifié pour augmenter ou diminuer la rigueur de la purification. Il est également possible d'effectuer la purification initiale en vertu de standards des conditions douces (décrit ci-dessus) mais par la suite diviser les billes de streptavidine (section 5.8) en aliquotes 4-5, qui peut ensuite être lavées sous con ionique croissanteconditions. Cela peut permettre à l'utilisateur d'identifier les conditions optimales qui éliminent les protéines non spécifiques qui interagissent. L'ensemble du processus peut également être effectuée à l'aide de transfection transitoire où les partenaires d'interaction sont connus et leur présence ou leur absence déterminée par western blot ultérieure seulement. Dans ce cas, nous suggérons deux 100cm 2 plats de cellules transfectées avec chaque 8 pg de plasmide d'expression. Cette procédure modifié est particulièrement utile lors de l'examen de la capacité des mutants dérivés de la protéine TAP-fusion d'interagir avec des partenaires de liaison connus.
Analyser les échantillons 1 à 8 sur argent colorées gels SDS-PAGE (ou par western blot si un anticorps est disponible) est un excellent moyen de dépannage, la technique devrait ne parviennent pas à générer des résultats acceptables. L'efficacité de chaque précipitation par affinité et élution à base spécifique peut être analysée à l'aide de cette méthode d'échantillonnage systématique. Les échantillons de un à huit doivent être prises lors optimsation du protocole pour chaque nouvel appât.
La technique de marquage TAP fournit une alternative puissante et robuste à d'autres approches telles que GST pull-bas, la levure deux essais hybrides etc purification par affinité double et étapes d'élution spécifiques (clivage TEV et l'élution biotine) de fournir la spécificité et la rigueur doit être maintenue à un niveau élevé tout au long de la procédure de purification. Critique cette technique peut être appliquée à n'importe quelle protéine et représente donc une excellente méthode pour identifier des partenaires de liaison pour une protéine cible d'intérêt.
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Cette recherche a été financée par une bourse du Wellcome Trust principal décerné au Dr Ian Goodfellow.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | |
| Hygromycine B | Roche | 10843555001 | |
| Lapin IgG Agarose | Sigma | A2909 | |
| AC-VET protéase | Invitrogen | 12575-015 | |
| Cocktail d'inhibiteurs de protéase | Calbiochem | 539134 | |
| Ultralink immobilisée streptavidine perles Plus | Percer | 53116 | |
| Vivaspin 500 concentrateur centrifuge (5KDa) | Vivaspin | V50112 | |
| Kit SilverQuest coloration à l'argent | Invitrogen | LC6070 | |
| Novex kit colloïdal coloration au bleu de Coomassie | Invitrogen | LC6025 | |
| 1,7 ml tubes prélubrifiés | Costar | 3207 | |
| Microcapillaire pointes de pipette | VWR | 37001-150 | |
| NuPage 4-12% Bis-Tris gels de gradient | Invitrogen | NP0322BOX | |
| CdCl 2 | Sigma | 202908 | |
| 5x tampon d'échantillon SDS | Pêcheur | PN39000 |
Références
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem. Soc. Trans. 38 (4), 875-878 (2010).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17 (10), 1030-1032 (1999).
- Blackwell, C., Brown, J. D. The application of tandem-affinity purification to Candida albicans. Methods Mol. Biol. 499, 133-148 (2009).
- Cochrane, A. Stable complex formation between HIV Rev and the nucleosome assembly protein, NAP1, affects Rev function. Virology. 388 (1), 103-111 (2009).
- Fernandez, E. Targeted tandem affinity purification of PSD-95 recovers core postsynaptic complexes and schizophrenia susceptibility proteins. Mol. Syst. Biol. 5, 269-269 (2009).
- Gloeckner, C. J. Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag. Methods Mol. Biol. 564, 359-372 (2009).
- Holowaty, M. N. Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7. J. Biol. Chem. 278 (32), 29987-29994 (2003).
- Burckstummer, T. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3 (12), 1013-1019 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4 E. EMBO Rep. 6 (10), 968-972 (2005).
- Goodfellow, I. G., Roberts, L. O. Eukaryotic initiation factor 4E. Int. J. Biochem. Cell Biol. 40 (12), 2675-2680 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon