Method Article
Individuazione di partner proteine interagenti mediante Tandem Affinity Purification
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Tandem purificazione per affinità è un valido approccio per l'individuazione di partner di legame proteico. Come prova di concetto, questa metodologia è stata applicata al ben caratterizzato eIF4E fattore di inizio traduzione di co-precipitato i fattori coinvolti in cellule ospiti di inizio di traduzione. Questo metodo è facilmente adattabile a qualsiasi proteina cellulare o virale.
Abstract
Un punto critico e spesso limitante comprendere la funzione di host e proteine virali è l'identificazione di parti interagenti proteine cellulari o virali. Ci sono molti approcci che permettono l'identificazione di partner interagenti, compreso il sistema del doppio ibrido di lievito, nonché abbassarle saggi utilizzando proteine ricombinanti e di immunoprecipitazione di proteine endogene seguita dalla identificazione spettrometria di massa 1. Recenti studi hanno evidenziato l'utilità del doppio affinità purificazione tag mediata, accoppiato con due passi di eluizione specifici l'identificazione di proteine che interagiscono. Questo approccio, definito Tandem purificazione per affinità (TAP), è stata inizialmente utilizzata nel lievito 2,3 ma più recentemente è stato adattato da utilizzare in cellule di mammifero 4-8.
Come proof-of-concept che abbiamo stabilito una purificazione per affinità tandem (TAP) il metodo con il ben caratterizzato eucariotica traduzione iniziazione factor eIF4E 9,10. il cellulare eIF4E fattore di traslazione è una componente critica del complesso eIF4F cellulari coinvolti nella cap-dipendente inizio della traduzione 10. Il tag TAP utilizzato in questo studio si compone di due unità proteina G e un peptide streptavidina vincolante separati da un virus Tobacco Etch (TEV) clivaggio sequenza proteasi. Il tag TAP utilizzato in questo studio si compone di due unità proteina G e un peptide streptavidina vincolante separati da un virus Tobacco Etch (TEV) clivaggio sequenza proteasi 8. Per rinunciare la necessità per la generazione di linee cellulari clonali, abbiamo sviluppato un sistema rapido che si basa sull'espressione del TAP-tag proteina esca da un plasmide episomally mantenuto base pMEP4 (Invitrogen). Espressione di targhetta eIF4E murino da questo plasmide è stato controllato mediante il cloruro di cadmio promotore inducibile metallotioneina.
Lisi delle cellule esprimenti e successiva purificazione per affinità tramite legame rabbit IgG agarosio, clivaggio della proteasi TEV, l'associazione a streptavidina legata agarosio e successiva eluizione biotina identificato numerose proteine apparentemente specifiche per il eIF4E pull-down (rispetto ai controlli linee cellulari che esprimono il tag TAP solo). Le identità delle proteine sono stati ottenuti mediante escissione delle bande da 1D spettrometria di SDS-PAGE e successiva massa. I componenti individuati incluse le proteine eIF4E note vincolanti eIF4G e 4EBP-1. In aggiunta, altri componenti del complesso eIF4F, di eIF4E che è un componente sono stati identificati, cioè eIF4A e Poly-A proteina legante. La capacità di identificare non solo i noti partner diretti vincolanti così come secondari proteine interagenti, evidenzia ulteriormente l'utilità di questo approccio nella caratterizzazione di proteine di funzione sconosciuta.
Protocollo
1. Generazione di linee cellulari: pMEP4 trasfezione / espressione
- Trasfezione delle cellule con il vettore di espressione pMEP4 e selezionare con 100 microgrammi di igromicina B / ml (Roche) fino a quando tutte le cellule mock trasfettate sono state uccise. Il plasmide viene mantenuta pMEP4 episomally quindi non è necessario isolare cloni specifici.
- Le cellule contenenti il vettore pMEP4 può essere indotta mediante trattamento con 10 pM CDCl 2 per 16 ore. Espressione e inducibilità dovrebbe essere confermato su piccola scala prima amplificazione delle linee cellulari. Il TAP-tag proteina può essere facilmente rilevato come i domini della proteina G si legano agli anticorpi da quasi tutte le specie. Per grandi purificazioni tipicamente 10 confluenti 175 cm 2 flaconi di cellule sono obbligatori. Ciò equivale a circa 2 X10 8 cellule che esprimono.
2. Cella lisati
- Raschiare le cellule in PBS e combinare in un unico tubo da 50 ml. Spin 1200X gfo 5 minuti (questo dovrebbe tradursi in 2-3 ml di emazie concentrate per cominciare, che si riduce a circa 1,5 ml dopo il lavaggio).
- Lavare le cellule per tre volte in PBS ghiacciato (50 ml ogni volta).
- Lisare le cellule in 5 ml di tampone di lisi (50 mM Tris-HCl (pH 7,5), 125 mM NaCl, 5% glicerolo, 0,2% NP-40, 1,5 mM MgCl 2, 25 mM NaF, 1 mM Na 3 VO 4 e inibitore della proteasi ). Nota: NaF, Na 3 VO 4 e inibitori di proteasi dovrebbero essere aggiunti fresco. Pipettare su e giù per 10 volte prima di partire per 5 minuti su ghiaccio. Siringa volte su e giù per 5-10 con un ago senza punta prima di ripartire in ghiaccio per 5 minuti su ghiaccio. Ripetere siringare e lasciare per altri 5 minuti in ghiaccio.
- Freeze-scongelare il lisato due volte (ghiaccio liquido N 2 / o secco e etanolo). Non permettere che il campione raggiunga la maggiore di 4 ° C. Nota: è possibile memorizzare il campione a -80 ° C fino a quando non ha il tempo di elaborare.
- Cellule aliquote del campione in provette da 1,5 ml e rimuovere non lisate e debris per centrifugazione (10 minuti a 4 ° C, X 16.000 g).
- Recuperare il surnatante, si fondono in un tubo di 15 ml Falcon e (opzionalmente) passano attraverso un filtro da 0,45 micron prima di prendere un campione di 20 microlitri per la quantificazione delle proteine rendimento. Rimuovere una ulteriore aliquota 50 microlitri per successiva analisi (Campione 1).
3. Associazione a coniglio IgG-agarosio
(Nota: Tutti i giri deve essere effettuata a 1200X g in una centrifuga refrigerata a 4 ° C per 1 minuto, se non diversamente indicato)
- Risospendere delicatamente il IgG-agarosio coniglio soluzione (Sigma-Aldrich) agitando bottiglia. Prendete 380 microlitri di IgG agarosio (con un taglio 1ml punta della pipetta) e rimuovere la distribuzione / soluzione conservante per centrifugazione per 1 minuto. Lavare le tre volte agarosio in tampone di lisi freddo (4 ° C) mediante centrifugazione per chiarire. La resa finale di agarosio dovrebbe essere di circa 250 pl di perline confezionati.
- Aggiungere il lisato cellulare eliminato dal 2,6 (in una vasca da 15 ml Falcone) la lavata-IgG di coniglio agarosio ed incubare per 3 ore (o per una notte) a 4 ° C usando un miscelatore rotante.
- Centrifugare le perline di agarosio per 5 minuti a 4 ° C e rimuovere il supernatante per la successiva analisi (Campione 2). Nota: Il TAP tag proteine dovrebbe essere associato con le sfere.
4. Proteasi TEV cleavage
(Nota: Tutti i giri deve essere effettuata a 1200X g in una centrifuga refrigerata a 4 ° C per 1 minuto, se non diversamente indicato)
- Lavate il coniglio IgG perle di agarosio tre volte in tampone di lisi freddo (4 ° C) mediante centrifugazione per chiarire (nota: il buffer di lisi non deve contenere inibitori della proteasi). Si deve prestare attenzione a non rimuovere o perdere eventuali microsfere durante le fasi di lavaggio.
- Lavare le perline altre due volte con tampone TEV-proteasi clivaggio (10 mM Tris-HCl (pH 7,5), NaCl 100 mM, e 0,2% NP-40) mediante centrifugazione per chiarire. Dopo l'ultimo lavaggio rimuovere con attenzione tutti i liquid dalle perline.
- Preparare una TEV mix clivaggio della proteasi, per ogni campione sono 467,5 ul H 2 O, 25 pl 20x TEV Buffer (Invitrogen), 5 pl 0,1 M DTT e 2,5 microlitri (25 U) proteasi TEV (Invitrogen). Aggiungere 500 microlitri di questo mix scissione TEV ad ogni campione di perline confezionati e trasferire l'intero mix da ml 1,7 pre-lubrificati tubo (Costar). Prima di rimuovere incubazione un'aliquota di 30 microlitri per successiva analisi (campione 3).
- Incubare la reazione TEV proteasi notte a 4 ° C usando un miscelatore rotante. Si noti che i tempi più brevi incubazioni possono essere utilizzate a seconda della natura della proteina esca e l'accessibilità del sito di clivaggio TEV proteasi, ma il tempo di incubazione minimo dovrebbero essere determinate empiricamente.
5. Associazione a Ultralink Streptavidina immobilizzata Plus. perline
(Nota: Tutti i giri deve essere effettuata a 1200X g in una centrifuga refrigerata a 4 ° C per 1 minuto, se non diversamente indicato)
- Centrifugare la reazione di scissione TEV contenente le IgG di coniglio perline di agarosio per 5 minuti a 1200X g (4 ° C). Rimuovere un'aliquota da 20 pl del supernatante chiarito per analisi (Campione 4)
- Trasferire il surnatante rimanente (circa 480 pl) in una nuova 1,5 ml pre-lubrificati tubo (Costar) e lasciarlo su ghiaccio. Non buttare via questo surnatante - questo contiene la proteina esca TEV spaccati e le eventuali proteine associate vincolanti.
- Aggiungere 500 microlitri di tampone di lisi refrigerati (sezione 2.3) per i restanti coniglio IgG perle di agarosio e risospendere. Centrifugare questa miscela di chiarire le perline prima di rimuovere il surnatante e combinandolo con quello dal punto 5.2. Lasciare la provetta in ghiaccio (dovrebbe ora contenere ~ 980 pl di campione).
- Conservare le IgG di coniglio perle di agarosio per l'analisi successiva (campione 5). Nota: se si esegue questo esempio su un gel SDS-PAGE (o Western Blot) darà un sacco di fondo dovuto alla presenza di IgG pesante e leggeracatene.
- Nel frattempo Risospendere delicatamente il Ultralink immobilizzata streptavidina Plus perle di agarosio (Pierce) agitando la bottiglia. Utilizzando una punta di 200 microlitri pipetta con il taglio finale, aliquota 70 microlitri di perle streptavidina per campione in pre-lubrificati provette da 1,5 ml e rimuovere la distribuzione / soluzione conservante per centrifugazione. Nota: queste perle sono molto piccole e quindi "becco d'anatra" o appartamento / narrow-ended suggerimenti possono essere utilizzati per ridurre al minimo la perdita di tallone durante il lavaggio.
- Lavare le perle streptavidina tre volte in tampone di lisi freddo (4 ° C) mediante centrifugazione per chiarire. Ciò dovrebbe lasciare circa il 45-50 microlitri di perle confezionate per tubo.
- Trasferire la TEV-proteasi supernatante clivaggio (cioè il 980 microlitri di campione dalla fase 5.3) per il tubo contenente le microsfere streptavidina lavate e incubare per 3 ore (o per una notte) a 4 ° C usando un miscelatore rotante.
- Centrifugare le perline streptavidina per 5 minuti a 4 ° C e rimuovere il supernatante per successiva analisi (Sampio 6). Lavare le perle streptavidina tre volte in tampone di lisi freddo (4 ° C) mediante centrifugazione per chiarire. Dopo l'ultimo lavaggio, rimuovere con cura tutti i buffer residuo di lavaggio.
6. Eluizione del peptide biotina streptavidina vincolante e proteina esca
(Nota: Tutti i giri deve essere effettuata a 1200X g in una centrifuga refrigerata a 4 ° C per 1 minuto, se non diversamente indicato)
- Eluire proteine marcate dalle perline streptavidina aggiunta di 500 pl di PBS: Biotina (1 mM D-biotina) e incubando a 4 ° C per 3 ore (o tutta la notte) utilizzando un miscelatore rotante.
- Spin le perline streptavidina per 5 minuti a 4 ° C e rimuovere il surnatante in una provetta 1,5 ml pre-lubrificati (Nota: questo è il tuo campione finale / eluizione (Sample 7).
- Aggiungere 500 pl altro Biotina: PBS alle perle streptavidina rimanenti e incubare a 4 ° C per 2-3 ore (o tutta la notte) utilizzando un miscelatore rotante.
- Retorba punto 6.2 e combinare i due eluizioni (campione 7). Tale eluizione finale può essere conservato a -80 ° C.
- Per valutare l'efficienza del eluizione biotina, streptavidina congelare le perle restanti e far bollire poi in tampone campione SDS (Sample 8) (raccomandazioni: LaneMarker buffer 5x campione di ridurre da Fisher).
7. La concentrazione di proteine
- L'eluato finale (Campione 7) deve essere concentrato prima dell'analisi causa della bassa concentrazione di proteine e volume relativamente elevato. Ciò può ottenere utilizzando a basso peso molecolare (<5 KDa) colonne spin Vivaspin (Vivascience). Intende concentrare il volume a meno di 100 ul. Il campione finale può essere bollito e conservato in tampone proteine campione (raccomandazioni: LaneMarker buffer 5x campione di ridurre da Fisher).
8. Analisi
- 1-8 I campioni possono essere analizzati mediante western blot per determinare l'efficienza di tirare verso il basso. Nota: La maggior parte degli anticorpi secondaricross-reagiscono con le proteine G domini della proteina di fusione, ma questo viene rimosso dopo clivaggio proteasi TEV.
- Campione 7 possono essere analizzati mediante 1D o 2D SDS-PAGE. La colorazione può essere eseguita utilizzando argento o Coomassie (raccomandazioni: colorazione argento colloidale SilverQuest e colorazione con Coomassie Blue kit da Invitrogen). L'identificazione di proteine può essere effettuata utilizzando la spettrometria di massa.
9. Risultati rappresentativi
Un esempio di 1D analisi SDS-PAGE di eluizione finale (Campione 7) da questo protocollo per individuare partner di legame di eIF4E TAP marcato viene fornita in Figura 2. Questo gel rappresentante riflette la natura complessa e ricca di interazioni eIF4E con altre proteine all'interno della cellula. Il confronto con il controllo negativo anche illustrato nella figura 2, generato da una linea cellulare che esprime solo il tag TAP, illustra la specificità di questa eIF4E esca pull-down. In questo caso 15% del concentrato finale eluizione (Sample7) è stata analizzata mediante 1D SDS-PAGE usando commercialmente disponibili prefabbricati gel gradiente prima di essere colorati con il kit di colorazione d'argento Silverquest da Invitrogen.
Tipicamente 50-85% del restante eluizione concentrato finale (Campione 7) viene analizzata con Coomassie colloidale macchia (Invitrogen). Campioni (fette di gel / bande) dalla corsia intero sono stati poi estratti e analizzati mediante spettrometria di massa.
Proteine identificate nel eIF4E pull-down sono stati filtrati contro i partner di legame dal controllo negativo per individuare partner di legame non specifici. Le proteine finali identificate utilizzando questo processo di eIF4E TAP tagging può essere visto in figura 2, che è rappresentativo delle normali proteine identificate utilizzando questa tecnica. Inoltre, un certo numero di eIF3 subunità sono state anche identificate (dati non mostrati).
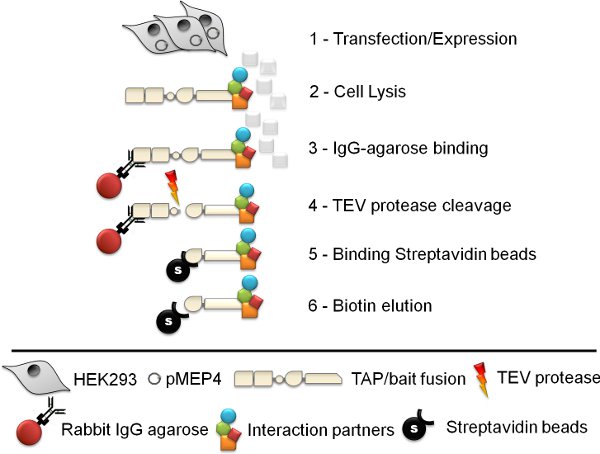
Figura 1. Schematica dellatandem procedura di purificazione per affinità. I sei tandem passo affinità di purificazione (TAP) il protocollo prevede la linea di generazione delle cellule, lisi cellulare, Rabbit IgG agarosio immuno-precipitazione, clivaggio della proteasi TEV, streptavidina purificazione per affinità tallone e infine eluizione biotina.
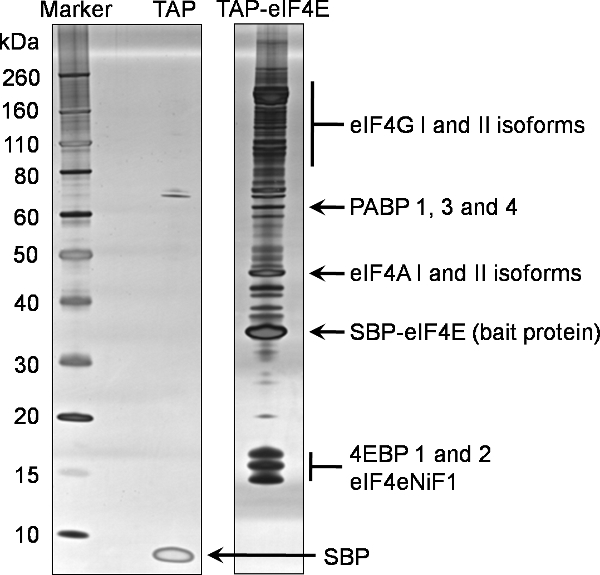
Figura 2. Tandem purificazione per affinità della proteina eIF4E murina. Interagire partner del terminale N TAP tag eIF4E sono stati purificati da cellule eucariotiche HEK293 che utilizzano il protocollo allegato. Una frazione del 20% della eluizione finale (Campione 7) è stata analizzata mediante SDS-PAGE su un gel pre-cast gradiente 4-12% (TAP-eIF4E corsia). L'analisi è stata effettuata equivalente per il tag da solo (corsia TAP). Le proteine sono state identificate usando colorazione argentica. Proteine successivamente identificati mediante spettrometria di massa del campione stesso sono evidenziati in questo gel.
Abbreviazioni: eIF4G; eucariotica traduzione apertura f4 attore gamma, PABP; proteine polyA vincolante, eIF4A: fattore di inizio eucariotico traduzione 4 alpha, SBP-eIF4E, il restante proteina esca TAP contenente il peptide streptavidina vincolante fusa alla eucariotico 4E fattore di inizio della traduzione, 4EBPs; inizio eucariotico 4E traduzione fattore vincolante proteine, eIF4eNiF1l eucariotico di inizio della traduzione fattore 4E fattore nucleare di importazione 1, SBP, il peptide streptavidina rimane vincolante dal peptide TEV TAP non fusa.
Discussione
La tecnica di codifica TAP illustrata qui viene illustrato un metodo altamente specifico e rigoroso per isolare i partner di legame delle proteine esca in cellule eucariotiche. Questo approccio può essere applicato sia alle proteine cellulari e virali. A nostra conoscenza, questa è la prima volta una tale tecnica è stata applicata alla eIF4E fattore di iniziazione traduzione. Identificazione del noto eIF4G proteine di legame eIF4E e le 4EBPs utilizzando questa tecnica conferma la validità di tale approccio. Inoltre, l'identificazione del restante componente del complesso eIF4F, cioè eIF4A, e PABP conferma che interazione indiretta e complessi terziari rimanere intatto durante il processo di purificazione. L'identificazione di isoforme multiple dei canonici eIF4E proteine di legame era anche evidente. Questi sono descritti in maggiore dettaglio nella figura 2.
Per quanto riguarda i limiti del metodo, certa attenzione dovrebbe essere presa per quanto riguarda la sceltadi proteine e se esca o meno per posizionare l'etichetta TAG alla N-o C-terminale. Potrebbe essere consigliabile effettuare un saggio funzionale o esaminare la localizzazione della proteina di fusione mediante microscopia prima della purificazione scala per garantire il derivato etichettato è funzionale. Integrali di membrana o proteine nucleari non possono necessariamente essere rilasciato dalle condizioni relativamente blande detergenti descritte nella fase di lisi. Come con tutti immunoprecipitazioni e simili pull-down saggi, modifiche possono essere apportate alla natura e concentrazione del detersivo nel tampone di lisi per aumentare l'efficienza di lisi. La concentrazione ionica dei buffer di lisi e lavaggio possono anche essere modificati per aumentare o ridurre la severità della purificazione. E 'anche possibile eseguire la purificazione iniziale in normali condizioni blande (sopra descritto), ma successivamente dividere i talloni sezione streptavidina (5,8) in aliquote 4-5, che possono successivamente essere lavate sotto crescente con ionicazioni. Ciò può consentire all'utente di identificare le condizioni ottimali per la rimozione di proteine interagenti non-specifici. L'intero processo può essere effettuata anche mediante trasfezione transiente in cui sono note le parti interagenti e la loro presenza o assenza determinata dalla successiva western blot solo. In questo caso vi suggeriamo due 100cm due piatti di cellule trasfettate ciascuna con 8 microgrammi di plasmide di espressione. Questa procedura modificata è particolarmente utile quando esaminando la capacità dei derivati mutanti del TAP-proteina di fusione per interagire con i noti partner di legame.
Analizzando i campioni da 1 a 8 su Silver macchiati SDS-PAGE gel (o western blot se un anticorpo è disponibile) è un ottimo modo di risoluzione dei problemi, se la tecnica non riescono a produrre risultati accettabili. L'efficienza di ciascuna precipitazione affinità basata e eluizione specifica possono essere analizzati utilizzando questo approccio sistematico campionamento. I campioni da uno a otto deve essere assunto durante Optimzazione del protocollo per ogni nuova esca.
La tecnica di tagging TAP offre un'alternativa potente e robusto per altre vie, come GST pull-down, lievito due saggi ibridi ecc purificazione per affinità doppia e specifiche fasi di eluizione (TEV cleavage eluizione e biotina) forniscono specificità e rigore deve essere mantenuta ad un livello alto durante la procedura di purificazione. Criticamente questa tecnica può essere applicata a qualsiasi proteina e rappresenta quindi un metodo eccellente per individuare partner di legame per una proteina bersaglio di interesse.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Questa ricerca è stata finanziata da una borsa di studio Wellcome Trust senior assegnato al Dr. Ian Goodfellow.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| Hygromycin B | Roche | 10843555001 | |
| IgG di agarosio | Sigma | A2909 | |
| AC-proteasi TEV | Invitrogen | 12575-015 | |
| Cocktail di inibitori della proteasi | Calbiochem | 539134 | |
| Ultralink immobilizzato streptavidina perline Plus | Perforare | 53116 | |
| Vivaspin 500 concentratore centrifugo (5KDa) | Vivaspin | V50112 | |
| SilverQuest silver Kit di colorazione | Invitrogen | LC6070 | |
| Novex Blu Coomassie colloidale Kit di colorazione | Invitrogen | LC6025 | |
| 1.7ml tubi prelubrificati | Costar | 3207 | |
| Microcapillare puntali | VWR | 37001-150 | |
| NuPage 4-12% Bis-Tris gradiente di gel | Invitrogen | NP0322BOX | |
| CDCl 2 | Sigma | 202908 | |
| 5x SDS Sample Buffer | Pescatore | PN39000 |
Riferimenti
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem. Soc. Trans. 38 (4), 875-878 (2010).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17 (10), 1030-1032 (1999).
- Blackwell, C., Brown, J. D. The application of tandem-affinity purification to Candida albicans. Methods Mol. Biol. 499, 133-148 (2009).
- Cochrane, A. Stable complex formation between HIV Rev and the nucleosome assembly protein, NAP1, affects Rev function. Virology. 388 (1), 103-111 (2009).
- Fernandez, E. Targeted tandem affinity purification of PSD-95 recovers core postsynaptic complexes and schizophrenia susceptibility proteins. Mol. Syst. Biol. 5, 269-269 (2009).
- Gloeckner, C. J. Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag. Methods Mol. Biol. 564, 359-372 (2009).
- Holowaty, M. N. Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7. J. Biol. Chem. 278 (32), 29987-29994 (2003).
- Burckstummer, T. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3 (12), 1013-1019 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4 E. EMBO Rep. 6 (10), 968-972 (2005).
- Goodfellow, I. G., Roberts, L. O. Eukaryotic initiation factor 4E. Int. J. Biochem. Cell Biol. 40 (12), 2675-2680 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon