Method Article
蛍光性DNAzymesを使用した細菌の検出
要約
我々は最近、単純な細菌検出用蛍光アッセイを "ミックス·アンド·読む"を設定するために適用できる蛍光DNAザイムプローブを生成するための斬新なアプローチが報告されています。これらの特殊なDNAプローブは、それによって蛍光シグナルの生成に細菌検出を翻訳する特定の細菌によって産原油の細胞外液(CEM)の存在下で発色団修飾DNA-RNAキメラ基質の切断を触媒する。本報告では、特定のDNAザイムプローブは、 "RFD-EC1"を表すモデルの細菌の検出のために採用されている重要な実験手順を説明します。大腸菌(Escherichia coli)。
要約
発生はそれぞれ毎年死亡と入院の何百万人と同様に巨大な経済的損失のために食品を媒介と院内感染病原体のアカウントにリンクされています。進行中の流行の場所の影響を正確に早い段階で犯人の病原体を識別することができる分析法のためにますます需要のような流行と最小化の防止。高い特異性(興味のある唯一の細菌を検出)、高感度(低として検出することができる:病原体を検出するための効果的な方法の大きな配列があるが、それらのどれも理想的な検出方法のために具現すべて次の5つの最初の要件を満たすことはできません単一のライブ細菌細胞など)を、短時間-結果に(数分から数時間)、偉大な運用の簡素化(長いサンプリング手順や特殊な装置の使用の必要はありません)、および費用対効果。例えば、古典的な微生物学的方法は非常に特異的であるが、長い時間(DAを必要とする週間YS)が1 PCRおよび抗体ベースの技術は短い待機時間(数時間から数日)を提供しています。決定的な結果を取得するために、彼らは高価な試薬および/ または洗練された装置の使用。結果2-4を必要とする、まだある前述の要件の1つまたは複数の特性の改善を提供する革新的な細菌の検出方法を開発に向けた科学研究のための大きな需要。当研究室では、細菌の検出を含むバイオセンシングアプリケーションのための分子プローブの新規クラスとしてDNAzymesの可能性を調べるに興味がある5。
DNAzymes(またdeoxyribozymesまたはDNA酵素として知られている)は人工の化学反応を触媒する機能を備えた一本鎖DNA分子を6-8、これらの分子は、多くの10とを含む広大なランダムシーケンスDNAプール(から単離することができる16個の配列)o "はin vitroでの選択で "として知られるプロセスによってR "SELEX"(指数関数的濃縮によるリガンドの系統的進化)。9月16日これらの特別なDNA分子は、広くバイオセンシングアプリケーションのための分子ツールとして近年検討されている6-8。
我々の研究室ではRNA-切断蛍光DNAzymes(RFDs、図1)を単離するためのインビトロでの選択の手順で確立したと分析のツールとしてRFDsの使用を検討した17から29 RFDsは、単一のリボヌクレオチドでDNA-RNAキメラ基質の切断を触媒する。蛍光体(F)およびクエンチャー(Q)によって挟まれているジャンクション(R)。 FとQの近くには未開基板最小限の蛍光をレンダリングします。しかし、切断イベントは、蛍光強度の顕著な増加を伴っているFとQの分離につながる。
最近では、我々は、細菌を検出するためのRFDsを単離する方法を開発しました。5これらの特別なRFDsは"に分離された粗混合物を使用すると、浄化の退屈なプロセスを回避します。自分の環境で、またはそれらが培養されているメディアの中の細菌の特定のタイプ( 図1)によって残された原油の細胞外液(CEM)の存在下で、 "点灯し、バイオセンサーの開発(完了するまで数ヶ月または数年かかる可能性がある)の対象となる微生物から適切なターゲットを識別します。外のターゲットを使用すると、細胞内のターゲットを取得する手順については、必要はありませんので、検定の手順はシンプルであることを意味します。
のみE.によって生成されたCEMの存在下で、上記のアプローチを使用して、我々は( 図2A。FS1)は、その基板を切断するRFDを導出した大腸菌 (CEM-EC)。5このE. RFD-EC1( 図2A)という名前の大腸菌検出RFDは、厳密にCEM-ECへの応答が他の細菌のホスト( 図3)からのCEMに応答しなくなることが判明した。
ここでは、presaの複数形NT E.を設定するための重要な実験手順RFD-EC1と代表的な結果を用いて大腸菌の検出アッセイ。
プロトコル
1。化学溶液の調製
- 0.5 Mエチレンジアミン四酢酸(EDTA)は:2リットル(L)プラスチック製のビーカーでは、186.1グラムのEDTAを量る(EMサイエンス)とオートクレーブ脱イオン蒸留水(DDH 2 O)の800ミリリットル(mL)を追加します。 NaOHペレット(EMサイエンス)を用いてpHを8.0に調整します。のddH 2 Oを用いて1 Lに最終的なボリュームを作る4℃でガラスびん、オートクレーブとストアにソリューションを転送
- 10×Tris-ホウ酸EDTA溶液(10×TBE、89 mMトリス、89 mMのホウ酸、2mMのEDTA、pH7.5)中:トリス塩基(BioShopカナダ)とホウ酸(BioShopカナダ)の220グラムの432グラムを計量、および4 Lのプラスチックビーカーにそれぞれ追加します。 0.5 M EDTA(pH 8.0)を80 mLを測定し、ビーカーに加える。コンポーネントが完全に溶解されるまで、徹底的に磁気撹拌棒溶液を混合4 Lの最後のボリュームにDDH 2 Oを追加します。 4℃でガラスびん、オートクレーブとストアにソリューションを転送
- 10%がpolyacrylami変性デ·ゲル在庫:4 Lのプラスチックビーカーに、尿素の1681.7グラム(BioShopカナダ)、10 400 mLの×TBE、40%アクリルアミド/ビスアクリルアミド(29:1)溶液(BioShopカナダ)の1 Lを追加します。のddH 2 Oで4 Lに音量を調整撹拌しながら尿素を溶かす。 4℃で1リットルの琥珀色のガラスびんとストアにソリューションを転送(それは重合の前に神経毒であるので注意!アクリルアミドは手袋、マスク、ゴーグルと白衣で処理する必要があります)。
- 2×ゲルローディングバッファー(2×GLB):200mLのガラスビーカーには、尿素44グラム、ショ糖の8グラム(BioShopカナダ)、ブロモフェノールブルー(BioShopカナダ)10mgを、xylenecyanol FFの10mgを(シグマ追加-Aldrich)を、10%ドデシル硫酸ナトリウム(SDS 400μL、BioShopカナダ)、10×TBE 4 mLの。 DDH 2 O 40 mLに、最終的な音量を調節して穏やかに加熱(50℃)と磁気バーで撹拌しながら固形物を溶解する。 4℃で1.5 mlのマイクロチューブやストアに1mLのアリコートを転送℃に私たちのに基づいて経験は、それが2×GLBは、ストレージ条件の下で固化するために使用することが°Cの前に90℃の熱2×GLBを簡潔にすることが必要である。
- 1 Mトリス - 塩酸(pH7.5)を200 mLのガラス製ビーカーにトリス塩基の12.1 gを秤量する。 DDH 2 O 60 mLを加えてマグネチックスターラーで攪拌して固体を溶解する。 1 M塩酸(Sigma-Aldrich社)を用いてpHを7.5に調整します。のddH 2 Oで100mlにボリュームを構成し、ガラスの瓶とオートクレーブに転送します。 4℃で保管してください
- 5 M NaClを:ガラスビーカー中でのNaCl 58.4グラム(BioShopカナダ)重さのddH 2 Oを150 mLで溶解のddH 2 Oで200mlに音量を調整する4℃でガラスの瓶、オートクレーブ、店舗への解決策を転送
- DNAの溶出緩衝液:ガラスビーカーでは、1 Mトリス - 塩酸(pH7.5)、5 M NaClを8 mLの0.5 M EDTA(pH8.0)を0.4mLの2 mLを混和します。のddH 2 Oで200mlに音量を調整する4℃でオートクレーブや店舗
- 2×反応緩衝液(2×RB、100 mMのHEPES、300mMのNaCl、30 mMのMgCl 2):50 mlのFalconチューブ(BDファルコン)へ、HEPESの1.2グラム(BioShopカナダ)を追加し、NaClとMgCl 2の0.30グラムの0.88グラム•6H 2 O (EMDケミカルズ)とのddH 2 Oの30 mLのコンポーネントが完全に溶解するまで穏やかに振とうして混合します。 10 N NaOH溶液を加えることによってpHを7.5に調整し、DDH 2 Oで50 mLに、最終的な音量を調節4℃でシリンジ駆動型フィルタユニット(0.22μmのミリポア)とストアを使用してソリューションを清める
- ルリアベルターニ(LB)培地:ビーカーでは、DDH 2 Oの1 Lを添加した(Sigma-Aldrich)をLB粉末20.0グラムの重量を量る磁気撹拌棒ソリューションを徹底的に混合し、三角フラスコに溶液を移します。室温で溶液とストアをオートクレーブします。
- 1.5%LB寒天は、250 mLのフラスコ中で寒天1.5gの(BioShopカナダ)を秤量し、液体LB 100 mLを加える。室温で混合し、ストアをオートクレーブします。
- 寒天めっき:メルトン電子レンジでLB寒天と〜50℃まで冷却ソリューション炎の下にペトリ皿に溶液(フィッシャーサイエンティフィック)を注ぐ。我々の経験では、LB寒天培地100 mLのは5-6プレートを得ることができます。
2。テンプレートによるRFD-EC1とRFSS1の建設は酵素ライゲーションを媒介
RFD-EC1( 図2A)は機能DNAザイムです。それは、触媒のシーケンスEC1と基板シーケンスFS1( 図2Aの黒と緑の線で示される)で構成されています。 RFSS1( 図2A)は 、触媒のシーケンスEC1が部分的にSS1にシャッフルがFS1の部分が変更されませんRFD-EC1のスクランブルバージョンです。 RFD-EC1とRFSS1は、ライゲーションのテンプレートとしてLT1の存在下でオリゴヌクレオチドEC1またはSS1( 図2Aに挿入されたボックスを参照)を有するオリゴヌクレオチドFS1のテンプレートを介した酵素ライゲーションによって行われた。ライゲーション反応を行うための手順を以下に提供されています。FS1は、以前に確立されたプロトコルに従って、イェール大学でケックオリゴ合成施設から得られた脱保護し、ゲル電気泳動により精製した。EC1 17から24、SS1とLT1が統合されたDNA Technologiesから購入し、ゲル電気泳動により精製した。
- FS1、EC1、SS1とのddH 2 Oを使用してLT1、100μMのストック溶液を調製使用するまで-20℃で保存することができます。
- チューブ1とチューブ2としてマークされた2つの1.5μLのマイクロチューブにFS1ストック溶液を5μLを転送します。各チューブに、DDH 2 Oの38.5μL、10の後、5μL×T4ポリヌクレオチドキナーゼ(PNK)反応バッファーを追加する500mMのトリス-HCl(pHは7.6、25°C)を含んでいる(MBI Fermentas)、100 mMののMgCl 2、50mMのDTT、1.0 mMのスペルミジン。 (ピペット上下ソリューションを数回)、ピペッティングにより各溶液を混和します。
- ATP(100 mMの、MBI Fermentas)を1μl加え、ピペッティングにより混和します。
- T4ポリヌクレオチドキナーゼ(Pの0.5μLを追加します。NK、10単位/μL、MBI Fermentas)とピペッティングにより混和する。 30分間37℃で反応混合物をインキュベートします。フルオロフォアの光退色最小限に抑えるためにアルミホイルでチューブをカバーすることを確認してください。
- 5分間90℃で加熱することにより反応を停止。 10分間室温で反応混合物を冷却する。
- EC1の5μLとチューブ1とチューブ2にSS1ストック溶液を、それぞれ追加します。
- ピペッティングすることにより、各チューブ、ミックスにLT1ストック溶液を5μLを追加します。 1分間、10分間室温に冷却し、90℃で反応混合物を加熱する。
- のddH 2 Oの118μLとし、10の20μL×400 mMのトリス-HCl(25℃でpH 7.8)、100 mMのMgCl 2、100mMのDTT、および5が含まれているT4 DNAリガーゼ緩衝液(MBI Fermentas)を追加mMのATP。ピペッティングで溶液を混合。
- ピペッティングにより、ミックス、T4 DNAリガーゼ(MBI Fermentas 5単位/μL)2μLを追加します。 1時間室温で反応混合物をインキュベートします。
- 、各チューブに3 M NaOAc(pH7.0)に20μlの渦とスピンダウンする。各チューブに冷100%エタノールを500μLを追加し、ボルテックスで溶液を混合し、30分間-20℃の冷凍庫に管を配置します。
- °C冷却遠心機(アレグラX22-R、ベックマン·コールター)の4で20分間、11,000 gで混合物を遠心分離し、注意深くピペッティングにより上清を除去します。
- 10分間のDNAコンセントレータ(SavantのDNA Speedvac、サーモフィッシャーサイエンティフィック)を使用してDNAペレットを乾燥させます。
- 30 1μL×ゲルローディングバッファー(GLB)、簡単にボルテックスし、卓上型遠心機(Minicentrifuge、VWRサイエンティフィック)とダウンスピンに再懸濁し、DNAのペレット。連結したDNAサンプルは10%dPAGEゲル上にロードするための準備が整いました。
3。 10%のdPAGEゲルの調製
次の手順では、簡単にゲル電気泳動し、そのセット·アップの装置について説明します。装置の設定と処理についてのより詳細については、tを参照してください私たちの以前に発行されたプロトコルをO。30,31
- 2枚のガラス板、2 0.75ミリメートルスペーサーと16ウェルコームを洗浄し、乾燥させます。クリップでガラス板とスペーサをアセンブルし、上向きにノッチガラス板の平らな面に水平に横たわっていた。
- 150 mLのビーカーに10%dPAGEミックス40 mLを転送します。ピペッターで渦巻くことで、10%APSの400μL、ミックス、テトラメチルエチレンジアミンの40μL(BioshopカナダTEMED)を追加します。
- プレートの間に慎重に混合物を注ぎ、すぐにコームを挿入します。混合物は10から20分間重合することができます。重合は、ビーカーに残された残留ゲルミックスをチェックすることによって確認することができます。
- 一度重合し、穏やかに櫛を削除してウエルに残ったゲル溶液を除去するためのddH 2 Oでウェルをすすぎます。
- 外向きにノッチ長方形のプレートとゲル電気泳動装置にプレートをマウントし、ノッチ板の背後に金属板を配置します。金属PLの使用食べた、ガラス板を割ることができる過熱を防ぐことができます。
- 装置の上部と下部のチャンバーに1×TBEを追加します。井戸は、バッファが充填され、ゲルの下端には、バッファに浸漬されていることを確認します。
- 40ミリアンペア(または750 V)は、現在の10〜15分間の事前実行を適用します。
4。 10%のdPAGEジェルで連結RFD-EC1とRFSS1の精製
- 3.7の手順に従って、1×TBEは、注射器と注射針を使用してウェルを洗浄してください。
- ピペッターとゲルローディングチップ(Diamed)を使用して2ウェル(各1)にRFD-EC1のとRFSS1そのライゲーション混合物を(2.13から)をロードします。プレートの下端上約5cmである底部色素(ブロモフェノールブルー)まで40ミリアンペア(750 V)の電流を適用します。
- ゲル実行している装置からガラス板を取り外し、平らな作業台の上に横になると慎重にスペーサーを削除します。
- 慎重にゲルから上部ガラス板を削除し、プラスチック製のラップ(ゲル上でラップのしわや折りたたみを回避しようとする)を持つゲルをラップします。
- 連結された製品は、UVシャドウ(260 nm)をしたり、数センチEC1またはSS1(EC1またはSS1 100pmolの上に見えるのDNAバンドが生成されます。透視(360 nm)で、いずれかによって可視化することができるマーカーとして使用することができ、 )4.2の段階でうまくロードされます。マーカーと目的のDNAバンドをマークします。
- 滅菌したカミソリの刃を持つ消費税のDNAバンドが、新しい1.5 mLのマイクロ遠心チューブに小さい部分と転送にゲルを切った。
- 滅菌したピペットチップ(200μLチップサイズ)を使用して、マイクロ遠心チューブ内でゲル片をつぶす。
- 各チューブに500μLのDNA溶出バッファーを追加し、光から蛍光体を保護するためにアルミ箔でそれらをカバーしています。 10分間ボルテックスのサンプルを。
- °C冷却遠心機(アレグラX22-R、ベックマン·コールター社)で4℃で4分間11000グラムでサンプルを遠心分離し、慎重にsの350μLを転送新しい1.5 mlのマイクロ遠心チューブ(必要に応じて、2回目の溶出は、別の10分間新鮮な溶出バッファー350μLで行うことができます)にupernatant。
- 、各チューブに3 M酢酸ナトリウム(NaOAc、pH7.0)を35μL(サンプル量0.1×)を追加しボルテックスで混和してスピンダウンする。各チューブに冷100%エタノールを900 uLのを追加します。数秒間チューブを手で振盪することにより、各サンプルを混ぜる。少なくとも1時間-20℃でチューブを置きます。
- 冷却遠心機で4℃20分間11000グラム°Cでサンプルを遠心分離し、注意深くピペッティングにより上清を除去します。
- 冷70%エタノールを100μLを加え、穏やかにチューブ全体の内壁を洗浄するために使用します。 4時7分11,000 gで再遠心分離℃に上清を除去し、10分間のDNAコンセントレータを使用して、ペレットを乾燥させます。
- DDH 2 Oと渦の100μLのDNAペレットを溶解する。 260nmでのUV吸光度に基づいてDNA濃度を決定します。 -20店のサンプル°C使用時まで。
5。細菌の調製
- 標的細菌株E.大腸菌 K12(MG1655)と関連する制御系統は、まず、グリセロールストックからLB寒天プレート上に播種されています。炎の下、または生物学的安全キャビネット内で、LB寒天培地の損傷を防ぐために、プレート表面に滅菌したピペットチップをゆっくりとストリークを有する細菌のグリセロールストックをタッチします。
- 14時間37℃で縞板とインキュベートを反転します。インキュベーション後、4℃でパラ(Pechineyプラスチック包装)とストアでプレート全体の周囲をシールこれらのプレートは、4週間の最大のために保存することができます。
6。原油外混合物の調製(CEMS)
- ピペットガン(コーニング)を使用して滅菌14 mlの培養チューブ(BDファルコン)にLBの2 mLを分注します。
- 滅菌したピペットチップを用いて、5.2の工程で調製寒天プレートから単一コロニーをピックアップし、AC電源に挿入しますultureチューブ。インキュベーター(ニューブランズウィックサイエンティフィック)の場所チューブは37℃に設定し、14時間、250 rpmで振とうする。
- 1パーセントの再接種の文化:ステップ6.2で調製した細菌培養液の20μLを14 mLの培養管とスパイクに新鮮なLBの2 mLを分注します。それぞれの菌液が約1のOD 600(600 nmで測定光学密度)に達するまで、250 rpmで振盪しながら37℃でチューブをインキュベートします。 OD 600を測定するために、UV分光光度計(UVジェネシス10、サーモフィッシャーサイエンティフィック)と600nmでの使い捨てキュベットを測定吸光度に各培養液1 mLを転送します。
- 室温で5分間、11,000 gで遠心分離して、新しい1.5 mlのマイクロ遠心チューブ、ペレット細胞に各培養液1 mLを転送します。
- 直ちに使用しない場合は-20℃で新鮮な1.5 mlのマイクロ遠心チューブとストアに透明な上清を転送します。
7。蛍光分光光度計を用いた検出
- 蛍光分光光度計(キャリーのEclipse、バリアン社)をオンにすると520 nmで488 nmの放射で励起してデータ収集パラメータを設定します。測定値は1時間分ごとに撮影することができます。
- 100%エタノール、続いてのddH 2 O 3水晶キュベット(バリアンキャリー)、洗浄します。窒素ガスを点滅させることで、キュベットを乾燥させます。ラベルキュベットC1(コントロール1)、C2(コントロール2)とT(テスト)。
- C1とC2とT.、各キュベットと蛍光分光光度計で行われ、それらを、2の25μL×RBを追加する方法CEM-ECの24μLにDDH 2 Oの24μLを転送します。最初の5分間蛍光データの収集を開始します。
- C2とTおよびC1〜RFD-EC1の1μL(5μMストック溶液から)にRFSS1の1μL(5μMストック溶液から)を追加します。ピペッティングにより各溶液を混和します。これは慎重に蛍光の読み取りが中断されないように開始する必要があります。反応は1時間の取得時間の残りの部分を継続することができます。
- 保存Excelのファイル形式のデータは、パーソナルコンピュータやグラフィカルなイメージを作成するプロセスデータにデータを転送します。
8。ゲル電気泳動による検出
ステップ7.4で調製した同一の反応混合物をゲル電気泳動による分析のために使用することができます。代わりに新たな反応が同様に調製し、1.5 mLのマイクロ遠心チューブ中でインキュベートすることができます。どちらの場合:
- 3 M NaOAc、100%エタノールを125μLの5μLを加えることによってクエンチ反応(1時間後)。ボルテックスすることにより、各溶液を混合し、1時間-20℃の冷凍庫に管を配置します。
- 4℃で20分間11000グラムで反応混合物を遠心分離℃、注意深くピペッティングにより上清を除去します。
- 10分間、DNAコンセントレータを使用して、ペレットを乾燥させます。
- 1の20μLに再懸濁したペレット×簡単にボルテックスGLB。卓上型遠心機(Minicentrifuge、VWRサイエンティフィック)と非常に簡単にチューブをスピンダウンする。これらのサンプルが読み込まれますdPAGEゲルにロードするのにyと入力します。
- 3.1から3.7で説明したように10%dPAGEゲルを準備します。ピペッターとゲルローディングチップ(Diamed)を使用してウェルに反応サンプルを(ステップ8.4)をロードします。プレートの下端上約5cmである底部色素(ブロモフェノールブルー)まで40ミリアンペア(750 V)の電流を適用します。
- ガラス板を取り出して、ゲル片を除去するために水道水で十分に洗浄してください。キムワイプ(キンバリークラークプロフェッショナル)でプレートを拭きます。
- 台風スキャナ(台風9200、可変モードでは、GEヘルスケア)を使用して、蛍光のゲルプレートをスキャンします。 ImageQuantソフトウェア(分子動力学)を使用してデータを分析します。
9。検出特異性
- 細菌の特異性をテストするために、同じ手順では、培養E.について上述した大腸菌は、そのCEMを準備し、開裂反応アッセイを行っては、例えばBと異なる菌株の数を実行することができます枯草菌、P. PELI、Y. ruckeri、L. Planturum、P. acidilactici( 図3Aに示すように)。
10。単一細胞の検出
1 mLのE.を準備します。大腸菌 2 CFU / mLのグリセロールストック(CFU:コロニー形成単位)。希釈によって、めっきによりCFUの濃度を確認する5この株式は0.2 CFU/100μLを含める必要があります。 -80℃で保存、使用するまで。
- LBの2 mLを含む10培養管を準備します。
- 250 rpmで振盪しながら37℃で2 CFU / mLのグリセロールストックとインキュベートし、100μLの各文化を接種する。全体のグリセロールストックの使用(1 mL)溶液に、10の文化を生成する必要があります。
- 4、8、12、16および24時間以下の時点でそれぞれ接種した培養管から収穫300μL。 24時間成長し、残りの文化にしておきます。
- OD 600を測定し、5分間、11,000 gで遠心して細胞を沈殿させる。
- -20&Dで新しい1.5 mlのマイクロチューブやストアにのCEMを転送例えば、使用するまでのC。
注:時間のポイント4、8と12で採取したサンプルについては、検出可能なOD 600は存在しないかもしれないので、残りの培養は、細菌を(濁度とOD測定により決定)を含む培養物を識別するために、24時間成長させることができます。 10文化の1つまたは2つだけは、 大腸菌が含まれる場合があります接種後、大腸菌 、残りのチューブは、任意のセルが含まれていません。 - RFD-EC1で開裂反応を準備するために指定された時点において培養陽性(-20℃で保存)から回収されたのCEMを使用し、8章で説明したようにdPAGEゲル電気泳動を用いて反応混合物を分析します。
11。概念と代表的な結果
細菌検出のためのRNA切断蛍光DNAザイム(RFD)を悪用の概念図に示されています。 1。 RFDは、ラベルが付いた2つのヌクレオチドによって隣接唯一のRNA結合(青R)のキメラDNA / RNA基質を切断する蛍光体(F)およびクエンチャー(Q)と、それぞれ。興味のある細菌( 大腸菌など)のメディアになるにつれ、それは原油の細胞外液(CEM)を残します。全体として、このCEMは、その後CEMに特異的に反応するRFDを取得するためにin vitroでの選択実験でで使用され、おそらくRFDは、細菌の署名分子であるCEMの特定の分子(紫星)と対話します。 CEMは、RFDを含む反応溶液に添加されるとき、それはRFDのRNA切断活性をトリガします。切断イベントは、蛍光計を使用するか、ゲル電気泳動のいずれかによって検出することができ、蛍光シグナルの結果、QからFを分離しています。
上記の概念の実験的検証は、EからCEMで行われました大腸菌 (CEM-EC)。我々はin vitroでの選択に介して3 RFD分子を取得し、最も効率的なものがRFD-EC1( 図2A)に指定されています。5 Weは、CEM-ECへの応答としてRFD-EC1の切断活性(RFSS1という変異配列と一緒に)テストされています。両方RFD-EC1とRFSS1は、基板FS1(すべてのシーケンスを図に示されています。2A)にDNAザイム部分の酵素的ライゲーションすることにより調製した。蛍光測定の実験( 図2B)では、CEM-ECは、RFD-EC1またはRFSS1を加えた後5分間、および55分間さらにインキュベートすることにより単独でインキュベートした。溶液の蛍光強度が連続的に分ごとに読み込まれた、データは(RF、時刻tでの蛍光強度の比と時間0での蛍光強度として計算して)相対的な蛍光を計算するために使用されていました。インキュベーションの時間とRFの値が図としてプロットされています。 2B。対照的に、RFSS1は強い蛍光シグナルを生成しませんでした。それはRFD-EC1は、CEM-ECの添加により蛍光シグナルの高レベルを生成することがわかった。したがって、蛍光産FUCEM-ECの接触時にRFD-EC1のnctionは、配列特異的である。
その観測された蛍光の増加はRNA結合の開裂によるものであるかどうかを確認するために、我々はdPAGEによって反応混合物を分析した。 RFD-EC1の切断は、二つのDNA断片を、5 '蛍光体を保持する断片と3'断片消光剤を保持を生成することが期待されています。唯一の未開RFD-EC1(unclv)と5 '断片(CLV)が蛍光イメージングによって検出される可能性がある。 dPAGE結果は図に示す。 RFSS1/CEM-EC混合物がなかった間、 図2Cは 、RFD-EC1とCEM-ECの反応混合物は確かに予想される切断産物を生産していることがわかります。
RFD-EC1の特異性は、他のいくつかのグラム陰性およびグラム陽性細菌から収集したデータを図に示されているのCEMを用いて検討した。 3A。 CEM-EC(青色の曲線)を含む唯一のサンプルは、蛍光の増加を生じた。のCEMとの交差反応性の欠如他の細菌からRFD-EC1は、E.のために非常に選択的であることを示しています大腸菌 。
また、培養し、単一のE.に必要な時間を検討大腸菌細胞RFD-EC1の切断を誘導することができる十分なCEMを生成するため。この実験で、 大腸菌のために定義されたCFUを(コロニー形成単位)を含む大腸菌のサンプルが十分に1 CFU / mLの濃度を達成するために希釈した。これは、4、8、12、16、24時間増殖培地と培養それで希釈した細菌サンプルの100μLを混合することによって続いた。のCEMは、その後各時点を収集し、RFD-EC1の切断活性を誘導するための試験を行った。 dPAGE結果は図に示す。図3Bは、12時間の培養時間が必要であることを示します。
最初の小信号の増加はCEM-EC( 図2BにRFSS1シーケンス(ネガティブコントロールとして)を添加した後、蛍光測定で観察されたことに注意することは重要です。赤いcurve)または他の細菌のCEM( 図3BにRFD-EC1、青以外のすべての曲線)はFRQモジュールの本質的な蛍光(QによるFの不完全焼入れによる)に起因しています。したがって、それはF-およびQ-標識配列の付加は、初期の蛍光の増加を生み出すことが期待されています。ただし、RFD-EC1/CEM-EC混合物は、時間の経過とともに蛍光の高いレベルを産生することができる。

図1 RNA-切断蛍光DNAザイム(RFD)プローブの模式図その関心のある特定の細菌の細胞によって産生される粗製細胞混合物(CEM)と接触すると蛍光を発する。 RFDは、それぞれ蛍光体(F)およびクエンチャー(Q)とラベルが付いた2つのヌクレオチドによって隣接唯一のRNA結合(青R)のキメラDNA / RNA基質を切断する。開裂反応の前に、RFDの蛍光レベルに近い近接のために最小限に抑えられます切断の際、FとQのあるImityは、QはFから出発し、結果として、強い蛍光シグナルが生成されます。
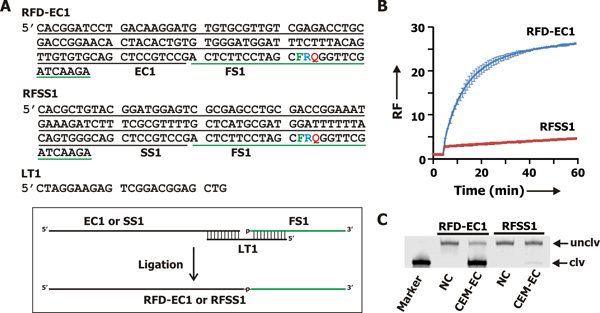
図2 E.大腸菌検出RFDします。 (A)RFD-EC1は、CEM-ECによって活性化することができるDNAザイムプローブです。 RFSS1は、コントロールとして用いRFD-EC1のスクランブルシーケンスです。 RFD-EC1とRFSS1は、テンプレートとしてLT1の存在下で、それぞれ、EC1とSS1とFS1を連結することにより製造された。 F:フルオレセイン修飾デオキシチミジン。 Q:DABCYL変性デオキシチミジン。 R:アデニンリボヌクレオチド。 (B)蛍光は、CEM-ECの存在下でRFD-EC1とRFSS1のプロファイルをシグナリング。 Bの開裂反応混合物(C)dPAGE解析(反応時間:60分)。写真は台風スキャナによって得られたdPAGEゲルの蛍光画像である。レーンNC:単独で反応緩衝液中RFD-EC1またはRFSS1、レーンCEM-EC:CEM-ECを含む反応緩衝液でRFD-EC1またはRFSS1。マーカー:RFD-CE1 0.25 NのNaOH、RNAの完全切断を引き起こすことが知られている手順で処理した。 unclv:RFD-EC1未開。 CLV:蛍光体を含む切断断片。
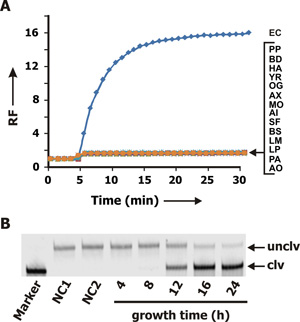
図3種々の細菌細胞から調製されたのCEMのRFD-EC1の(A)蛍光シグナル伝達プロフィール。 EC: 大腸菌 K12、PP: 膿PELI、BD:ブレバンディモナスdiminuta、HA:ハフニアalvei。YR:エルシニアruckeri、OG:Ochrobactrum grignonese。AX:アクロxylosoxidans、MO:モラクセラosloensis、AI:アシネトバクターlwoffi、SF:セラチアfonticola、BS:枯草菌(Bacillus subtilis)、LM:ロイコノストックのmesenteroides、LP:乳酸菌planturum、PA:乳酸菌acidilactici、AO:放線菌オリエンタリス 。各CEMサンプルはRFD-EC1の加え、続いて5分間インキュベートした。 (B)dPAGE60分反応後RFD-EC1/CEM-EC混合物の分析。レーンNC1:単独で反応緩衝液中RFD-EC1。レーンNC2:CEM-BS( 枯草菌から調製されたCEM)を含む反応緩衝液中RFD-EC1。シングルE.を含む細菌培養から採取したCEM-ECを含む反応緩衝液中でRFD-EC1:4、8、12、16、24で標識されたレーン4成長期後に大腸菌細胞、8、12、16および24時間であった。
ディスカッション
今日一般的な細菌の検出方法のほとんどは、低速(古典的な微生物)または技術的に(抗体、PCR)を求めています。そこで、検出ツールの次の世代は、スピードとシンプルさに向かって応えるべきだと思います。この目的のために、我々は、RNA切断し、蛍光シグナルの生成を介して細菌の存在を報告するためにシンプルなアッセイを開発するために使用することができ、蛍光シグナルDNAザイムを作成しました。注目のDNAザイムプローブ、RFD-EC1は、E.の成長時に生成CEMによって活性化される培養培地中の大腸菌 。提案手法は、検出の対象として細菌の粗細胞混合物を使用して、面倒なターゲットの抽出と増幅のステップをバイパスしているので、それは細菌検出のためのアッセイの非常に単純な、 "ミックス·アンド·読む"のタイプを設定するために使用することができます。私たちのDNAザイムの使用は、蛍光ベースの検出方法に限定されません。同じDNAザイムシステムアッセイcを使用して、例えば、比色検出有機色素と一緒にローリングサークル増幅を利用し、以前に報告された方法を用いて設計することができます。32我々は、より大きな運用の簡素化と新たな細菌のバイオセンサーを生成するための魅力的な道のような細菌を検出するためのDNAzymesの使用を予見する。
開示事項
利害の衝突が宣言されません。
謝辞
この仕事のための資金は、自然科学とカナダの工学研究会(NSERC)とSentinel生理活性紙ネットワークによって提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号またはモデル | |
| 寒天 | BioShopカナダ | AGR003 | |
| 過硫酸アンモニウム(APS) | BioShopカナダ | AMP001 | |
| アクリルアミド/ビスアクリルアミド(40%、29:1) | BioShopカナダ | ACR004 | |
| ホウ酸 | BioShopカナダ | BOR001 | |
| ブロモフェノールブルー | Sigma-Aldrich社 | B8026 | |
| EDTA | EMサイエンス | EXO539-1 | |
| 塩酸 | Sigma-Aldrich社 | 38281 | |
| HEPES | Bioshopカナダ | HEP001 | |
| LBブロス | Sigma-Aldrich社 | L3022 | |
| MgCl 2を | EMD化学品 | B10149-34 | |
| NaClを | BioShopカナダ | SOD002 | |
| NaOAc | EMDケミカルズ | SXO255-1 | |
| NaOHで | EMDケミカルズ | SXO590-1 | |
| SDS | BioShopカナダ | SDS001 | |
| TEMED | BioShopカナダ | TEM001 | |
| トリス塩基 | BioShopカナダ | BST666 | |
| Tween 20を | Sigma-Aldrich社 | P9416 | |
| 尿素 | BioShopカナダ | URE001 | |
| Xylenecyanol FF | Sigma-Aldrich社 | X4126 | |
| DNAコンセントレータ | サーモフィッシャーサイエンティフィック | SavantのDNA SpeedVac 120 | |
| マイレクスフィルタユニット | ミリポア | SLGP033RS | |
| ゲルローディングのヒント | Diamed | TEC200EX-K | |
| ImageQuantソフトウェア | 分子動力学 | バージョン5.0 | |
| キムワイプ | キンバリークラークプロフェッショナル | 34705 | |
| ミニボルテックス | VWR | 58816-121 | |
| パラフィルム | Pechineyプラスチック包装 | PM996 | |
| ペトリ皿 | フィッシャー·サイエンティフィック | Fisherbrand 08-757-12 | |
| Stripettorプラス(ピペットガン) | コーニング | 07764714 | |
| 石英キュベット | バリアン社 | 66-100216-00 | |
| シェーカー/インキュベーター | ニューブランズウィックサイエンティフィック | クラシックシリーズC24 | |
| 台風スキャナ | GEヘルスケア | 9200可変モード | |
| 遠心分離 | ベックマン·コールター | アレグラX22-R | |
| UV分光光度計 | サーモフィッシャーサイエンティフィック | GenesysUV 10 | |
| 蛍光分光光度計 | バリアン社 | ケアリーのEclipse |
参考文献
- Zourob, M., Elwary, S., Truner, A. . Principles of Bacterial Detection: Biosensors, Recognition Receptors and Microsystems. , (2008).
- Call, D. R. Challenges and Opportunities for Pathogen Detection Using DNA Microarrays. Crit. Rev. Microbiol. 31, 91-99 (2005).
- Lazcka, O., Campo, D. e. l., J, F., Muñoz, F. X. Pathogen detection: A perspective of traditional methods and biosensors. Biosens. Bioelectron. 22, 1205-1217 (2007).
- Velusamy, V. An overview of foodborne pathogen detection: In the perspective of biosensors. Biotechnol. Adv. 28, 232-254 (2010).
- Ali, M. M. Fluorogenic DNAzyme Probes as Bacterial Indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Liu, J., Cao, Z., Lu, Y. Functional Nucleic Acid Sensors. Chem. Rev. 109, 1948-1998 (2009).
- Li, Y., Lu, Y. . Functional Nucleic Acids for Analytical Applications. , (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Joyce, G. F. Forty Years of In Vitro Evolution. Angew. Chem. Int. Ed. 46, 6420-6436 (2007).
- Breaker, R. R., Joyce, G. F. A DNA enzyme that cleaves RNA. Chem. Biol. 1, 223-229 (1994).
- Cuenoud, B., Szostak, J. W. A DNA metalloenzyme with DNA ligase activity. Nature. 375, 611-614 (1995).
- Chinnapen, D. J., Sen, D. A deoxyribozyme that harnesses light to repair thymine dimers in DNA. Proc. Natl. Acad. Sci. U. S. A. 101, 65-69 (2004).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Silverman, S. K. DNA as a versatile chemical component for catalysis, encoding, and stereocontrol. Angew. Chem. Int. Ed. 49, 7180-7201 (2010).
- Mei, S. H. An efficient RNA-cleaving DNA enzyme that synchronizes catalysis with fluorescence signaling. J. Am. Chem. Soc. 125, 412-420 (2003).
- Liu, Z. Assemblage of signaling DNA enzymes with intriguing metal-ion specificities and pH dependences. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Kandadai, S. A., Li, Y. Characterization of a catalytically efficient acidic RNA-cleaving deoxyribozyme. Nucleic Acids Res. 33, 7164-7175 (2005).
- Rupcich, N. Quenching of fluorophore-labeled DNA oligonucleotides by divalent metal ions: implications for selection, design, and applications of signaling aptamers and signaling deoxyribozymes. J. Am. Chem. Soc. 128, 780-790 (2005).
- Shen, Y., Brennan, J. D., Li, Y. Characterizing the secondary structure and identifying functionally essential nucleotides of pH6DZ1, a fluorescence-signaling and RNA-cleaving deoxyribozyme. Biochemistry. 44, 12066-12076 (2005).
- Chiuman, W., Li, Y. Revitalization of six abandoned catalytic DNA species reveals a common three-way junction framework and diverse catalytic cores. J. Mol. Biol. 357, 748-754 (2006).
- Chiuman, W., Li, Y. Evolution of high-branching deoxyribozymes from a catalytic DNA with a three-way junction. Chem. Biol. 13, 1061-1069 (2006).
- Shen, Y. Catalysis and rational engineering of trans-acting pH6DZ1, an RNA-cleaving and fluorescence-signaling deoxyribozyme with a four-way junction structure. Chem BioChem. 7, 1343-1348 (2006).
- Ali, M. M., Kandadai, S. A., Li, Y. Characterization of pH3DZ1 - An RNA-cleaving deoxyribozyme with optimal activity at pH 3. Can. J. Chem. 85, 261-273 (2007).
- Chiuman, W., Li, Y. Efficient signaling platforms built from a small catalytic DNA and doubly labeled fluorogenic substrates. Nucleic Acids Res. 35, 401-405 (2007).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA MgZ based on a non-classic allosteric design. PLoS ONE. 2, e1224 (2007).
- Shen, Y. Entrapment of fluorescence signaling DNA enzymes in sol gel-derived materials for metal ion sensing. Anal. Chem. 79, 3494-3503 (2007).
- Kandadai, S. A. Characterization of an RNA-cleaving deoxyribozyme with optimal activity at pH 5. Biochemistry. 48, 7383-7391 (2009).
- Zhao, W., Brook, M. A., Li, Y. Periodic assembly of nanospecies on repetitive DNA sequences generated on gold nanoparticles by rolling circle amplification. Methods Mol. Biol. 474, 79-90 (2008).
- Navani, N. K., Mok, W. K., Li, Y. In vitro selection of protein-binding DNA aptamers as ligands for biosensing applications. Methods Mol. Biol. 504, 399-415 (2009).
- Ali, M. M., Li, Y. Colorimetric sensing by using allosteric-DNAzyme-coupled rolling circle amplification and a peptide nucleic acid-organic dye probe. Angew. Chem. Int. Ed. 48, 3512-3515 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved