Method Article
Nachweis von Bakterien mit fluorogenen DNAzyme
In diesem Artikel
Zusammenfassung
Wir haben vor kurzem einen neuartigen Ansatz zur Erzeugung von fluorogenen DNAzym Sonden, die angewendet zur Einrichtung eine einfache, "Mix-and-read" Fluoreszenz-Assay für die bakterielle Nachweis kann berichtet werden. Diese speziellen DNA-Sonden katalysieren die Spaltung eines Chromophors-modifizierten DNA-RNA-chimären Substrat in Gegenwart von rohem extrazellulären Mischung (CEM) von einem bestimmten Bakteriums fort, wodurch Nachweis von Bakterien in Fluoreszenz-Erzeugungseinheit erzeugt wird. In diesem Bericht beschreiben wir wichtigen experimentellen Verfahren, bei denen eine bestimmte DNAzym Sonde bezeichnet "RFD-EC1" für den Nachweis des Modells Bakterium verwendet wird, Escherichia coli (E. coli).
Zusammenfassung
Ausbrüche an lebensmittelbedingten und im Krankenhaus erworbene Erreger Konto für Millionen von Todesfällen und Hospitalisierungen sowie kolossale wirtschaftliche Verluste jedes Jahr verbunden. Prävention solcher Ausbrüche und die Minimierung der Auswirkungen einer laufenden Epidemie Ort eine ständig steigende Nachfrage nach analytischen Methoden, die genau zu identifizieren Täter Krankheitserreger im frühesten Stadium kann. Zwar gibt es eine große Palette von effektiven Methoden für den Erregernachweis, kann keiner von ihnen erfüllen auch die folgenden fünf führenden Anforderungen für einen idealen Nachweismethode verkörperte: eine hohe Spezifität (Erkennen nur das Bakterium von Interesse), hohe Empfindlichkeit (zum Aufspüren von so niedrig als eine einzige Live-Bakterienzelle), kurze Time-to-Ergebnisse (Minuten bis Stunden), große Einfachheit im Betrieb (keine langwierigen Probenahmeverfahren und den Einsatz von Spezialgeräten) und Wirtschaftlichkeit. So sind zum Beispiel klassischen mikrobiologischen Methoden hochspezifische erfordern aber eine längere Zeit (days bis Wochen), um eine definitive Resultat zu erwerben. 1 PCR-und Antikörper-basierte Techniken bieten kürzere Wartezeiten (Stunden bis Tage), aber sie erfordern den Einsatz von teuren Reagenzien und / oder die anspruchsvolle Ausrüstung. 2-4 Folglich gibt es immer noch eine große Nachfrage für die wissenschaftliche Forschung zur Entwicklung innovativer bakterielle Nachweismethoden, die verbesserten Eigenschaften in einem oder mehreren der oben genannten Anforderungen zu bieten. Unser Labor ist daran interessiert, Prüfung des Potenzials DNAzyme als einer neuen Klasse von molekularen Sonden für Biosensorik Anwendungen, einschließlich Nachweis von Bakterien. 5
DNAzyme (auch als Desoxyribozyme oder DNA-Enzyme bezeichnet) sind synthetische einzelsträngige DNA-Moleküle mit der Fähigkeit zur Katalyse chemischer Reaktionen. 6-8 Diese Moleküle aus einer großen Random-Sequenz-DNA-Pool (die nicht weniger als 10 enthält, kann isoliert werden 16 einzelne Sequenzen) durch ein Verfahren als "in-vitro-Selektion" o bekanntr "SELEX" (systematische Evolution von Liganden durch exponentielle Anreicherung). 16.9 Diese besonderen DNA-Moleküle sind weit verbreitet in den letzten Jahren als molekulare Werkzeuge für Biosensorik Anwendungen untersucht. 8.6
Unser Labor hat in vitro Selektion Verfahren zur Isolierung von RNA-spaltende fluoreszierende DNAzyme (. RFDs; Abb. 1) wurde untersucht und die Verwendung von RFDs als analytische Werkzeuge 17-29 RFDs katalysieren die Spaltung eines DNA-RNA-Chimären Substrat an einem einzelnen Ribonukleotid. Verbindung (R), die von einem Fluorophor (F) und einem Quencher (Q) flankiert wird. Die Nähe von F und Q macht die ungespaltenen Substrat minimale Fluoreszenz. Allerdings führt die Spaltung bei der Trennung von F und Q, die durch deutliche Erhöhung der Fluoreszenzintensität begleitet wird.
In jüngerer Zeit entwickelten wir ein Verfahren zur Isolierung von RFDs für Nachweis von Bakterien. 5 Diese speziellen RFDs wurden isoliert "leuchtet "in Anwesenheit des rohen extrazellulären Mischung (CEM) zurückgelassen von einer besonderen Art von Bakterien in ihrem Umfeld oder in den Medien werden sie kultiviert (Abb. 1). Die Verwendung von rohen Mischung umgeht das mühsame Prozess der Reinigung und Identifizierung eines geeigneten Ziels von der Mikrobe von Interesse für die Entwicklung von Biosensoren (was Monate oder Jahre in Anspruch nehmen). Der Einsatz von extrazellulären Ziele bedeutet, dass das Testen von Verfahren ist einfach, weil es keine Notwendigkeit für Schritte zur intrazellulären Ziele zu erreichen.
Mit dem obigen Ansatz abgeleitet wir eine RFD, das sein Substrat spaltet (FS1;. 2A) nur in Gegenwart des CEM hergestellt von E. coli (CEM-EG). 5 Diese E. coli-Sensing RFD, benannt RFD-EC1 (Abb. 2A), erwies sich als streng, die auf CEM-EG aber nicht reagierende zu CEMs aus einer Vielzahl von anderen Bakterien (Abb. 3).
Hier haben wir PreseNT die wichtigsten experimentellen Verfahren für die Einrichtung E. coli-Nachweis-Assays mit RFD-EC1 und repräsentative Ergebnisse.
Protokoll
1. Herstellung von chemischen Lösungen
- 0,5 M Ethylendiamintetraessigsäure (EDTA): In einem 2-Liter (L) Kunststoffbecher, wiegen 186,1 g EDTA (EM Science) und fügen Sie 800 Milliliter (ml) autoklaviertem deionisiertem destilliertem Wasser (ddH 2 O). Der pH-Wert 8,0 mit NaOH-Pellets (EM Science). Machen Sie das Endvolumen auf 1 L mit ddH 2 O. Die Lösung wird in Glasflaschen, Autoklaven und lagern bei 4 ° C.
- 10 × Tris-Borat-EDTA-Lösung (10 x TBE, 89 mM Tris, 89 mM Borsäure, 2 mM EDTA, pH 7,5): Wiegen Sie 432 g Tris-Base (BioShop Kanada) und 220 g Borsäure (BioShop Kanada) , und fügen Sie jeweils mit einem 4 L Plastikbecher. Messen Sie 80 ml 0,5 M EDTA (pH 8,0), und fügen Sie in das Becherglas. Fügen Sie ddH 2 O auf ein Gesamtvolumen von 4 L. gründlich mischen der Lösung mit einem Magnetrührer, bis Komponenten vollständig gelöst sind. Die Lösung wird in Glasflaschen, Autoklaven und lagern bei 4 ° C.
- 10% igen denaturierenden polyacrylamide Gel Lager: Zu einer 4 L Plastikbecher, fügen 1681,7 g Harnstoff (BioShop Kanada), 400 ml 10 x TBE, 1 L 40% Acrylamid / Bisacrylamid (29:1)-Lösung (BioShop Kanada). Stellen Sie die Lautstärke auf 4 L mit ddH 2 O. Das Auflösen der Harnstoff unter Rühren. Übertragen Sie die Lösung auf 1 L Braunglasflaschen und lagern bei 4 ° C. (Achtung! Acrylamid sollte daher mit Schutzhandschuhen, Mundschutz, Schutzbrillen und Kittel behandelt werden, weil es ein Nervengift ist vor der Polymerisation).
- 2 × Gelladepuffer (2 × GLB): In einen 200 ml-Becherglas, fügen Sie 44 g Harnstoff, 8 g Saccharose (BioShop Kanada), 10 mg Bromphenolblau (BioShop Kanada), 10 mg xylenecyanol FF (Sigma -Aldrich), 400 ul 10% Natriumdodecylsulfat (SDS; BioShop Kanada) und 4 ml 10 × TBE. Stellen Sie das Endvolumen auf 40 ml mit ddH 2 O lösen und die Feststoffe mit leichtem Erwärmen (50 ° C) und Rühren mit einem Magnetrührer bar. 1 ml Aliquots in 1,5 ml Reaktionsgefäße und lagern bei 4 ° C. Auf der Grundlage unsererErfahrung, ist es notwendig, Hitze 2 × GLB kurz bei 90 ° C vor dem Gebrauch, weil 2 × GLB erstarrt unter den Lagerungsbedingungen.
- 1 M Tris-HCl (pH 7,5): Wiegen 12,1 g Tris-Base in einem 200 ml Becherglas. 60 ml ddH 2 O lösen und die Feststoffe durch Rühren mit einem Magnetrührer. Der pH-Wert auf 7,5 mit 1 M HCl (Sigma-Aldrich). Machen Sie auf ein Volumen von 100 ml mit ddH 2 O und Transfer zu einer Glasflasche und Autoklaven. Lagerung bei 4 ° C.
- 5 M NaCl: Im Becherglas wiegen 58,4 g NaCl (BioShop Kanada) und löst mit 150 ml ddH 2 O. Stellen Sie die Lautstärke auf 200 ml mit ddH 2 O. Die Lösung wird in einer Glasflasche, Autoklaven und lagern bei 4 ° C.
- DNA Elutionspuffer: In einem Becherglas, mix 2 ml 1 M Tris-HCl (pH 7,5), 8 ml 5 M NaCl und 0,4 ml 0,5 M EDTA (pH 8,0). Stellen Sie die Lautstärke auf 200 ml mit ddH 2 O. Autoklavieren und bei 4 ° C.
- 2 × Reaktionspuffer (2 × RB; 100 mM HEPES, 300 mM NaCl, 30 mM MgCl 2): Zu einem 50 ml Falcon-Röhrchen (BD Falcon), fügen Sie 1,2 g HEPES (BioShop Kanada), 0,88 g NaCl und 0,30 g MgCl 2 • 6 H 2 O (EMD Chemicals) und 30 ml ddH 2 O. Mischen durch Schütteln leicht bis Komponenten vollständig gelöst sind. Den pH-Wert auf 7,5 durch Zugabe von 10 N NaOH-Lösung und stellen Sie den endgültigen Volumen von 50 ml mit ddH 2 O. Reinigen Sie die Lösung mit einer Spritze-driven Filtereinheit (0,22 um, Millipore) und bei 4 ° C.
- Luria Bertani (LB)-Bouillon: In einem Becherglas, wiegt 20,0 g LB-Pulver (Sigma-Aldrich), gefolgt von der Zugabe von 1 l ddH 2 O Gründlich mischen der Lösung mit einem Magnetrührer, die Lösung mit einem Erlenmeyerkolben. Autoklavieren Lösung und bei Raumtemperatur lagern.
- 1,5% LB-Agar: Wiegen 1,5 g Agar (BioShop Kanada) in einem 250 ml Kolben geben und 100 ml flüssiges LB. Autoklavieren Mischung und bei Raumtemperatur lagern.
- Agar Plating: Melt die LB-Agar in der Mikrowelle und die Lösung auf ~ 50 ° C. Füllen Sie die Lösung in Petrischalen (Fisher Scientific) unter einer Flamme. Nach unserer Erfahrung kann 100 ml LB-Agar-Platten liefern 6.5.
2. Bau der RFD-EC1 und RFSS1 durch Vorlage vermittelte enzymatische Ligation
RFD-EC1 (Abb. 2A) ist der Funktionsumfang DNAzym. Es besteht aus der katalytischen Sequenz EC1 und dem Substrat-Sequenz FS1 (angedeutet durch schwarze und grüne Linien in Abb.. 2A). RFSS1 (2A) eine verschlüsselte Version des RFD-EC1, wobei die katalytische Sequenz EC1 teilweise gemischt in SS1 aber die FS1 Abschnitt unverändert bleibt. RFD-EC1 und RFSS1 wurden durch Vorlage vorgenommen vermittelt enzymatische Ligation der Oligonukleotide FS1 mit Oligonukleotid EC1 oder SS1 in Gegenwart von LT1 als Ligationsmatrize (siehe Kasten eingefügt in Fig.. 2A). Das Verfahren für die Durchführung der Ligationsreaktion wird unten bereitgestellt.FS1 wurde von Keck Oligosynthese Einrichtungen an der Yale Universität erhalten, entschützt und gereinigt durch Gelelektrophorese nach einem vorher festgelegten Protokoll. 17-24 EC1, SS1 und LT1 von Integrated DNA Technologies erworben und mittels Gelelektrophorese.
- Bereiten Sie einen 100 pM Stammlösung von FS1, EC1, SS1 und LT1 mit ddH 2 O. Lagern Sie sie bei -20 ° C bis zur Verwendung.
- Übertragen Sie 5 ul der FS1 Stammlösung zu zwei 1,5 ul Mikrozentrifugenröhrchen als Tube 1 und Tube 2 markiert. Zu jedem Röhrchen, fügen 38,5 ul ddH 2 O und dann 5 ul 10 × T4 Polynukleotid-Kinase (PNK) Reaktionspuffer A (MBI Fermentas), die 500 mM Tris-HCl (pH 7,6, 25 ° C) enthält, 100 mM MgCl 2, 50 mM DTT, 1,0 mM Spermidin. Mischen Sie jede Lösung mit einer Pipette (Pipette die Lösung nach oben und unten ein paar Mal).
- Fügen Sie 1 ul ATP (100 mM; MBI Fermentas) und durch Pipettieren gründlich mischen.
- In 0,5 ul T4-Polynukleotidkinase (PNK; 10 Einheiten / ul; MBI Fermentas) und durch Pipettieren gründlich mischen. Inkubieren der Reaktionsmischung bei 37 ° C für 30 min. Vergewissern Sie sich, um die Rohre mit Alufolie abdecken zu minimieren Ausbleichen des Fluorophors.
- Die Reaktion wird durch Erhitzen bei 90 ° C für 5 min. Kühlen der Reaktionsmischung bei Raumtemperatur für 10 min.
- Fügen Sie 5 ul der EC1 und SS1-Stammlösung zu Tube 1 und Tube 2 haben.
- Fügen Sie 5 ul der LT1-Stammlösung in jedes Röhrchen, durch Pipettieren gründlich mischen. Erhitzen der Reaktionsmischung bei 90 ° C für 1 min und Abkühlen auf Raumtemperatur für 10 min.
- Fügen Sie 118 uL ddH 2 O und dann 20 ul 10 × T4-DNA-Ligase-Puffer (MBI Fermentas), die 400 mM Tris-HCl (pH 7,8 bei 25 ° C), 100 mM MgCl 2, 100 mM DTT und 5 enthält mM ATP. Mischen Sie die Lösung durch Pipettieren.
- Fügen Sie 2 ul T4 DNA-Ligase (5 Einheiten / ul; MBI Fermentas) und durch Pipettieren gründlich mischen. Inkubieren der Reaktionsmischung bei Raumtemperatur für 1 h.
- Je 20 ul 3 M NaOAc (pH 7,0) zu jedem Röhrchen, vortexen und Spin-Down. Fügen Sie 500 ul von kaltem 100% Ethanol zu jedem Röhrchen, mischen Sie die Lösung durch Vortexen und stellen Sie die Rohre in -20 ° C Gefrierschrank für 30 min.
- Zentrifugieren Sie die Mischungen bei 11.000 g für 20 min bei 4 ° C in einer Kühlzentrifuge (Allegra X22-R, Beckman Coulter) und entfernen Sie vorsichtig den Überstand mit einer Pipette.
- Trocknen der DNA-Pellet unter Verwendung eines DNA-Konzentrator (Savant DNA Speedvac Thermo Scientific) für 10 min.
- Resuspendieren DNA-Pellets in 30 ul 1 × Gelladepuffer (GLB), kurz vortexen und Spin-down mit einer Tischzentrifuge (Minizentrifuge, VWR Scientific). Die ligierte DNA Proben bereit zum Laden auf ein 10% DPAGE Gel.
3. Herstellung von 10% Gel DPAGE
Die folgenden Schritte beschreiben Sie kurz den Apparat der Gel-Elektrophorese und ihre Set-up. Für mehr Details über die Geräte, Einstellungen und Handhabung entnehmen Sie bitte to unsere bisher veröffentlichten Protokollen. 30,31
- Waschen und trocknen Sie zwei Glasplatten, zwei 0,75 mm Spacer und eine 16-Well-Kamm. Montieren Glasplatten und Abstandshalter mit Clips und lag horizontal auf einer ebenen Fläche mit gekerbten Glasplatte nach oben zeigt.
- Übertragen Sie 40 ml 10% DPAGE Mix in ein 150 mL Becherglas. In 40 ul Tetramethylethylendiamin (TEMED; Bioshop Kanada), 400 ul 10% APS, und durch Umschwenken mischen mit einer Pipette.
- Gießen Sie die Mischung vorsichtig zwischen den Platten und sofort den Kamm einsetzen. Man läßt das Gemisch für 10 bis 20 min polymerisiert. Die Polymerisation kann durch Überprüfung der Rest-Gel-Mischung im Becherglas verbleiben bestätigt werden.
- Einmal polymerisiert, nehmen Sie den Kamm vorsichtig und spülen Sie die Wells mit ddH 2 O, um das restliche Gel-Lösung in den Vertiefungen zu entfernen.
- Montieren Sie die Platten auf das Gel Elektrophorese-Vorrichtung mit dem ungekerbten rechteckigen Platte nach außen und legen Sie eine Metallplatte hinter dem Rastenplatte. Die Verwendung des Metalls plaß hilft, Überhitzung, die die Glasplatten knacken kann zu verhindern.
- In 1 × TBE, um den oberen und unteren Kammern der Vorrichtung. Stellen Sie sicher, dass die Vertiefungen mit dem Puffer gefüllt und die untere Kante des Gels wird in den Puffer eingetaucht.
- Tragen Sie einen 40 mA (oder 750 V) und Strom für 10 bis 15 min Vorlauf.
4. Die Reinigung von Liganden-RFD-EC1 und RFSS1 um 10% Gel DPAGE
- Nach dem Schritt von 3,7 gründlich Vertiefungen mit 1 × TBE Verwendung einer Spritze und einer Nadel.
- Laden Sie das Ligationsgemisch der RFD-EC1 und dass der RFSS1 (ab 2.13) in 2 Vertiefungen (eine für jeden) mit Hilfe einer Pipette und Gelbeladung Spitzen (DiaMed). Anwenden eines 40 mA (750 V) Strom bis zum unteren Farbstoff (Bromphenolblau) etwa 5 cm über dem unteren Rand der Platten.
- Entfernen Sie Glasplatten aus dem Gel laufende Apparatur, legen sich auf eine Flachbank oben und entfernen Sie vorsichtig die Abstandshalter.
- Entfernen Sie vorsichtig die obere Glasplatte aus dem Gel undWickeln Sie das Gel mit Plastikfolie (versuchen Sie, Fältchen und Falten der Wickel auf dem Gel zu vermeiden).
- Die ligierten Produkte visualisiert werden kann entweder durch UV-Schatten (260 nm) oder durch Durchleuchtung (360 nm), das eine DNA-Bande sichtbar wenige Zentimeter über EC1 oder SS1 (100 pmol EC1 oder SS1 produzieren kann als Marker verwendet werden und geladen in eine Vertiefung in dem Schritt von 4,2). Markieren Sie die gewünschten DNA-Banden mit einem Marker.
- Excise die DNA-Bande mit einem sterilen Rasierklingen, schneiden Sie das Gel in kleine Stücke schneiden und in ein frisches 1,5 ml Mikrozentrifugenröhrchen.
- Crush die Gel-Stücke innerhalb der Mikrozentrifugenröhrchen mit einer sterilen Pipettenspitze (200 &mgr; l Düsengröße).
- Fügen Sie 500 ul DNA-Elutionspuffer in jedes Röhrchen und bedecken Sie sie mit Alufolie, um die Fluorophore vor Licht zu schützen. Vortex die Proben 10 min.
- Zentrifugieren Sie die Proben bei 11.000 g für 4 min bei 4 ° C in einer Kühlzentrifuge (Allegra X22-R, Beckman Coulter) und transferieren es vorsichtig in 350 &mgr; l des supernatant in ein frisches 1,5 ml Mikrozentrifugenröhrchen (falls nötig, eine zweite Elution kann mit 350 ml frisches Elutionspuffer für weitere 10 Minuten durchgeführt werden).
- In 35 ul (0,1 × des Probenvolumens) von 3 M Natriumacetat (NaOAc, pH 7,0) zu jedem Röhrchen, vortexen und Spin-Down. Zusatz von 900 ul kaltem 100% Ethanol in jedes Röhrchen. Mischen jeder Probe durch Quittungsaustauschvorgang das Rohr für ein paar Sekunden. Die Röhrchen bei -20 ° C für mindestens 1 h.
- Zentrifugieren Sie die Proben bei 11.000 g für 20 min bei 4 ° C in einer Kühlzentrifuge und entfernen Sie vorsichtig den Überstand mit einer Pipette.
- Zugabe von 100 ul kaltem 70% igem Ethanol und verwenden, um vorsichtiges Abspülen die gesamte Innenwand des Rohres. Re-Zentrifuge bei 11.000 g für 7 min bei 4 ° C Entfernen Sie den Überstand und Pellet trocknen Sie die Verwendung des DNA-Konzentrator für 10 min.
- Lösen Sie die DNA-Pellets in 100 &mgr; l ddH 2 O und vortexen. Ermitteln Sie die DNA-Konzentration auf die UV-Absorption bei 260 nm, gemessen. Shop-Proben bei -20° C bis zur Verwendung.
5. Herstellung von Bakterien
- Das Ziel Bakterienstamm E. coli K12 (MG1655) und Steuereinrichtungen Stämme werden zunächst auf LB-Agarplatten ausplattiert aus Glycerin hat. Unter einer Flamme oder in einem biologischen Sicherheitsschrank, berühren Sie die bakteriellen Glycerin Lager mit einer sterilen Pipettenspitze vorsichtig und Streifen auf der Plattenoberfläche verhindert die Beschädigung des LB-Agar.
- Invert gestreift Platten und bei 37 ° C für 14 h. Nach der Inkubation versiegeln den gesamten Umfang der Platten mit Parafilm (Pechiney Plastic Packaging) und bei 4 ° C. Diese Platten können für maximal 4 Wochen gelagert werden.
6. Herstellung von rohem Extrazelluläre Mischungen (CEMs)
- Dosiernadel 2 ml LB in sterilen 14 ml Kulturröhrchen (BD Falcon) mit einer Pipette Pistole (Corning).
- Mit einer sterilen Pipettenspitze, wählen Sie eine einzelne Kolonie von einer Agarplatte in dem Schritt von 5,2 vorbereitet und setzen Sie sie in ACultur Rohr. Die Röhrchen in einem Inkubator (New Brunswick Scientific) bei 37 ° C eingestellt, und schütteln bei 250 rpm für 14 h.
- 1% Re-Impfung Kultur: Dispense 2 ml frisches LB in 14 ml Tuben und Kultur Spike mit 20 ul von Bakterienkulturen in Schritt 6.2 vorbereitet. Die Röhrchen bei 37 ° C unter Schütteln bei 250 Upm bis jede bakterielle Lösung erreicht ein OD 600 (optische Dichte bei 600 nm gemessen wird) von etwa 1. Um OD 600 messen, je 1 ml der jeweiligen Kultur zu einem Einweg umfüllen und messen Absorption bei 600 nm mit einem UV-Spektralphotometer (Genesys UV 10, Thermo Scientific).
- 1 ml von jeder Kultur in ein neues 1,5 ml Mikrozentrifugenröhrchen und Pellet Zellen durch Zentrifugation bei 11.000 g für 5 min bei Raumtemperatur.
- Übertragen Sie die klare Überstand in ein frisches 1,5 ml Mikrozentrifugenröhrchen und Lagerung bei -20 ° C, wenn nicht sofort verwendet wird.
7. Erkennung anhand der Fluoreszenz-Spektralphotometer
- Schalten Sie Fluoreszenz-Spektralphotometer (Cary-Eclipse, Varian Inc.) und richten Sie die Datenerfassung Parameter mit Anregung bei 488 nm und Emission bei 520 nm. Messwerte können jede Minute für 1 h eingenommen werden.
- Waschen 3 Quarz-Küvetten (Varian Cary) mit ddH 2 O, um 100% Ethanol. Trocknen Sie die Küvetten durch Blinken Stickstoffgas. Label-Küvetten C1 (Kontrolle 1), C2 (Kontrolle 2) und T (Test).
- Übertragung von 24 uL ddH 2 O bis C1 und 24 ul von CEM-EG bis C2 und T. hinzufügen 25 ul von 2 × RB zu jeder Küvette und legen sie in der Fluoreszenz-Spektralphotometer. Sammeln Fluoreszenzdaten für die ersten 5 Minuten.
- Fügen Sie 1 ul RFSS1 (aus einer 5 pM Stammlösung) bis C2 und 1 ul RFD-EC1 (von 5 pM Stammlösung) zu T und C1. Mischen Sie jede Lösung durch Pipettieren. Dies muss sorgfältig eingeleitet werden, so dass die Fluoreszenzmesswerte nicht unterbrochen werden. Lassen Sie die Reaktion für den Rest der 1 h Erfassungszeit fortzusetzen.
- Sparendie Daten im Excel-Dateiformat, übertragen die Daten an einen PC und Verarbeitung von Daten, um eine grafische Bild zu erzeugen.
8. Nachweis durch Gelelektrophorese
Die gleiche Reaktion in Schritt 7.4 Mischungen hergestellt werden können für die Analyse durch Gelelektrophorese verwendet werden, alternativ neue Reaktionen können ähnlich hergestellt werden gewaschen und in 1,5 ml Mikrozentrifugenröhrchen. In jedem Fall:
- Quench-Reaktionen (nach 1 h) durch Zugabe von 5 ul 3 M NaOAc und 125 ul 100% Ethanol. Mischen Sie jede Lösung durch Vortexen und stellen Sie die Rohre in -20 ° C Gefrierschrank für 1 h.
- Zentrifugieren Sie die Reaktionsmischungen bei 11.000 g für 20 min bei 4 ° C und entfernen Sie vorsichtig den Überstand mit einer Pipette.
- Trocknen der Pellets mit dem DNA-Konzentrator für 10 min.
- Resuspendieren Pellets in 20 ul 1 × GLB durch kurzes Vortexen. Spin down Röhren sehr kurz mit einer Tischzentrifuge (Minizentrifuge, VWR Scientific). Diese Proben werden geleseny zum Laden in eine DPAGE Gel.
- Eine 10% ige DPAGE Gel nach 3,1-3,7 beschrieben. Laden der Reaktion Proben (von Schritt 8.4) in die Vertiefungen mit einer Pipette und Ladepuffer Spitzen (Diamed). Anwenden eines 40 mA (750 V) Strom bis zum unteren Farbstoff (Bromphenolblau) etwa 5 cm über dem unteren Rand der Platten.
- Entfernen Sie Glasplatten und waschen gründlich mit Leitungswasser, um alle Gelstückchen entfernen. Wischen Sie mit einem Kimwipe Platten (Kimberly-Clark Professional).
- Scannen Sie das Gel für die Fluoreszenz-Platte mit einem Typhoon-Scanner (Typhoon 9200, Variable-Modus, GE Healthcare). Analysieren Sie die Daten mit Hilfe ImageQuant-Software (Molecular Dynamics).
9. Nachweis Spezifität
- Zum Testen bakterielle Spezifität, beschrieben die gleichen Verfahren zur Kultivierung von E. oben coli, kann die Herstellung CEM und Durchführung einer Spaltungsreaktion Assay für eine Reihe von verschiedenen Bakterienstämmen wie B. durchgeführt werden subtilis, P. Peli, Y. ruckeri, L. S.lanturum, P. acidilactici (gezeigt in. 3A).
10. Single Cell-Erkennung
Bereiten Sie eine 1 ml E. coli Glycerin Lager von 2 CFU / ml (KBE: Kolonie bildende Einheit) durch serielle Verdünnung und bestätigen CFU-Konzentration durch Plattieren 5 Diese Lager sollten 0,2 KBE/100 ul enthalten.. Bei -80 ° C bis zum Gebrauch.
- Bereiten 10 Kulturröhrchen mit 2 ml LB.
- Beimpfen jede Kultur mit 100 ul 2 CFU / ml Glycerin Lager und bei 37 ° C unter Schütteln bei 250 Umdrehungen pro Minute. Die Nutzung des gesamten Glycerin Lager (1 ml) sollte zu 10 Kulturen.
- Ernte 300 ul von jedem Impfkultur Rohrs an den folgenden Zeitpunkten: 4, 8, 12, 16 und 24 Stunden. Verlassen Sie die verbleibenden Kultur für 24 Stunden wachsen.
- Messen der OD 600 und fällt die Zellen durch Zentrifugation bei 11.000 g für 5 min.
- Übertragen Sie die CEMs an die frische 1,5 ml Reaktionsgefäße und bei -20 & DzB; C bis zur Verwendung.
Hinweis: Da es keine nachweisbare OD 600 sein kann, für die Proben zu den Zeitpunkten 4, 8 und 12 geerntet, damit das restliche Kultur für 24 Stunden wachsen, um Kulturen mit Bakterien (bestimmt durch Trübung und OD-Messung) zu identifizieren. Nur ein oder zwei der 10 Kulturen enthalten E. coli nach der Inokulation und die übrigen Rohre nicht enthalten keine Zellen. - Verwenden Sie die CEMs von positiven Kulturen (gelagert bei -20 ° C) an den dafür vorgesehenen Zeitpunkten wieder zu Spaltungsreaktionen mit RFD-EC1 vorzubereiten, und dann analysieren die Reaktionsgemische mit DPAGE Gelelektrophorese wie in Abschnitt 8 beschrieben.
11. Konzept und repräsentative Ergebnisse
Das Konzept der Nutzung einer RNA-spaltende fluoreszierende DNAzyme (RFD) für Nachweis von Bakterien ist in Abb.. 1. Die RFD spaltet einen chimären DNA / RNA-Substrates bei einer einsamen RNA-Bindung (blau R) von zwei Nukleotiden markiert flankiertmit einem Fluorophor (F) und einem Quencher (Q), jeweils. Als ein Bakterium von Interesse (wie zB E. coli) wächst in Medien, wird er hinter einem rohen extrazellulären Mischung (CEM) verlassen. Diese CEM als Ganze wird dann in einem in vitro-Selektion Experiment verwendet, um eine Reaktion, die RFD sich speziell auf das CEM zu erhalten, vermutlich die RFD wirkt mit einem spezifischen Moleküls (violetten Stern) in der CEM, die eine Signatur Molekül des Bakterium ist. Wenn der CEM der Reaktionslösung, enthaltend den RFD hinzugefügt wird, löst es die RNA-spaltende Aktivität des RFD. Die Spaltung bei trennt F aus F, wodurch man einen fluoreszierenden Signal, das entweder nachgewiesen werden kann mit einem Fluorimeter oder mittels Gelelektrophorese.
Die experimentelle Validierung der oben genannten Konzept wurde mit dem CEM von E. getan coli (CEM-EG). Wir erhalten 3 RFD Molekülen über In-vitro-Selektion, und die effizienteste wurde als RFD-EC1 (Abb. 2A) bezeichnet. 5 WE testeten die Spaltungsaktivität von RFD-EC1 (zusammen mit einer mutierten Sequenz mit dem Namen RFSS1) als Reaktion auf CEM-EG. Sowohl RFD-EC1 und RFSS1 wurden durch enzymatische Ligation der DNAzyme Portionen auf das Substrat FS1 (alle Sequenzen sind in Abb. 2A) vorbereitet. In der Fluoreszenzmessung Experiment (2B), wurde CEM-EG zu 5 min, durch die Zugabe von RFD-EC1 oder RFSS1 gefolgt, und von einer weiteren Inkubation für 55 weitere Minuten lang inkubiert. Die Fluoreszenzintensität der Lösung wurde kontinuierlich gelesen jede Minute und die Daten wurden verwendet, um relative Fluoreszenz berechnet (RF; als das Verhältnis der Fluoreszenzintensität zum Zeitpunkt t vs der Fluoreszenzintensität zum Zeitpunkt 0) berechnet. Die RF-Werte gegen die Zeit der Inkubation werden als Bild dargestellt. 2B. Es wurde festgestellt, dass RFD-EC1 ein hohes Maß an Fluoreszenz-Signal nach Zugabe von CEM-EG produziert; in krassem Gegensatz, RFSS1 führte nicht zu einer starken Fluoreszenz-Signal. Somit wird die Fluoreszenz erzeugenden function der RFD-EC1 bei Kontakt mit CEM-EG ist sequenzspezifisch.
Um zu überprüfen, dass die beobachtete Fluoreszenz Steigerungen sind aufgrund der Spaltung der RNA-Bindung, analysierten wir Reaktionsgemischen durch DPAGE. Spaltung von RFD-EC1 wird erwartet, dass zwei DNA-Fragmente, eine 5'-Fragment Halten des Fluorophor und ein 3'-Fragment Halten des Quencher zu erzeugen. Nur ungeschnitten RFD-EC1 (unclv) und die 5'-Fragment (CLV) konnte durch Fluoreszenz-Bildgebung erfasst werden. Die DPAGE Ergebnis gezeigt. 2C zeigt, dass das Reaktionsgemisch aus RFD-EC1 und CEM-EG tatsächlich das erwartete Spaltprodukt, während die Mischung RFSS1/CEM-EC nicht.
Die Spezifität der RFD-EC1 wurde unter Verwendung von CEM aus mehreren anderen Gram-negativen und gram-positive Bakterien gesammelt und die Daten werden in gezeigt. 3A. Nur die Probe mit CEM-EG (blaue Kurve) produzierte eine Zunahme der Fluoreszenz. Das Fehlen von Kreuzreaktivität mit CEMsvon den anderen Bakterien zeigt an, dass RFD-EC1 hochselektiv für E. ist coli.
Wir untersuchten auch die Zeit für das Kultivieren einer einzigen E. benötigt coli-Zelle, um eine ausreichende CEM, das die Spaltung RFD-EC1 induzieren kann zu erzeugen. Für dieses Experiment wurde ein E. coli enthaltenden Probe definiert KBE (koloniebildende Einheiten) wurde ausreichend verdünnt, um die Konzentration von 1 CFU / ml zu erreichen. Dies wurde durch Mischen von 100 ul der verdünnten Probe mit bakteriellen Wachstumsmedium und Kultivieren für 4, 8, 12, 16 und 24 h. CEMs wurden dann für jeden Zeitpunkt gesammelt und auf Induktion der Spaltungsaktivität der RFD-EC1. Die DPAGE Ergebnis gezeigt. 3B zeigt, dass ein Kultivieren von 12 Stunden benötigt wird.
Es ist wichtig zu beachten, dass die anfängliche kleines Signal Anstieg der Fluoreszenz-Messungen nach der Zugabe von RFSS1 Sequenz (als Negativkontrolle) an CEM-EC beobachtet (2B, rot Curve) oder RFD-EC1 andere bakterielle CEMs (3B; alle Kurven außer blau) mit der intrinsischen Fluoreszenz des FRQ-Modul (aufgrund unvollständiger Abschrecken von F von Q) zurückzuführen. Somit wird erwartet, dass die Zugabe von F-und Q-markierten Sequenzen würde eine erste Fluoreszenzanstieg zu erzeugen. Jedoch sind nur RFD-EC1/CEM-EC Mischungen in der Lage, ein hohes Maß an Fluoreszenz über die Zeit.

1. Schematische Darstellung der RNA-spaltende fluoreszierenden DNAzym (RFD)-Sonde, die fluoresziert bei Kontakt mit dem rohen extrazellulären Mischung (CEM) von spezifischen bakteriellen Zellen von Interesse produziert. Die RFD spaltet eine chimäre DNA / RNA-Substrat mit einer freien RNA Gestänge (Blau R) von zwei Nukleotiden mit einem Fluorophor (F) und einem Quencher (Q), die jeweils markierten flankiert. Vor der Spaltung ist die Fluoreszenz Niveau des RFD minimal durch die enge proximity von F und Q Nach der Spaltung von F Q abweicht, was dazu führt, eine starke Fluoreszenz erzeugt wird.
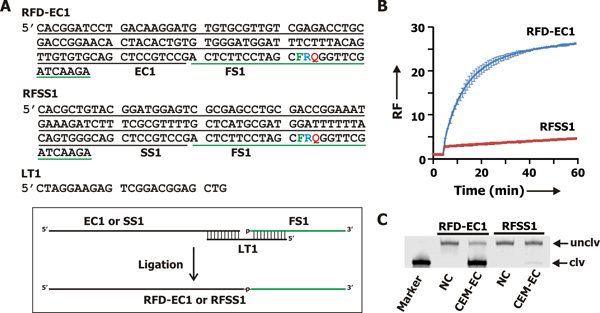
Abbildung 2. Die E. coli-Sensing-RFD. (A) RFD-EC1 ist die DNAzym Sonde, die durch CEM-EG aktiviert werden kann. RFSS1 ist eine Sequenz von verwürfelten RFD-EC1 als Kontrolle verwendet. RFD-EC1 und RFSS1 wurden durch Ligation FS1 mit EC1 bzw. SS1, in Gegenwart von LT1 als Matrize. F: Fluorescein-modifizierten desoxythymidin. Q: Dabcyl-modifizierte desoxythymidin. R: Adenin Ribonukleotid. (B) Fluoreszenzsignal Profile RFD-EC1 und RFSS1 in Gegenwart von CEM-EG. (C) DPAGE Analyse der Spaltprodukte Reaktionsgemische in B (Reaktionszeit: 60 min). Abgebildet ist ein Fluoreszenz-Bild des DPAGE erhaltene Gel mit von Typhoon-Scanner. Lane NC: RFD-EC1 oder RFSS1 im Reaktionspuffer allein; Lane CEM-EC: RFD-EC1 oder RFSS1 in der Reaktion Puffer mit CEM-EG. Marker: RFD-CE1 behandelt mit 0,25 N NaOH, ist ein Verfahren bekannt, um volle Spaltung von RNA führen kann. unclv: ungespaltenen RFD-EC1. CLV: die Spaltung Fragment, welches das Fluorophor.
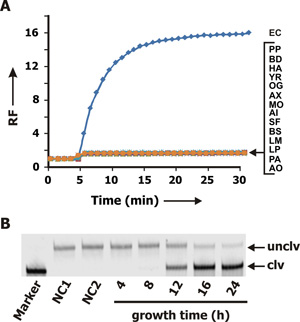
Abbildung 3. (A) Fluoreszenz-Signal-Profil von RFD-EC1 in CEMs von verschiedenen bakteriellen Zellen hergestellt. EG: Escherichia coli-K12; PP: Pseudomonas Peli; BD: Brevundimonas diminuta; HA: Hafnia alvei; YR: Yersinia ruckeri; OG: Ochrobactrum grignonese; AX: Achromobacter xylosoxidans; MO: Moraxella osloensis; KI: Acinetobacter lwoffi; SF: Serratia fonticola; BS: Bacillus subtilis; LM: Leuconostoc mesenteroides; LP: Lactobacillus planturum; PA: Pediococcus acidilactici; AO: Actinomyces orientalis. Jeder CEM Probe wurde für 5 min durch die Zugabe von RFD-EC1 inkubiert, gefolgt. (B) DPAGEAnalyse der RFD-EC1/CEM-EC Mischungen nach einer 60-min-Reaktion. Lane NC1: RFD-EC1 im Reaktionspuffer allein. Lane NC2: RFD-EC1 im Reaktionspuffer, CEM-BS (die CEM hergestellt aus Bacillus subtilis). Die Gassen mit 4, 8, 12, 16 und 24 beschriftet: RFD-EC1 in der Reaktion Puffer mit CEM-EG aus der Bakterienkultur mit einer einzigen E. genommen coli-Zelle nach einer Wachstumszeit von 4, 8, 12, 16 und 24 h beträgt.
Diskussion
Die meisten der häufigsten bakteriellen Nachweismethoden sind heute entweder langsam (klassische mikrobielle) oder technisch anspruchsvolle (Antikörper, PCR). Daher glauben wir, dass die nächste Generation von Tools zur Erkennung sollte nach Schnelligkeit und Einfachheit gerecht zu werden. Zu diesem Zweck haben wir eine RNA-spaltende und Fluoreszenz-Signal DNAzym, mit dem einfache Assays zu entwickeln, um die Anwesenheit von Bakterien durch die Erzeugung eines Fluoreszenzsignals berichten kann geschaffen werden. Die vorgestellten DNAzym Sonde RFD-EC1, wird durch die CEM während das Wachstum von E. hergestellt aktiviert coli in Kulturmedien. Da unsere Methode verwendet Rohöl extrazellulären Mischungen aus einem Bakterium als Ziel der Erkennung und umgeht das mühsame Ziel Extraktion und Amplifikation Schritte, es verwendet werden kann die Einrichtung sehr einfach werden, "Mix-and-read"-Typ von Tests zur Erkennung bakterieller. Die Nutzung unserer DNAzym ist nicht auf fluoreszenzbasierten Verfahren beschränkt. Zum Beispiel, mit kolorimetrischen Nachweis der gleichen DNAzym System Assay cein entworfen mit einer Methode, die bereits früher berichtet Rolling-Circle-Amplifikation nutzt in Verbindung mit einem organischen Farbstoff werden. 32 Wir sehen den Einsatz von DNAzymen für Nachweis von Bakterien als attraktiver Weg zu neuen bakteriellen Biosensoren mit größerer Einfachheit im Betrieb zu generieren.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Finanzierung dieser Arbeit wurde vom Natural Sciences and Engineering Research Council von Kanada (NSERC) und der Sentinel Bioaktive Paper Network zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer oder Modell | |
| Agar | BioShop Kanada | AGR003 | |
| Ammoniumpersulfat (APS) | BioShop Kanada | AMP001 | |
| Acrylamid / Bis-Acrylamid (40%, 29:1) | BioShop Kanada | ACR004 | |
| Borsäure | BioShop Kanada | BOR001 | |
| Bromphenolblau | Sigma-Aldrich | B8026 | |
| EDTA | EM Science | EXO539-1 | |
| HCl | Sigma-Aldrich | 38281 | |
| HEPES | Bioshop Kanada | HEP001 | |
| LB-Brühe | Sigma-Aldrich | L3022 | |
| MgCl 2 | EMDChemicals | B10149-34 | |
| NaCl | BioShop Kanada | SOD002 | |
| NaOAc | EMD Chemicals | SXO255-1 | |
| NaOH | EMD Chemicals | SXO590-1 | |
| SDS | BioShop Kanada | SDS001 | |
| TEMED | BioShop Kanada | TEM001 | |
| Tris-Base | BioShop Kanada | BST666 | |
| Tween 20 | Sigma-Aldrich | P9416 | |
| Harnstoff | BioShop Kanada | URE001 | |
| Xylenecyanol FF | Sigma-Aldrich | X4126 | |
| DNA-Konzentrator | Thermo Scientific | Savant DNA SpeedVac 120 | |
| Millex Filtereinheit | Millipore | SLGP033RS | |
| Tipps zum Einlegen von Gel | Diamed | TEC200EX-K | |
| ImageQuant-Software | Molecular Dynamics | Version 5.0 | |
| Kimwipes | Kimberly-Clark Professional | 34705 | |
| Mini Vortexer | VWR | 58816-121 | |
| Parafilm | Pechiney Plastic Packaging | PM996 | |
| Petrischalen | Fisher Scientific | Fisherbrand 08-757-12 | |
| Stripettor Plus (Pipetten-Pistole) | Corning | 07764714 | |
| Quarzküvetten | Varian Inc. | 66-100216-00 | |
| Shaker / Incubator | New Brunswick Scientific | Classic-Serie C24 | |
| Typhoon-Scanner | GE Healthcare | 9200 Variable-Modus | |
| Zentrifugieren | Beckman Coulter | Allegra X22-R | |
| UV-Spektrophotometers | Thermo Scientific | GenesysUV 10 | |
| Fluoreszenz-Spektralphotometer | Varian Inc. | Cary-Eclipse |
Referenzen
- Zourob, M., Elwary, S., Truner, A. . Principles of Bacterial Detection: Biosensors, Recognition Receptors and Microsystems. , (2008).
- Call, D. R. Challenges and Opportunities for Pathogen Detection Using DNA Microarrays. Crit. Rev. Microbiol. 31, 91-99 (2005).
- Lazcka, O., Campo, D. e. l., J, F., Muñoz, F. X. Pathogen detection: A perspective of traditional methods and biosensors. Biosens. Bioelectron. 22, 1205-1217 (2007).
- Velusamy, V. An overview of foodborne pathogen detection: In the perspective of biosensors. Biotechnol. Adv. 28, 232-254 (2010).
- Ali, M. M. Fluorogenic DNAzyme Probes as Bacterial Indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Liu, J., Cao, Z., Lu, Y. Functional Nucleic Acid Sensors. Chem. Rev. 109, 1948-1998 (2009).
- Li, Y., Lu, Y. . Functional Nucleic Acids for Analytical Applications. , (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Joyce, G. F. Forty Years of In Vitro Evolution. Angew. Chem. Int. Ed. 46, 6420-6436 (2007).
- Breaker, R. R., Joyce, G. F. A DNA enzyme that cleaves RNA. Chem. Biol. 1, 223-229 (1994).
- Cuenoud, B., Szostak, J. W. A DNA metalloenzyme with DNA ligase activity. Nature. 375, 611-614 (1995).
- Chinnapen, D. J., Sen, D. A deoxyribozyme that harnesses light to repair thymine dimers in DNA. Proc. Natl. Acad. Sci. U. S. A. 101, 65-69 (2004).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Silverman, S. K. DNA as a versatile chemical component for catalysis, encoding, and stereocontrol. Angew. Chem. Int. Ed. 49, 7180-7201 (2010).
- Mei, S. H. An efficient RNA-cleaving DNA enzyme that synchronizes catalysis with fluorescence signaling. J. Am. Chem. Soc. 125, 412-420 (2003).
- Liu, Z. Assemblage of signaling DNA enzymes with intriguing metal-ion specificities and pH dependences. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Kandadai, S. A., Li, Y. Characterization of a catalytically efficient acidic RNA-cleaving deoxyribozyme. Nucleic Acids Res. 33, 7164-7175 (2005).
- Rupcich, N. Quenching of fluorophore-labeled DNA oligonucleotides by divalent metal ions: implications for selection, design, and applications of signaling aptamers and signaling deoxyribozymes. J. Am. Chem. Soc. 128, 780-790 (2005).
- Shen, Y., Brennan, J. D., Li, Y. Characterizing the secondary structure and identifying functionally essential nucleotides of pH6DZ1, a fluorescence-signaling and RNA-cleaving deoxyribozyme. Biochemistry. 44, 12066-12076 (2005).
- Chiuman, W., Li, Y. Revitalization of six abandoned catalytic DNA species reveals a common three-way junction framework and diverse catalytic cores. J. Mol. Biol. 357, 748-754 (2006).
- Chiuman, W., Li, Y. Evolution of high-branching deoxyribozymes from a catalytic DNA with a three-way junction. Chem. Biol. 13, 1061-1069 (2006).
- Shen, Y. Catalysis and rational engineering of trans-acting pH6DZ1, an RNA-cleaving and fluorescence-signaling deoxyribozyme with a four-way junction structure. Chem BioChem. 7, 1343-1348 (2006).
- Ali, M. M., Kandadai, S. A., Li, Y. Characterization of pH3DZ1 - An RNA-cleaving deoxyribozyme with optimal activity at pH 3. Can. J. Chem. 85, 261-273 (2007).
- Chiuman, W., Li, Y. Efficient signaling platforms built from a small catalytic DNA and doubly labeled fluorogenic substrates. Nucleic Acids Res. 35, 401-405 (2007).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA MgZ based on a non-classic allosteric design. PLoS ONE. 2, e1224 (2007).
- Shen, Y. Entrapment of fluorescence signaling DNA enzymes in sol gel-derived materials for metal ion sensing. Anal. Chem. 79, 3494-3503 (2007).
- Kandadai, S. A. Characterization of an RNA-cleaving deoxyribozyme with optimal activity at pH 5. Biochemistry. 48, 7383-7391 (2009).
- Zhao, W., Brook, M. A., Li, Y. Periodic assembly of nanospecies on repetitive DNA sequences generated on gold nanoparticles by rolling circle amplification. Methods Mol. Biol. 474, 79-90 (2008).
- Navani, N. K., Mok, W. K., Li, Y. In vitro selection of protein-binding DNA aptamers as ligands for biosensing applications. Methods Mol. Biol. 504, 399-415 (2009).
- Ali, M. M., Li, Y. Colorimetric sensing by using allosteric-DNAzyme-coupled rolling circle amplification and a peptide nucleic acid-organic dye probe. Angew. Chem. Int. Ed. 48, 3512-3515 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten