Method Article
Détection des bactéries à l'aide ADNzymes fluorogènes
Dans cet article
Résumé
Nous avons récemment rapporté une nouvelle approche pour générer des sondes DNAzyme fluorogènes qui peuvent être appliqués à mettre en place un simple, «mix-and-lire" immunofluorescence pour la détection bactérienne. Ces sondes d'ADN spécifiques catalyser le clivage d'un chromophore de modification d'ADN-ARN substrat chimérique en présence d'un mélange brut extracellulaire (LEC) produite par une bactérie spécifique, traduisant ainsi la détection de bactéries dans la génération du signal de fluorescence. Dans ce rapport, nous allons décrire les principaux procédures expérimentales où une sonde DNAzyme spécifique notée "RFD-EC1" est employé pour la détection de la bactérie modèle, Escherichia coli (E. coli).
Résumé
Outbreaks linked to food-borne and hospital-acquired pathogens account for millions of deaths and hospitalizations as well as colossal economic losses each and every year. Prevention of such outbreaks and minimization of the impact of an ongoing epidemic place an ever-increasing demand for analytical methods that can accurately identify culprit pathogens at the earliest stage. Although there is a large array of effective methods for pathogen detection, none of them can satisfy all the following five premier requirements embodied for an ideal detection method: high specificity (detecting only the bacterium of interest), high sensitivity (capable of detecting as low as a single live bacterial cell), short time-to-results (minutes to hours), great operational simplicity (no need for lengthy sampling procedures and the use of specialized equipment), and cost effectiveness. For example, classical microbiological methods are highly specific but require a long time (days to weeks) to acquire a definitive result.1 PCR- and antibody-based techniques offer shorter waiting times (hours to days), but they require the use of expensive reagents and/or sophisticated equipment.2-4 Consequently, there is still a great demand for scientific research towards developing innovative bacterial detection methods that offer improved characteristics in one or more of the aforementioned requirements. Our laboratory is interested in examining the potential of DNAzymes as a novel class of molecular probes for biosensing applications including bacterial detection.5
DNAzymes (also known as deoxyribozymes or DNA enzymes) are man-made single-stranded DNA molecules with the capability of catalyzing chemical reactions.6-8 These molecules can be isolated from a vast random-sequence DNA pool (which contains as many as 1016 individual sequences) by a process known as "in vitro selection" or "SELEX" (systematic evolution of ligands by exponential enrichment).9-16 These special DNA molecules have been widely examined in recent years as molecular tools for biosensing applications.6-8
Our laboratory has established in vitro selection procedures for isolating RNA-cleaving fluorescent DNAzymes (RFDs; Fig. 1) and investigated the use of RFDs as analytical tools.17-29 RFDs catalyze the cleavage of a DNA-RNA chimeric substrate at a single ribonucleotide junction (R) that is flanked by a fluorophore (F) and a quencher (Q). The close proximity of F and Q renders the uncleaved substrate minimal fluorescence. However, the cleavage event leads to the separation of F and Q, which is accompanied by significant increase of fluorescence intensity.
More recently, we developed a method of isolating RFDs for bacterial detection.5 These special RFDs were isolated to "light up" in the presence of the crude extracellular mixture (CEM) left behind by a specific type of bacteria in their environment or in the media they are cultured (Fig. 1). The use of crude mixture circumvents the tedious process of purifying and identifying a suitable target from the microbe of interest for biosensor development (which could take months or years to complete). The use of extracellular targets means the assaying procedure is simple because there is no need for steps to obtain intracellular targets.
Using the above approach, we derived an RFD that cleaves its substrate (FS1; Fig. 2A) only in the presence of the CEM produced by E. coli (CEM-EC).5 This E. coli-sensing RFD, named RFD-EC1 (Fig. 2A), was found to be strictly responsive to CEM-EC but nonresponsive to CEMs from a host of other bacteria (Fig. 3).
Here we present the key experimental procedures for setting up E. coli detection assays using RFD-EC1 and representative results.
Protocole
1. Préparation de solutions chimiques
- 0,5 M d'acide éthylènediaminetétraacétique (EDTA): Dans un 2 litres (L) récipient en plastique, pèse 186,1 g d'EDTA (EM Science) et ajouter 800 ml (ml) de autoclavé désionisée de l'eau distillée (FD 2 O). Ajuster le pH à 8,0 à l'aide des pastilles de NaOH (EM Science). Faire le volume final à 1 L avec ddH 2 O. Transférer la solution dans des bouteilles en verre, autoclave et conserver à 4 ° C.
- 10 × Tris-borate de solution d'EDTA (10 × TBE; 89 mM de Tris, 89 mM d'acide borique, EDTA 2 mM, pH 7,5): peser 432 g de Tris-base (BioShop Canada) et 220 g d'acide borique (BioShop Canada) , et ajouter chacun à un récipient en plastique de 4 L. Mesurer 80 ml d'EDTA 0,5 M (pH 8,0) et ajouter dans le bécher. Ajouter ddH 2 O pour un volume final de 4 L. Bien mélanger la solution avec un agitateur magnétique jusqu'à composants soient complètement dissous. Transférer la solution dans des bouteilles en verre, autoclave et conserver à 4 ° C.
- 10% de dénaturation polyacrylamide gel de stock: Pour un bécher en plastique de 4 L, ajouter 1681,7 g d'urée (BioShop Canada), 400 ml de 10 × TBE, 1 L de 40% d'acrylamide / bisacrylamide (29:1) solution (BioShop Canada). Réglez le volume à 4 L avec ddH 2 O. Dissoudre l'urée en agitant. Transférer la solution dans des bouteilles de 1 L en verre ambré et conserver à 4 ° C. (Attention! L'acrylamide doit être manipulé avec des gants, un masque, des lunettes et une blouse de laboratoire, car elle est une neurotoxine avant la polymérisation).
- 2 × tampon de chargement de gel (2 × GLB): Pour un bécher de 200 ml en verre, ajouter 44 g d'urée, 8 g de saccharose (BioShop Canada), 10 mg de bleu de bromophénol (BioShop Canada), 10 mg de xylenecyanol FF (Sigma -Aldrich), 400 ul de dodécylsulfate de sodium 10% (SDS; BioShop Canada), et 4 ml de 10 × TBE. Réglez le volume final à 40 ml avec ddH 2 O et de dissoudre les solides avec un chauffage modéré (50 ° C) et en agitant avec un barreau magnétique. Transférer 1 ml aliquote dans 1,5 ml microtubes et conserver à 4 ° C. Sur la base de notrel'expérience, il est nécessaire à la chaleur 2 x brièvement GLB à 90 ° C avant de les utiliser parce que 2 × GLB se solidifie sous la condition de stockage.
- 1 M Tris-HCl (pH 7,5): Peser 12,1 g de Tris-base dans un bécher de 200 ml en verre. Ajouter 60 ml de ddH 2 O et de dissoudre les solides par agitation avec un agitateur magnétique. Ajuster le pH à 7,5 en utilisant HCl 1 M (Sigma-Aldrich). Compléter le volume à 100 ml avec ddH 2 O et de transférer à une bouteille en verre et autoclave. Conserver à 4 ° C.
- 5 M de NaCl: En récipient en verre pèse 58,4 g de NaCl (BioShop Canada) et de dissoudre avec 150 ml d'ddH 2 O. Réglez le volume à 200 ml avec ddH 2 O. Transférer la solution dans une bouteille en verre, dans un autoclave et conserver à 4 ° C.
- Tampon d'élution d'ADN: Dans un bécher en verre, mélanger 2 ml de 1 M Tris-HCl (pH 7,5), 8 ml de NaCl 5 M et 0,4 ml d'EDTA 0,5 M (pH 8,0). Réglez le volume à 200 ml avec ddH 2 O. Autoclave et conserver à 4 ° C.
- 2 × tampon de réaction (2 × RB; 100 mM d'HEPES, 300 mM NaCl, 30 mM MgCl 2): pour un tube de 50 ml Falcon (BD Falcon), ajouter 1,2 g d'HEPES (BioShop Canada), 0,88 g de NaCl et 0,30 g de MgCl 2 • 6H 2 O (EMD Chemicals) et 30 ml de ddH 2 O. Mélanger en secouant légèrement jusqu'à ce que les composants soient complètement dissous. Ajuster le pH à 7,5 par ajout de NaOH 10 N solution et régler le volume final de 50 mL avec ddH 2 O. Purifier la solution en utilisant une unité à seringue filtre (0,22 um, Millipore) et conserver à 4 ° C.
- Luria Bertani (LB) Bouillon: Dans un bécher, peser 20,0 g de poudre de LB (Sigma-Aldrich), suivie par l'addition de 1 L de ddH 2 O. Bien mélanger la solution avec un agitateur magnétique, transférer la solution dans une fiole conique. Autoclaver la solution et stocker à température ambiante.
- LB agar 1,5%: Peser 1,5 g d'agar-agar (BioShop Canada) dans un flacon de 250 ml et ajouter 100 ml de LB liquide. Autoclaver le mélange et le conserver à température ambiante.
- Placage Agar: Melt la gélose LB dans un micro-ondes et laisser refroidir la solution à ~ 50 ° C. Versez la solution dans des boîtes de Pétri (Fisher Scientific) en vertu d'une flamme. Dans notre expérience, 100 ml de gélose LB peut donner 5-6 plaques.
2. Construction de RFD-EC1 et RFSS1 par modèle Médiation ligature enzymatique
RFD-EC1 (Fig. 2A) est la vedette DNAzyme. Il se compose de l'EC1 séquence catalytique et la séquence du substrat FS1 (indiquées par des lignes noires et vertes de la Fig. 2A). RFSS1 (Fig. 2A) est une version brouillée de RFD-EC1 où le EC1 séquence catalytique est partiellement mélangé dans SS1, mais la partie FS1 reste inchangé. RFD-EC1 et RFSS1 sont faites par l'intermédiaire de modèle de ligature enzymatique de l'oligonucléotide avec FS1 EC1 oligonucléotide ou SS1, en présence d'LT1 que le modèle de ligature (voir la zone inséré dans la Fig. 2A). La procédure à suivre pour effectuer la réaction de ligature est fourni ci-dessous.FS1 a été obtenue à partir des installations de synthèse Keck Oligo à l'Université Yale, déprotégé et purifié par électrophorèse sur gel en suivant un protocole déjà établi. 17-24 EC1, SS1 et LT1 ont été achetés auprès de Integrated DNA Technologies et purifié par électrophorèse sur gel.
- Préparer une solution 100 uM stock de FS1, EC1, SS1 et LT1 utilisant ddH 2 O. Rangez-les à -20 ° C jusqu'à son utilisation.
- Transférer 5 ul de la solution mère FS1 à deux microtubes 1,5 ul marqués comme Tube Tube 1 et 2. Pour chaque tube, ajouter 38,5 ul de ddH 2 O, puis 5 pi de 10 × T4 polynucléotide kinase (PNK) Un tampon de réaction (MBI Fermentas), qui contient 500 mM Tris-HCl (pH 7,6, 25 ° C), 100 mM MgCl 2, 50 mM DTT, 1,0 mM spermidine. Mélanger chaque solution par pipetage (pipeter la solution de haut en bas à quelques reprises).
- Ajouter 1 ul d'ATP (100 mM; MBI Fermentas) et mélanger par pipetage.
- Ajouter 0,5 ul de la T4 polynucléotide kinase (PNK; 10 unités / ul; MBI Fermentas) et mélanger par pipetage. Incuber les mélanges réactionnels à 37 ° C pendant 30 min. Assurez-vous que pour couvrir les tubes de papier d'aluminium afin de minimiser photoblanchiment du fluorophore.
- Stopper la réaction par chauffage à 90 ° C pendant 5 min. Refroidir les mélanges de réaction à température ambiante pendant 10 min.
- Ajouter 5 pi de l'EC1 et SS1 solution mère dans le tube 1 et le tube 2, respectivement.
- Ajouter 5 ul de la solution mère LT1 à chaque tube, mélanger par pipetage. Chauffer les mélanges réactionnels à 90 ° C pendant 1 min et laisser refroidir à température ambiante pendant 10 min.
- Ajouter 118 ul de ddH 2 O puis 20 ul de 10 × T4 ADN ligase tampon (MBI Fermentas), qui contient 400 mM Tris-HCl (pH 7,8 à 25 ° C), 100 mM MgCl 2, 100 mM de DTT, et 5 mM d'ATP. Mélanger la solution par pipetage.
- Ajouter 2 ul d'ADN ligase T4 (5 unités / pl; MBI Fermentas) et mélanger par pipetage. Incuber les mélanges réactionnels à température ambiante pendant 1 h.
- Ajouter 20 ul de NaOAc 3 M (pH 7,0) dans chaque tube, vortex et spin down. Ajouter 500 ul d'éthanol à 100% à froid dans chaque tube, mélanger la solution au vortex et placer les tubes dans -20 ° C congélateur pendant 30 min.
- Centrifuger les mélanges à 11 000 g pendant 20 min à 4 ° C dans une centrifugeuse réfrigérée (Allegra X22-R, Beckman Coulter) et retirez soigneusement le surnageant par pipetage.
- Sécher le culot d'ADN à l'aide d'un concentrateur d'ADN (ADN Savant Speedvac, Thermo Scientific) pendant 10 min.
- Remettre les culots d'ADN dans 30 pl de tampon de charge 1 x gel (GLB), homogénéiser brièvement au vortex et spin down avec une centrifugeuse de paillasse (Minicentrifuge, VWR Scientific). Les échantillons d'ADN ligaturés sont prêts pour le chargement sur un gel dPAGE 10%.
3. Préparation du gel dPAGE 10%
Les étapes suivantes décrivent brièvement l'appareil d'électrophorèse sur gel et de son set-up. Pour plus de détails sur les appareils, les réglages et la manipulation, s'il vous plaît se référer to nos protocoles publiés précédemment. 30,31
- Lavez et séchez les deux plaques de verre, deux espaceurs 0,75 mm et d'un peigne de 16 puits. Monter les plaques de verre et les entretoises avec des clips et de jeter à l'horizontale sur une surface plane avec plaque de verre entaillé vers le haut.
- Transfert de 40 ml de mélange dPAGE 10% dans un bécher de 150 ml. Ajouter 40 ul de tétraméthyléthylènediamine (TEMED; Bioshop Canada), 400 ul de l'APS 10%, et le mélange en remuant avec une pipette.
- Verser le mélange soigneusement entre les plaques et insérez immédiatement le peigne. Laisser le mélange à polymériser de 10 à 20 min. La polymérisation peut être confirmée en vérifiant la composition de gel résiduel dans le bécher.
- Une fois polymérisé, enlever le peigne doucement et rincer le puits avec ddH 2 O pour éliminer la solution de gel résiduel dans le puits.
- Monter les plaques sur l'appareil d'électrophorèse sur gel avec la plaque rectangulaire entaille vers l'extérieur et placer une plaque de métal derrière la plaque crantée. L'utilisation de la pl métalliquemangé aide à éviter la surchauffe qui peut casser les plaques de verre.
- Ajouter une TBE × aux chambres supérieure et inférieure de l'appareil. Vérifier que les puits sont remplis avec le tampon et le bord inférieur du gel est immergé dans le tampon.
- Appliquer une mA 40 (ou 750 V) en cours et de pré-exécution de 10 à 15 min.
4. Purification de ligaturé RFD-EC1 et RFSS1 par Gel dPAGE 10%
- Après l'étape de 3,7, rincer les puits avec 1 TBE × aide d'une seringue et une aiguille.
- Chargez le mélange de ligature des RFD-EC1 et celle de RFSS1 (de 2,13) dans 2 puits (un pour chaque) en utilisant une pipette et des conseils de chargement de gel (DiaMed). Appliquer une 40 mA (750 V) en cours jusqu'à ce que le colorant de fond (bleu de bromophénol) est d'environ 5 cm au-dessus du bord inférieur des plaques.
- Retirer des plaques de verre de l'appareil marche gel, s'allonger sur une paillasse à plat et retirez soigneusement les entretoises.
- Retirez délicatement les plaques de verre haut du gel etenvelopper le gel d'une pellicule plastique (essayez d'éviter les rides et de pliage de la enveloppements sur le gel).
- Les produits ligaturés peut être visualisée par ombrage soit UV (260 nm) ou par transillumination (360 nm), qui produisent une bande d'ADN visibles quelques centimètres au-dessus EC1 ou SS1 (100 pmol de EC1 ou SS1 peut être utilisée comme marqueur et chargée dans un puits dans l'étape consistant à 4,2). Marquer les bandes d'ADN souhaités avec un marqueur.
- D'accise de la bande d'ADN avec des lames de rasoir stériles, couper le gel en petits morceaux et le transfert dans une nouvelle tube de 1,5 ml de microcentrifugation.
- Ecrasez les morceaux de gel dans le microtube l'aide d'un embout stérile pipette (200 pi taille de la pointe).
- Ajouter 500 pi de tampon d'élution d'ADN dans chaque tube et les couvrir avec une feuille d'aluminium pour protéger les fluorophores de la lumière. Vortex les échantillons pendant 10 min.
- Centrifuger les échantillons à 11.000 g pendant 4 min à 4 ° C dans une centrifugeuse réfrigérée (Allegra X22-R, Beckman Coulter) et transférer soigneusement 350 pi de l'artupernatant à une nouvelle tube de 1,5 ml de microcentrifugation (si nécessaire, une seconde élution peut être fait avec 350 ul de tampon d'élution frais pendant 10 min).
- Ajouter 35 ul (0,1 × du volume de l'échantillon) acétate de sodium 3 M (NaOAc, pH 7,0) dans chaque tube, mélanger au vortex et centrifuger. Ajouter 900 uL d'éthanol à 100% à froid dans chaque tube. Mélanger chaque échantillon par se serrer la main le tube pendant quelques secondes. Placer les tubes à -20 ° C pendant au moins 1 h.
- Centrifuger les échantillons à 11.000 g pendant 20 min à 4 ° C dans une centrifugeuse réfrigérée et retirez soigneusement le surnageant par pipetage.
- Ajouter 100 ul d'éthanol à 70% froid et l'utiliser pour rincer doucement toute la paroi intérieure du tube. Re-centrifuger à 11000 g pendant 7 min à 4 ° C. Eliminer le surnageant et sécher le culot à l'aide du concentrateur d'ADN pour 10 min.
- Dissoudre les culots d'ADN dans 100 ul de ddH 2 O et vortex. Déterminer la concentration en ADN sur la base de l'absorbance UV à 260 nm. Conserver les échantillons à -20° C jusqu'à utilisation.
5. Préparation des bactéries
- L'objectif souche bactérienne E. coli K12 (MG1655) et des souches de contrôle pertinentes sont d'abord étalées sur des plaques d'agar LB à partir de stocks de glycérol. En vertu d'une flamme ou au sein d'une enceinte de sécurité biologique, toucher le stock de glycérol bactérienne avec une pointe de pipette stérile et doucement strie sur la surface de la plaque pour éviter d'endommager la gélose LB.
- Inverser les plaques striées et incuber à 37 ° C pour 14 h. Après incubation, sceller le pourtour des plaques avec du Parafilm (Pechiney Plastic Packaging) et conserver à 4 ° C. Ces plaques peuvent être stockées pour une durée maximale de 4 semaines.
6. Préparation de mélanges bruts extracellulaires (CEM)
- Distribuer 2 ml de LB en stériles tubes 14 ml de culture (BD Falcon) en utilisant une arme à feu pipette (Corning).
- En utilisant une pointe de pipette stérile, prélever une colonie isolée à partir d'une plaque de gélose préparé à l'étape de 5,2 et l'insérer dans actube de ulture. Placer les tubes dans un incubateur (New Brunswick Scientific) réglé à 37 ° C, et agiter à 250 rpm pendant 14 h.
- 1% réensemencement de la culture: Distribuer 2 ml de LB frais en 14 tubes de culture mL et pointes avec 20 ul de cultures bactériennes préparées à l'étape 6.2. Incuber les tubes à 37 ° C avec agitation à 250 rpm jusqu'à ce que chaque solution bactérienne a atteint une DO 600 (densité optique mesurée à 600 nm) d'environ 1. Pour mesurer DO 600, transférer 1 ml de chaque culture à une cuvette jetable et mesurer l'absorbance à 600 nm avec un spectrophotomètre UV (UV Genesys 10, Thermo Scientific).
- Transférer 1 ml de chaque culture à une nouvelle tube de 1,5 ml et centrifuger les cellules par centrifugation à 11.000 g pendant 5 min à température ambiante.
- Transférer le surnageant clair pour un nouveau tube de 1,5 ml et centrifuger à -20 ° C si elle n'est pas utilisée immédiatement.
7. Détection à l'aide spectrophotomètre à fluorescence
- Allumez le spectrophotomètre à fluorescence (Cary Eclipse, Varian Inc) et de définir les paramètres d'acquisition de données avec une excitation à 488 nm et émission à 520 nm. Les lectures peuvent être prises toutes les minutes pendant 1 h.
- Laver 3 cuvettes de cristal de quartz (Varian Cary) avec ddH 2 O, suivie par l'éthanol 100%. Sécher les cuvettes par le clignotement de l'azote gazeux. Étiquette cuvettes C1 (contrôle 1), C2 (contrôle 2) et T (test).
- Transfert 24 uL de ddH 2 O à C1 et 24 pi de CEM-CE à C2 et T. Ajouter 25 uL de 2 × RB à chaque cuvette et les place dans le spectrophotomètre à fluorescence. Commencez à collecter des données de fluorescence pour les 5 premières minutes.
- Ajouter 1 ul de RFSS1 (à partir d'une solution à 5 uM stock) à C2 et 1 pl de RFD-EC1 (à partir d'une solution à 5 uM stock) à T et C1. Mélanger chaque solution par pipetage. Ce doit être soigneusement lancé afin que les lectures de fluorescence ne sont pas interrompus. Laisser la réaction se poursuivre pendant le reste du temps d'acquisition 1 h.
- Sauverles données dans le format de fichier Excel, transférer les données vers un ordinateur personnel et les données de processus pour créer une image graphique.
8. Détection par électrophorèse sur gel
Les mélanges réactionnels mêmes préparés à l'étape 7.4 peut être utilisé pour l'analyse par électrophorèse sur gel; alternativement de nouvelles réactions peuvent être préparés de manière similaire et incubés dans des tubes de 1,5 mL à centrifuger. Dans les deux cas:
- Réactions de trempe (après 1 h) par l'addition de 5 pi de NaOAc 3 M et 125 ul d'éthanol à 100%. Mélanger chaque solution en vortex et placer les tubes dans les congélateur à -20 ° C pendant 1 h.
- Centrifuger les mélanges de réaction à 11.000 g pendant 20 min à 4 ° C et retirer délicatement le surnageant par pipetage.
- Sécher les granulés en utilisant le concentrateur d'ADN pour 10 min.
- Pellets Remettre en suspension dans 20 pl de 1 × GLB brièvement au vortex. Centrifuger les tubes très brièvement avec une centrifugeuse de paillasse (Minicentrifuge, VWR Scientific). Ces échantillons sont lusy pour le chargement dans un gel dPAGE.
- Préparer un gel dPAGE 10% tel que décrit dans 3.1 à 3.7. Charger les échantillons de réaction de l'étape (8.4) dans les puits à l'aide d'une pipette et des conseils de chargement de gel (DiaMed). Appliquer une 40 mA (750 V) en cours jusqu'à ce que le colorant de fond (bleu de bromophénol) est d'environ 5 cm au-dessus du bord inférieur des plaques.
- Retirer des plaques de verre et se laver soigneusement avec de l'eau du robinet pour enlever les morceaux de gel. Essuyez les plaques avec un Kimwipe (Kimberly-Clark Professional).
- Numérisation la plaque de gel pour la fluorescence à l'aide d'un scanner Typhoon (Typhoon 9200, le mode Variable, GE Healthcare). Analyser les données en utilisant le logiciel ImageQuant (Molecular Dynamics).
9. Spécificité de détection
- Pour tester la spécificité bactérienne, les mêmes procédures décrites ci-dessus pour la culture E. coli, la préparation de son CEM et de mener une analyse réaction de clivage peut être effectuée pour un certain nombre de différentes souches bactériennes telles que B. subtilis, P. Peli, Y. ruckeri, L. planturum, P. acidilactici (montré dans la Fig. 3A).
10. Détection Single Cell
Préparer un 1 ml E. stock de glycérol coli de 2 UFC / ml (UFC: unité formant colonie) par dilution en série et confirmer la concentration UFC par placage 5 Ce stock devrait contiennent 0,2 UFC/100 mL.. Conserver à -80 ° C jusqu'à son utilisation.
- Préparer 10 tubes de culture contenant 2 mL de LB.
- Inoculer chaque culture avec 100 uL de 2 stock de glycérol UFC / ml et incuber à 37 ° C avec agitation à 250 rpm. L'utilisation de l'ensemble du stock de glycérol (1 ml) devrait donner 10 cultures.
- Récolte 300 ul de chaque tube de culture inoculé à des points de temps suivants: 4, 8, 12, 16 et 24 h. Laissez le reste de la culture de se développer pendant 24 h.
- Mesurer la DO 600 et précipiter les cellules par centrifugation à 11.000 g pendant 5 min.
- Transférer les CEM à de nouvelles 1,5 microtubes ml et conserver à -20 & dpar exemple en C jusqu'à utilisation.
Remarque: Comme il peut y avoir aucune détectable OD 600 pour des échantillons récoltés au moment des points 4, 8 et 12, permettent la culture restant à croître pendant 24 h afin d'identifier les cultures contenant des bactéries (déterminée par la turbidité et OD mesure). Seuls un ou deux des 10 cultures peut contenir E. coli après l'inoculation et les tubes restants ne contiennent pas toutes les cellules. - Utilisez les CEM récupérés à partir de cultures positives (stocké à -20 ° C) à des points dans le temps désignés pour préparer réactions de clivage avec RFD-EC1, puis d'analyser les mélanges de réaction par électrophorèse en gel dPAGE tel que décrit dans la Section 8.
11. Résultats Concept et Représentant
Le concept de l'exploitation d'une DNAzyme ARN-clivage fluorescent (RFD) pour la détection bactérienne est illustré dans la figure. 1. Le RFD clive un ADN chimère / ARN substrat à une seule liaison d'ARN (bleu R) flanquée de deux nucléotides marquésavec un fluorophore (F) et un agent d'extinction (Q), respectivement. Comme une bactérie d'intérêt (comme E. coli) pousse dans les médias, il laissera derrière lui un mélange brut extracellulaire (LEC). Ce CEM dans son ensemble est ensuite utilisé dans une expérience dans la sélection in vitro pour obtenir une RFD qui est sensible spécifiquement à la CEM; sans doute le RFD interagit avec une molécule spécifique (violet étoiles) dans le CEM qui est une molécule signature de la bactérie. Lorsque le LEC est ajouté à la solution réactionnelle contenant le RFD, il déclenche l'activité ARN-clivage de l'RFD. L'événement de clivage sépare F à partir de Q, résultant en un signal fluorescent qui peut être détecté soit en utilisant un fluorimètre ou par électrophorèse sur gel.
La validation expérimentale du concept ci-dessus a été fait avec le CEM de E. coli (CEM-CE). Nous avons obtenu 3 molécules RFD via la sélection in vitro, et la plus efficace a été désigné comme RFD-EC1 (Fig. 2A). 5 We testé l'activité de clivage de RFD-EC1 (avec une séquence mutante nommée RFSS1) en réponse au LEC-CE. Les deux RFD-EC1 et RFSS1 ont été préparés par ligature enzymatique des parties DNAzyme au substrat FS1 (toutes les séquences sont présentés dans la figure. 2A). Dans l'expérience de mesure de fluorescence (figure 2B), CEM-CE a été incubé seul pendant 5 min, suivie par l'addition de RFD-EC1 ou RFSS1, et par une nouvelle incubation de 55 min plus. L'intensité de fluorescence de la solution a été continuellement lire toutes les minutes et les données ont été utilisées pour calculer fluorescence relative (RF; calculé comme le rapport de l'intensité de fluorescence à l'instant t par rapport à l'intensité de fluorescence au temps 0). Les valeurs Rf vs le temps d'incubation sont tracés comme Fig. 2B. Il a été constaté que RFD-EC1 produit un niveau élevé du signal de fluorescence lors de l'addition de la CGE-CE; à l'opposé, RFSS1 n'a pas produit un signal fort de fluorescence. Ainsi, la fluorescence fu-productionnction de RFD-EC1 au contact CEM-CE est une séquence spécifique.
Afin de vérifier que les augmentations observées de fluorescence sont dues à la coupure de la liaison d'ARN, nous avons analysé les mélanges réactionnels par dPAGE. Le clivage de RFD-EC1 devrait générer deux fragments d'ADN, un fragment 5 'en conservant le fluorophore et un fragment 3' en conservant l'extincteur. Seulement clivée RFD-EC1 (unclv) et le fragment 5 '(CLV) a pu être détecté par imagerie de fluorescence. Le résultat dPAGE représenté dans la Fig. 2C révèle que le mélange réactionnel de RFD-EC1 et CEM-CE effet produit le produit de clivage prévue, tandis que le mélange ne RFSS1/CEM-EC.
La spécificité de RFD-EC1 a été examiné en utilisant CEM recueillies à partir de plusieurs autres Gram-négatives et gram positives bactéries et les données sont affichées dans la figure. 3A. Seul l'échantillon contenant CEM-CE (courbe bleue) a entraîné une augmentation de la fluorescence. Le manque de réactivité croisée avec CEMdes autres bactéries indique que RFD-EC1 est hautement sélectif pour E. coli.
Nous avons également examiné le temps nécessaire pour la culture d'une seule E. coli cellule afin de produire suffisamment de LEC qui peut induire le clivage du RFD-EC1. Pour cette expérience, un E. coli contenant l'échantillon défini UFC (unités formant colonie) a été suffisamment dilué pour obtenir la concentration de 1 UFC / ml. Elle a été suivie en mélangeant 100 ul de l'échantillon dilué bactérienne avec les médias et la culture de la croissance il pour 4, 8, 12, 16 et 24 h. CEM ont ensuite été recueillies pour chaque point de temps et testé pour induire l'activité de clivage de la RFD-EC1. Le résultat dPAGE représenté dans la Fig. 3B indique qu'un temps de culture de 12 h est nécessaire.
Il est important de noter que l'augmentation du signal initial petite observées dans les mesures de fluorescence après l'addition de RFSS1 séquence (comme contrôle négatif) à la CEM-CE (Fig. 2B, rouge curve) ou RFD-EC1 à d'autres systèmes de SCE bactériennes (fig. 3B; toutes les courbes à l'exception bleu) est attribuée à la fluorescence intrinsèque du module FRQ (raison de trempe incomplète de F par Q). Ainsi, on s'attend à ce que l'addition de F-et Q-étiquetés séquences produirait une augmentation initiale de la fluorescence. Cependant, seuls mélanges RFD-EC1/CEM-EC sont capables de produire un niveau élevé de la fluorescence au cours du temps.

Figure 1. Illustration schématique de l'ARN-clivage fluorescent DNAzyme (RFD) sonde fluorescente au contact avec le mélange brut extracellulaire (LEC) produite par des cellules bactériennes spécifiques d'intérêt. Le RFD clive un ADN chimère / ARN substrat à une seule liaison d'ARN (bleu R) flanquée de deux nucléotides marqués avec un fluorophore (F) et un agent d'extinction (Q), respectivement. Avant la réaction de clivage, le niveau de fluorescence du RFD est minime en raison de la proximité proxmité de F et Q. A clivage, Q s'écarte F; à la suite, un signal fort de fluorescence est produite.
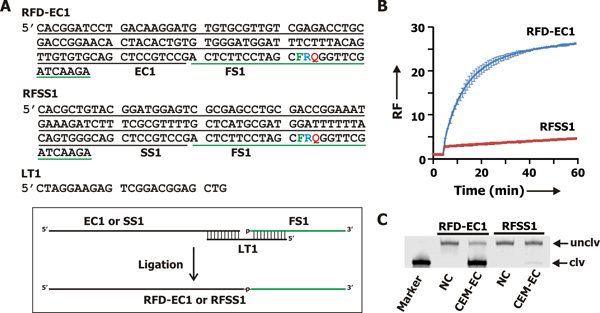
Figure 2. Le E. coli-détection RFD. (A) RFD-EC 1 est la sonde DNAzyme qui peut être activé par LEC-CE. RFSS1 est une séquence brouillée de RFD-EC1 utilisé comme un contrôle. RFD-EC1 et RFSS1 ont été produites par ligation avec FS1 EC1 et SS1, respectivement, en présence d'LT1 comme matrice. F: fluorescéine-désoxythymidine modifié. Q: Dabcyl modifié désoxythymidine. R: ribonucléotide adénine. (B) Fluorescence de signalisation profils de RFD-EC1 et RFSS1 dans la présence de CEM-CE. Analyse dPAGE (C) des mélanges réactionnels de clivage dans B (temps de réaction: 60 min). Sur la photo est une image de fluorescence du gel obtenu avec dPAGE par scanner Typhoon. Lane NC: RFD-EC1 ou RFSS1 dans le tampon de réaction seul; Lane CEM-CE: RFD-EC1 ou RFSS1 dans le tampon de réaction contenant CEM-CE. Marqueur: RFD-CE1 traité avec 0,25 N NaOH, une méthode connue de provoquer un clivage complet de l'ARN. unclv: non clivée RFD-EC1. clv: le fragment de clivage contenant le fluorophore.
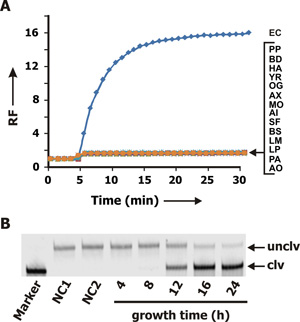
Figure 3. (A) Fluorescence de signalisation profil de RFD-EC1 en CEM préparés à partir de diverses cellules bactériennes. CE: Escherichia coli-K12; PP: Pseudomonas peli; BD: Brevundimonas diminuta; HA: Hafnia alvei; YR: Yersinia ruckeri; OG: Ochrobactrum grignonese; AX: Achromobacter xylosoxidans; MO: Moraxella osloensis; AI: Acinetobacter lwoffi; SF: Serratia fonticola; BS: Bacillus subtilis; LM: Leuconostoc mesenteroides; LP: planturum Lactobacillus; PA: Pediococcus acidilactici; AO: Actinomyces orientalis. Chaque échantillon a été CEM incubé pendant 5 min, suivie par l'addition de RFD-EC1. (B) dPAGEanalyse de mélanges RFD-EC1/CEM-EC après une réaction de 60 min. Lane NC1: RFD-EC1 dans le tampon de réaction seul. Lane NC2: RFD-EC1 dans le tampon de réaction contenant CEM-BS (le CEM préparé à partir de Bacillus subtilis). Les voies marquées avec 4, 8, 12, 16 et 24: RFD-EC1 dans le tampon de réaction contenant CEM-CE prise partir de la culture bactérienne contenant un seul E. cellule d'Escherichia la suite d'une période de croissance de 4, 8, 12, 16 et 24 h, respectivement.
Discussion
La plupart des méthodes courantes de détection bactérienne aujourd'hui sont soit lent (microbienne classique) ou techniquement exigeants (anticorps, PCR). Ainsi, nous pensons que la prochaine génération d'outils de détection doit répondre vers la vitesse et la simplicité. À cette fin, nous avons créé un ARN-clivage et la fluorescence de signalisation DNAzyme qui peut être utilisé pour développer des essais simples pour signaler la présence de bactéries à travers la génération d'un signal de fluorescence. La vedette DNAzyme sonde, RFD-EC1, est activé par le CEM produite pendant la croissance de E. coli dans les milieux de culture. Depuis notre méthode utilise des mélanges bruts extracellulaires d'une bactérie que l'objectif de détection et de court-circuite l'extraction laborieuse cible et d'amplification, il peut être utilisé pour mettre en place très simple, "mix-and-lire" type de tests pour la détection bactérienne. L'utilisation de notre DNAzyme ne se limite pas à la méthode basée sur la détection par fluorescence. Par exemple, la détection colorimétrique en utilisant le même système de dosage DNAzyme cune être conçus en utilisant une méthode précédemment rapporté que exploite amplification par cercle roulant en conjonction avec un colorant organique 32. Nous prévoyons l'utilisation de ADNzymes pour la détection bactérienne comme une séduisante avenue pour générer de nouveaux biocapteurs bactériens avec une plus grande simplicité opérationnelle.
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Le financement de ce travail a été fourni par les sciences naturelles et en génie du Canada (CRSNG) et le Réseau de papier bioactif Sentinel.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue ou de modèle | |
| Gélose | BioShop Canada | AGR003 | |
| Persulfate d'ammonium (APS) | BioShop Canada | AMP001 | |
| L'acrylamide / bis-acrylamide (40%, 29:1) | BioShop Canada | ACR004 | |
| L'acide borique | BioShop Canada | BOR001 | |
| Bleu de bromophénol | Sigma-Aldrich | B8026 | |
| EDTA | EM Science | EXO539-1 | |
| HCl | Sigma-Aldrich | 38281 | |
| HEPES | Bioshop Canada | HEP001 | |
| Bouillon LB | Sigma-Aldrich | L3022 | |
| MgCl 2 | EMDProduits chimiques | B10149-34 | |
| NaCl | BioShop Canada | SOD002 | |
| NaOAc | EMD Chemicals | SXO255-1 | |
| NaOH | EMD Chemicals | SXO590-1 | |
| SDS | BioShop Canada | SDS001 | |
| TEMED | BioShop Canada | TEM001 | |
| Tris-base | BioShop Canada | BST666 | |
| Tween 20 | Sigma-Aldrich | P9416 | |
| Urée | BioShop Canada | URE001 | |
| Xylenecyanol FF | Sigma-Aldrich | X4126 | |
| Concentrateur d'ADN | Thermo Scientific | Savant ADN SpeedVac 120 | |
| Millex unité de filtre | Millipore | SLGP033RS | |
| Conseils pour le chargement de gel | DiaMed | TEC200EX-K | |
| Logiciel ImageQuant | Dynamique Moléculaire | Version 5.0 | |
| Kimwipes | Kimberly-Clark Professional | 34705 | |
| Mini Vortexer | VWR | 58816-121 | |
| Parafilm | Pechiney Plastic Packaging | PM996 | |
| Des boîtes de Pétri | Fisher Scientific | Fisherbrand 08-757-12 | |
| Stripettor Plus (pipette arme à feu) | Corning | 07764714 | |
| Cuves en quartz | Varian Inc | 66-100216-00 | |
| Shaker / Incubateur | New Brunswick Scientific | Classic Series C24 | |
| Typhoon Scanner | GE Healthcare | 9200 mode Variable | |
| Centrifuger | Beckman Coulter | X Allegra22-R | |
| Spectrophotomètre UV | Thermo Scientific | GenesysUV 10 | |
| Spectrophotomètre à fluorescence | Varian Inc | Cary Eclipse |
Références
- Zourob, M., Elwary, S., Truner, A. . Principles of Bacterial Detection: Biosensors, Recognition Receptors and Microsystems. , (2008).
- Call, D. R. Challenges and Opportunities for Pathogen Detection Using DNA Microarrays. Crit. Rev. Microbiol. 31, 91-99 (2005).
- Lazcka, O., Campo, D. e. l., J, F., Muñoz, F. X. Pathogen detection: A perspective of traditional methods and biosensors. Biosens. Bioelectron. 22, 1205-1217 (2007).
- Velusamy, V. An overview of foodborne pathogen detection: In the perspective of biosensors. Biotechnol. Adv. 28, 232-254 (2010).
- Ali, M. M. Fluorogenic DNAzyme Probes as Bacterial Indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Liu, J., Cao, Z., Lu, Y. Functional Nucleic Acid Sensors. Chem. Rev. 109, 1948-1998 (2009).
- Li, Y., Lu, Y. . Functional Nucleic Acids for Analytical Applications. , (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Joyce, G. F. Forty Years of In Vitro Evolution. Angew. Chem. Int. Ed. 46, 6420-6436 (2007).
- Breaker, R. R., Joyce, G. F. A DNA enzyme that cleaves RNA. Chem. Biol. 1, 223-229 (1994).
- Cuenoud, B., Szostak, J. W. A DNA metalloenzyme with DNA ligase activity. Nature. 375, 611-614 (1995).
- Chinnapen, D. J., Sen, D. A deoxyribozyme that harnesses light to repair thymine dimers in DNA. Proc. Natl. Acad. Sci. U. S. A. 101, 65-69 (2004).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Silverman, S. K. DNA as a versatile chemical component for catalysis, encoding, and stereocontrol. Angew. Chem. Int. Ed. 49, 7180-7201 (2010).
- Mei, S. H. An efficient RNA-cleaving DNA enzyme that synchronizes catalysis with fluorescence signaling. J. Am. Chem. Soc. 125, 412-420 (2003).
- Liu, Z. Assemblage of signaling DNA enzymes with intriguing metal-ion specificities and pH dependences. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Kandadai, S. A., Li, Y. Characterization of a catalytically efficient acidic RNA-cleaving deoxyribozyme. Nucleic Acids Res. 33, 7164-7175 (2005).
- Rupcich, N. Quenching of fluorophore-labeled DNA oligonucleotides by divalent metal ions: implications for selection, design, and applications of signaling aptamers and signaling deoxyribozymes. J. Am. Chem. Soc. 128, 780-790 (2005).
- Shen, Y., Brennan, J. D., Li, Y. Characterizing the secondary structure and identifying functionally essential nucleotides of pH6DZ1, a fluorescence-signaling and RNA-cleaving deoxyribozyme. Biochemistry. 44, 12066-12076 (2005).
- Chiuman, W., Li, Y. Revitalization of six abandoned catalytic DNA species reveals a common three-way junction framework and diverse catalytic cores. J. Mol. Biol. 357, 748-754 (2006).
- Chiuman, W., Li, Y. Evolution of high-branching deoxyribozymes from a catalytic DNA with a three-way junction. Chem. Biol. 13, 1061-1069 (2006).
- Shen, Y. Catalysis and rational engineering of trans-acting pH6DZ1, an RNA-cleaving and fluorescence-signaling deoxyribozyme with a four-way junction structure. Chem BioChem. 7, 1343-1348 (2006).
- Ali, M. M., Kandadai, S. A., Li, Y. Characterization of pH3DZ1 - An RNA-cleaving deoxyribozyme with optimal activity at pH 3. Can. J. Chem. 85, 261-273 (2007).
- Chiuman, W., Li, Y. Efficient signaling platforms built from a small catalytic DNA and doubly labeled fluorogenic substrates. Nucleic Acids Res. 35, 401-405 (2007).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA MgZ based on a non-classic allosteric design. PLoS ONE. 2, e1224 (2007).
- Shen, Y. Entrapment of fluorescence signaling DNA enzymes in sol gel-derived materials for metal ion sensing. Anal. Chem. 79, 3494-3503 (2007).
- Kandadai, S. A. Characterization of an RNA-cleaving deoxyribozyme with optimal activity at pH 5. Biochemistry. 48, 7383-7391 (2009).
- Zhao, W., Brook, M. A., Li, Y. Periodic assembly of nanospecies on repetitive DNA sequences generated on gold nanoparticles by rolling circle amplification. Methods Mol. Biol. 474, 79-90 (2008).
- Navani, N. K., Mok, W. K., Li, Y. In vitro selection of protein-binding DNA aptamers as ligands for biosensing applications. Methods Mol. Biol. 504, 399-415 (2009).
- Ali, M. M., Li, Y. Colorimetric sensing by using allosteric-DNAzyme-coupled rolling circle amplification and a peptide nucleic acid-organic dye probe. Angew. Chem. Int. Ed. 48, 3512-3515 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon