Method Article
형광 DNAzymes을 사용하여 박테리아의 탐지
요약
Google은 최근은 간단 세균 검출을위한 형광 분석 "을 믹스 앤 읽기"를 설정하는 데 적용할 수있는 형광 DNAzyme 프로브를 생성하기위한 새로운 접근 방식을보고했습니다. 이러한 특별한 유전자 프로브는이를 형광 신호 생성에 세균 검출을 번역 특정 세균에 의해 생산되는 원유 세포외 혼합물 (CEM)의 앞에서 발색단 - 수정의 DNA-RNA 키메라 기판의 절단을 catalyze. 이 보고서에서 우리는 "RFD-EC1"표시된 특정 DNAzyme 프로브는 모델 박테리아의 탐지를 위해 고용되는 핵심 실험 절차를 설명합니다 대장균 (E. 대장균).
초록
발병은 사망과 입원의 수백만뿐만 아니라 어마어마한 경제적 손실을 각각 매년 식인과 병원 - 인수 병원균 계정에 연결되어. 지속적인 유행병 장소 정확하게 초기 단계에서 범인의 병원균을 식별할 수있는 분석 방법에 대한 늘어나는 수요의 영향 등의 발생과 최소화의 예방. 높은 특이성 (이익만을 세균 검출), 고감도 (낮은 검출 능력 : 병원균 검출을위한 효과적인 방법의 큰 배열이있다지만, 그들 모두가 이상적인 검출 방법에 대한 구현 모두 다음과 같은 다섯 최고의 요건을 충족 없습니다 하나의 살아있는 세균 세포 등), 짧은 시간 - 결과 (시간 분), 훌륭한 운영 단순 (긴 샘플링 절차 및 전문 장비의 사용에 대한 필요도), 그리고 비용 효율성. 예를 들어, 고전적인 미생물 방법은 매우 구체적인 것이지만 오랜 시간 (다를 필요로주 YS는) 1 PCR과 항체 기반 기술을 짧은 대기 시간 (일 시간) 제공합니다. 최종 결과를 얻기 위해,하지만 그들은 값비싼 시약 및 / 또는 정교한 장비의 사용. 따라서 2-4 요구, 아직 거기있다 상기 요건 중 하나 이상의 향상된 특징을 제공하는 혁신적인 세균의 검출 방법을 개발하는 방향으로 과학 연구에 대한 큰 수요. 우리 연구실은 세균 검출 포함한 biosensing 어플 리케이션을위한 분자 프로브의 소설 클래스로 DNAzymes의 가능성을 검사에 관심있다 5.
DNAzymes는 (또한 deoxyribozymes 또는 DNA의 효소라고도 함)가 사람이 만든 화학 반응을 catalyzing의 기능이 단일 좌초된 DNA의 분자를. 6-8 이들 분자가 많은 10대로 들어있는 광대한 임의 시퀀스의 DNA 풀 (격리 수 과정에 의한 16 개별 시퀀스)가 O "체외 선택에서"로 알려진R "SELEX"(지수 농축에 의한 리간드의 체계적 진화). 9-16 이러한 특수한 DNA 분자 널리 biosensing 응용 프로그램을위한 분자 도구로 최근 몇 년 동안 검사되었습니다. 6-8
우리 연구실은 RNA-cleaving 형광 DNAzymes (. RFDs; 그림 1)을 분리을위한 체외 선택 절차에 설립되었으며 분석 도구로 RFDs의 사용을 조사 17-29 RFDs가 단일 ribonucleotide에서 DNA-RNA 키메라 기판의 절단을 catalyze. 형광단 (F)과 끄는 (Q)의 어귀되어 접합 (R). F와 Q의 가까운 거리는 uncleaved 기판 최소한의 형광을 렌더링. 그러나 절단 이벤트는 형광 강도의 상당한 증가와 함께되어 F와 Q의 분리로 안내합니다.
최근, 우리는 박테리아 감지를위한 RFDs를 분리하는 방법을 개발 5.이 특별한 RFDs은 "으로 고립되었다자신의 환경이나 그들이 배양해되는 미디어 박테리아의 특정 유형 (그림 1)에 의해 남겨진 원유 세포외 혼합물 (CEM)의 면전에서 "점등. 원유 혼합물의 사용이 정화의 지루한 과정을 circumvents 및 바이오 센서 개발 (완료하는 데 몇 달 또는 몇 년이 걸릴 수도있는)에 대한 관심 미생물의 적합한 대상을 식별. 세포외 대상의 사용은 세포내 표적을 구하는 단계 필요가 없기 때문에 시금 절차는 간단하다 의미합니다.
오직 E. 제작한 CEM의 면전에서, 위의 접근 방식을 사용하여, 우리는 (그림 2A. FS1)은 기판 클리브 RFD를 파생 대장균 (CEM-EC) 5.이 E. RFD-EC1 (그림 2A)이라는 대장균 감지 RFD는, 엄격하게 CEM-EC에 대응하지만 다른 박테리아의 호스트 (그림 3)에서 CEMs에 대한 의식이된다는 것을 발견했다.
여기 preseE.을 설정하기위한 주요 실험 절차를 NT RFD-EC1 및 담당자 결과를 이용하여 대장균 검출 assays.
프로토콜
1. 화학 솔루션의 작성
- 0.5 M 에틸렌 디아 민 테트라 아세트산 (EDTA (에틸렌 다이아 민 테트라 초산)) : 2리터에서 (L) 플라스틱 비이커는 186.1 g EDTA (에틸렌 다이아 민 테트라 초산) (EM 과학) 무게와 autoclaved deionized - 증류수 (ddH 2 O) 800 밀리리터를 (ML) 추가합니다. NaOH 쥐똥 (EM 과학)를 사용하여 8.0으로 산도를 조정합니다. ddH 2 O를 사용하는 한 패로 최종 볼륨 만들기 4 ° C.에 유리 병, 압력솥 및 저장소 솔루션을 전송
- 10 × 트리스-borate EDTA (에틸렌 다이아 민 테트라 초산) 용액 (10 × TBE, 89 MM 트리스, 89 MM 붕산, 2 밀리미터 EDTA (에틸렌 다이아 민 테트라 초산), 산도 7.5) : 432 트리스베이스의 G (BioShop 캐나다)와 붕산 (BioShop 캐나다)의 220g의 무게를 그리고 4 패 플라스틱 비커에 각각 추가하십시오. 0.5 M EDTA (에틸렌 다이아 민 테트라 초산) (산도 8.0)의 80 ML을 측정하고 비커에 추가합니다. 구성 요소가 완전히 해소되기 전까지는 철저히 자기 볶음 바와 솔루션을 섞어 4 L.의 최종 볼륨 ddH 2 O를 추가합니다. 4 ° C.에 유리 병, 압력솥 및 저장소 솔루션을 전송
- 10% polyacrylami를 denaturing드 겔 재고 : 4 패 플라스틱 비이커에게, 우레아의 1681.7 G (BioShop 캐나다), 10의 400 ML × TBE, 40 % 아크릴 아미드 / bisacrylamide (29:1) 용액 (BioShop 캐나다) 1 L를 추가합니다. ddH 2 O를 포함해서 4 패로 볼륨을 조정 감동과 우레아를 해산. 4 1 패 호박 유리병 및 매장 ° C.에 솔루션을 전송 (이것은 이전 중합에 neurotoxin 때문에 Caution!의 아크릴 아미드는 장갑, 마스크, 고글 및 실험 복으로 취급해야합니다.)
- 2 × 겔 로딩 버퍼 (2 × GLB) : 200 ML 유리 비커 위하여는, 우레아의 44 G, 자당 8 G (BioShop 캐나다), bromophenol 블루 (BioShop 캐나다)의 10 밀리그램, xylenecyanol FF 10 밀리그램을 (시그마 추가 - 올드 리치), 10 %의 나트륨 dodecyl 황산 400 μL (SDS; BioShop 캐나다), 10 × TBE 4 ML. ddH 2 O 40 ML에 최종 볼륨을 조정하고 온화한 가열 (50 ℃) 및 자석 막대와 함께 감동과 고체를 해산. 4에서 1.5 ML의 microcentrifuge 튜브 및 매장에 한 ML의 나누어지는 전송 ° C. 우리 바탕으로경험은, 그것은 2 × GLB는 저장 조건 하에서 굳은하기 때문에 사용하기 ° C에서 사전에 90 시에 열 두 × GLB를 간략하게 필요합니다.
- 1 M 트리스-HCL (산도 7.5) : 200 ML 유리 비커에 트리스베이스의 12.1 g의 무게를. ddH 2 O의 60 ML을 추가하고 자기 활동가로 교반하여 고체를 해산. 1 M HCL (시그마 - 올드 리치)를 사용하여 7.5으로 산도를 조정합니다. ddH 2 O 100 ML에 볼륨을 만들어 유리병과 압력솥에 전송할 수 있습니다. 4 ° C.에 저장
- 5 M NaCl : 유리 비커에는 NaCl의 58.4 G (BioShop 캐나다)를 무게와 ddH 2 O에 150 ML로 분해 ddH 2 O. 200 ML에 볼륨을 조정 4 ° C.에 유리병, 압력솥 및 매장에 대한 해결책을 전송
- DNA를 용출 버퍼 : 유리 비커에 2 ML를 섞어 1 M 트리스-HCL (산도 7.5), 5 M NaCl 8 ML과 0.5 M EDTA (에틸렌 다이아 민 테트라 초산) (산도 8.0)를 0.4 ML. ddH 2 O. 200 ML에 볼륨을 조정 4 ° C.에서 압력솥 및 매장
- 2 × 반응 버퍼 (2 × RB; 100 밀리미터 HEPES, 300 MM NaCl, 30 밀리미터 MgCl 2) : 50 ML 팔콘 튜브 (BD 팔콘)에는 HEPES 1.2 G (BioShop 캐나다), NaCl의 0.88 g과 MgCl 2의 0.30 g • 6H 2 O를 추가 (EMD 화학)와 ddH 2 O를 30 ML 구성 요소가 완전히 해소되기 전까지는 약간 떨고로 섞는다. 10 N NaOH 용액을 추가하여 7.5로 산도를 조절하고 ddH 2 O를 함께 50 ML에 최종 볼륨을 조정 4 ° C.에 주사기 기반의 필터 유니트 (0.22 μm의, Millipore)와 저장소를 사용하여 솔루션을 정화
- Luria Bertani (LB) 국물 : 비커에 ddH 2 O를 1 L의 추가 다음에 (시그마 - 올드 리치) LB 분말의 20.0 g의 무게를 자기 볶음 바와 솔루션을 철저를 섞어, 원뿔형 플라스크에 솔루션을 전송합니다. 상온에서 솔루션 및 저장소를 압력솥.
- 1.5 %의 LB 한천 배지가 : 250 ML 플라스크에 한천 1.5 G (BioShop 캐나다)를 체중과 액체 LB 100 ML에 추가합니다. 상온에서 혼합 및 저장소를 압력솥.
- 한천 도금 : 멜t 전자렌지에 LB 한천과 ~ 50 ° C.에 대한 시원한 해결책 화염 하에서 배양 접시 (피셔 과학)에 솔루션을 하거라. 우리의 경험에서, LB 한천의 100 ML은 5-6 접시를 얻을 수 있습니다.
2. 템플릿에 의한 RFD-EC1 및 RFSS1 건설 효소 결합을 매개
RFD-EC1 (그림 2A)는 기능 DNAzyme입니다. 그것은 촉매 시퀀스 EC1 및 기판 순서 FS1 (그림. 2A의 검은색과 녹색 선으로 표시됨)로 구성되어 있습니다. RFSS1 (그림 2A)는 촉매 시퀀스 EC1은 일부가 SS1으로 단행했지만 FS1 부분은 변경되지 않습니다 RFD-EC1의 불 가능한 버전입니다. RFD-EC1 및 RFSS1은 (그림. 2A에 삽입된 상자 참조) 내고 템플릿으로 LT1의 면전에서 oligonucleotide EC1이나 SS1과 oligonucleotide FS1의 효소 결합을 매개 템플릿에 의해 만들어 졌 었죠. 결합 반응을 실시를위한 절차는 아래에 제공됩니다.FS1는 이전에 설립 프로토콜에 따라, 예일 대학에서 중사님 Oligo 합성 시설에서 얻은 deprotected 및 겔 전기 영동에 의해 정화되었다. 17-24 EC1, SS1과 LT1은 통합의 DNA 기술에서 구입하여 겔 전기 영동에 의해 정화되었다.
- ddH 2 O를 사용 FS1, EC1, SS1과 LT1의 100 μm의 주식 솔루션을 준비 사용할 때까지 -20 ° C에서 그들을 저장합니다.
- 관 1 관 2로 표시된 두 1.5 μL microcentrifuge 튜브에 FS1 주식 솔루션의 5 μL를 전송합니다. 각각의 튜브로 ddH 2 O의 38.5 μL와 10 다음 5 μL × T4 폴리 뉴클레오 타이드의 키나제 (PNK) 반응 완충액을 추가 500 MM 트리스-HCL (산도 7.6, 25 ° C)를 포함하는 (MBI Fermentas), 100 MM MgCl 2, 50 밀리미터 DTT, 1.0 밀리미터 spermidine. (몇 번 다운 용액을 피펫과) pipetting하여 각 솔루션을 섞는다.
- ATP (100 MM, MBI Fermentas) 1 μL를 추가하고 pipetting하여 섞는다.
- T4 폴리 뉴클레오 타이드의 키나제 0.5 μL (P 추가북한, pipetting하여 MBI Fermentas)와 믹스, 10 단위 / μL. 30 분 동안 37 ° C에서 반응 섞어 알을 품다. 형광단의 photobleaching 최소화하기 위해 알루미늄 호일로 튜브를 포함해야합니다.
- 5 분 90 ° C에서 가열하여 반응을 끄다. 10 분 동안 상온에서 반응 섞어 시원한 시간을 보내세요.
- EC1 5 μL와 튜브 1과 튜브 2 ~ SS1 주식 용액을 각각 추가합니다.
- pipetting하여 각 튜브, 믹스로 LT1 주식 솔루션의 5 μL를 추가합니다. 1 분 및 10 분 실온에 시원한에 대해 90 ° C에서 반응 섞어 가열.
- ddH 2 O의 118 μL 후 10 20 μL × 400 MM 트리스-HCL (° C 25 산도 7.8), 100 밀리미터 MgCl 2, 100 밀리미터 DTT, 5를 포함 T4 DNA ligase 버퍼 (MBI Fermentas)를 추가 MM ATP. pipetting하여 솔루션을 섞는다.
- 그리고 pipetting하여 혼합, 2 T4의 DNA ligase의 μL를 (MBI Fermentas 5 단위 / μL) 추가합니다. 1 H 위해 상온에서 반응 섞어 알을 품다.
- 각 튜브, 와동에 3 M NaOAc (산도 7.0) 20 μL을 추가하고 다운 스핀. 각 튜브에 차가운 100 % 에탄올 500 μL를 추가, vortexing하여 솔루션을 섞어 30 분 -20 ° C의 냉장고에 튜브를 넣습니다.
- ° C 냉장 원심 분리기 (Allegra X22-R, Beckman 보습 바로 앞에 달린 풀베는 날) 4시 20 분 동안 1만1천그램에 섞어 원심 조심스럽게 pipetting하여 뜨는을 제거합니다.
- 10 분 동안 DNA 농축기 (학자의 DNA Speedvac, 써모 과학)를 사용하여 DNA를 펠렛을 건조합니다.
- Resuspend의 DNA를 한 후 30 μL × 간략 겔 로딩 버퍼 (GLB), 와동에 쥐똥하고 benchtop의 원심 분리기 (Minicentrifuge, VWR 과학)와 다운 스핀. 출혈도 잡았 DNA 샘플은 10 % dPAGE 젤로 로딩을위한 준비가되어 있습니다.
3. 10 % dPAGE 젤의 작성
다음 단계는 간략 겔 전기 영동과 그 설정의 장치에 대해 설명합니다. 장치, 설정 및 취급에 관한보다 자세한 내용은 T를 참조하시기 바랍니다우리 이전에 다음 ID로 출판 프로토콜을 O. 30,31
- 워시와 두 유리 접시, 두 0.75 mm 스페이서와 16 잘 빗을 건조. 클립 유리 접시와 스페이서를 조립 및 최대 마주 노치 유리 플레이트와 평평한 표면에 수평으로 누워.
- 150 ML의 비커에 10 % dPAGE 믹스의 40 ML을 전송합니다. pipettor으로 소용돌이에 의해, 10 % APS 400 μL와 혼합, tetramethylethylenediamine 40 μL을 (Bioshop 캐나다 TEMED)를 추가합니다.
- 접시 사이에 조심스럽게 혼합 붓고 즉시 빗을 넣습니다. 혼합물은 10 분에서 20 분 동안 중합하도록 허용합니다. 중합은 비커에 남은 잔류 겔 믹스를 선택하여 확인하실 수 있습니다.
- 일단 polymerized, 부드럽게 빗질을 제거하고 우물에 잔류 겔 용액을 제거 ddH 2 O와 우물 린스.
- 밖으로 향하게 unnotched 사각 접시와 겔 전기 영동 장치에 접시를 탑재하고 노치 플레이트 뒤에있는 금속 접시를 놓습니다. 금속 PL의 사용먹고는 유리 접시를 깰 수있는 과열을 방지하는 데 도움이됩니다.
- 장치의 위쪽과 아래쪽 챔버로 1 × TBE 프로그램을 추가합니다. 우물은 버퍼 충만하고 젤의 아래쪽 가장자리가 버퍼에 잠겨 있도록하십시오.
- 40mA (또는 750 V) 전류 10 ~ 15 분 동안 미리 실행을 적용합니다.
4. 10 % dPAGE 젤에 의한 출혈도 잡았 RFD-EC1 및 RFSS1의 정제
- 3.7 단계에 따라 1 × TBE는 주사기와 바늘을 이용하여 우물 린스.
- pipettor 및 겔 로딩 팁 (Diamed)을 사용하여 두 우물 (하나씩)으로 RFD-EC1 및 RFSS1의 결합의 혼합 (2.13부터)로드합니다. 하단 염료까지 전류 40mA를 (750 V) 적용 (bromophenol 파란색) 접시의 아래쪽 가장자리 위에 약 5cm입니다.
- 겔 실행 장치에서 유리 조각을 제거, 플랫 벤치 위에 누워 조심스럽게 스페이서를 제거합니다.
- 조심스럽게 겔에서 최고의 유리 접시를 제거하고플라스틱 랩 (겔의 랩의 주름과 접힘을 방지하려고)와 젤 싸다.
- 출혈도 잡았 제품은 자외선 미행 (260 NM)에 의해 또는 몇 cm EC1 또는 SS1 (EC1 또는 SS1 100 pmol 위에 보이는 DNA 밴드를 얻을 수 transillumination (360 NM)에 의해 어느 시각이 될 수있는 마커로 사용될 수 있으며, ) 4.2 단계에서 잘에 로드된. 마커를 사용하여 원하는 DNA 밴드를 표시한다.
- 멸균 면도날과 소비세의 DNA 밴드, 신선한 1.5 ML의 microcentrifuge 튜브로 작은 조각 및 전송로 젤을 했네요.
- 멸균 피펫 팁을 (200 μL 팁 크기)를 사용 microcentrifuge 튜브 내에서 겔 조각을 크러시.
- 각 튜브에 500 μL의 DNA 용리 버퍼를 추가하고 빛으로부터 fluorophores을 보호하기 위해 알루미늄 호일로 그들을 포괄합니다. 10 분 대한 와동 샘플을.
- ° C 냉장 원심 분리기 (Allegra X22-R, Beckman 보습 바로 앞에 달린 풀베는 날) 4시 4 분 동안 1만1천그램에서 샘플을 원심 조심스럽게 s의 350 μL를 전송신선한 1.5 ML의 microcentrifuge 튜브 (필요한 경우, 두 번째 용출이 다른 10 분 화사하게 용출 버퍼 350 μL와 함께 할 수있는)에 upernatant.
- 각 튜브에 3 M의 아세트산 나트륨 (NaOAc, 산도 7.0) 35 μL (샘플 볼륨의 0.1 ×)를 추가 vortexing에 의해 믹스와 다운 스핀. 각 튜브에 차가운 100 % 에탄올 900 UL을 추가합니다. 각 샘플을 섞어서 몇 초 동안 튜브를 손으로 흔들어. 최소 1 H를 위해 -20 ° C에서 튜브를 놓습니다.
- 4에 20 분 ° C에서 냉장 원심 분리기에 대해 11,000g에서 샘플을 원심 조심스럽게 pipetting하여 뜨는을 제거합니다.
- 차가운 70 % 에탄올 100 μL를 추가하고 부드럽게 튜브의 전체 내벽을 씻어하기 위하여 그것을 사용합니다. 4에 7 분을위한 1만1천g에서 다시 원심 ° C. 표면에 뜨는를 제거하고 10 분 동안 DNA 농축기를 사용하여 펠렛을 건조.
- ddH 2 O와 와동 100 μL의 DNA의 알약을 분해. 260 NM에서 자외선 흡광도에 따라 DNA의 농도를 결정합니다. 스토어 샘플 -20시° C에서 사용까지.
5. 박테리아의 작성
- 목표는 세균 변형 E. 대장균 K12 (MG1655) 및 관련 제어 계통은 첫째 글리세롤 주식에서 LB 한천 플레이트에 도금하고 있습니다. 불꽃에서 또는 생물 학적 안전 캐비닛 내의, LB 한천 손상을 방지하기 위해 플레이트 표면 멸균 피펫 팁 부드럽게 탄력과 세균성 글리세롤 주식을 만져.
- 14 H에 대해 37 ° C에서 streaked 접시와 부화 반전. 부화 후 4 ° C.에 Parafilm (Pechiney 플라스틱 포장) 및 매장과 접시의 전체 주변을 봉인 이 번호판은 4 주간 최대 저장할 수 있습니다.
6. 조잡 세포외 혼합물의 준비 (CEMs)
- 피펫 총 (코닝)을 사용하여 무균 14 ML 문화 튜브 (BD 팔콘)에 LB 2 ML 하는걸.
- 멸균 피펫 팁을 사용하여 5.2 단계에서 준비 한천 플레이트에서 하나의 식민지를 선택하고 AC에 삽입ulture 튜브. 인큐베이터 (뉴 브 런 즈윅 과학)에서 개최되는 튜브는 37 ° C로 설정, 14 H 250 rpm으로 흔들.
- 1% 다시 접종 문화 : 6.2 단계에서 준비 세균성 문화의 20 μL와 14 ML 문화 튜브와 스파이크로 신선한 LB 2 ML 하는걸. 각 세균 솔루션은 약 1 OD 600 (600 NM에서 측정 광학 밀도)에 도달할 때까지 250 rpm으로 흔들어으로 37 ° C에서 튜브를 품어. OD 600을 측정하려면, 자외선 분광 광도계 (Genesys 자외선 10, 써모 과학)과 600 nm의에서 일회용 cuvette하고 측정하는 흡광도 각 문화의 한 ML을 전송.
- 상온에서 5 분 동안 1만1천그램에서 원심 분리하여 새 1.5 ML의 microcentrifuge 튜브 및 펠렛 세포에 각 문화의 한 ML을 전송합니다.
- 즉시 사용하지 않을 경우 -20 ° C에서 신선한 1.5 ML의 microcentrifuge 튜브 및 저장소에 명확한 뜨는을 전송합니다.
7. 형광 분광을 이용하여 감지
- 형광 분광 광도계를 켭니다 (캐리 이클립스, Varian INC)와 520 nm의에서 488 nm의 및 방사에서 여기로 데이터 수집 매개 변수를 설정합니다. 수치는 1 H를 위해 매 순간을 촬영 할 수 있습니다.
- 100 % 에탄올 뒤에 ddH 2 O, 3 개의 석영 크리스탈 cuvettes (Varian 캐리)를 씻으십시오. 질소 가스를 점멸하여 cuvettes 말리. 라벨 cuvettes C1 (제어 1), C2 (제어 2)와 T (테스트).
- C1과 C2와 T. 각 cuvette 및 형광 분광 광도계에 장소를로이 중 25 μL × RB를 추가로 CEM-EC 24 μL로 ddH 2 O 24 μL를 전송합니다. 처음 5 분 형광 데이터 수집을 시작합니다.
- C2와 T와 C1까지 RFD-EC1 1 μL (5 μm의 주식 솔루션에서)에 RFSS1 1 μL를 (5 μm의 주식 용액에서) 추가합니다. pipetting하여 각 솔루션을 섞는다. 이것은 신중하게 형광 수치가 중단되지 않도록 시작되어야합니다. 한 H 수집 시간의 나머지를 계속 반응을 허용합니다.
- 저장엑셀 파일 형식의 데이터는 개인용 컴퓨터와 그래픽 이미지를 만드는 프로세스 데이터에 데이터를 전송합니다.
8. 겔 전기 영동에 의해 감지
7.4 단계에서 준비한 동일한 반응 혼합물은 겔 전기 영동하여 분석을 위해 사용될 수 있으며, 또한 새로운 반응이 비슷하게 준비와 1.5 ML의 microcentrifuge 튜브에 incubated 수 있습니다. 두 경우 :
- 3 M NaOAc 및 100 % 에탄올 125 μL의 5 μL를 추가하여 끄지 반응 (1 H 이후). vortexing하여 각 솔루션을 섞어서 1 H를 위해 -20 ° C의 냉장고에서 튜브를 넣습니다.
- 4에서 20 분 동안 11,000그램에 반응 섞어 원심 ° C와 조심스럽게 pipetting하여 뜨는을 제거합니다.
- 10 분 동안 DNA 농축기를 사용하여 알약을 건조합니다.
- 한 20 μL의 Resuspend 쥐똥 × 간략 vortexing하여 GLB. benchtop의 원심 분리기 (Minicentrifuge, VWR 과학)와 매우 간략하게 튜브를 봐. 이러한 샘플 읽기dPAGE 젤로로드하는 Y.
- 같은 3.1-3.7에 설명된 10 % dPAGE 젤을 준비합니다. pipettor 및 겔 로딩 팁 (Diamed)를 사용하는 우물에 반응 샘플을 (단계 8.4에서)로드합니다. 하단 염료까지 전류 40mA를 (750 V) 적용 (bromophenol 파란색) 접시의 아래쪽 가장자리 위에 약 5cm입니다.
- 유리 접시를 제거하고 모든 겔 조각을 제거하려면 수돗물로 깨끗이 씻는다. kimwipe (킴벌리 - 클라크 프로 페셔널)와 접시를 닦아주십시오.
- 태풍 스캐너 (태풍 9200, 가변 모드, GE 헬스케어)를 사용하여 형광을위한 겔 접시를 검사합니다. ImageQuant 소프트웨어 (분자 역학)를 사용하여 데이터를 분석.
9. 감지 특이성
- 세균성 특이성을 테스트 들어, 동일한 절차 culturing E. 위해 위에서 설명한 대장균은 그 CEM을 준비하고 절단 반응 분석을 실시 같은 B. 등 다양한 세균 변종의 번호를 수행할 수 subtilis, P. peli, Y. ruckeri, L. Planturum, P. acidilactici (그림. 3A 참조).
10. 단일 셀 감지
1 ML E. 준비 대장균의 글리세롤의 2 CFU / ML (CFU : 식민지 - 성형 장치)의 주식 시리얼 희석하여하고 도금하여 CFU 농도를 확인 다섯이 주식 0.2 CFU/100 μL를 포함해야합니다.. -80시 매장 ° C에서 사용까지.
- LB 2 ML을 포함하는 10 문화 튜브를 준비합니다.
- 250 rpm으로 흔들어으로 37 ° C에서 2 CFU / ML 글리세롤 재고와 부화 100 μL으로 각 문화의 예방. 전체 글리세롤 주식의 이용 (1 ML은) 10 문화를 양보해야합니다.
- 4, 8, 12, 16 및 24 H : 다음 시간 지점에서 각각 주사 문화 튜브에서 수확 300 μL. 24 H 위해 성장 남아있는 문화를 둡니다.
- OD 600을 측정하고 5 분 대한 1만1천g에서 원심 분리하여 세포를 침전.
- -20 & D에 신선한 1.5 ML의 microcentrifuge 튜브 및 저장소에 CEMs 전송예를 들어, 사용까지 C #.
참고 : 시간이 포인트 4, 8, 12에서 수확한 시료에 대해 전혀 감지 OD 600이 없을 수도 있기 때문에, 남은 문화가 박테리아를 (탁도와 OD 측정에 의해 결정) 포함하는 문화를 식별하기 위해 24 H 위해 성장할 수 있습니다. 10 문화의 단 하나 또는 두 E.를 포함할 수 있습니다 접종 후 대장균과 남은 튜브는 어떤 세포를 포함하지 않습니다. - 섹션 8에 설명되어있는대로 dPAGE 겔 전기 영동을 사용하여 반응 혼합물을 분석 후 RFD-EC1로 절단 반응을 준비하도록 지정 timepoints에 긍정적인 문화 (-20 ° C에 저장된)에서 발견한 CEMs를 사용합니다.
11. 개념과 대표 결과
세균 검출을위한 RNA-cleaving 형광등의 DNAzyme (RFD)를 이용하는 개념은 그림 그림한다. 1. RFD는 레이블이 세포핵의 어귀 고독한 RNA 결합 (파란색 R)에서 키메라의 DNA / RNA 기판을 클리브형광단 (F) 각각 끄는 (Q)와. 관심 세균 (예 : E. 대장균 등) 미디어에서 자랍니다로서 원유 세포외 혼합물 (CEM) 뒤에 떠날 것이다. 전체 이것은 CEM 그런 다음 CEM에 특히 민감하게 반응하는 것입니다 RFD를 얻기 위해 체외 선택 실험에 사용되는, 아마도 RFD는 세균의 서명 분자이다 CEM의 특정 분자 (보라색 성급)와 상호 작용합니다. CEM은 RFD를 포함하는 반응 용액에 추가되면, 그것은 RFD의 RNA-cleaving 활동을 트리거합니다. 절단 이벤트 fluorimeter이나 겔 전기 영동에 의한를 사용하거나 감지할 수 형광 신호가 발생, Q에서 F를 분리시킵니다.
위의 컨셉의 실험적 검증은 E.에서 CEM으로 이루어졌다 대장균 (CEM-EC). 우리는 체외 선택에 통해 3 RFD 분자를 취득하고, 가장 효율적인 하나는 RFD-EC1 (그림 2A)로 지정되었다. 5 W를e는 CEM-EC에 대한 응답으로 RFD-EC1의 절단 작업을 (RFSS1라는 돌연변이 시퀀스와 함께) 테스트. 두 RFD-EC1 및 RFSS1는 기판 FS1 (모든 시퀀스는 그림에 표시됩니다. 2A)에 DNAzyme 부분 효소 결합에 의해 준비되었다. 형광 측정 실험 (그림 2B)에서 CEM-EC는 RFD-EC1 또는 RFSS1의 추가 다음에 5 분, 위해 55 분 이상에 대한 부화하여 혼자 incubated되었다. 솔루션의 형광 강도는 지속적으로 매 순간을 읽고되었고 데이터 (RF, 시간 t에서의 형광 강도의 비율 대 시간 0 형광 강도로 계산) 상대 형광을 계산하는 데 사용되었습니다. 부화의 시간 대 RF 값은 그림으로 꾸몄다됩니다. 2B. 그것은 RFD-EC1은 CEM-EC의 추가시 형광 신호의 높은 수준의 생산 사실을 발견했다; 스탁 대조적으로, RFSS1는 강한 형광 신호를 생성하지 않았습니다. 따라서 형광 생산 부CEM-EC 문의시 RFD-EC1의 nction는 순서 특정이다.
그 관찰된 형광 증가는 RNA의 결합의 절단에 의한 위치 확인하기 위해, 우리는 dPAGE하여 반응 혼합물을 분석했다. RFD-EC1의 절단은 두 개의 DNA 조각, 5 '형광단을 유지 조각과 3'조각 끄는을 유지를 생성할 것으로 예상된다. 오직 RFD-EC1 (unclv)를 uncleaved하고 5 '조각 (clv)는 형광 이미징에 의해 감지 수 있습니다. dPAGE 결과는 그림. 2C는 RFSS1/CEM-EC 혼합하지 않았 반면 RFD-EC1 및 CEM-EC의 반응 혼합물은 실제로 예상 절단 제품을 생산 것을 보여준다.
RFD-EC1의 특이성은 여러 가지 다른 그램 음성과 그램 양성 세균에서 수집된 데이터를 그림에 표시됩니다 CEMs을 사용하여 조사되었다. 3A. CEM-EC (파란색 곡선)를 포함하는 단 샘플은 형광의 증가를 생산. CEMs와 교차 반응의 부족다른 세균으로부터 RFD-EC1은 E.위한 매우 선택적임을 나타냅니다 대장균.
우리는 또한 culturing 단일 E.에 필요한 시간을 조사 대장균 세포 RFD-EC1의 절단을 일으킬 수있는 충분한 CEM을 생산 순서를 유지해야합니다. 본 실험, E. 위해 대장균 샘플 (식민지 - 성형 단위) CFU을 정의 포함하는 것이 적절 한 CFU / ML의 농도를 달성하기 위해 희석되었다. 이것은 4, 8, 12, 16 및 24 H을위한 성장 매체와 culturing 그것으로 희석 세균 시료 100 μL를 혼합하여 다음되었다. CEMs는 다음 각 timepoint에 대한 수집 및 RFD-EC1의 절단 활동을 유도를 위해 테스트되었습니다. dPAGE 결과는 그림. 3B 12 H의 culturing 시간이 필요했음을 나타냅니다.
빨간색 curv, 그것은 초기 작은 신호 증가 (그림 2B CEM-EC에 RFSS1 시퀀스 (부정적인 컨트롤 등)의 추가 후 형광 측정에서 관찰 점에 유의하는 것이 중요하다E)이나 기타 세균 CEMs (그림 3B까지 RFD-EC1, 파란색을 제외한 모든 곡선)은 FRQ 모듈의 본질적인 형광 (Q에 의한 F의 불완전 담금질에 의한)에 따라 결정됩니다. 그러므로, 그것은 F-및 Q-레이블 시퀀스의 추가는 초기 형광 증가를 생산할 것이라고 예상된다. 단, RFD-EC1/CEM-EC 혼합물은 시간에 따른 형광 높은 수준의 제작이 가능합니다.

그림 1. RNA-cleaving 형광 DNAzyme (RFD) 탐침의 도식 그림 관심있는 특정 박테리아 세포에 의해 생산되는 원유 세포외 혼합물 (CEM)와 접촉시 fluoresces. RFD는 각각 형광단 (F)과 끄는 (Q)로 표시된 두 세포핵의 어귀 고독한 RNA 결합 (파란색 R)에서 키메라의 DNA / RNA 기판을 클리브. 절단 반응하기 전에 RFD의 형광 수준은 가까운 prox로 인해 최소한의 것입니다절단시 F와 질문의 imity은 Q를 F 출발, 결과, 강한 형광 신호가 생산됩니다.
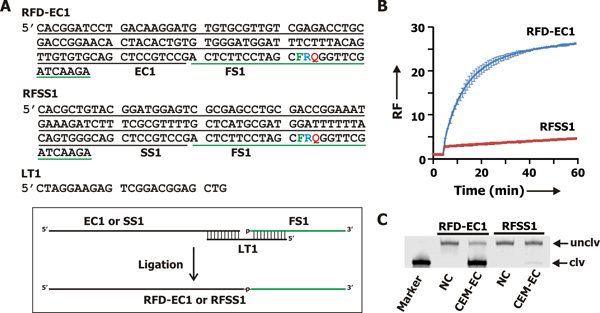
그림 2. E. 대장균 - 센싱은 RFD. () RFD-EC1은 CEM-EC에 의해 활성화될 수 DNAzyme 프로브입니다. RFSS1는 컨트롤로 사용 RFD-EC1의 불 가능한 시퀀스입니다. RFD-EC1 및 RFSS1은 템플릿으로 LT1의 면전에서, 각각 EC1 및 SS1으로 FS1을 ligating에서 생산되었다. F : 플루오레신으로 수정한의 deoxythymidine. Q : Dabcyl으로 수정한의 deoxythymidine. R : 아데닌 ribonucleotide. (B) 형광은 CEM-EC의 면전에서 RFD-EC1 및 RFSS1의 프로파일을 신호. B의 절단 반응 혼합물의 (C) dPAGE 분석 (반응 시간 : 60 분). 사진은 태풍 스캐너가 함께 얻은 dPAGE 겔의 형광 이미지입니다. 레인 NC : 혼자 반응 버퍼에 RFD-EC1 또는 RFSS1, 차선 CEM-EC : CEM-EC를 포함하는 반응 버퍼에 RFD-EC1 또는 RFSS1. 마커: RFD-CE1 0.25 N NaOH, RNA의 전체 절단을 일으키는 것으로 알려진 프로 시저로 치료. unclv : RFD-EC1 uncleaved. clv : 형광단을 포함 절단 조각.
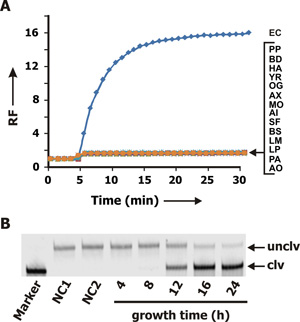
그림 3. () 형광 각종 세균 세포에서 준비 CEMs에 RFD-EC1의 프로필을 신호. EC : 에스 대장균-K12, PP : 모나스 peli; BD : Brevundimonas diminuta; HA : Hafnia alvei; YR : Yersinia ruckeri; OG : Ochrobactrum grignonese; AX : Achromobacter xylosoxidans, 미주리 : Moraxella osloensis; AI : Acinetobacter lwoffi; SF : Serratia fonticola, BS : 바실러스 subtilis, LM : Leuconostoc mesenteroides의, LP : 락토 planturum, PA : Pediococcus acidilactici, AO : Actinomyces orientalis. 각 CEM 샘플은 RFD-EC1의 추가 다음 5 분 동안 incubated되었다. (B) dPAGE60 분 반응 후 RFD-EC1/CEM-EC 혼합의 분석. 레인 NC1 : 반응 완충액 혼자의 RFD-EC1. 레인 NC2 : CEM-BS (바실러스 subtilis에서 준비한 CEM)를 포함하는 반응 버퍼에 RFD-EC1. 단일 E를 포함하는 세균성 문화에서 가져온 CEM-EC를 포함하는 반응 버퍼에 RFD-EC1 : 4, 8, 12, 16 및 24으로 표시 차선 4의 성장 기간에 따라 대장균 세포, 8, 12, 16, 24 H, 각각.
토론
일반 세균의 검출 방법의 대부분은 오늘도 느려 (클래식 미생물) 또는 기술적 요구 (항체, PCR)입니다. 따라서, 우리는 탐지 도구의 차세대 속도와 단순으로 수용해야한다고 믿습니다. 이를 위해, 우리는 RNA-cleaving 및 형광 신호의 생성을 통해 박테리아의 존재를보고 간단한 assays을 개발하는 데 사용할 수있는 형광 - 신호 DNAzyme을 만들었습니다. 기능 DNAzyme 프로브, RFD-EC1은 E.의 성장 기간 동안 생산 CEM에 의해 활성화됩니다 문화 미디어의 대장균. 우리의 방법은 감지의 대상으로 세균의 원유 세포외 혼합물을 사용하며, 힘드는 대상 추출 및 증폭 단계를 무시하기 때문에, 그것은 매우 간단한 설정하는 데 사용할 수 있습니다, 세균 검출을위한 assays의 유형을 '믹스 앤 읽기 ". 우리 DNAzyme의 사용은 형광 기반의 검출 방법에 제한되지 않습니다. 예를 들어, colorimetric 탐지 같은 DNAzyme 시스템 분석 C를 사용은 유기 염료와 함께 압연 원형 증폭을 이용 이전에 보고된 방법을 사용하여 설계한다. 32 우리는 더 큰 운영 단순 새로운 세균 biosensors를 생성하는 매력적인 가로수와 같은 세균 검출을위한 DNAzymes의 사용을 예견한다.
공개
관심의 어떠한 충돌 선언 없습니다.
감사의 말
이 작품에 대한 자금 지원은 자연 과학 및 캐나다의 공학 연구 협의회 (NSERC)과 센티넬 Bioactive 종이 네트워크에 의해 제공되었다.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 또는 모델 | |
| 한천 | BioShop 캐나다 | AGR003 | |
| 암모늄 persulfate (APS) | BioShop 캐나다 | AMP001 | |
| 아크릴 아미드 / 비스 아크릴 아미드 (40 %, 29:1) | BioShop 캐나다 | ACR004 | |
| 붕산 | BioShop 캐나다 | BOR001 | |
| Bromophenol 블루 | 시그마 - 올드 리치 | B8026 | |
| EDTA (에틸렌 다이아 민 테트라 초산) | EM 과학 | EXO539-1 | |
| HCL | 시그마 - 올드 리치 | 38,281 | |
| HEPES | Bioshop 캐나다 | HEP001 | |
| LB의 국물 | 시그마 - 올드 리치 | L3022 | |
| MgCl 2 | EMD화학 제품 | B10149-34 | |
| NaCl | BioShop 캐나다 | SOD002 | |
| NaOAc | EMD 화학 | SXO255-1 | |
| NaOH | EMD 화학 | SXO590-1 | |
| SDS | BioShop 캐나다 | SDS001 | |
| TEMED | BioShop 캐나다 | TEM001 | |
| 트리스베이스 | BioShop 캐나다 | BST666 | |
| 십대 초반 20 | 시그마 - 올드 리치 | P9416 | |
| 요소 | BioShop 캐나다 | URE001 | |
| Xylenecyanol FF | 시그마 - 올드 리치 | X4126 | |
| DNA 농축기 | 써모 과학 | 학자의 DNA SpeedVac 120 | |
| Millex 필터 유닛 | Millipore | SLGP033RS | |
| 젤로드 도움말 | Diamed | TEC200EX-K | |
| ImageQuant 소프트웨어 | 분자 역학 | 버전 5.0 | |
| Kimwipes | 킴벌리 - 클락 전문 | 34,705 | |
| 미니 Vortexer | VWR | 58816-121 | |
| Parafilm | Pechiney 플라스틱 포장 | PM996 | |
| 배양 접시 | 피셔 과학 | Fisherbrand 08-757-12 | |
| Stripettor 플러스 (피펫 총) | 코닝 | 07764714 | |
| 석영 cuvettes | Varian INC | 66-100216-00 | |
| 흔드는 / 보육 | 뉴 브 런 즈윅 과학 | 클래식 시리즈 C24 | |
| 태풍 스캐너 | GE 헬스케어 | 9200 가변 모드 | |
| 원심 분리기 | Beckman 보습 바로 앞에 달린 풀베는 날 | Allegra X22-R | |
| 자외선 분광 | 써모 과학 | GenesysUV 10 | |
| 형광 분광 | Varian INC | 캐리 이클립스 |
참고문헌
- Zourob, M., Elwary, S., Truner, A. . Principles of Bacterial Detection: Biosensors, Recognition Receptors and Microsystems. , (2008).
- Call, D. R. Challenges and Opportunities for Pathogen Detection Using DNA Microarrays. Crit. Rev. Microbiol. 31, 91-99 (2005).
- Lazcka, O., Campo, D. e. l., J, F., Muñoz, F. X. Pathogen detection: A perspective of traditional methods and biosensors. Biosens. Bioelectron. 22, 1205-1217 (2007).
- Velusamy, V. An overview of foodborne pathogen detection: In the perspective of biosensors. Biotechnol. Adv. 28, 232-254 (2010).
- Ali, M. M. Fluorogenic DNAzyme Probes as Bacterial Indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Liu, J., Cao, Z., Lu, Y. Functional Nucleic Acid Sensors. Chem. Rev. 109, 1948-1998 (2009).
- Li, Y., Lu, Y. . Functional Nucleic Acids for Analytical Applications. , (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Joyce, G. F. Forty Years of In Vitro Evolution. Angew. Chem. Int. Ed. 46, 6420-6436 (2007).
- Breaker, R. R., Joyce, G. F. A DNA enzyme that cleaves RNA. Chem. Biol. 1, 223-229 (1994).
- Cuenoud, B., Szostak, J. W. A DNA metalloenzyme with DNA ligase activity. Nature. 375, 611-614 (1995).
- Chinnapen, D. J., Sen, D. A deoxyribozyme that harnesses light to repair thymine dimers in DNA. Proc. Natl. Acad. Sci. U. S. A. 101, 65-69 (2004).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Silverman, S. K. DNA as a versatile chemical component for catalysis, encoding, and stereocontrol. Angew. Chem. Int. Ed. 49, 7180-7201 (2010).
- Mei, S. H. An efficient RNA-cleaving DNA enzyme that synchronizes catalysis with fluorescence signaling. J. Am. Chem. Soc. 125, 412-420 (2003).
- Liu, Z. Assemblage of signaling DNA enzymes with intriguing metal-ion specificities and pH dependences. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Kandadai, S. A., Li, Y. Characterization of a catalytically efficient acidic RNA-cleaving deoxyribozyme. Nucleic Acids Res. 33, 7164-7175 (2005).
- Rupcich, N. Quenching of fluorophore-labeled DNA oligonucleotides by divalent metal ions: implications for selection, design, and applications of signaling aptamers and signaling deoxyribozymes. J. Am. Chem. Soc. 128, 780-790 (2005).
- Shen, Y., Brennan, J. D., Li, Y. Characterizing the secondary structure and identifying functionally essential nucleotides of pH6DZ1, a fluorescence-signaling and RNA-cleaving deoxyribozyme. Biochemistry. 44, 12066-12076 (2005).
- Chiuman, W., Li, Y. Revitalization of six abandoned catalytic DNA species reveals a common three-way junction framework and diverse catalytic cores. J. Mol. Biol. 357, 748-754 (2006).
- Chiuman, W., Li, Y. Evolution of high-branching deoxyribozymes from a catalytic DNA with a three-way junction. Chem. Biol. 13, 1061-1069 (2006).
- Shen, Y. Catalysis and rational engineering of trans-acting pH6DZ1, an RNA-cleaving and fluorescence-signaling deoxyribozyme with a four-way junction structure. Chem BioChem. 7, 1343-1348 (2006).
- Ali, M. M., Kandadai, S. A., Li, Y. Characterization of pH3DZ1 - An RNA-cleaving deoxyribozyme with optimal activity at pH 3. Can. J. Chem. 85, 261-273 (2007).
- Chiuman, W., Li, Y. Efficient signaling platforms built from a small catalytic DNA and doubly labeled fluorogenic substrates. Nucleic Acids Res. 35, 401-405 (2007).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA MgZ based on a non-classic allosteric design. PLoS ONE. 2, e1224 (2007).
- Shen, Y. Entrapment of fluorescence signaling DNA enzymes in sol gel-derived materials for metal ion sensing. Anal. Chem. 79, 3494-3503 (2007).
- Kandadai, S. A. Characterization of an RNA-cleaving deoxyribozyme with optimal activity at pH 5. Biochemistry. 48, 7383-7391 (2009).
- Zhao, W., Brook, M. A., Li, Y. Periodic assembly of nanospecies on repetitive DNA sequences generated on gold nanoparticles by rolling circle amplification. Methods Mol. Biol. 474, 79-90 (2008).
- Navani, N. K., Mok, W. K., Li, Y. In vitro selection of protein-binding DNA aptamers as ligands for biosensing applications. Methods Mol. Biol. 504, 399-415 (2009).
- Ali, M. M., Li, Y. Colorimetric sensing by using allosteric-DNAzyme-coupled rolling circle amplification and a peptide nucleic acid-organic dye probe. Angew. Chem. Int. Ed. 48, 3512-3515 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유