Method Article
Rilevazione di batteri utilizzando DNAzimi fluorogenici
In questo articolo
Riepilogo
Abbiamo recentemente riportato un nuovo approccio per generare sonde DNAzyme fluorogenici che possono essere applicati per impostare un semplice, "mix-e-leggere" test di fluorescenza per la rilevazione batterica. Queste sonde di DNA specifiche catalizzare il clivaggio di un cromoforo-modificato DNA-RNA chimerico substrato in presenza della miscela grezza extracellulare (CEM) prodotta da un batterio specifico, così traducendo rilevamento batterica nella generazione del segnale di fluorescenza. In questa relazione si descrivono principali procedure sperimentali in cui è occupato di una sonda specifica indicata DNAzyme "RFD-EC1" per il rilevamento del batterio del modello, Escherichia coli (E. coli).
Abstract
Le epidemie legate al cibo-borne e acquisita in ospedale conto patogeni per milioni di morti e ricoveri, nonché colossali perdite economiche ogni anno. La prevenzione di tali epidemie o minimizzare l'impatto di un luogo epidemia in corso una sempre crescente domanda di metodi analitici in grado di identificare con precisione i patogeni colpevole nella fase più precoce. Anche se vi è una vasta gamma di metodi efficaci per l'individuazione degli agenti patogeni, nessuno di loro può soddisfare tutte le seguenti cinque requisiti premier incarnati per un metodo ideale di rilevazione: alta specificità (la rilevazione solo il batterio di interesse), elevata sensibilità (in grado di rilevare il più basso come una singola cellula batterica dal vivo), a breve time-to-risultati (minuti a ore), grande semplicità operativa (senza bisogno di lunghe procedure di campionamento e l'uso di attrezzature specializzate), e l'efficacia dei costi. Ad esempio, classici metodi microbiologici sono altamente specifici, ma richiedono un lungo tempo (days a settimana) per acquisire un risultato definitivo. 1 PCR e tecniche basate su anticorpi offrono brevi tempi di attesa (ore o giorni), ma richiedono l'uso di reagenti costosi e / o apparecchiature sofisticate. 2-4 conseguenza, vi è ancora una grande richiesta per la ricerca scientifica verso lo sviluppo di innovativi metodi di rilevazione di batteri in grado di offrire caratteristiche migliorate in uno o più dei requisiti di cui sopra. Il nostro laboratorio è interessati ad esaminare il potenziale di DNAzimi come una nuova classe di sonde molecolari per applicazioni di biosensori inclusa l'individuazione di batteri. 5
DNAzimi (noto anche come deoxyribozymes enzimi o DNA) sono artificiali elica singola molecole di DNA con la capacità di catalizzare reazioni chimiche. 6-8 Queste molecole possono essere isolato da una vasta casuale sequenza di DNA piscina (che contiene fino a 10 16 singole sequenze) mediante un processo noto come "selezione in vitro" or "SELEX" (evoluzione sistematica di ligandi di arricchimento esponenziale). 9-16 Queste molecole di DNA speciali sono stati ampiamente esaminati negli ultimi anni, come strumenti molecolari per applicazioni di biosensori. 6-8
Il nostro laboratorio ha stabilito nelle procedure di selezione in vitro per l'isolamento di RNA-cleaving DNAzimi fluorescenti (RFD;. Fig. 1) e ha studiato l'uso di RFD come strumenti analitici 17-29 RFD catalizzare la scissione di un substrato di DNA-RNA chimerico in un ribonucleotide singolo. giunzione (R) che è affiancato da un fluoroforo (F) e un quencher (Q). La vicinanza di F e Q rende la fluorescenza non scissa substrato minimo. Tuttavia, l'evento scissione porta alla separazione di F e Q, che è accompagnato da significativo aumento dell'intensità di fluorescenza.
Più recentemente, abbiamo sviluppato un metodo per isolare RFD per la rilevazione dei batteri. 5 Questi RFD speciali sono stati isolati a "illuminare "in presenza della miscela grezza extracellulare (CEM) lasciata da un tipo specifico di batteri nel loro ambiente o nei mezzi di coltura sono essi (Fig. 1). L'uso della miscela grezza aggira il processo di purificazione e tedioso identificazione di un bersaglio adatto dal microbo di interesse per lo sviluppo biosensore (che potrebbe richiedere mesi o anni per completare). L'uso di obiettivi extracellulari: la procedura è semplice saggio perché non vi è alcuna necessità di attivarsi per ottenere bersagli intracellulari.
Utilizzando l'approccio di cui sopra, abbiamo derivato uno RFD che fende il suo substrato (FS1;. Fig. 2A) solo in presenza del CEM prodotta da E. coli (CEM-CE). 5 Questa E. coli-sensing RFD, di nome RFD-EC1 (Fig. 2A), è stato ritenuto strettamente rispondenti alle CEM-CE, ma che non risponde a CEM da una serie di altri batteri (Fig. 3).
Qui Present le procedure chiave sperimentale per la costituzione di E. coli test di rilevazione utilizzando i risultati RFD-EC1 e rappresentativo.
Protocollo
1. Preparazione di soluzioni chimiche
- 0,5 M di etilene diaminetetraacetic acido (EDTA): In un litro 2 (L) bicchiere di plastica, peso 186,1 g EDTA (EM Science) e aggiungere 800 millilitri (ml) di autoclavato deionizzata-acqua distillata (DDH 2 O). Regolare il pH a 8,0 con NaOH pellets (EM Science). Portare al volume finale di 1 L con ddh 2 O. Trasferire la soluzione in bottiglie di vetro, autoclave e conservare a 4 ° C.
- 10 × Tris-borato EDTA soluzione (10 x TBE, 89 mM Tris, 89 mM di acido borico, 2 mM EDTA, pH 7,5): Pesare 432 g di Tris-base (Bioshop Canada) e 220 g di acido borico (Bioshop Canada) , e aggiungere a ciascuno di un bicchiere di plastica 4 L. Misurare 80 ml di 0,5 M EDTA (pH 8,0) e aggiungere al becher. Aggiungere ddh 2 O per un volume finale di 4 L. Mescolare la soluzione con un agitatore magnetico fino a quando i componenti sono completamente disciolti. Trasferire la soluzione in bottiglie di vetro, autoclave e conservare a 4 ° C.
- 10% denaturazione polyacrylamide gel magazzino: per un bicchiere di plastica 4 L, aggiungere 1681,7 g di urea (Bioshop Canada), 400 ml di 10 × TBE, 1 L di 40% di acrilammide / bisacrilammide (29:1) soluzione (Bioshop Canada). Regolare il volume a 4 L con ddh 2 O. Sciogliere l'urea con agitazione. Trasferire la soluzione in 1 L flaconi di vetro ambrato e conservare a 4 ° C. (Attenzione! L'acrilamide deve essere maneggiato con guanti, maschera, occhiali e camice da laboratorio, perché è una neurotossina prima della polimerizzazione).
- 2 × gel loading buffer (2 × GLB): In un becher da 200 ml in vetro, aggiungere 44 g di urea, 8 g di saccarosio (Bioshop Canada), 10 mg di blu di bromofenolo (Bioshop Canada), 10 mg di xylenecyanol FF (Sigma -Aldrich), 400 pl di 10% solfato di sodio dodecil (SDS; Bioshop Canada), e 4 ml di 10 x TBE. Regolare il volume finale a 40 mL con ddh 2 O e sciogliere i solidi con blando riscaldamento (50 ° C) e con una barra di agitazione magnetica. Trasferire 1 mL aliquota in provette per microcentrifuga da 1,5 ml e conservare a 4 ° C. Sulla base della nostraesperienza, è necessario calore 2 × brevemente GLB a 90 ° C prima dell'uso perché 2 × GLB solidifica sotto le condizioni di conservazione.
- 1 M Tris-HCl (pH 7,5): Pesare 12,1 g di Tris-base in un becher di 200 ml in vetro. Aggiungere 60 mL di ddh 2 O e sciogliere i solidi da agitazione con un agitatore magnetico. Aggiustare il pH a 7,5 con 1 M HCl (Sigma-Aldrich). Portare il volume a 100 mL con ddh 2 O e versare in un bottiglia di vetro e di autoclave. Conservare a 4 ° C.
- 5 M NaCl: In bicchiere di vetro peso 58,4 g di NaCl (Bioshop Canada) e sciogliere con 150 mL di ddh 2 O. Regolare il volume a 200 mL con ddh 2 O. Trasferire la soluzione ad un bottiglia di vetro, autoclave e conservare a 4 ° C.
- DNA tampone di eluizione: In un bicchiere di vetro, mescolare 2 ml di 1 M Tris-HCl (pH 7,5), 8 ml di NaCl 5 M e 0,4 mL di 0,5 M EDTA (pH 8,0). Regolare il volume a 200 mL con ddh 2 O. Autoclave e conservare a 4 ° C.
- 2 × buffer di reazione (2 × RB, 100 mM di HEPES, 300 mM NaCl, 30 mM MgCl 2): Ad una provetta Falcon 50 mL (BD Falcon), aggiungono 1,2 g di HEPES (Bioshop Canada), g 0,88 di NaCl e 0,30 g di MgCl 2 • 6H 2 O (EMD Chemicals) e 30 ml di ddh 2 O. Mescolare agitando leggermente fino a quando i componenti sono completamente sciolto. Aggiustare il pH a 7,5 con l'aggiunta di soluzione di NaOH 10 N e regolare il volume finale a 50 mL con ddh 2 O. Purificare la soluzione utilizzando una siringa-driven gruppo filtro (0,22 micron, Millipore) e conservare a 4 ° C.
- Luria Bertani (LB) Broth: In un becher, pesa 20,0 g di polvere LB (Sigma-Aldrich), seguita dalla aggiunta di 1 L di ddh 2 O. Mescolare la soluzione con una ancoretta magnetica, trasferire la soluzione in una beuta. Autoclavare la soluzione e conservare a temperatura ambiente.
- LB agar 1,5%: Pesare 1,5 g di agar (Bioshop Canada) in un pallone da 250 mL e aggiungere 100 ml di LB liquido. Sterilizzare il composto e conservare a temperatura ambiente.
- Agar placcatura: Melt l'agar LB in un forno a microonde e raffreddare la soluzione a ~ 50 ° C. Versare la soluzione in piastre Petri (Fisher Scientific) sotto una fiamma. Nella nostra esperienza, 100 ml di agar LB può produrre 5-6 piatti.
2. Costruzione di RFD-EC1 e RFSS1 da Template Mediata Ligation enzimatico
RFD-EC1 (Fig. 2A) è il DNAzyme featured. Essa consiste nel EC1 sequenza catalitico e il substrato sequenza FS1 (indicato da linee nere e verdi in Fig. 2A). RFSS1 (Fig. 2A) è una versione di scrambled RFD-EC1 dove la sequenza EC1 catalitico viene parzialmente rimescolato nel SS1 ma porzione FS1 rimane invariata. RFD-EC1 e RFSS1 sono state fatte da modello mediata ligazione enzimatica del FS1 oligonucleotide con EC1 oligonucleotide o SS1 in presenza di LT1 come templato legatura (vedere la scatola inserita in Fig. 2A). La procedura per condurre la reazione di ligazione è fornita di seguito.FS1 è stato ottenuto da Keck Servizi sintesi Oligo alla Yale University, deprotetti e purificati mediante elettroforesi su gel a seguito di un protocollo stabilito in precedenza. 17-24 EC1, SS1 e LT1 sono stati acquistati da Technologies DNA integrato e purificato mediante elettroforesi su gel.
- Preparare una soluzione stock 100 micron di FS1, EC1, SS1 e LT1 con ddh 2 O. Conservare a -20 ° C fino al momento dell'uso.
- Trasferire 5 microlitri di soluzione di riserva FS1 a due provette da microcentrifuga 1,5 microlitri contrassegnati come provetta 1 e tubo 2. Per ogni provetta, aggiungere 38,5 pl di ddh 2 O e poi 5 microlitri di 10 × polinucleotide chinasi T4 (PNK) Un tampone di reazione (MBI Fermentas), che contiene 500 mM Tris-HCl (pH 7,6, 25 ° C), 100 mM MgCl 2, 50 mM DTT, 1,0 mM spermidina. Mescolare ogni soluzione pipettando (pipetta la soluzione su e giù un paio di volte).
- Aggiungere 1 ml di ATP (100 mM; MBI Fermentas) e mescolare pipettando.
- Aggiungere 0,5 pl di polinucleotide chinasi T4 (PNK, 10 unità / pl; Fermentas MBI) e mescolare pipettaggio. Incubare le miscele di reazione a 37 ° C per 30 min. Assicurati di coprire le provette con foglio di alluminio per ridurre al minimo photobleaching del fluoroforo.
- Raffreddare bruscamente la reazione mediante riscaldamento a 90 ° C per 5 min. Raffreddare le miscele di reazione a temperatura ambiente per 10 min.
- Aggiungere 5 microlitri della EC1 e SS1 soluzione madre alla fermata della metropolitana 1 e tubo 2, rispettivamente.
- Aggiungere 5 microlitri di soluzione di riserva LT1 a ciascuna provetta, pipettando. Riscaldare le miscele di reazione a 90 ° C per 1 min e raffreddare a temperatura ambiente per 10 min.
- Aggiungere 118 pl di ddh 2 O e poi 20 pl di 10 x tampone di T4 DNA ligasi (MBI Fermentas), che contiene 400 mM Tris-HCl (pH 7,8 a 25 ° C), 100 mM MgCl 2, 100 mM DTT, e 5 mM ATP. Mescolare la soluzione pipettando.
- Aggiungere 2 microlitri di T4 DNA ligasi (5 unità / microlitro; MBI Fermentas) e mescolare pipettaggio. Incubare le miscele di reazione a temperatura ambiente per 1 h.
- Aggiungere 20 microlitri di 3 M NaOAc (pH 7,0) a ciascun vortice tubo e spin down. Aggiungere 500 pl di etanolo 100% freddo a ciascun tubo, miscelare la soluzione con vortex e posizionare i tubi in congelatore a -20 ° C per 30 min.
- Centrifugare le miscele a 11.000 g per 20 minuti a 4 ° C in una centrifuga refrigerata (Allegra X22-R, Beckman Coulter) e rimuovere il supernatante pipettando.
- Essiccare il pellet di DNA utilizzando un concentratore DNA (Savant DNA SpeedVac, Thermo Scientific) per 10 min.
- Pellets Risospendere DNA in 30 microlitri di 1 × tampone di caricamento gel (GLB), brevemente vortice e spin down, con una centrifuga da banco (Minicentrifuge, VWR Scientific). I campioni di DNA legatura sono pronti per il caricamento su un gel dPAGE 10%.
3. Preparazione del gel dPAGE 10%
Le seguenti operazioni brevemente descrive l'apparecchio di elettroforesi su gel e il set-up. Per maggiori dettagli sulle impostazioni, le apparecchiature e la gestione, fare riferimento tO i nostri protocolli precedentemente pubblicati. 30,31
- Lavare e asciugare due lastre di vetro, due distanziatori 0,75 mm e uno di 16 e pettine. Montare lastre di vetro e distanziali con clip e laici orizzontalmente su una superficie piana con lastra di vetro intagliato verso l'alto.
- Trasferire 40 ml di miscela dPAGE 10% in un becher da 150 mL. Aggiungere 40 microlitri di tetrametiletilendiammina (TEMED; Bioshop Canada), 400 microlitri di APS al 10%, e mescolare con una pipetta.
- Versate il composto con cura tra le piastre e inserire immediatamente il pettine. Permettere alla miscela di polimerizzazione per 10 a 20 min. La polimerizzazione può essere confermata controllando la miscela residua gel lasciata nel becher.
- Una volta polimerizzato, rimuovere il pettine con delicatezza e sciacquare i pozzetti con ddh 2 O per rimuovere la soluzione residua gel nei pozzetti.
- Montare le piastre sul dispositivo gel elettroforesi con la piastra senza intaglio rettangolare rivolto verso l'esterno e mettere una placca di metallo dietro la piastra dentata. L'uso del metallo plmangiato aiuta a prevenire il surriscaldamento che può rompere le lastre di vetro.
- Aggiungere 1 × TBE alle camere superiore ed inferiore dell'apparato. Verificare che i pozzetti vengono riempiti con il tampone e il bordo inferiore del gel viene immerso nel buffer.
- Applicare un 40 mA (o 750 V) corrente e pre-run da 10 a 15 min.
4. Purificazione di ligato RFD-EC1 e RFSS1 da Gel dPAGE 10%
- Dopo il passo di 3,7, sciacquare i pozzetti con 1 x TBE utilizzando una siringa e un ago.
- Caricare la miscela di ligazione di RFD-EC1 e quella di RFSS1 (da 2,13) in 2 pozzi (uno per ciascuno) con una pipetta e punte di carico gel (DiaMed). Applicare a 40 mA (750 V) finché la corrente inferiore colorante (blu di bromofenolo) è circa 5 cm sopra il bordo inferiore delle piastre.
- Rimozione lastre di vetro dall'apparecchio funzionamento gel, sdraiarsi su un piano di panca piana e rimuovere con attenzione i distanziali.
- Rimuovere accuratamente le lastre di vetro migliori dal gel eavvolgere il gel con pellicola trasparente (cercare di evitare rughe e pieghevoli dei rivestimenti in gel).
- I prodotti ligati possono essere visualizzate mediante shadowing UV (260 nm) o mediante transilluminazione (360 nm), che produrrà una banda visibile DNA pochi centimetri EC1 o SS1 (100 pmol di EC1 o SS1 può essere utilizzato come marcatore e caricati in un pozzo nella fase di 4,2). Contrassegnare le bande di DNA desiderati con un pennarello.
- Excise la banda di DNA con lamette sterili, tagliare il gel in piccoli pezzi e il trasferimento in una nuova provetta 1,5 ml microcentrifuga.
- Schiacciare i pezzi gel all'interno della provetta con una punta di pipetta sterile (200 size punta mL).
- Aggiungere 500 microlitri di buffer di eluizione del DNA per ogni provetta e coprirli con un foglio di alluminio per proteggere i fluorofori dalla luce. Vortex i campioni per 10 minuti.
- Centrifugare i campioni a 11.000 g per 4 minuti a 4 ° C in una centrifuga refrigerata (Allegra X22-R, Beckman Coulter) e trasferire con cautela 350 microlitri della supernatant in una nuova provetta da microcentrifuga da 1,5 mL (se necessario, una seconda eluizione può essere fatto con 350 pl di tampone di eluizione fresco per altri 10 minuti).
- Aggiungere 35 microlitri (0,1 × del volume del campione) di acetato di sodio 3 M (NaOAc, pH 7,0) in ciascuna provetta, mescolare con vortex e spin down. Aggiungere 900 uL di freddo etanolo al 100% ad ogni provetta. Mescolare ciascun campione mano agitando la provetta per alcuni secondi. Porre i tubi a -20 ° C per almeno 1 h.
- Centrifugare i campioni a 11.000 g per 20 minuti a 4 ° C in una centrifuga refrigerata e rimuovere il supernatante pipettando.
- Aggiungere 100 pl di etanolo freddo al 70% e utilizzarlo per risciacquare delicatamente l'intera parete interna del tubo. Re-centrifuga a 11.000 g per 7 minuti a 4 ° C. Rimuovere il surnatante e asciugare il pellet usando il concentratore DNA per 10 min.
- Sciogliere il pellet di DNA in 100 pl di ddh 2 O e vortex. Determinare la concentrazione di DNA basata sulla assorbanza UV a 260 nm. Conservare i campioni a -20° C fino all'uso.
5. Preparazione di batteri
- L'obiettivo ceppo batterico E. coli K12 (MG1655) e ceppi di controllo rilevanti sono da un lato piastrati su piastre di agar LB dalle scorte di glicerolo. Sotto una fiamma o all'interno di una cappa di sicurezza biologica, toccare il brodo batterica glicerolo con una pipetta sterile e delicatamente striscia sulla superficie della lastra per evitare di danneggiare l'agar LB.
- Invertire le piastre striato e incubare a 37 ° C per 14 h. Dopo l'incubazione, sigillare tutto il perimetro delle piastre con Parafilm (Pechiney imballaggi in plastica) e conservare a 4 ° C. Queste piastre possono essere conservato per un massimo di 4 settimane.
6. Preparazione del greggio miscele extracellulari (CEM)
- Dispensare 2 ml di LB in provette di coltura sterili 14 ml (BD Falcon) utilizzando una pistola pipetta (Corning).
- Utilizzando una punta di pipetta sterile, scegliere una singola colonia da una piastra di agar preparata nella fase di 5.2 e inserirlo in acultura tubo. Porre le provette in un incubatore (New Brunswick Scientific) impostato a 37 ° C, e agitare a 250 rpm per 14 h.
- 1% re-inoculo cultura: Dispensare 2 ml di LB fresco in 14 ml provette di coltura e spike con 20 microlitri di colture batteriche preparati in fase 6.2. Incubare le provette a 37 ° C con agitazione a 250 rpm fino ciascuna soluzione batterica raggiunge un OD 600 (densità ottica misurata a 600 nm) di circa 1. Per misurare OD 600, trasferire 1 ml di ogni cultura di una cuvetta monouso e misura di assorbanza a 600 nm con uno spettrofotometro UV (UV Genesys 10, Thermo Scientific).
- Trasferire 1 ml di ciascuna coltura ad una nuova provetta 1,5 mL microcentrifuga e cellule pellet mediante centrifugazione a 11.000 g per 5 min a temperatura ambiente.
- Trasferire il surnatante limpido in una nuova provetta 1,5 ml microcentrifuga e conservare a -20 ° C se non utilizzato immediatamente.
7. Rilevamento mediante spettrofotometro a fluorescenza
- Accendere spettrofotometro a fluorescenza (Cary Eclipse, Varian Inc) e impostare i parametri di acquisizione dati con eccitazione a 488 nm ed emissione a 520 nm. Le letture possono essere prese ogni minuto per 1 h.
- Lavare 3 cuvette di quarzo (Varian Cary) con ddh 2 O, seguito da 100% di etanolo. Asciugare le cuvette facendo lampeggiare gas azoto. Etichetta cuvette C1 (controllo 1), C2 (controllo 2) e T (test).
- Trasferire 24 microlitri di ddh 2 O di C1 e 24 pl di CEM-CE, in C2 e T. Aggiungere 25 microlitri di 2 × RB ad ogni cuvetta e metterli nello spettrofotometro a fluorescenza. Inizia raccolta dati di fluorescenza per i primi 5 minuti.
- Aggiungere 1 ml di RFSS1 (da una soluzione al 5 mM stock) in C2 e 1 ml di RFD-EC1 (da una soluzione al 5 mM stock) a T e C1. Mescolare ogni soluzione pipettando. Questo deve essere attentamente avviato in modo che le letture della fluorescenza non vengano interrotti. Lasciare la reazione di continuare per il resto del tempo di acquisizione 1 h.
- Salvarei dati in formato di file Excel, trasferire i dati ad un personal computer ed elaborare i dati per creare una immagine grafica.
8. Rilevazione mediante elettroforesi su gel
Le miscele di reazione stesse preparate nel passaggio 7,4 può essere utilizzato per l'analisi mediante elettroforesi su gel, in alternativa nuove reazioni può essere preparata in maniera simile e incubate in provette per microcentrifuga da 1,5 ml. In entrambi i casi:
- Reazioni di estinzione (dopo 1 h) aggiungendo 5 pl di NaOAc 3 M e 125 pl di etanolo al 100%. Miscelare ciascuna soluzione con vortex e posizionare i tubi in congelatore a -20 ° C per 1 h.
- Centrifugare le miscele di reazione a 11.000 g per 20 minuti a 4 ° C e rimuovere il supernatante pipettando.
- Essiccare i granuli utilizzando il concentratore DNA per 10 min.
- Pellets Risospendere in 20 microlitri di 1 × GLB brevemente vortex. Spin down tubi molto brevemente con una centrifuga da banco (Minicentrifuge, VWR Scientific). Questi campioni vengono lettiy per il caricamento in un gel dPAGE.
- Preparare un gel dPAGE del 10% come indicato in 3,1-3,7. Caricare i campioni di reazione (dal punto 8.4) nei pozzetti con una pipetta e punte di carico gel (DiaMed). Applicare a 40 mA (750 V) finché la corrente inferiore colorante (blu di bromofenolo) è circa 5 cm sopra il bordo inferiore delle piastre.
- Rimozione lastre di vetro e lavare accuratamente con acqua del rubinetto per rimuovere eventuali pezzi di gel. Pulire le piastre con un Kimwipe (Kimberly-Clark Professional).
- Scansione la piastra gel per fluorescenza utilizzando uno scanner Typhoon (Tifone 9200, Modalità variabile, GE Healthcare). Analizzare i dati utilizzando un software ImageQuant (Molecular Dynamics).
9. Rilevamento Specificità
- Per testare specificità batterica, le stesse procedure sopra descritte per la coltura E. coli, la sua preparazione CEM e conducendo un test di reazione di scissione può essere eseguita per un certo numero di diversi ceppi batterici come B. subtilis, P. Peli, Y. ruckeri, L. planturum, P. acidilactici (mostrato in Fig. 3A).
10. Cellula di rilevamento singolo
Preparare una mL 1 E. glicerolo magazzino coli di 2 CFU / ml (CFU: unità formanti colonia) per diluizione seriale e confermare la concentrazione di CFU da placcatura 5 Questa riserva dovrebbe contenere ufc/100 0,2 microlitri.. Conservare a -80 ° C fino all'utilizzo.
- Preparare 10 provette di coltura contenente 2 ml di LB.
- Inoculare ogni coltura con 100 microlitri di 2 CFU / mL archivio glicerolo e incubare a 37 ° C con agitazione a 250 rpm. L'uso del magazzino glicerolo intero (1 mL) dovrebbe dare 10 culture.
- Harvest 300 microlitri da ogni provetta di coltura inoculato nei seguenti punti temporali: 4, 8, 12, 16 e 24 h. Lasciare la cultura rimanente di crescere per 24 h.
- Misurare la OD 600 e precipitare le cellule mediante centrifugazione a 11.000 g per 5 min.
- Trasferire i CEM a tubi da microcentrifuga da 1,5 ml freschi e conservare a -20 & deg; C fino all'utilizzo.
Nota: Poiché non ci può essere rilevabile OD 600 per i campioni raccolti in fase di punti 4, 8 e 12, permettono la cultura rimanente di crescere per 24 ore al fine di individuare colture contenenti batteri (determinata dalla torbidità e misurazione OD). Solo uno o due dei 10 colture possono contenere E. coli dopo l'inoculazione e dei tubi rimanenti non conterrà tutte le celle. - Utilizzare i CEM recuperate da culture positive (conservati a -20 ° C) ai tempi di valutazione designati per preparare le reazioni di dissociazione con RFD-EC1, e quindi analizzare le miscele di reazione con elettroforesi su gel dPAGE come descritto nella Sezione 8.
11. Concetto e Rappresentante Risultati
Il concetto di sfruttare una RNA-scissione DNAzyme fluorescente (RFD) per il rilevamento batterica è illustrato in Fig. 1. Il RFD fende un DNA chimerico / substrato di RNA in un RNA unico collegamento (blu R) affiancato da due nucleotidi marcaticon un fluoroforo (F) e un quencher (Q), rispettivamente. Come un batterio di interesse (ad esempio E. coli) cresce in media, si lascia dietro una miscela grezza extracellulare (CEM). Questo CEM nel suo complesso viene poi utilizzato in un esperimento di selezione in vitro per ottenere un RFD che risponde specificamente alla CEM, presumibilmente dell'RFD interagisce con una molecola specifica (viola stella) nel CEM che è una molecola firma del batterio. Quando il CEM viene aggiunto alla soluzione di reazione contenente il RFD, si innesca l'RNA-clivaggio attività del RFD. L'evento clivaggio separa da Q F, risultando in un segnale fluorescente che può essere rilevato sia utilizzando un fluorimetro o mediante elettroforesi su gel.
La verifica sperimentale del concetto sopra è stato fatto con il CEM da E. coli (CEM-CE). Abbiamo ottenuto 3 molecole RFD tramite selezione in vitro, e il più efficiente è stato designato come RFD-EC1 (Fig. 2A). 5 We verificato l'attività di scissione RFD-EC1 (insieme a una sequenza mutante denominato RFSS1) in risposta a CEM-CE. Sia RFD-EC1 e RFSS1 sono stati preparati mediante ligazione enzimatica delle porzioni DNAzyme al substrato FS1 (tutte le sequenze sono mostrate nella fig. 2A). Nell'esperimento di misurazione di fluorescenza (Fig. 2B), CEM-CE è stata incubata per 5 minuti sola, seguita dall'aggiunta di RFD-EC1 o RFSS1, e da un'ulteriore incubazione per 55 min più. L'intensità di fluorescenza della soluzione è stata continuamente ogni minuto e leggere i dati è stato utilizzato per calcolare fluorescenza relativa (RF, calcolato come rapporto tra l'intensità di fluorescenza al tempo t contro l'intensità di fluorescenza a tempo 0). I valori RF contro il tempo di incubazione sono tracciate come Fig. 2B. Si è constatato che RFD-EC1 prodotto un elevato livello di segnale di fluorescenza dopo l'aggiunta di CEM-CE, in contrasto, RFSS1 non produrre un forte segnale di fluorescenza. Così, la fluorescenza produce function di RFD-EC1 venendo a contatto con CEM-EC è sequenza-specifica.
Al fine di verificare che aumenti osservati fluorescenza sono dovuti al clivaggio del legame dell'RNA, abbiamo analizzato miscele di reazione da dPAGE. La scissione di RFD-EC1 dovrebbe generare due frammenti di DNA, un 5 'frammento mantenendo il fluoroforo e 3' frammento mantenendo il quencher. Solo non scissa RFD-EC1 (unclv) e il 5 'frammento (CLV) potrebbe essere rilevato da immagini a fluorescenza. Il risultato dPAGE mostrato nella fig. 2C rivela che la miscela di reazione di RFD-EC1 e CEM-EC infatti prodotto il prodotto atteso scissione, mentre la miscela RFSS1/CEM-EC no.
La specificità di RFD-EC1 è stata esaminata usando CEM raccolti da diversi altri batteri gram negativi e gram positivi ei dati è mostrato in fig. 3A. Solo il campione contenente CEM-CE (blu curva) ha prodotto un aumento della fluorescenza. La mancanza di reattività crociata con CEMdagli altri batteri indica che RFD-EC1 è altamente selettivo per E. coli.
Abbiamo anche analizzato il tempo necessario per la coltura di una singola E. coli cella per produrre CEM sufficiente che può indurre la scissione di RFD-EC1. Per questo esperimento, uno E. coli campione contenente definito CFU (unità formanti colonia) è stato adeguatamente diluita per ottenere la concentrazione di 1 CFU / ml. Questo è stato seguito da miscelazione 100 pl del campione diluito con mezzi di crescita batterica e coltura per 4, 8, 12, 16 e 24 h. CEM sono stati poi raccolti per ciascun tempo di valutazione e testati per indurre l'attività di scissione di RFD-EC1. Il risultato dPAGE mostrato nella fig. 3B indica che un tempo di coltura 12 h è necessaria.
È importante notare che l'aumento iniziale piccolo segnale osservato in misurazioni di fluorescenza dopo l'aggiunta di RFSS1 sequenza (come controllo negativo) a CEM-CE (Fig. 2B; rosso curve) o RFD-EC1 ad altri CEM batteriche (Fig. 3B, tutte le curve eccetto blu) è attribuita alla fluorescenza intrinseca del modulo FRQ (a causa di quenching incompleto di F da Q). Pertanto, si prevede che l'aggiunta di F e Q-marcati sequenze produrrebbe un aumento iniziale fluorescenza. Tuttavia, solo miscele RFD-EC1/CEM-EC sono in grado di produrre un elevato livello di fluorescenza nel tempo.

Figura 1. Illustrazione schematica del RNA-tagliente fluorescente DNAzyme (RFD) sonda che reagisce al contatto con la miscela grezza extracellulare (CEM) prodotta da particolari cellule batteriche di interesse. Il RFD fende un DNA chimerico / RNA substrato a RNA isolato collegamento (blu R) affiancato da due nucleotidi marcati con un fluoroforo (F) e un quencher (Q), rispettivamente. Prima della reazione di scissione, il livello di fluorescenza della RFD è minima a causa della stretta PROXimity di F e Q. Al clivaggio, Q parte da F, come risultato, un segnale forte fluorescenza viene prodotto.
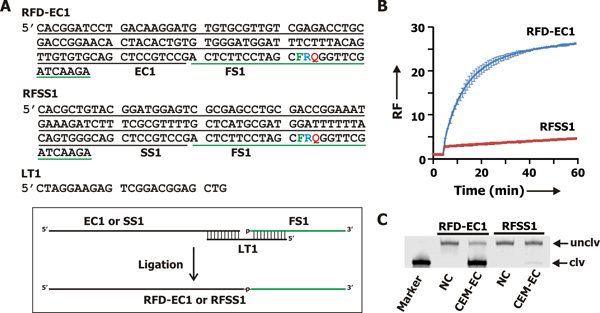
Figura 2. La E. coli-sensing RFD. (A) RFD-EC1 è la sonda DNAzyme che può essere attivato da CEM-EC. RFSS1 è codificato sequenza di RFD-EC1 usato come controllo. RFD-EC1 RFSS1 e sono state prodotte legando FS1 con EC1 e SS1, rispettivamente, in presenza di LT1 come templato. F: fluoresceina-deossitimidina modificato. Q: DABCYL-modified deossitimidina. R: ribonucleotide adenina. (B) fluorescenza segnalazione profili di RFD-EC1 e RFSS1 in presenza di CEM-CE. (C) analisi dPAGE delle miscele di reazione dei scissione in B (tempo di reazione: 60 min). Nella foto è una immagine di fluorescenza del gel ottenuto con dPAGE da scanner Typhoon. Lane, NC: RFD-EC1 o RFSS1 nel tampone di reazione solo; Lane, CEM-EC: RFD-EC1 o RFSS1 nel buffer di reazione contenente CEM-CE. Marcatore: RFD-CE1 trattata con 0,25 N NaOH, un procedimento noto per provocare scissione completa di RNA. unclv: non scissa RFD-EC1. CLV: il frammento di clivaggio contenente fluoroforo.
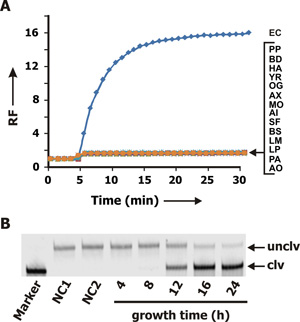
Figura 3. (A) fluorescenza segnalazione profilo di RFD-EC1 in CEM preparati da varie cellule batteriche. EC: Escherichia coli K12, PP: Pseudomonas Peli; BD: Brevundimonas diminuta; HA: Hafnia alvei; YR: Yersinia ruckeri; OG: Ochrobactrum grignonese; AX: xylosoxidans Achromobacter; MO: Moraxella osloensis; AI: Acinetobacter lwoffi; SF: Serratia fonticola; BS: Bacillus subtilis; LM: Leuconostoc mesenteroides; LP: planturum Lactobacillus; PA: Pediococcus acidilactici; AO: Actinomyces orientalis. Ogni campione CEM è stata incubata per 5 minuti seguita dalla aggiunta di RFD-EC1. (B) dPAGEanalisi di miscele RFD-EC1/CEM-EC dopo 60 min di reazione. Corsia NC1: RFD-EC1 nel buffer solo per reazione. Corsia NC2: RFD-EC1 nel tampone di reazione contenente CEM-BS (CEM preparato da Bacillus subtilis). Le corsie marcate con 4, 8, 12, 16 e 24: RFD-EC1 nel tampone di reazione contenente CEM-EC prelevato dalla coltura batterica contenente un singolo E. coli cellule dopo un periodo di crescita di 4, 8, 12, 16 e 24 ore, rispettivamente.
Discussione
La maggior parte dei metodi comuni di rilevazione batteriche oggi sono o lento (microbica classico) o tecnicamente impegnativo (anticorpi, PCR). Pertanto, riteniamo che la prossima generazione di strumenti di rilevazione devono provvedere verso la velocità e la semplicità. A questo scopo, abbiamo creato un RNA-congiunzione e fluorescenza DNAzyme segnalazione che può essere usato per sviluppare saggi semplici per segnalare la presenza di batteri attraverso la generazione di un segnale di fluorescenza. La funzionalità DNAzyme sonda, RFD-EC1, viene attivato dal CEM prodotto durante la crescita di E. coli in terreni di coltura. Poiché il nostro metodo utilizza miscele grezze extracellulari di un batterio come destinazione di rilevamento e bypassa l'estrazione laboriosa bersaglio e passaggi di amplificazione, può essere utilizzato per impostare molto semplice, "mix-e-leggere" tipo di analisi per la scoperta batterica. L'utilizzo del nostro DNAzyme non è limitato a fluorescenza metodo di rilevamento basato. Ad esempio, il rilevamento colorimetrico utilizzando lo stesso sistema di dosaggio DNAzyme cuno essere realizzato utilizzando un metodo precedentemente riportato che sfrutta amplificazione laminazione cerchio in combinazione con un colorante organico. 32 Si prevede l'uso di DNAzimi per la rivelazione batterica come un attraente via per generare nuovi biosensori batterici con maggiore semplicità operativa.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Il finanziamento per questo lavoro è stato fornito dal Natural Sciences and Engineering Research Council of Canada (NSERC) e il Network Bioactive Paper Sentinel.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo o modello | |
| Agar | Bioshop Canada | AGR003 | |
| Ammonio persolfato (APS) | Bioshop Canada | AMP001 | |
| L'acrilamide / bis-acrilammide (40%, 29:1) | Bioshop Canada | ACR004 | |
| Acido borico | Bioshop Canada | BOR001 | |
| Blu di bromofenolo | Sigma-Aldrich | B8026 | |
| EDTA | EM Science | EXO539-1 | |
| HCl | Sigma-Aldrich | 38281 | |
| HEPES | Bioshop Canada | HEP001 | |
| LB brodo | Sigma-Aldrich | L3022 | |
| MgCl 2 | EMDProdotti chimici | B10149-34 | |
| NaCl | Bioshop Canada | SOD002 | |
| NaOAc | EMD Chemicals | SXO255-1 | |
| NaOH | EMD Chemicals | SXO590-1 | |
| SDS | Bioshop Canada | SDS001 | |
| TEMED | Bioshop Canada | TEM001 | |
| Tris-base | Bioshop Canada | BST666 | |
| Tween 20 | Sigma-Aldrich | P9416 | |
| Urea | Bioshop Canada | URE001 | |
| Xylenecyanol FF | Sigma-Aldrich | X4126 | |
| DNA concentratore | Thermo Scientific | Savant DNA SpeedVac 120 | |
| Millex Unità di filtraggio | Millipore | SLGP033RS | |
| Gel di carico suggerimenti | DiaMed | TEC200EX-K | |
| Software ImageQuant | Dinamica Molecolare | Versione 5.0 | |
| Kimwipes | Kimberly-Clark Professional | 34705 | |
| Mini vortex | VWR | 58816-121 | |
| Parafilm | Pechiney Plastic Packaging | PM996 | |
| Scatole Petri | Fisher Scientific | FISHERBRAND 08-757-12 | |
| Stripettor Plus (Pipette pistola) | Corning | 07764714 | |
| Cuvette di quarzo | Varian Inc | 66-100216-00 | |
| Shaker / Incubatore | New Brunswick Scientific | Classic Series C24 | |
| Typhoon Scanner | GE Healthcare | 9200 Modalità variabile | |
| Centrifuga | Beckman Coulter | Allegra X22-R | |
| Spettrofotometro UV | Thermo Scientific | GenesysUV 10 | |
| Spettrofotometro a fluorescenza | Varian Inc | Cary Eclipse |
Riferimenti
- Zourob, M., Elwary, S., Truner, A. . Principles of Bacterial Detection: Biosensors, Recognition Receptors and Microsystems. , (2008).
- Call, D. R. Challenges and Opportunities for Pathogen Detection Using DNA Microarrays. Crit. Rev. Microbiol. 31, 91-99 (2005).
- Lazcka, O., Campo, D. e. l., J, F., Muñoz, F. X. Pathogen detection: A perspective of traditional methods and biosensors. Biosens. Bioelectron. 22, 1205-1217 (2007).
- Velusamy, V. An overview of foodborne pathogen detection: In the perspective of biosensors. Biotechnol. Adv. 28, 232-254 (2010).
- Ali, M. M. Fluorogenic DNAzyme Probes as Bacterial Indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Liu, J., Cao, Z., Lu, Y. Functional Nucleic Acid Sensors. Chem. Rev. 109, 1948-1998 (2009).
- Li, Y., Lu, Y. . Functional Nucleic Acids for Analytical Applications. , (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Joyce, G. F. Forty Years of In Vitro Evolution. Angew. Chem. Int. Ed. 46, 6420-6436 (2007).
- Breaker, R. R., Joyce, G. F. A DNA enzyme that cleaves RNA. Chem. Biol. 1, 223-229 (1994).
- Cuenoud, B., Szostak, J. W. A DNA metalloenzyme with DNA ligase activity. Nature. 375, 611-614 (1995).
- Chinnapen, D. J., Sen, D. A deoxyribozyme that harnesses light to repair thymine dimers in DNA. Proc. Natl. Acad. Sci. U. S. A. 101, 65-69 (2004).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Silverman, S. K. DNA as a versatile chemical component for catalysis, encoding, and stereocontrol. Angew. Chem. Int. Ed. 49, 7180-7201 (2010).
- Mei, S. H. An efficient RNA-cleaving DNA enzyme that synchronizes catalysis with fluorescence signaling. J. Am. Chem. Soc. 125, 412-420 (2003).
- Liu, Z. Assemblage of signaling DNA enzymes with intriguing metal-ion specificities and pH dependences. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Kandadai, S. A., Li, Y. Characterization of a catalytically efficient acidic RNA-cleaving deoxyribozyme. Nucleic Acids Res. 33, 7164-7175 (2005).
- Rupcich, N. Quenching of fluorophore-labeled DNA oligonucleotides by divalent metal ions: implications for selection, design, and applications of signaling aptamers and signaling deoxyribozymes. J. Am. Chem. Soc. 128, 780-790 (2005).
- Shen, Y., Brennan, J. D., Li, Y. Characterizing the secondary structure and identifying functionally essential nucleotides of pH6DZ1, a fluorescence-signaling and RNA-cleaving deoxyribozyme. Biochemistry. 44, 12066-12076 (2005).
- Chiuman, W., Li, Y. Revitalization of six abandoned catalytic DNA species reveals a common three-way junction framework and diverse catalytic cores. J. Mol. Biol. 357, 748-754 (2006).
- Chiuman, W., Li, Y. Evolution of high-branching deoxyribozymes from a catalytic DNA with a three-way junction. Chem. Biol. 13, 1061-1069 (2006).
- Shen, Y. Catalysis and rational engineering of trans-acting pH6DZ1, an RNA-cleaving and fluorescence-signaling deoxyribozyme with a four-way junction structure. Chem BioChem. 7, 1343-1348 (2006).
- Ali, M. M., Kandadai, S. A., Li, Y. Characterization of pH3DZ1 - An RNA-cleaving deoxyribozyme with optimal activity at pH 3. Can. J. Chem. 85, 261-273 (2007).
- Chiuman, W., Li, Y. Efficient signaling platforms built from a small catalytic DNA and doubly labeled fluorogenic substrates. Nucleic Acids Res. 35, 401-405 (2007).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA MgZ based on a non-classic allosteric design. PLoS ONE. 2, e1224 (2007).
- Shen, Y. Entrapment of fluorescence signaling DNA enzymes in sol gel-derived materials for metal ion sensing. Anal. Chem. 79, 3494-3503 (2007).
- Kandadai, S. A. Characterization of an RNA-cleaving deoxyribozyme with optimal activity at pH 5. Biochemistry. 48, 7383-7391 (2009).
- Zhao, W., Brook, M. A., Li, Y. Periodic assembly of nanospecies on repetitive DNA sequences generated on gold nanoparticles by rolling circle amplification. Methods Mol. Biol. 474, 79-90 (2008).
- Navani, N. K., Mok, W. K., Li, Y. In vitro selection of protein-binding DNA aptamers as ligands for biosensing applications. Methods Mol. Biol. 504, 399-415 (2009).
- Ali, M. M., Li, Y. Colorimetric sensing by using allosteric-DNAzyme-coupled rolling circle amplification and a peptide nucleic acid-organic dye probe. Angew. Chem. Int. Ed. 48, 3512-3515 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon