このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
全細胞集団の単一細胞レベルでサイクリンBの分解を勉強
要約
後期促進複合体(APC / C)依存性ユビキチン化とここサイクリンBのその後の破壊によってトリガされ移行を後期にかけてする中期、我々は、パルスチェイスラベルに続いて、システムを構築し、全体の細胞集団でサイクリンB蛋白質分解を監視することができ、有糸分裂チェックポイントによる干渉の検出を容易にする。
要約
細胞分裂時に2つの娘細胞の間で染色体の均等分配は遺伝的安定性1を保証するための前提条件です。染色体分離時の不正確さは、悪性腫瘍の特徴であり、進行性の疾患で2月4日に関連付けられている。スピンドルアセンブリチェックポイント(SAC)は、一つ一つの染色体が紡錘体1への安定したバイポーラ·アタッチメントを決めるまで戻って中期の細胞を保持分裂サーベイランス·メカニズムです。 SACはこうしてsecurinとサイクリンBの分解と染色体分離と有糸分裂の出口をブロックするために活性化APC / CサブユニットCdc20との干渉によって、その機能を発揮する。染色体の不適切な添付ファイルは、SACシグナルのサイレンシングを防止し、問題が染色体missegregation、異数性および悪性増殖1を回避するために解決されるまで、APC / C Cdc20の継続的な阻害を引き起こす。
私宛のほとんどの研究APC / C依存性タンパク質分解に不適切な染色体付着のnfluenceは、微小管への染色体付着を妨害する薬剤を解重合または微小管安定化を用いたスピンドルの混乱を利用しました。微小管の動態との干渉が重要な調節因子の輸送と局在に影響を与えることができるので、これらの手順は、人工的な効果5を誘発するリスクを負うものとします。
SACは細胞集団を動じに有糸分裂時にAPC /サイクリンBのC依存性タンパク質分解を妨害する方法を勉強するために、我々は、有糸分裂染色体とサイクリンB 6のタンパク質分解の中期アライメントの同時モニタリングを許可されたヒストンH2-GFPベースのシステムを確立。
タンパク質分解プロファイルを表現するために、我々は、C末端SNAPの部分6( 図1)とキメラサイクリンBレポーター分子を生成しました。自己標識反応では、SNAP-部分はと共有結合を形成することが可能であるグアニンキャリア(SNAP基板)7,8( 図1)。 SNAPの基質分子は容易に入手可能であり、異なる蛍光色素の広いスペクトルを運ぶ。キメラサイクリンB-SNAPの分子は、成長培地7( 図1)への膜透過性のSNAP基質の添加により標識されたようになります。標識反応に続いて、サイクリンB-SNAPの蛍光強度は、パルスチェイス反応のような方法と蛍光強度の低下サイクリンB劣化6( 図1)のレベルを反映しています。我々のシステムは並行してセル(またはいくつかの細胞集団)の多数の分裂APC / C依存性タンパク質分解のモニタリングを容易にする。これにより、システムは、エージェント/遷移をする中期後期にかけてでタンパク質分解活性を妨害することができる小分子を同定するための貴重なツールであるかもしれません。また、有糸分裂時のサイクリンBの合成として最近重要mechanisとして示唆されている9,10安定したサイクリンBの発現レベルを維持することによって、マウスとヒトの分裂ブロックの育成にmが、このシステムは、バランスのとれた均衡6の一つの要素として、サイクリンB蛋白質分解を分析することができました。
プロトコル
1。顕微鏡チャンバースライド上U2OSベースサイクリンB-SNAPのレポーター細胞(クローン11細胞6)の播種
- 少なくとも48時間のログフェーズで非同期的に増殖させたサブコンのSNAPレポーター細胞をトリプシン処理。
- 8よく顕微鏡室(細胞の分布定数)での作業。
10%ウシ胎児血清、ペニシリン/ストレプトマイシンを添加したフェノールレッドを含まない通常の増殖培地(350μlの中に顕微鏡室の表面全体にわたって一定の分布で8ウェル顕微鏡室への細胞、遠心分離機10,000細胞を再懸濁しの播種用およびピルビン酸ナトリウム)。顕微鏡室( 図2)に細胞懸濁液を移す。
8よく顕微鏡室(中央の最大細胞密度)での作業。
顕微鏡CHAMBの中心に高い密度で細胞の播種のためにERは、フェノールレッドを含まない通常の増殖培地300μlのチャンバーをロードします。顕微鏡室( 図2)の中央に慎重に5,000細胞を追加します。
96ウェル特殊光学プレート(細胞の分布定数)での作業。
井戸の表面全体にわたって一定の分布で96ウェルプレートに細胞の播種は、5000細胞を遠心分離し、フェノールレッドを含まない通常の増殖培地を300μlに再懸濁する。 96ウェルプレートに細胞懸濁液を移す。必要な細胞を含むウェルの総数に応じて、細胞数および懸濁培地( 図2)の総量を調整します。
96ウェル特殊光学プレート(中央の最大細胞密度)での作業。
96ウェルプレートに細胞の播種については、慎重にSmalの中のフェノールレッドを含まない通常の増殖培地を15μlに1500セルを追加ウェルの中央( 図2)細胞増殖の制限を達成するために、各ウェルの中央にLのドロップ。
- 播種された細胞は、標準的な細胞培養条件(37℃、100%の空気の湿度、5%のCO 2)下で少なくとも18時間増殖させる。
2。 SNAPの基板とレポーター細胞の染色
- 染色手順の開始前に30分は、フェノールレッドを含まない通常の増殖培地のアリコートは、37℃に温まるまで
- SNAPの基板の取り扱いを容易にするため(私たちの場合、TMRスターで)-20℃で保存することができ、400μmの原液の濃度を得るためにDMSOにTMRのスターを溶かす
- 事前の染色に、1μMの最終ラベリング濃度を得るために、フェノールレッドを含まない通常の増殖培地200μl(37℃)で、TMRスターストック溶液0.5μlを希釈します。
- 非同期的に増殖している細胞から、正常な増殖培地を除去し、リットルでインキュベート標準的な培養条件下で25分間媒体をabeling。
- フェノールレッドを含まない通常の増殖培地(37℃)で培地および細胞洗浄を4回ラベリング削除します。 30分間、フェノールレッドを含まない通常の増殖培地(37℃)300μlの細胞をインキュベートします。事前の顕微鏡への輸送に残留アンバウンドSNAPの基板を除去するために新鮮なフェノールレッドを含まない通常の増殖培地(37℃)で培地を交換してください。
- あらかじめ温めておいた上で発泡スチロールの箱の顕微鏡への輸送細胞(37℃)の温度変化を最小限に抑えるために、ヒートブロック。
3。蛍光強度の測定
- 分析前に2時間は、所望の温度に、そのすべてのコンポーネントと全体の顕微鏡を持って来るために℃のドライモードで37に人工気候室の空気温度を調整します。予熱は、湿度を設定する前に、顕微鏡に結露やその後の損傷を避けるために重要です。
- 空気湿度トンを調整O 60%、分析の開始に先立って、CO 2から5パーセント。
- スキャン^ R·アクイジション·ソフトウェアを起動し、標準設定を定義します( 表1を参照)。
- 分析する井戸の位置を定義します。
- ΔT(取得サイクル時間)と取得サイクルの絶対数を定義します。
- 井戸の数が多いの解析が必要な場合は、ハードウェアのオートフォーカスを選択し、それ以外の場合は、ソフトウェアのみのオートフォーカスを使用すれば十分です。
- 収集を開始して、最初の2つの取得サイクルのために監督する。フィルタが変更され、対応するTMRのスターイメージが( 図3)が取得される前に、顕微鏡は、そのチャネルの最初の画像のその後の買収により、ヒストンH2-GFPシグナルに焦点を当てます。これは、再度次のサイクルを繰り返す前に、井戸内および検査対象の各ウェルのためのすべてのポジションのために繰り返される。
4。タンパク質分解プロファイルUの分析スキャン^ Rを歌う
- スキャン^ R解析ソフトウェアを起動し、細胞核を使用して画像を解析するなどの信号強度及び隣接セルを分離を支援するために、流域のアルゴリズムに基づいてしきい値を使用して、メインオブジェクトとして定義されたヒストンH2-GFPによって可視化した。細胞質と核から成るサブオブジェクトはTMRstar分析のために作成する必要があります。 (メイン·オブジェクトの重要な性質は、XおよびYの位置、時間、および最大であり、主となるオブジェクトとTMRstarサブオブジェクトの面積で割った強度のGFPの強さを意味します)この分析プロセスは、大規模なデータのために数時間かかることがあります数量。
- 分析の主要なオブジェクト( 図4)に割り当てられ、(セルのトレース)は時間の経過とともに、TMRスター平均蛍光強度subobjectを可視化するモードをトレースするように変更します。検査すべき細胞の数が少なくとも140サイクルを持続させる、それらの測定上のゲーティングによっておよびH2-GFPの高い最大強度を絞り込むことができる。見える細胞の大きい数字で最初の代表と客観的なビューが表示されます( 図4)が可能になります。
- 単一細胞レベル( 図5A)で同時にヒストンH2-GFPおよびTMRスター蛍光を可視化するために、目的の細胞のトレースを選択します。
- 目的の細胞に、マウスの右クリックを使用しており、ヒストンH2-GFPおよびサイクリンB-SNAP毎回ポイントのTMRのスターの図については、輸出可能な画像ギャラリーを生成します。
- スキャン^ R解析ソフトウェアおよびゲート、目的の細胞をX対Yのドットプロット( 図5B)で表される地域の人口·モードに変更します。
- ゲート領域に新しいドットプロットウィンドウを適用し、時間をかけて、TMRスター蛍光強度( 図5C)を意味して可視化する。
- さらに計算のためのMicrosoft Excelへのデータのエクスポート(時間と蛍光強度)。
5。代表的な結果
イチジクURE 5D及び5Eは、染色体のずれ( 図5E)の兆候がない通常の有糸分裂の進行しているセルのTMRのスター蛍光強度曲線によって表されるサイクリンB動態を、描いています。核膜破壊(NEBD、赤い三角形によって示される)に続く細胞質の圧縮時に、TMRのスター蛍光強度は、同型のウィンドウ(図中の明るい部分)までは急激な上昇を示しているセルが前中期6を入力したときに到達した。蛍光強度が前期と中期を介して細胞が進行する限り安定したレベルに留まり、その後の染色体のすべてが安定中期板( 図5D及び5E)を設立してしまえば急速に低下し始める。この低下は後期(曲線上の青い点)の間に染色体分離の前にあります。有糸分裂後期でクロマチンはdecondenseを開始(青色の棒グラフ)と蛍光強度曲線に近づく一方セルは相間の形態を採用しています有糸分裂の前に高原( 図5D及び5E)より低い台地。
| オートフォーカスの設定 | ヒストンH2-GFP(メインオブジェクト取得の設定) | TMRのスターで標識サイクリンB-SNAP (取得の設定) | 取得サイクルタイム繰返し |
| 粗いオートフォーカス+ / -39μmの24層 ファインオートフォーカス + / -5.4μmの14層 | GFPフィルターセット: 露光時間:100ミリ秒 光強度:25% | TRITCフィルターセット: 露光時間:150ミリ秒 光強度:33.3% | 2〜5分。 |
| GFPフィルターセット: 露光時間:12ミリ秒 光強度:12.5% | 最大継続分析の48時間(60%の減少、空気湿度によって制限される) |
表1。サイクリンB蛋白質分解の分析のために使用される標準的な設定にします。ヒストンは、H2-GFPは、サイクリンB-SNAPの蛍光強度を測定するための焦点面を定義するための基準構造として使用した。累積毒性を避けるために、画像取得の設定に比べて焦点手順の間に光強度が低くなっています。
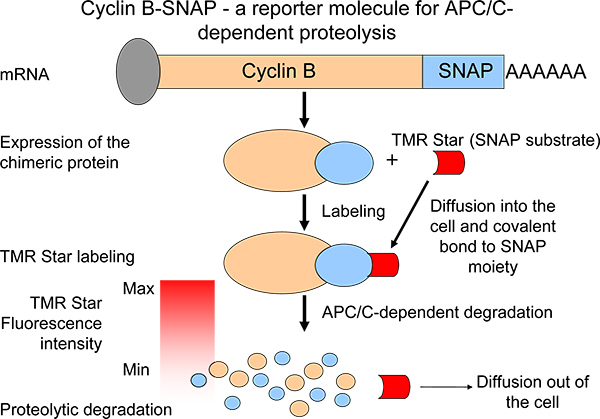
図1。内在性タンパク質に似て分解特性を有するキメラサイクリンBの誘導体の発現を介しサイクリンB蛋白質分解の測定の模式図 。パルスチェイス標識後、蛍光強度の減少は、APC / C活性の尺度である。
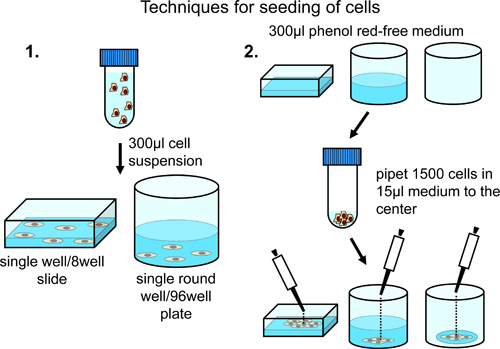
図2。 8ウェルスライドまたは96ウェルプレート上の細胞の解析。の正規分布ウェルの表面を横切って細胞は300μlの最終容量に再懸濁することにより達成されると、単一または少数の位置を手動で分析のために定義されている場合に、推奨されます。中心領域における細胞の富化はプレフィルドまたは空のウェルの中心に細胞を添加することによって達成される。この手法は、異なるウェルで自動化された画像取得の場合に推奨されます。

図3。データ収集のシーケンス。H2-GFPヒストンの)検出が有糸分裂時に染色体の整列の焦点面の定義やモニタリングに使用されます。 B)は、TMRスター蛍光強度の蛍光強度は、サイクリンB-SNAPのラベルされたパルスチェイスの絶対量の尺度である。
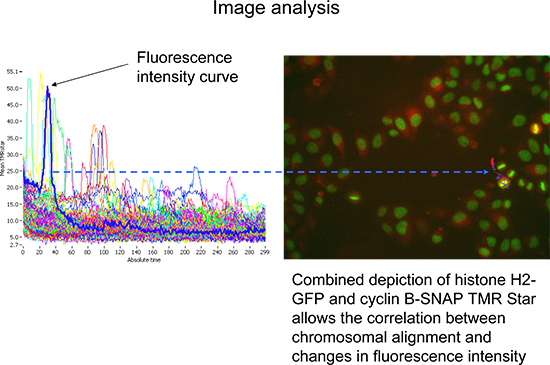
図4。平均TMRスターの代表的なスキャン^ R解析ソフトウェアベースの描写時間の経過とともに蛍光強度(面積で割った総TMRstar強度)。代表的な細胞のトレース(青)の選択。対応する画像(右図)は、ヒストンH2-GFP(緑色)とサイクリンB-SNAP(赤)の同時可視化を可能に。 拡大図を表示するには、ここをクリックしてください 。
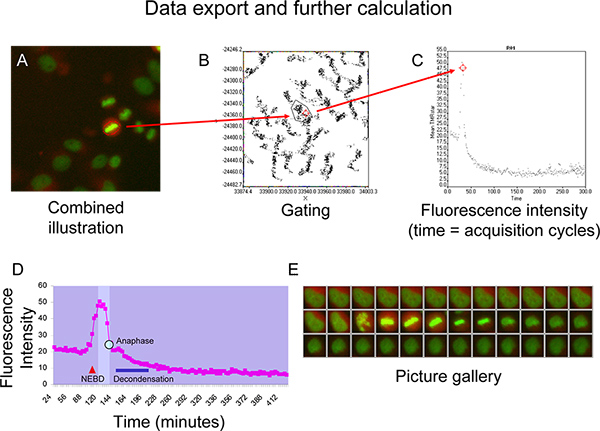
図5 A + B)は、XYドットプロット上の関心のあるセルを表すドットのゲーティングの例。ドットプロットを使用して時間をかけて平均TMRstarの蛍光強度(面積で割った総TMRstar強度)のC)の可視化。 D + E)は、Microsoft Excelで生成されるようなNEBD(核膜破壊)とクロマチン脱凝縮(青いバー)の間に同型ウィンドウ(図上のbrigherフィールド)とサイクリンB-SNAPの代表的な蛍光強度曲線。後期はtによって示され曲線上の彼の青いドット。 拡大図を表示するには、ここをクリックしてください 。
映画は1。 補足ムービーを見るにはここをクリック 。
ディスカッション
我々はここでサイクリンB蛋白質分解と染色体のアライメントの同時モニタリングを促進するライブセルイメージングベースのアプローチを提示します。このアプローチは、単一細胞レベルでの細胞集団を非摂動の分裂制御の研究を可能にします。サイクリンBの劣化曲線は、APC / Cの活性に直接的な洞察を促進するため、間接的にSAC 6のステータスを反映します。
開示事項
特別な利害関係は宣言されません。
謝辞
我々はpLPCX-ヒストンH2-GFPプラスミドを提供するため、S.テイラーに感謝しています。我々は継続的な支援のためにR. Mertelsmannに感謝します。この作品は、ドイツ学術振興によってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| 品名 | 会社 | カタログ番号 | コメント(オプション) |
| レポーター細胞株 | 6に記載のように社内で発生した | 11レポーター細胞のクローンを作成する (U2OSベースサイクリンB-SNAPの発現細胞) | |
| レトロサイクリンB-SNAPの発現ベクター | 6に記載のように社内で発生した | pLNCX2-サイクリンB mut5-SNAP | |
| Phenolredを含まないDMEM | ギブコ | 21063-029 | FCSは、ピルビン酸ナトリウム、ペニシリン/ストレプトマイシンマイシンの補充が必要 |
| SNAP-セルのTMR-スター | ニューイングランドバイオラボ | S9105S | DMSO中の原液400μMの |
| 特別Opsticsプレート、96ウェル | コスター | 3720 | |
| μ-スライド8よく、ibiTreat | Ibidi | 80826 | |
| 顕微鏡ユニット | オリンポス | 人工気象室と、IX-81逆顕微鏡 | |
| 客観 | オリンポス | UPLSAPO 20倍対物レンズ(NA 0.75) | |
| 取得ソフトウェア | オリンポス | スキャン^ R取得ソフトウェア(v.2.2.09) | |
| 解析ソフトウェア | オリンポス | スキャン^ R解析ソフトウェア(v.1.2.0.6) |
参考文献
- Musacchio, A., Salmon, E. D. The spindle-assembly checkpoint in space and time. Nat. Rev. Mol. Cell Biol. 8, 379-393 (2007).
- Nasmyth, K. Segregating sister genomes: the molecular biology of chromosome separation. Science. 297, 559-565 (2002).
- Cahill, D. P. Mutations of mitotic checkpoint genes in human cancers. Nature. 392, 300-303 (1998).
- Lengauer, C., Kinzler, K. W., Vogelstein, B. Genetic instabilities in human cancers. Nature. 396, 643-649 (1998).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463, 485-4892 (2010).
- Schnerch, D. Monitoring APC/C activity in the presence of chromosomal misalignment in unperturbed cell populations. Cell Cycle. 11, (2012).
- Keppler, A. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat. Biotechnol. 21, 86-869 (2003).
- Jansen, L. E., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J. Cell. Biol. 176, 795-805 (2007).
- Malureanu, L. Cdc20 hypomorphic mice fail to counteract de novo synthesis of cyclin B1 in mitosis. J. Cell Biol. 191, 313-329 (2010).
- Mena, A. L., Lam, E. W., Chatterjee, S. Sustained spindle-assembly checkpoint response requires de novo transcription and translation of cyclin B1. PLoS One. 5, (2010).
- Wolthuis, R. Cdc20 and Cks direct the spindle checkpoint-independent destruction of cyclin. A. Mol. Cell. 30, 290-302 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved