Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
L'étude de la protéolyse de la cycline B au niveau de la cellule unique dans les populations de cellules entières
Dans cet article
Résumé
Métaphase anaphase de transition est déclenchée par l'anaphase de promotion de complexe (APC / C) en fonction de l'ubiquitination et la destruction subséquente de la cycline B. Ici, nous avons mis en place un système qui, à la suite pulse-chase étiquetage, permet de suivre la cycline B protéolyse dans des populations de cellules entières et facilite la détection des interférences par le checkpoint mitotique.
Résumé
Répartition égale des chromosomes entre les cellules filles deux au cours de la division cellulaire est une condition sine qua non pour garantir la stabilité génétique 1. Inexactitudes lors de la séparation des chromosomes sont une caractéristique de malignité et associée à une maladie progressive 2-4. Le point de contrôle de groupe de broches (CSC) est un mécanisme de surveillance mitotique qui retient les cellules de métaphase jusqu'à ce que chaque chromosome unique a établi une fixation bipolaire stable à la broche 1 mitotique. Le CSC exerce sa fonction par interférence avec l'activation APC / C Cdc20 sous-unité pour bloquer la protéolyse de Securin et la cycline B et donc la séparation des chromosomes mitotiques et de sortie. Une mauvaise fixation des chromosomes empêche de faire taire SAC signalisation et provoque une inhibition continue de l'APC / C Cdc20 jusqu'à ce que le problème soit résolu pour éviter missegregation chromosomes, aneuploïdie et tumeurs malignes 1.
La plupart des études portant sur les influence de l'attachement chromosomique indue sur APC / C en fonction de la protéolyse a profité de perturbation broche à l'aide dépolymérisation des microtubules ou de stabilisation des médicaments pour interférer avec l'attachement aux microtubules chromosomique. Depuis interférence avec la cinétique des microtubules peut affecter le transport et la localisation des régulateurs critiques, ces procédures comportent un risque d'induire des effets artificiels 5.
Pour étudier comment le SAC interfère avec APC / C-protéolyse dépendante de la cycline B lors de la mitose dans imperturbable populations de cellules, nous avons établi un système d'histone H2-GFP-base qui a permis le contrôle simultané de l'alignement des chromosomes mitotiques en métaphase et la protéolyse de la cycline B 6 .
Pour décrire les profils protéolytiques, nous avons généré une molécule chimérique cycline B journaliste avec un fragment C-terminal SNAP 6 (figure 1). Dans une réaction d'auto-étiquetage, le SNAP-fragment est capable de former des liaisons covalentes avecalkylguanine-supports (substrat SNAP) 7,8 (figure 1). Molécules de substrat SNAP sont facilement disponibles et effectuer un large éventail de différents fluorochromes. Chimères cycline B-SNAP molécules marquées devenir lors de l'addition du substrat de SNAP-membrane perméable au milieu de croissance 7 (figure 1). À la suite de la réaction de marquage, l'intensité cycline B-SNAP fluorescence diminue dans une réaction de type pulse-chase manière intensités de fluorescence et de tenir compte des niveaux de dégradation de la cycline B 6 (figure 1). Notre système facilite le suivi des mitotique APC / C en fonction de la protéolyse dans un grand nombre de cellules (ou plusieurs populations cellulaires) en parallèle. Ainsi, le système peut être un outil précieux pour identifier des agents / petites molécules qui sont capables d'interférer avec l'activité protéolytique à la métaphase anaphase de transition. En outre, comme la synthèse de cycline B lors de la mitose a été récemment suggéré comme un mechanis importantsm dans la promotion d'un bloc mitotique chez la souris et chez l'homme en maintenant les niveaux d'expression de cycline B 9,10 stables, ce système nous a permis d'analyser la protéolyse B cycline comme un élément d'un équilibre harmonieux 6.
Protocole
1. Ensemencement des cellules à base de cycline U2OS Reporter B-SNAP (clone 11 Cellules 6) sur diapositives chambre du microscope
- Trypsiniser subconfluentes cellules reporters SNAP qui ont été autorisés à se développer de manière asynchrone en phase logarithmique pendant au moins 48 heures.
- Travailler avec 8 chambres microscope puits (distribution constante des cellules).
Pour l'ensemencement de cellules sur 8 chambres microscope ainsi à une distribution constante sur toute la surface de la chambre de microscope, centrifugeuse 10.000 cellules et remettre en suspension dans 350 ul de rouge de phénol libre milieu de croissance normale (supplémenté avec 10% de sérum bovin foetal, de pénicilline / streptomycine et le pyruvate de sodium). Transférer la suspension cellulaire à la chambre de microscope (Figure 2).
Travailler avec 8 chambres microscope ainsi (densité cellulaire maximale au centre).
Pour l'ensemencement de cellules à une densité plus élevée au centre de la chamb microscopeer, charger la chambre avec 300 ul de phénol rouge sans milieu de croissance normal. Ajouter 5.000 cellules soigneusement au centre de la chambre de microscope (Figure 2).
Travailler avec des plaques de 96 puits optiques spéciales (distribution constante des cellules).
Pour l'ensemencement de cellules sur des plaques de 96 puits à une répartition constante sur toute la surface du puits, centrifuger 5.000 cellules et remettre en suspension dans 300 ul de rouge de phénol libre milieu de croissance normale. Transférer la suspension cellulaire sur une plaque de 96 puits. En fonction du nombre total de cellules contenant du puits nécessaire, ajuster le nombre de cellules et le volume total du milieu de suspension (figure 2).
Travailler avec des plaques de 96 puits optiques spéciales (densité cellulaire maximale au centre).
Pour les semis de cellules sur des plaques 96 puits, ajouter prudemment 1500 cellules dans 15 ul de phénol milieu dépourvu de rouge croissance normale dans un smalL goutte au centre de chaque puits pour atteindre restriction de la croissance des cellules par rapport au centre du puits (figure 2).
- Laisser les cellules ensemencées à croître pendant au moins 18 heures dans des conditions standard de culture cellulaire (37 ° C, humidité de l'air 100%, 5% de CO 2).
2. La coloration des cellules avec Substrat Reporter SNAP
- 30 min avant le début de la procédure de coloration permettent aliquotes de rouge de phénol libre milieu de croissance normal de se réchauffer à 37 ° C.
- Pour une manipulation aisée du substrat SNAP (dans notre cas TMR Star) dissoudre étoiles TMR dans le DMSO pour obtenir une concentration dans la solution mère de 400 uM, ce qui peut être conservé à -20 ° C.
- Avant la coloration, diluer 0,5 pi de solution TMR actions étoile dans 200 ul de phénol milieu dépourvu de rouge croissance normale (37 ° C) pour obtenir une concentration d'étiquetage finale de 1 uM.
- Retirer milieu de croissance normal des cellules de culture et incuber de manière asynchrone dans lAbeling moyen pendant 25 min dans des conditions de culture standard.
- Retirez le marquage de cellules moyennes et lavage quatre fois avec du phénol milieu dépourvu de rouge croissance normale (37 ° C). Incuber les cellules dans 300 ul de phénol milieu dépourvu de rouge croissance normale (37 ° C) pendant 30 min. Avant le transport du microscope remplacer le milieu frais avec rouge de phénol libre milieu de croissance normale (37 ° C) pour éliminer les résidus non lié substrat SNAP.
- Transport cellules au microscope dans une boîte en polystyrène sur un pré-chauffée (37 ° C) bloc de chauffage pour minimiser les variations de température.
3. Mesure de l'intensité de fluorescence
- Deux heures avant l'analyse de régler la température de l'air de la chambre climatique à 37 ° C en mode sec afin d'amener le microscope complet avec tous ses composants à la température voulue. Préchauffage avant de l'humidité est important de ne pas endommager la condensation et à la suite de la loupe.
- Réglez t humidité de l'airo 60% et CO 2 à 5%, avant le début de l'analyse.
- Démarrez le logiciel de numérisation ^ R acquisition et de définir des réglages standard (voir le tableau 1).
- Définir les positions des puits à analyser.
- Définir l'At (temps de cycle d'acquisition) et le nombre absolu de cycles d'acquisition.
- Si l'analyse d'un plus grand nombre de puits est souhaitée, sélectionnez l'autofocus matériel, sinon il suffit d'utiliser l'autofocus logiciel seul.
- Lancement de l'acquisition et de superviser les deux premiers cycles d'acquisition. Le microscope se concentrera sur la histone H2-GFP signal, à la suite de l'acquisition d'une première image en ce que le canal en amont du filtre est modifiée et l'image étoile correspondant est acquis TMR (figure 3). Cet article est alors répété pour toutes les positions à l'intérieur d'un puits et pour chacun des puits à examiner, avant de répéter à nouveau au cycle suivant.
4. L'analyse des profils u protéolytiquechanter scan ^ R
- Démarrer le logiciel d'analyse de numérisation ^ R et analyser les images avec les noyaux des cellules, comme visualisé par l'histone H2-GFP, défini comme l'objet principal, à l'aide d'un seuil basé sur l'intensité du signal et d'un algorithme de bassins versants afin d'aider à séparer les cellules voisines. Un sous-objet constitué d'un noyau avec un cytoplasme doit être créé pour l'analyse TMRstar. (Les propriétés importantes de l'objet principal est positions X et Y, l'heure et maximale et moyenne intensités de GFP pour l'objet principal et l'intensité totale divisée par la surface de la sous-objet TMRstar.) Ce processus d'analyse peut prendre plusieurs heures en raison de données volumineuses quantités.
- Changer de retracer le mode de visualiser la moyenne sous-objet TMR étoiles intensité de la fluorescence au cours du temps (traces de cellules), affectée aux objets analysés principales (figure 4). Le nombre de cellules à examiner peut être réduite par le déclenchement de ces mesures d'une durée d'au moins 140 cycles et à une forte intensité maximale de H2-GFP. Je suis làau plus grand nombre de cellules permet une vue représentant première et d'objectif (figure 4).
- Sélectionnez une trace de cellule d'intérêt pour visualiser histone H2-GFP et TMR étoiles fluorescence simultanément au niveau de la cellule unique (figure 5A).
- En utilisant le clic droit de la souris sur une cellule d'intérêt et de générer une galerie de photos exportable à titre d'illustration de l'histone H2-GFP et de la cycline B-SNAP étoiles TMR pour chaque point de temps unique.
- Changer le mode de la population du logiciel de numérisation ^ Analyse R et la porte de la région où la cellule d'intérêt est représenté sur la parcelle X vs Y point (figure 5B).
- Appliquer une fenêtre nouveau point complot pour la région fermée et visualiser dire TMR étoiles fluorescence d'intensité au fil du temps (figure 5C).
- Exporter des données (durée et l'intensité de fluorescence) vers Microsoft Excel pour le calcul ultérieur.
5. Les résultats représentatifs
Figueure 5D et 5E représentent cycline B cinétique, représenté par une courbe d'intensité de fluorescence TMR étoile, d'une cellule qui produit au moyen d'une mitose ordinaire sans signe de désalignement chromosomique (figure 5E). Après compactage du cytoplasme suite de la rupture de l'enveloppe nucléaire (NEBD, comme indiqué par le triangle rouge), TMR étoiles fluorescence d'intensité montre une augmentation brutale jusqu'à ce que la fenêtre isomorphe (zone plus claire sur le schéma) est atteint lorsque la cellule entre prométaphase 6. L'intensité de fluorescence reste à un niveau stable tant que le produit des cellules à travers la prophase et la métaphase, puis commence à chuter rapidement une fois que tous les chromosomes ont établi une plaque métaphasique stable (figure 5D et 5E). Cette baisse précède la séparation des chromosomes lors de l'anaphase (point bleu sur la courbe). Dans la mitose fin de la chromatine commence à decondense (barres bleues) et la cellule adopte la morphologie interphase tandis que la courbe d'intensité de fluorescence se rapproche d'uneplateau qui est plus faible que le plateau avant la mitose (figure 5D et 5E).
| Réglages autofocus | Histone H2-GFP (principaux paramètres d'acquisition d'objets) | Cycline B-SNAP étiquetés avec TMR étoiles (Paramètres d'acquisition) | Répétitions d'acquisition temps de cycle |
| Grossiers autofocus + / -39 um 24 couches Fine mise au point automatique + / -5,4 Um 14 couches | GFP jeu de filtres: Temps de pose: 100 ms Intensité lumineuse: 25% | TRITC jeu de filtres: Temps de pose: 150 ms Intensité lumineuse: 33,3% | 2 à 5 min. |
| GFP jeu de filtres: Temps de pose: 12 ms Intensité lumineuse: 12,5% | Jusqu'à 48 h d'analyse continue (limitée par l'humidité de l'air réduit de 60%) |
Tableau 1. Les paramètres standard tels qu'ils sont utilisés pour l'analyse de la cycline B protéolyse. Histones H2-GFP a été utilisé comme une structure de référence pour définir le plan focal pour la mesure de l'intensité de fluorescence cycline B-SNAP. Les intensités lumineuses au cours de la procédure de mise au point sont bas par rapport aux paramètres d'acquisition d'images pour éviter de phototoxicité cumulatif.
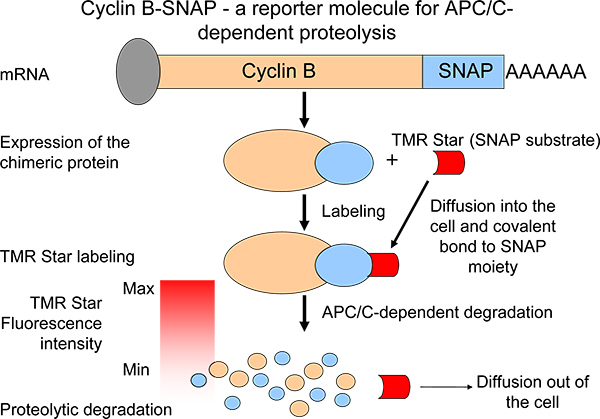
Figure 1. Schématique de la mesure de la cycline B protéolyse par l'expression de la cycline B d'un dérivé chimérique ayant des caractéristiques semblables à la dégradation de la protéine endogène. La baisse de l'intensité de fluorescence après pulse-chase étiquetage est une mesure de l'APC / C-activité.
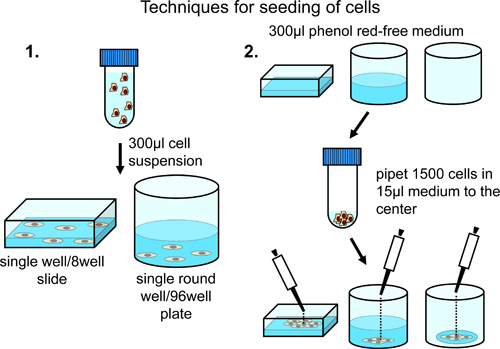
Figure 2. L'analyse des cellules sur 8 toboggans ainsi ou plaques de 96 puits. La distribution régulière de l'cellules sur la surface du puits est réalisé par remise en suspension dans un volume final de 300 ul et il est recommandé, si seulement un seul ou quelques positions sont définies manuellement pour analyse. L'enrichissement des cellules dans la zone centrale est réalisée par addition de cellules au centre de puits préremplies ou vide. Cette technique est recommandée en cas d'acquisition automatique d'images dans différents puits.

Figure 3. Séquence d'acquisition de données. A) Détection de l'histone H2-GFP est utilisée pour la définition de son plan focal et le suivi de l'alignement chromosomique lors de la mitose. B) L'intensité de fluorescence de l'intensité de fluorescence TMR étoile est une mesure de la quantité absolue de pulse-chase étiquetés cycline B-SNAP.
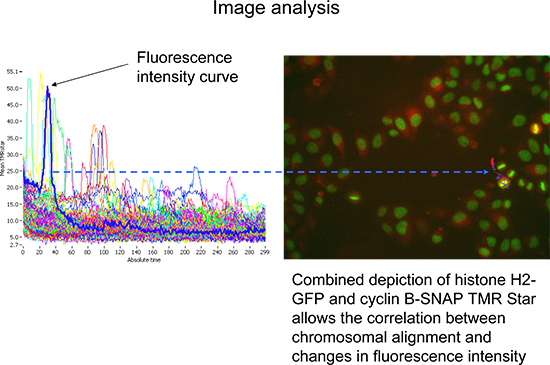
Figure 4. Représentant de numérisation représentation ^ R Analyse basée sur les logiciels de la moyenne TMR étoilesintensités de fluorescence (intensité TMRstar totale divisée par la superficie) au fil du temps. Sélection d'une trace de cellules représentant (bleu). Les images correspondantes (illustré à droite) permet la visualisation simultanée de l'histone H2-GFP (vert) et de la cycline B-SNAP (rouge). Cliquez ici pour agrandir la figure .
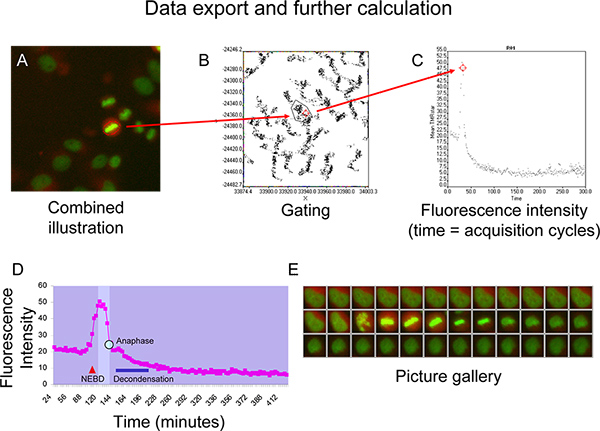
Figure 5. A + B) Exemple de déclenchement de points représentant une cellule d'intérêt sur le graphique XY-point. C) La visualisation de l'intensité moyenne de fluorescence TMRstar (intensité TMRstar totale divisée par la superficie) dans le temps en utilisant le tracé de points. D + E) représentant la courbe d'intensité de fluorescence de la cycline B-SNAP avec fenêtre isomorphe (champ brigher sur le schéma) entre NEBD (répartition de l'enveloppe nucléaire) et de la chromatine décondensation (barre bleue) tel que généré dans Microsoft Excel. Anaphase est indiqué par til point bleu sur la courbe. Cliquez ici pour agrandir la figure .
Discussion
Nous présentons ici une approche imagerie des cellules vivantes basée faciliter la surveillance simultanée de la cycline B protéolyse et l'alignement des chromosomes. Cette approche permet l'étude du contrôle mitotique dans imperturbable populations de cellules au niveau de la cellule unique. Courbes de dégradation des cyclines B faciliter aperçu direct sur l'activité de l'APC / C et donc indirectement refléter l'état de la SAC 6.
Cette approch...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Nous sommes reconnaissants à S. Taylor pour assurer la pLPCX-Histone H2-GFP plasmide. Nous remercions R. Mertelsmann pour leur soutien continu. Ce travail a été soutenu par la Deutsche Forschungsgemeinschaft.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du produit | Entreprise | Numéro de catalogue | Commentaires (optionnel) |
| Lignée cellulaire Reporter | généré en interne tel que décrit 6 | cloner 11 cellules reporters (U2OS à base de cycline B-SNAP cellules exprimant) | |
| Rétroviral vecteur d'expression de cycline B-SNAP | généré en interne tel que décrit 6 | pLNCX2-cycline B mut5-SNAP | |
| PHENOLRED DMEM sans | Gibco | 21063-029 | La supplémentation avec du FCS, le pyruvate de sodium, pénicilline / streptomycine nécessaire |
| SNAP-Cell TMR-Star | New England Biolabs | S9105S | Solution mère 400 uM dans du DMSO |
| Plaque spéciale Opstics, 96 puits | Costar | 3720 | |
| μ-Slide 8 puits, ibiTreat | Ibidi | 80826 | |
| Unité de microscopie | Olympe | IX-81 microscope inverse avec la chambre climatique | |
| Objectif | Olympe | UPLSAPO objectif 20x (NA 0,75) | |
| Logiciel d'acquisition | Olympe | Scan ^ R acquisition de logiciels (v.2.2.09) | |
| Logiciel d'analyse | Olympe | Scan ^ Analyse logiciel R (v.1.2.0.6) |
Références
- Musacchio, A., Salmon, E. D. The spindle-assembly checkpoint in space and time. Nat. Rev. Mol. Cell Biol. 8, 379-393 (2007).
- Nasmyth, K. Segregating sister genomes: the molecular biology of chromosome separation. Science. 297, 559-565 (2002).
- Cahill, D. P. Mutations of mitotic checkpoint genes in human cancers. Nature. 392, 300-303 (1998).
- Lengauer, C., Kinzler, K. W., Vogelstein, B. Genetic instabilities in human cancers. Nature. 396, 643-649 (1998).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463, 485-4892 (2010).
- Schnerch, D. Monitoring APC/C activity in the presence of chromosomal misalignment in unperturbed cell populations. Cell Cycle. 11, (2012).
- Keppler, A. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat. Biotechnol. 21, 86-869 (2003).
- Jansen, L. E., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J. Cell. Biol. 176, 795-805 (2007).
- Malureanu, L. Cdc20 hypomorphic mice fail to counteract de novo synthesis of cyclin B1 in mitosis. J. Cell Biol. 191, 313-329 (2010).
- Mena, A. L., Lam, E. W., Chatterjee, S. Sustained spindle-assembly checkpoint response requires de novo transcription and translation of cyclin B1. PLoS One. 5, (2010).
- Wolthuis, R. Cdc20 and Cks direct the spindle checkpoint-independent destruction of cyclin. A. Mol. Cell. 30, 290-302 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon