Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Studieren Proteolyse von Cyclin B an der Single Cell Level in Whole Cell Populationen
In diesem Artikel
Zusammenfassung
Metaphase zur Anaphase Übergang durch Anaphase-promoting complex (APC / C)-abhängige Ubiquitinierung und anschließende Zerstörung von Cyclin B. Hier haben wir ein System, das Pulse-Chase-Kennzeichnung nach, ermöglicht die Überwachung Cyclin B Proteolyse im gesamten Zellpopulationen und wird ausgelöst, erleichtert den Nachweis von Störungen durch den mitotischen Checkpoint.
Zusammenfassung
Gleichmäßige Verteilung der Chromosomen auf die beiden Tochterzellen bei der Zellteilung ist eine Voraussetzung für die Gewährleistung der genetischen Stabilität 1. Ungenauigkeiten bei der Trennung der Chromosomen sind ein Markenzeichen der Bösartigkeit und mit fortschreitender Erkrankung 2-4. Die Spindelanordnung Prüfpunkt (SAC) eine mitotische Überwachungsmechanismus zurückhält, die Zellen in der Metaphase bis jeder einzelnen Chromosom eine stabile Befestigung an der bipolaren mitotischen Spindel 1 hergestellt wurde. Der SAC übt seine Funktion durch Interferenz mit der aktivierenden APC / C-Untereinheit Cdc20 Proteolyse des Securin und Cyclin B und damit Trennung der Chromosomen und mitotische Ausfahrt blockieren. Bei unsachgemäßer Befestigung von Chromosomen verhindert Silencing SAC-Signalisierung und führt fort Hemmung der APC / C Cdc20, bis das Problem behoben ist auf Chromosom missegregation, Aneuploidie und bösartigen Neubildungen 1 zu vermeiden.
Die meisten Studien, die die i adressiertnfluence unsachgemäßer chromosomale Befestigung an APC / C-abhängige Proteolyse nutzte der Spindel Störungen mit Depolymerisation oder Mikrotubuli-stabilisierende Medikamente mit chromosomalen Bindung an Mikrotubuli stören. Da Störungen Mikrotubuli Kinetik des Transports und Lokalisierung der kritischen Regulatoren beeinflussen können, tragen diese Verfahren ein Risiko der Induktion künstliche Effekte 5.
Um zu untersuchen, wie die SAC mit APC / C-abhängige Proteolyse von Cyclin B stört während der Mitose in ungestörten Zellpopulationen, haben wir ein Histon H2-GFP-basiertes System, das erlaubt die gleichzeitige Überwachung von Metaphase Ausrichtung der mitotischen Chromosomen und Proteolyse von Cyclin B 6 .
Einen proteolytischen Profile darstellen, erzeugten wir eine chimäre Cyclin B Reportermolekül mit einem C-terminalen SNAP-Einheit 6 (Abbildung 1). In einem Self-Labelling Reaktion ist der SNAP-Einheit in der Lage, kovalente Bindungen mit bildenAlkylguanin-Trägern (SNAP Substrat) 7,8 (Abbildung 1). SNAP Substratmolekülen sind leicht verfügbar und führen ein breites Spektrum verschiedener Fluorochrome. Chimäres Cyclin B-SNAP-Moleküle geworden markierten bei Zugabe des Membran-permeablen Substrat SNAP zum Wachstumsmedium 7 (Abbildung 1). Nach der Markierungsreaktion sinkt die Cyclin B-SNAP Fluoreszenzintensität in einem Puls-Chase-Reaktion-artig und Fluoreszenzintensitäten reflektieren Ebenen von Cyclin B Abbau 6 (Abbildung 1). Unserem System erleichtert die Überwachung der mitotischen APC / C-abhängige Proteolyse in einer großen Anzahl von Zellen (oder mehrere Zellpopulationen) parallel. Dabei kann das System ein wertvolles Werkzeug, um Agenten / kleine Moleküle, die in der Lage, mit proteolytischer Aktivität in der Metaphase stören den Übergang Anaphase zu identifizieren sein. Darüber hinaus hat die Synthese von Cyclin B während der Mitose kurzem als wichtiges mechanis vorgeschlagen wordenm bei der Förderung eines mitotischen Block in Mäusen und Menschen, indem sie Cyclin B Expression stabiler 9,10, aktiviert dieses System uns Cyclin B Proteolyse als ein Element eines ausgewogenen Gleichgewichts 6 analysieren.
Protokoll
Ein. Seeding der U2OS-basierte Cyclin B-SNAP Reporter Cells (Clone 11 Cells 6) auf Mikroskop Chamber Slides
- Trypsinieren subkonfluenten SNAP Reporter Zellen, die erlaubt, asynchron wachsen in log-Phase für mindestens 48 h wurden.
- Arbeiten mit 8 gut Mikroskop Kammern (konstante Verteilung der Zellen).
Zum Beimpfen von Zellen auf 8-Well-Mikroskop-Kammern mit einer konstanten Verteilung über die gesamte Fläche des Mikroskops Kammer Zentrifuge 10.000 Zellen und Resuspendieren in 350 ul Phenolrot-freiem normalem Wachstumsmedium (ergänzt mit 10% fötalem Rinderserum, Penicillin / Streptomycin und Natriumpyruvat). Übertragen Zellsuspension zum Mikroskop Kammer (Abbildung 2).
Arbeiten mit 8 gut Mikroskop Kammern (maximale Zelldichte in der Mitte).
Zum Beimpfen von Zellen in einer höheren Dichte im Zentrum des Mikroskops chambäh, laden Sie die Kammer mit 300 ul Phenolrot-freiem normalem Wachstumsmedium. Hinzufügen 5.000 Zellen vorsichtig auf die Mitte des Mikroskops Kammer (Abbildung 2).
Arbeiten mit 96 auch spezielle Optik Platten (konstante Verteilung der Zellen).
Zum Beimpfen von Zellen auf 96-Well-Platten bei einer konstanten Verteilung über die gesamte Oberfläche des Bohrlochs, zentrifugieren 5.000 Zellen und Resuspendieren in 300 ul Phenolrot-freiem normalen Wachstumsmedium. Übertragen der Zellsuspension auf eine 96-Well-Platte. Abhängig von der Gesamtzahl der Zellen enthaltende Bohrungen Bedarf die Zellzahl und das Gesamtvolumen des Suspensionsmediums (Abbildung 2).
Arbeiten mit 96 auch spezielle Optik Platten (maximale Zelldichte in der Mitte).
Für die Aussaat von Zellen auf 96-Well-Platten, vorsichtig 1.500 Zellen in 15 ul Phenolrot-freiem normalem Wachstumsmedium in einem small Tropfen auf die Mitte jeder Vertiefung zur Beschränkung Zellwachstum zu erreichen, um die Mitte der Wanne (Abbildung 2).
- Lassen ausgesäten Zellen für mindestens 18 Stunden unter Standardbedingungen Zellkultur Bedingungen (37 ° C, 100% Luftfeuchtigkeit, 5% CO 2) zu wachsen.
2. Die Färbung der Reporter Zellen mit SNAP Substrate
- 30 Minuten vor dem Beginn des Färbeverfahrens ermöglichen Aliquots von Phenolrot-frei normalen Wachstumsmedium zum Aufwärmen auf 37 ° C.
- Für die einfache Handhabung des SNAP Substrat (in unserem Fall TMR Star) aufzulösen TMR Star in DMSO, um eine Konzentration in der Stammlösung von 400 uM, die bei -20 ° C gelagert werden erhalten
- Vor dem Färben, verdünnte 0,5 ul TMR-Sterne-Stammlösung in 200 ul Phenolrot-freiem normalem Wachstumsmedium (37 ° C), um eine endgültige Kennzeichnung Konzentration von 1 uM erhalten.
- Entfernen normalem Wachstumsmedium von den asynchron wachsenden Zellen und Inkubation in lAbeling Medium für 25 min unter Standard-Kulturbedingungen.
- Entfernen Etikettieren Medium und Waschen Zellen viermal mit Phenolrot-freiem normalem Wachstumsmedium (37 ° C). Inkubieren Zellen in 300 ul Phenolrot-freiem normalem Wachstumsmedium (37 ° C) für 30 min. Vor dem Transport zu dem Mikroskop ersetzen das Medium mit frischem Phenolrot-freiem normalem Wachstumsmedium (37 ° C), um restliche ungebundenen SNAP Substrat zu entfernen.
- Transport-Zellen mit dem Mikroskop in einem Styropor-Box auf einem vorgewärmten (37 ° C) Heizblock gegen Temperaturschwankungen zu minimieren.
3. Messung der Fluoreszenzintensität
- Zwei Stunden vor der Analyse einstellen Lufttemperatur der Klimakammer bei 37 ° C in trockenem Modus, um das gesamte Mikroskop mit all seinen Komponenten auf die gewünschte Temperatur zu bringen. Vorheizen, bevor Sie die Luftfeuchtigkeit ist wichtig, Kondensation und anschließende Beschädigung des Mikroskops zu vermeiden.
- Passen Luftfeuchtigkeit to 60% und CO 2 bis 5% vor dem Beginn der Analyse.
- Starten Sie den Scan ^ R Acquisition Software und definieren Standard-Einstellungen (siehe Tabelle 1).
- Werden die Stellen der Vertiefungen analysiert werden.
- Definieren Sie die At (Übernahme Zykluszeit) und die absolute Zahl der Erwerbs-Zyklen.
- Wenn die Analyse einer größeren Anzahl von Brunnen gewünscht wird, wählen Sie Hardware Autofokus, sonst ist es ausreichend, um Software-Autofokus-alone verwenden.
- Starten Sie die Aufnahme und Überwachung in den ersten beiden Erfassungszyklen. Das Mikroskop ist am Histon H2-GFP-Signal zu konzentrieren, mit anschließender Übernahme eines ersten Bildes in diesem Kanal, bevor der Filter gewechselt wird und der entsprechende TMR Stern Bild aufgenommen wird (Abbildung 3). Dies wird dann für alle Positionen innerhalb einer Vertiefung und für jede der Mulden zu untersuchenden wiederholt, vor der Wiederholung erneut den nächsten Zyklus.
4. Analyse der proteolytischen Profile usingen Scan ^ R
- Starten Sie den Scan ^ R-Analyse-Software und analysieren die Bilder mit den Zellkernen, wie visualisiert Histon H2-GFP, als Haupt-Objekt definiert, mit einer Schwelle von Signalintensität und einer Wasserscheide-Algorithmus, um die Trennung benachbarter Zellen unterstützen basiert. Ein Unterobjekt, bestehend aus einem Kern mit Zytoplasma sollte TMRstar Analyse erstellt werden. (Wichtige Eigenschaften des Hauptobjekts sind X und Y-Positionen, Zeit und maximalen und mittleren Intensitäten der GFP für die Haupt-Objekt und die gesamte Intensität Bereich für die TMRstar Unterobjekt unterteilt.) Diese Analyse kann einige Stunden wegen großer Datenmengen Mengen.
- Ändern Sie den Modus zu verfolgen, um die subobject mittlere TMR-Sterne Fluoreszenzintensität über die Zeit (Zelle Spuren), zugeordnet zu den analysierten wichtigsten Objekte (Abbildung 4) zu visualisieren. Die Anzahl der zu untersuchenden Zellen lassen sich nach Gating auf diesen Messungen Laufzeit von mindestens 140 Zyklen und mit einer hohen maximalen Lichtstärke von H2-GFP verengt werden. Suchenbei größeren Anzahlen von Zellen ermöglicht eine erste repräsentative und objektive Sicht (Abbildung 4).
- Wählen Sie eine Zelle Spur von Interesse Histon H2-GFP-und TMR-Sterne-Fluoreszenz gleichzeitig an der einzelnen Zelle (5A) zu visualisieren.
- Mit der rechten Maustaste auf eine Zelle von Interesse und erzeugen eine ausführbare Bildergalerie zur Illustration von Histon H2-GFP und Cyclin B-SNAP TMR Star für jeden einzelnen Zeitpunkt.
- Wechseln Sie in der Bevölkerung Modus des Scan ^ R Analysesoftware und Tor in der Region, wo die Zelle von Interesse auf der X vs Y dot plot (5B) vertreten ist.
- Tragen Sie einen neuen Punkt Plot-Fenster auf die gated Region und visualisieren meine TMR-Sterne Fluoreszenzintensität über die Zeit (Abbildung 5C).
- Exportieren von Daten (Zeit und Fluoreszenz-Intensität), um Microsoft Excel zur weiteren Berechnung.
5. Repräsentative Ergebnisse
Feigeure 5D und 5E zeigen Cyclin B Kinetik, durch einen Stern TMR Fluoreszenzintensität Kurve dargestellt, mit einer Zelle, die durch einen regelmäßigen verläuft Mitose ohne Anzeichen einer chromosomalen Fehlausrichtung (5E). Nach der Verdichtung des Cytoplasmas nach Kernhülle Aufteilung (NEBD, wie durch den roten Dreieck gekennzeichnet) zeigt TMR-Sterne Fluoreszenzintensität einen abrupten Anstieg bis die isomorph Fenster (heller Bereich in der Abbildung) ist erreicht, wenn die Zelle in Prometaphase 6. Fluoreszenzintensität bleibt auf einem stabilen Niveau, solange die Zelle durchläuft und Prophase Metaphase und dann beginnt schnell einmal fallen alle Chromosomen eines stabilen Metaphaseplatte festgelegt (Figur 5D und 5E). Dieser Rückgang voraus Trennung der Chromosomen während der Anaphase (blauer Punkt auf der Kurve). Im späten Mitose das Chromatin beginnt decondense (blaue Balken) und die Zelle nimmt Interphase Morphologie während die Fluoreszenzintensität Kurve nähert sich einPlateau, das niedriger als das Plateau vor der Mitose (5D und 5E) ist.
| Autofokus-Einstellungen | Histone H2-GFP (Hauptobjekt Akquisition Einstellungen) | Cyclin B-SNAP mit TMR Stern markiert (Erwerb Einstellungen) | Erfassungszykluszeit Wiederholungen |
| Grob Autofokus + / -39 um 24 Layers Feine Autofokus + / -5,4 Um 14 Layers | GFP Filter gesetzt: Belichtungszeit: 100 ms Lichtstärke: 25% | TRITC Filter gesetzt: Belichtungszeit: 150 ms Lichtstärke: 33,3% | 2 bis 5 min. |
| GFP Filter gesetzt: Belichtungszeit: 12 ms Lichtstärke: 12,5% | Bis zu 48 h an fortgesetzte Analyse (begrenzt durch reduzierte Luftfeuchte von 60%) |
Tabelle 1. Standardeinstellungen für Analysen von Cyclin B Proteolyse verwendet. Histone H2-GFP wurde als Referenz verwendet, um die Struktur Fokusebene zur Messung von Cyclin B-SNAP Fluoreszenzintensität definieren. Lichtintensitäten während der Fokussierung Verfahrens sind niedriger als bei der Bildaufnahme Einstellungen gegenüber kumulativen Phototoxizität zu vermeiden.
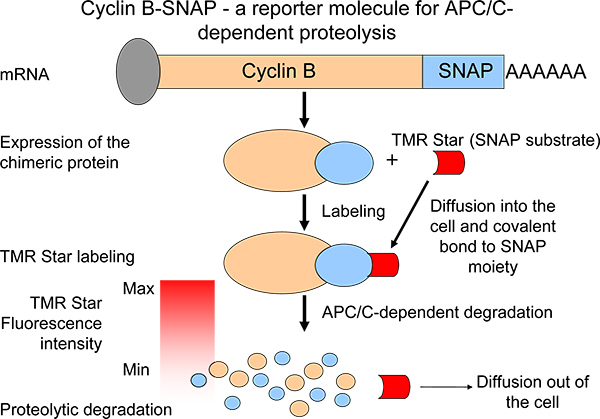
Abbildung 1. Schematische Darstellung der Messung von Cyclin B Proteolyse durch Expression eines chimären Cyclin B Derivates Abbaueigenschaften ähnlich dem endogenen Protein. Der Rückgang der Fluoreszenzintensität nach Pulse-Chase-Markierung ist ein Maß für APC / C-Aktivität.
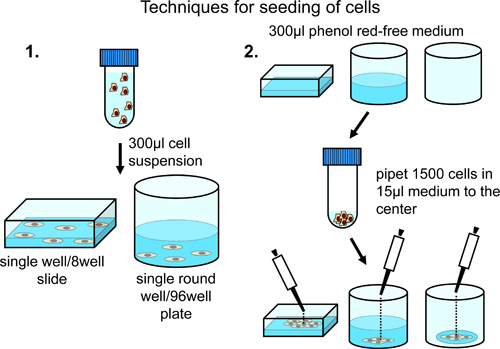
Abbildung 2. Analyse von Zellen auf 8 gut Dias oder 96-Well-Platten. Regelmäßige Verteilung derZellen in der Oberfläche des Bohrlochs durch Resuspension in einem Endvolumen von 300 ul erreicht und wird empfohlen, wenn nur ein einzelner oder weniger Positionen für eine Analyse manuell definiert werden. Anreicherung von Zellen im mittleren Bereich wird durch Zugabe von Zellen zu dem Zentrum der vorgefüllten oder leeren Vertiefungen erreicht. Diese Technik wird im Falle einer automatisierten Bildaufnahme in verschiedenen Vertiefungen empfohlen.

Abbildung 3. Ablauf der Datenerfassung. A) Nachweis von Histon-H2-GFP ist für Fokusebene Definition und Überwachung von chromosomalen Ausrichtung während der Mitose verwendet. B) Fluoreszenzintensität des TMR Stern Fluoreszenzintensität ist ein Maß für den absoluten Betrag der Pulse-Chase-markierten Cyclin B-SNAP.
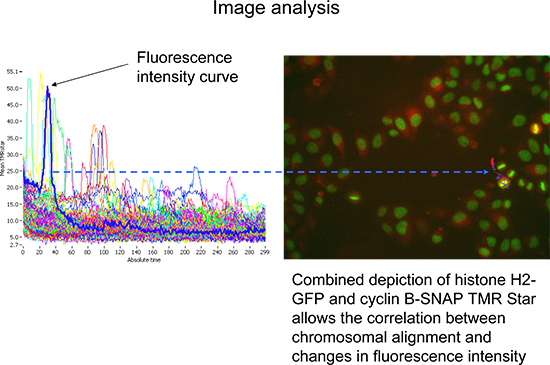
Abbildung 4. Vertreter Scan ^ R Analysis Software-basierte Darstellung der mittleren TMR SternFluoreszenzintensitäten (insgesamt TMRstar Intensität geteilt durch Fläche) über die Zeit. Auswahl einer repräsentativen Zelle Spur (blau). Die entsprechenden Bilder (siehe rechts) ermöglichen die gleichzeitige Darstellung von Histon H2-GFP (grün) und Cyclin B-SNAP (rot). Klicken Sie hier für eine größere Abbildung zu sehen .
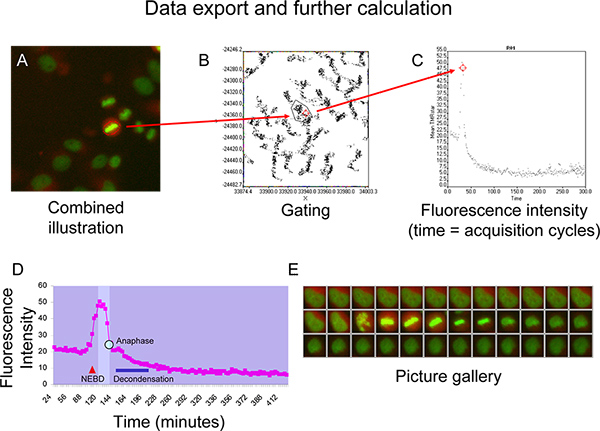
Abbildung 5. A + B) Beispiel für Gating von Punkten, die eine Zelle von Interesse auf der XY-Punkt-Diagramm. C) Visualisierung der mittleren TMRstar Fluoreszenzintensität (insgesamt TMRstar Intensität geteilt durch Fläche) im Laufe der Zeit mit der Dot-Plot. D + E) Vertreter Fluoreszenzintensität Kurve von Cyclin B-SNAP mit isomorph Fenster (brigher Feld auf dem Diagramm) zwischen NEBD (Kernhülle Aufteilung) und Chromatindekondensation (blauer Balken) als in Microsoft Excel erzeugt. Anaphase durch t angedeutetEr blauen Punkt auf der Kurve. Klicken Sie hier für eine größere Abbildung zu sehen .
Diskussion
Wir stellen Ihnen hier eine Live-Cell-Imaging-basierten Ansatz erleichtert die gleichzeitige Überwachung von Cyclin B Proteolyse und Chromosom Ausrichtung. Dieser Ansatz ermöglicht die Untersuchung der mitotischen Kontrolle in ungestörten Zellpopulationen an der einzelnen Zelle. Cyclin B Abbaukurven erleichtern direkten Einblick in die Aktivität des APC / C und damit indirekt geben den Status des SAC 6.
Dieser Ansatz, obwohl gegründet basierend auf dem Scan ^ R-Software, kann...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir sind dankbar, S. Taylor für die Bereitstellung der pLPCX-Histone H2-GFP-Plasmid. Wir danken R. Mertelsmann für die kontinuierliche Unterstützung. Diese Arbeit wurde von der Deutschen Forschungsgemeinschaft unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Produkts | Firma | Katalog-Nummer | Kommentare (optional) |
| Reporter-Zelllinie | generiert in-house wie beschrieben 6 | Klon 11 Reporterzellen (U2OS basierenden Cyclin B-SNAP exprimierende Zellen) | |
| Retrovirale Cyclin B-SNAP Expressionsvektor | generiert in-house wie beschrieben 6 | pLNCX2-Cyclin B mut5-SNAP | |
| Phenolrot-freiem DMEM | Gibco | 21063-029 | Supplementierung mit FCS, Natriumpyruvat, Penicillin / Streptomycin-mycin erforderlich |
| SNAP-Cell TMR-Star | New England Biolabs | S9105S | Stammlösung 400 uM in DMSO |
| Besondere Opstics Plate, 96 well | Costar | 3720 | |
| μ-Slide 8 gut, ibiTreat | Ibidi | 80826 | |
| Mikroskopie-Einheit | Olymp | IX-81 inverse Mikroskop mit Klimakammer | |
| Ziel | Olymp | UPLSAPO 20x-Objektiv (NA 0,75) | |
| Acquisition Software | Olymp | Scan ^ R Acquisition Software (v.2.2.09) | |
| Analyse-Software | Olymp | Scan ^ R Analysis Software (v.1.2.0.6) |
Referenzen
- Musacchio, A., Salmon, E. D. The spindle-assembly checkpoint in space and time. Nat. Rev. Mol. Cell Biol. 8, 379-393 (2007).
- Nasmyth, K. Segregating sister genomes: the molecular biology of chromosome separation. Science. 297, 559-565 (2002).
- Cahill, D. P. Mutations of mitotic checkpoint genes in human cancers. Nature. 392, 300-303 (1998).
- Lengauer, C., Kinzler, K. W., Vogelstein, B. Genetic instabilities in human cancers. Nature. 396, 643-649 (1998).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463, 485-4892 (2010).
- Schnerch, D. Monitoring APC/C activity in the presence of chromosomal misalignment in unperturbed cell populations. Cell Cycle. 11, (2012).
- Keppler, A. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat. Biotechnol. 21, 86-869 (2003).
- Jansen, L. E., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J. Cell. Biol. 176, 795-805 (2007).
- Malureanu, L. Cdc20 hypomorphic mice fail to counteract de novo synthesis of cyclin B1 in mitosis. J. Cell Biol. 191, 313-329 (2010).
- Mena, A. L., Lam, E. W., Chatterjee, S. Sustained spindle-assembly checkpoint response requires de novo transcription and translation of cyclin B1. PLoS One. 5, (2010).
- Wolthuis, R. Cdc20 and Cks direct the spindle checkpoint-independent destruction of cyclin. A. Mol. Cell. 30, 290-302 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten