JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
전체 세포 인구의 단일 셀 수준에서 Cyclin B의 Proteolysis 유학
요약
전환이 anaphase-추진 단지 (APC / C)에 의존 ubiquitination과 여기 cyclin B.의 후속 파괴를, 우리는, 펄스 - 체이스 라벨에 따라, 시스템을 설립 전체 세포 인구에서 cyclin B의 proteolysis를 모니터링 할 수 있으며를 통해 실행됩니다 anaphase에 Metaphase mitotic 검문소에 의한 간섭의 검색을 용이하게한다.
초록
세포 분열 동안 두 딸 세포 사이의 염색체의 동등한 배포 유전자 안정성 1 보장을위한 전제 조건입니다. 염색체 분리시 부정확 한이 종의 특징이며 진행성 질환 2-4와 관련된. 스핀들 조립 검사 점 (SAC)는 모든 단일 염색체는 mitotic 스핀들 1 안정적인 바이폴라 첨부 파일을 확립 할 때까지 다시 metaphase에서 세포를 보유하고 mitotic 감시 메커니즘입니다. SAC는 활성화와 간섭에 의해 그 기능을 발휘 APC / C securin와 cyclin B의 proteolysis하므로 염색체 분리와 mitotic 출구를 차단하는 subunit Cdc20. 염색체의 부적절한 첨부 파일 SAC 신호의 입을 방지하고 문제가 염색체 missegregation, aneuploidy와 악성 growths 일을 피하기 위해 해결 될 때까지 APC / C Cdc20의 지속적인 억제가 발생합니다.
내가 연설을 대부분의 연구APC / C에 의존 proteolysis에 부적절한 염색체 첨부 파일의 nfluence는 미소로 염색체 첨부 파일을 방해하는 약물을 depolymerizing 또는 미세 소관 - 안정화를 사용하여 스핀들 혼란을 이용했다. 미세 소관의 동력학과 간섭이 교통 및 중요한 규제의 국산화에 영향을 미칠 수 있기 때문에, 이러한 절차는 인공 효과, 5를 일으킬 위험을 맺습니다.
SAC는에서 유사 분열 동안 APC / cyclin B의 C-의존 proteolysis을 방해하는 방법 공부하는 세포 집단을 교란되지 않은, 우리는 허용 히스톤 H2-GFP-기반 시스템을 설립 metaphase mitotic 염색체의 정렬 및 cyclin B 6 proteolysis의 동시 모니터링 .
proteolytic 프로필을 묘사하기 위해, 우리는 C-터미널 SNAP 잔기 6 (그림 1) 키메라 cyclin의 B 기자 분자를 생성. 자체 라벨 반응에서 SNAP-잔기가 함께 공유 결합 채권을 형성 할 수 있습니다alkylguanine - 반송파 (SNAP 기판) 7,8 (그림 1). SNAP 기판 분자 쉽게 사용할 수 있으며 다른 fluorochromes의 폭 넓은 스펙트럼을 가지고 다니십시오. 키메라 cyclin B-SNAP 분자는 성장 매체 7 (그림 1)에 막 투과성 SNAP 기판의 추가에 표시됩니다. 라벨 반응 후, cyclin B-SNAP 형광 강도는 펄스 추격전 반응과 같은 방식으로 형광 강도 cyclin B 저하 6 (그림 1)의 수준을 반영에 떨어. Google 시스템은 병렬 셀 (또는 여러 세포 집단)의 큰 숫자 mitotic APC / C에 의존 proteolysis의 모니터링을 용이하게한다. 따라서, 시스템은 에이전트 / 전환을 anaphase 할 metaphase에서 proteolytic 활동을 방해 할 수있는 작은 분자를 식별 할 수있는 귀중한 도구가 될 수 있습니다. 또한, 유사 분열 동안 cyclin B의 합성으로 최근 중요한 mechanis으로 제안 된안정적인 cyclin B 표현 수준 9,10를 유지하여 마우스와 인간의 mitotic 블록을 조성하는데있어,이 시스템은 우리가 균형 잡힌 평형 6 항 중 어느 한 요소로 cyclin의 B의 proteolysis를 분석 할 수있게.
프로토콜
1. 현미경 상공 회의소 슬라이드에 U2OS 기반 Cyclin B-SNAP 리포터 세포 (클론 11 셀 6) 심는
- 최소 48 시간의 로그 단계에서 비동기 적으로 성장 할 수 있었다 subconfluent SNAP 기자 세포를 Trypsinize.
- 8 잘 현미경 챔버 (세포의 지속적인 배포)과 함께 일하는 것.
현미경 챔버의 전체 표면 원심 분리기 만 세포에서 일정한 분포 8 잘 현미경 챔버에 세포를 퍼 뜨리고 및 페놀 붉은 무료로 정상적인 성장 매체의 350 μl (10 % 태아 소 혈청, 페니실린 / 스트렙토 마이신과 보충에 resuspend에 대한 나트륨 pyruvate). 현미경 챔버 (그림 2)에 세포 현탁액을 전송합니다.
8 잘 현미경 챔버 (중앙에 최대 세포 밀도)의 협력을 참조하시기 바랍니다.
현미경 chamb의 중심에 높은 밀도로 세포의 퍼 뜨리고 들어음, 페놀 붉은 무료로 정상적인 성장 매체 300 μl로 챔버를로드합니다. 현미경 챔버의 중심 (그림 2)주의 깊게 5,000 셀을 추가합니다.
96 잘 특별 광학 플레이트 (세포의 지속적인 배포)과 함께 일하는 것.
잘의 전체 표면에 걸쳐 일정한 분포에서 96 잘 플레이트로 세포의 퍼 뜨리고를 들어, 5,000 세포를 원심 분리기와 페놀 붉은 무료로 정상적인 성장 매체 300 μl에 resuspend. 96 - 웰 플레이트에 세포 현탁액을 전송합니다. 필요한 세포 함유 우물의 총 수에 따라 휴대폰 번호 및 정지 매체의 총 부피 (그림 2)를 조정할 수 있습니다.
96 잘 특별 광학 플레이트 (중앙에 최대 세포 밀도)의 협력을 참조하시기 바랍니다.
96 잘 플레이트로 세포의 시딩를 들어, 신중하게 smal에 페놀 붉은 무료로 정상적인 성장 매체의 15 μl에 1,500 셀을 추가잘의 중심 (그림 2)에 세포의 성장에 제한을 달성하기위한 각도의 중심부에 난 드롭.
- 시드 세포 표준 세포 배양 조건 (37 ° C, 100 % 공기 습도, 5 % CO 2)에 따라 최소 18 시간에 성장 할 수 있습니다.
2. SNAP 기판과 리포터 세포의 착색
- 전에 착색 절차의 시작 부분에 30 분은 페놀 붉은 무료로 정상적인 성장 매체의 aliquots 37까지 따뜻하게 할 수 있습니다 ° C.
- SNAP 기판의 취급이 용이합니다 (우리의 경우 TMR 스타에서) -20 ° C.에 저장할 수 있습니다 400 μm의 주식 솔루션에 집중을 얻기 위해 DMSO에 TMR 스타를 해산
- 이전 착색에, 1 μm의 최종 라벨 농도를 얻기 위해 페놀 붉은 무료로 정상적인 성장 매체 (37 ° C) 200 μl에 TMR 스타 주식 솔루션의 0.5 μl를 희석.
- 비동기 적으로 성장하는 세포의 정상적인 성장 매체를 제거하고 리터에 품다표준 문화 조건에서 25 분을 위해 매체를 abeling.
- 페놀 붉은 무료로 정상적인 성장 매체 (37 ° C)와 중간 및 세척 세포 네 번 라벨 제거합니다. 30 분을위한 페놀 붉은 무료로 정상적인 성장 매체 (37 ° C) 300 μl에 세포를 배양. 이전 현미경을 운반에 잔류 언 바운드 SNAP 기판을 제거하는 신선한 페놀 붉은 무료로 정상적인 성장 매체 (37 ° C)와 매체를 교체하십시오.
- 미리 예열에 스티로폼 상자에 현미경으로 전송 셀 (37 ° C) 온도 편차를 최소화하기 위해 열 블록.
3. 형광 강도 측정
- 전에 분석에 두 시간 ° C 건조 모드에서 원하는 온도로 모든 구성 요소를 사용하여 전체 현미경을 가져다하기 위해 37 기후 챔버의 공기 온도를 조정합니다. 사전 가열 습도를 설정하기 전에 현미경에 결로 및 후속 손상을 방지하는 것이 중요합니다.
- 공기 습도 t을 조정O 60% 및 분석의 시작하기 전에 CO 2-5%.
- 스캔 ^ R 취득 소프트웨어를 시작하고 표준 설정 (표 1 참조) 정의합니다.
- 분석 할 우물의 위치를 정의합니다.
- Δt (수집 사이클 타임) 및 취득주기의 절대 수를 정의합니다.
- 우물의 더 많은 분석이 필요하다면, 하드웨어 자동 초점을 선택, 그렇지 않으면 혼자 소프트웨어 자동 초점을 사용하기에 충분합니다.
- 인수를 시작하고 처음 두 인수 사이클에 대해 감독한다. 필터가 변경되어 해당 TMR 스타 이미지 (그림 3) 인수되기 전에 현미경 해당 채널의 첫 번째 이미지의 후속 취득과 함께 히스톤 H2-GFP 신호에 초점을 맞출 것이다. 이것은 다시 다음주기를 반복하기 전에 잘 내의 검사 할 우물의 각에 대한 모든 위치에 대해 반복됩니다.
4. Proteolytic 프로필 U 분석스캔 ^ R을 노래
- 스캔 ^ R 분석 소프트웨어를 시작하고 세포 핵으로 이미지를 분석과 같은 신호 강도와 인근 세포를 분리하는 데 큰 도움이 분수령 알고리즘을 기반으로하는 임계 값을 사용하여, 주요 개체로 정의 된 히스톤 H2-GFP에 의해 시각화. 세포질과 핵으로 구성된 subobject는 TMRstar 분석을 위해 생성되어야합니다. (주 객체의 중요 속성 X와 Y 위치, 시간, 및 최대이며 주요 개체와 TMRstar subobject을위한 영역으로 나눈 전체 강도에 대한 GFP의 농도를 의미합니다.)이 분석 과정은 큰 데이터로 인해 몇 시간이 걸릴 수 있습니다 수량.
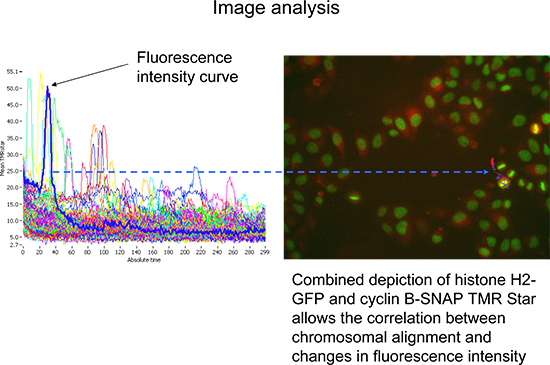
- 분석 주요 개체 (그림 4)에 할당 된 시간이 지남에 따라 subobject 말은 TMR 별 형광 강도를 (셀 추적), 시각화 모드를 추적 변경합니다. 검사 할 셀의 수는 적어도 140 사이클을 지속 해당 측정에 게이팅 의해 H2-GFP의 높은 최대 강도로 좁혀 할 수 있습니다. 찾고세포의 큰 숫자에 첫 번째 대표하고 객관적인보기 (그림 4) 있습니다.
- 단일 세포 수준 (그림 5A)에서 동시에 히스톤 H2-GFP 및 TMR 스타 형광를 시각적으로 관심 셀 추적을 선택합니다.
- 관심있는 셀을 마우스 오른쪽 클릭을 사용하고 히스톤 H2-GFP 및 cyclin B-SNAP 매번 지점에 대한 TMR 스타의 그림에 내보낼 사진 갤러리를 생성합니다.
- 스캔 ^ R 분석 소프트웨어 및 게이트 관심 셀이 X 대 Y 점 플롯 (그림 5B)에 표현된다 지역의 인구 모드로 변경합니다.
- 게이트 지역에 새로운 점 플롯 창을 적용하고 시간이 지남에 따라 TMR 스타 형광 강도 (그림 5C)를 의미 표시합니다.
- 자세한 계산 용 Microsoft Excel로 내보내기 데이터 (시간과 형광 강도).
5. 대표 결과
무화과우레 5D와 5E 호야는 셀의 TMR 별 형광 강도 곡선으로 표현 cyclin B 동력학을, 묘사 그 염색체 misalignment의 흔적 (그림 5E 호야)없이 일반 유사 분열을 통해 진행. 셀 prometaphase 6 입력 할 때 isomorphic 창 (다이어그램에서 밝은 영역)에 도달 할 때까지 핵 봉투 고장 (NEBD 등 빨간색 삼각형으로 표시)에 따라 세포질의 압축되면, TMR 스타 형광 강도는 갑작스런 증가를 보여줍니다. 형광 강도는 prophase와 metaphase를 통해 셀 수익금만큼 안정적인 수준으로 유지 한 후 염색체의 모든 (그림 5D와 5E 호야) 안정적인 metaphase 플레이트를 설립 한 후 빠른 속도로 떨어지기 시작합니다. 이 드롭 anaphase (곡선에 파란색 점) 동안 염색체 분리를 앞에. 후반 유사 분열에서 염색질은 decondense 시작 (파란색 바)과 형광 강도 곡선이 접근하는 동안 셀은 계면 형태를 채택유사 분열 (그림 5D와 5E 호야) 전 고원보다 낮은 고원.
| 자동 초점 설정 | 히스톤 H2-GFP (주 객체 수집 설정) | TMR 스타 라벨이 Cyclin B-SNAP (수집 설정) | 취득주기 시간 반복 |
| 거친 자동 초점 + / -39 μm 24 층 정밀 자동 초점 + / -5.4 μm 14 층 | GFP 필터 설정 : 노출 시간 : 100 밀리 초 빛의 세기 : 25 % | TRITC 필터 설정 : 노출 시간 : 150 밀리 초 빛의 세기 : 33.3 % | 2-5 분. |
| GFP 필터 설정 : 노출 시간 : 12 밀리 초 빛의 세기 : 12.5 % | 최대 지속적인 분석의 48 시간 (60 %의 감소 공기 중 습도에 의해 제한)에 |
표 1. cyclin B의 proteolysis의 분석에 사용 된 표준 설정입니다. 히스톤은 H2-GFP는 cyclin B-SNAP 형광 강도의 측정을 위해 초점 비행기를 정의하는 참조 구조로 사용되었다. 누적 광독성을 피하기 위해 이미지 수집 설정에 비해 초점 절차를 수행하는 동안 광도가 낮은 있습니다.
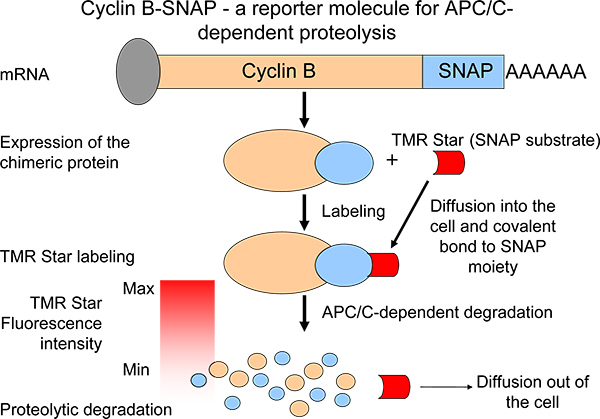
1 그림. 내생 단백질과 유사한 열화 특성과 키메라 cyclin의 B 유도체의 표현을 통해 cyclin B의 proteolysis의 측정의 개략도. 펄스 - 체이스 라벨 후 형광 강도의 하락은 APC / C-활동을 측정하기위한 한 방법입니다.
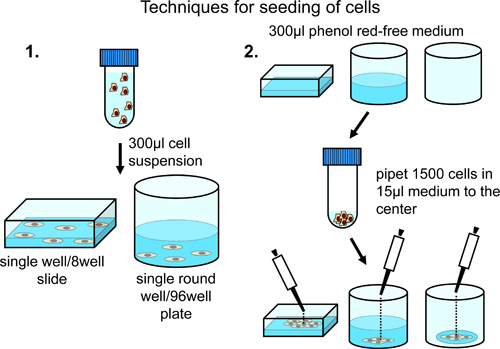
그림 2. 8도 슬라이드 또는 96 잘 접시에 세포의 분석.의 정기 배포우물의 표면에 걸쳐 셀 300 μl의 최종 볼륨에 resuspension에 의해 달성된다 만 단일 또는 몇 위치를 수동으로 분석을 위해 정의되는 경우 권장합니다. 중심 지역에서 세포의 농축은 prefilled 또는 빈 우물의 중심에 셀의 추가에 의해 달성된다. 이 기술은 다른 우물의 자동 이미지 수집의 경우 권장합니다.

그림 3. 데이터 수집의 순서가. 히스톤 H2-GFP의 A) 검색이 유사 분열 동안 염색체 정렬의 초점 평면 정의 및 모니터링에 사용됩니다. B) TMR 스타 형광 강도의 형광 강도는 cyclin B-SNAP 표시된 펄스 체이스의 절대 금액을 측정하기위한 한 방법입니다.
4 그림. 평균 TMR 별 대표 스캔 ^ R 분석 소프트웨어 기반의 묘사시간이 지남에 따라 형광 강도 (면적으로 나눈 총 TMRstar 강도). 대표 셀 추적 (파란색)의 선택. 해당 이미지 (오른쪽에 표시)의 동시 시각화를 허용 히스톤 H2-GFP (녹색)와 cyclin B-SNAP (빨간색). 더 큰 그림을 보려면 여기를 클릭하십시오 .
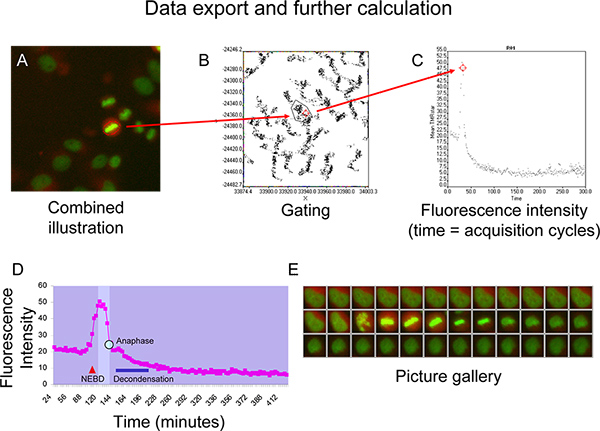
그림 5. A + B) XY-점 음모에 관심있는 셀을 나타내는 점의 게이팅을위한 예. C) 시간이 지남에 따라 평균 TMRstar의 형광 강도 (면적으로 나눈 총 TMRstar 강도)의 시각화는 점 플롯을 사용합니다. D + E) Microsoft Excel에서 생성으로 NEBD (핵 봉투 고장)와 염색질 decondensation (파란색 막대) 사이 isomorphic 창 (그림에서 brigher 필드)와 cyclin B-SNAP 대표 형광 강도 곡선. Anaphase는 t로 표시됩니다곡선에서 그는 파란색 점은. 큰 그림을 보려면 여기를 클릭하십시오 .
영화는 1. 보완 동영상을 보려면 여기를 클릭하십시오 .
토론
우리는 여기 cyclin B의 proteolysis 및 염색체 정렬의 동시 모니터링을 용이하게 라이브 셀 이미징 기반의 접근 방식을 제시한다. 이 방법은에서 mitotic 컨트롤의 연구는 단일 세포 수준에서 세포 인구를 교란되지 않은 수 있습니다. Cyclin B 저하 곡선은 APC / C의 활동에 직접 통찰력을 촉진하므로 간접적으로 SAC 6의 상태를 반영합니다.
이 방법은 비록 스캔 ^ R 소프트웨어를 ...
공개
관심 없음 충돌이 선언 없습니다.
감사의 말
우리는 pLPCX-히스톤 H2-GFP 플라스미드를 제공 S. 테일러에게 감사합니다. 우리는 지속적인 지원 R. Mertelsmann 감사드립니다. 이 작품은 독일 Forschungsgemeinschaft에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| 제품의 이름 | 회사 | 카탈로그 번호 | 코멘트 (선택 사항) |
| 리포터 세포 라인 | 생성 자체는 6 설명 | 11 기자 세포를 복제 (U2Os 기반 cyclin B-SNAP 표현 세포) | |
| Retroviral cyclin B-SNAP 표현 벡터 | 생성 자체는 6 설명 | pLNCX2-cyclin B mut5-SNAP | |
| Phenolred 무료 DMEM | Gibco | 21063-029 | FCS, 나트륨 pyruvate, 페니실린 / strepto-mycin과 보완 필요 |
| SNAP-세포 TMR-스타 | 뉴 잉글랜드 Biolabs | S9105S | DMSO의 주식 솔루션을 400 μM |
| 특별 Opstics 판, 잘 96 | Costar | 3720 | |
| μ-슬라이드 8 잘 ibiTreat | Ibidi | 80,826 | |
| 현미경 단위 | 하늘 | 기후 챔버와 IX-81 역 현미경 | |
| 목표 | 하늘 | UPLSAPO 20x 목적 (NA 0.75) | |
| 수집 소프트웨어 | 하늘 | 스캔 ^ R 취득 소프트웨어 (v.2.2.09) | |
| 분석 소프트웨어 | 하늘 | 스캔 ^ R 분석 소프트웨어 (v.1.2.0.6) |
참고문헌
- Musacchio, A., Salmon, E. D. The spindle-assembly checkpoint in space and time. Nat. Rev. Mol. Cell Biol. 8, 379-393 (2007).
- Nasmyth, K. Segregating sister genomes: the molecular biology of chromosome separation. Science. 297, 559-565 (2002).
- Cahill, D. P. Mutations of mitotic checkpoint genes in human cancers. Nature. 392, 300-303 (1998).
- Lengauer, C., Kinzler, K. W., Vogelstein, B. Genetic instabilities in human cancers. Nature. 396, 643-649 (1998).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463, 485-4892 (2010).
- Schnerch, D. Monitoring APC/C activity in the presence of chromosomal misalignment in unperturbed cell populations. Cell Cycle. 11, (2012).
- Keppler, A. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat. Biotechnol. 21, 86-869 (2003).
- Jansen, L. E., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J. Cell. Biol. 176, 795-805 (2007).
- Malureanu, L. Cdc20 hypomorphic mice fail to counteract de novo synthesis of cyclin B1 in mitosis. J. Cell Biol. 191, 313-329 (2010).
- Mena, A. L., Lam, E. W., Chatterjee, S. Sustained spindle-assembly checkpoint response requires de novo transcription and translation of cyclin B1. PLoS One. 5, (2010).
- Wolthuis, R. Cdc20 and Cks direct the spindle checkpoint-independent destruction of cyclin. A. Mol. Cell. 30, 290-302 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유