このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
DNAグリコシラーゼ活性の定常状態、プレ定常状態、およびシングルターンオーバー速度論的測定
要約
8 - オキソグアニンDNAグリコシラーゼのグリコシラーゼ活性のための時間のコースには、製品の形成と線形定常段階のバーストを示す二相性である。クエンチフロー法を利用して、バースト定常状態速度は、それぞれ、製品のDNAグリコシラーゼからの8 - オキソグアニンおよび放出の切除に対応する、測定することができる。
要約
ヒト8 - オキソグアニンDNAグリコシラーゼ(OGG1)は、DNAから8 - オキソ-7,8 - dihydroguanine(8 - oxoG)変異原性酸化的DNA損傷部を切除。 OGG1の運動特性評価は8 oxoG切除と製品リリースの速度を測定するために行われる。 OGG1濃度は、基質DNAよりも低い場合には、生成物形成の経時変化は二相であり、生成物形成の迅速な指数期( すなわちバースト)直鎖定常相が続く。生成物形成の初期バーストが正しく基板上に係酵素の濃度に相当し、バースト振幅は、酵素の濃度に依存する。一次速度バーストの定数は8-oxoG切除の極限速度に対応しており、遅い定常状態速度は製品リリースの速度(製品DNA解離速度定数、kは オフ ) を測定。ここでは、属を分離して測定するために、定常状態、予め定常状態、およびシングルターンオーバーのアプローチを説明OGG1触媒サイクリング中ecific手順。蛍光ラベルされた病変を含むオリゴヌクレオチドおよび精製OGG1は、正確な運動の測定を容易にするために使用されています。低酵素濃度は、定常状態測定値は、反応の試薬 の混 合を手動焼入れを行うために使用されるので、定常状態速度(kは オフ )を確認するために行うことができる。さらに、ゼロ時間で、縦軸上の点に定常速度の外挿は、製品の形成のバーストが最初のターンオーバー( つまり、y切片が正である)中に発生したことを示します。指数バースト位相の一定の一次速度は、定常状態位相の前に短い時間間隔(<1秒)に形成された生成物の量を検査し、8のレートに対応する迅速な混合および急冷法を用いて測定することができる-oxoG切除( すなわち化学)。触媒サイクルがどこにある化学的工程は、シングルターンオーバーアプローチを用いて測定することができる酵素飽和基質DNA(E> S)によって阻止。これらのアプローチは、DNA損傷の除去効率に影響を及ぼすの基本速度定数を測定することができる。
概要
好気性の環境は、ゲノムの不安定性を早める。酸化ストレスに起因する大規模な前変異DNAの病変は、7,8 - ジヒドロ-8 - オキソグアニン(8-oxoG)です。これは8-oxoGの曖昧なコーディングの可能性があるためです。ヒト8 - オキソグアニンDNAグリコシラーゼ(OGG1)は、8 oxoGの塩基除去修復を開始するための責任があります。アプリンサイト(AP-サイト)で、製品のDNAの結果8-oxoGベースOGG1消費税のグリコシラーゼ活性。 OGG1の弱いリアーゼ活性は、いくつかの例では、AP-サイトを切開することができます。
のDNAグリコシラーゼの運動特性評価は、一般的に、彼らは二相性のタイムコースを示すことを発見。生成物形成の初期段階に高速( すなわちバースト)後、線形定常状態位相は1-3観察される。この動作は、いがのに対し、タイムコースの直線部分の間に律速されている化学( すなわちコンホメーション変化や製品のリリース)は、次の手順を示すものである多くの場合、一過性相と称される第相は、反応の最初のサイクルの間に酵素活性部位での生成物の形成に相当する。製品のリリースは、定常状態フェーズにおいて律速である場合には、活性測定は、製品のDNA結合親和性の定性的な指標を提供しますが、酵素活性部位( すなわち化学)でのイベントに関する運動情報を提供していません。したがって、指数前定常状態バースト位相を分離して測定するための方法は、酵素の活性部位4で最初の酵素ターンオーバー時にイベントを調査するために必要とされる。
(1)定常状態、(2)予め定常状態、(3)シングルターンオーバーOGG1の触媒挙動を特徴付けるために3つの標準的な運動のアプローチが存在する。これらのアプローチは、反応混合物中の酵素の濃度と各アプローチにおいて利用基質比の酵素によって互いに異なる。典型的な定常状態のアプローチでは、時には、複数の回転動力学と呼ばれる酵素の低濃度は、製品の形成に従うために使用される。複数の酵素のターンオーバーが大幅に基質濃度に影響を与えないように、基質濃度が大幅に酵素濃度を超えています。この状況では、経時変化は、線形である必要があり、それがバーストこの方法で使用される低酵素濃度に起因する第ターンオーバー中に発生したかどうかを識別することは困難であり、バースト振幅は酵素濃度と等価であることに注意してください。これは、より高い酵素濃度を用いて第一の酵素のターンオーバーを迅速に発生したかどうかを検出するゼロ時間への線形時間経過を外挿することによって克服することができる。縦軸の切片(y軸)は酵素濃度に比例して、積極的に基板と係酵素の尺度を提供するべきである。このアプローチでは、原理的にはバースト位相の存在の証拠、DIFを提供することができますがferentアプローチは、バースト位相の速度を測定する必要がある。多くの場合、バースト位相は、手動混合焼入れの技術によって測定することが速すぎる。この状況では、前定常状態およびシングルターンオーバー運動( すなわち一過性運動)は、しばしば反応5の初期の時点に従わ急速混合および消光楽器を必要と近づく。予め定常状態のアプローチでは、高濃度の酵素製品は、かなりの量の第一の代謝回転の間に形成されるように使用される。複数のターンオーバー、触媒サイクリング( すなわちバーストに続く直線位相)を観察するために続いているので、基質濃度は、酵素濃度([酵素] <[基板])より大きい。触媒循環せず、酵素の活性部位でのイベントを分離するために、シングルターンオーバー条件が利用される。この場合、基板は、基板の全ては、私に参加するように、(E >> S)酵素で飽和されているn 'はシングルターンオーバー "とは、一般に単一指数関数時間経過を示す。
バースト位相は、製品のリリース(K オフ ) を示す酵素を上記のように、しばしば時間コースの定常段階のレートを制限します。製品リリース(V SS、濃/時間)の速度は、線形定常状態位相の傾きから決定することができる。活性酵素濃度(E)は、一定の内在的速度に製品リリースのレートを変換するために必要とされるここで、k オフ = V SS / [E]。重要なことに、活性酵素の濃度は、典型的に不純物、不活性酵素、非生産的に基材に結合した酵素、およびタンパク質濃度を決定するために使用される方法に起因する測定されたタンパク質濃度よりも低い。製品リリースが遅いとき活性酵素濃度は、バースト振幅から決定することができる。したがって、定常状態の時間経過にを外挿するゼロ時間は、観測された定常状態速度からK オフ (製品リリース)を計算するために必要な活性酵素の推定値を提供します。
バーストの動態を測定するには、あらかじめ定常状態アプローチは線形定常段階の前に発生最初離職中に製品の形成に従うことが必要である。バースト動態は、酵素製品中間体の形成に従う。反応は基質との混合酵素によって開始された後に反応が定常状態位相に達するまで、酵素生成物の量が急激に増加する。触媒作用ははるか迅速な製品リリースより長い場合は、バーストの振幅が想定して、製品への基質の化学変換のレートに対応均衡に積極的に従事し、酵素と観測された指数関数的なアプローチ(K OBS)に等しいと逆率化学はごくわずかです。
場合によっては、触媒CYでしがみつくような化学と製品リリースの速度の大きさが大きく異なっていない場合など、事前定常解析を妨害する。この場合、基板への過剰な酵素を用いた相対的な、単一の代謝回転の酵素と結合した触媒循環を制限基板を防止する。一次速度定数(kは OBS)などに応じて、反応の第一の化学的工程を単離することができ、正確に判定する。この速度定数は、上記プレ定常状態アプローチから決定OBS kのようになります。
ここでは、これらの運動のアプローチはOGG1のグリコシラーゼ活性を分析するために使用することができる方法について説明します。
プロトコル
1。酵素とDNA基質の調製
- E.におけるGST-融合タンパク質として過剰発現OGG1 大腸菌は 、精製の ためのGST-タグを利用した後、HRV-3Cプロテアーゼ( 図1)6裂によりGST-タグを削除。
- シングル8-oxoG残基を含む化学的に合成された5'-6 - カルボキシフルオレセイン(6-FAM)標識オリゴヌクレオチドおよびその相補未標識オリゴヌクレオチド鎖を購入します。 34-merのオリゴヌクレオチドは、5 '末端から17位の8-oxoGが含まれています。ポリアクリルアミドゲル電気泳動によりこれらのオリゴヌクレオチドを精製する。
- アニーリングバッファー中で1:1.2のモル比(10mMのトリス-HCl、pH7.5で、50mMのNaCl、1におけるその相補鎖と混合5'-6-FAM標識オリゴヌクレオチドた8 oxoGを含む二本鎖DNA基質を調製1.5 mlのマイクロチューブ内のEDTA)。 5分間沸騰したお湯でのフロートチューブを置きます。 Rにゆっくりと水が冷えて、次に場所にチューブを残して、OOM温度(これは約2時間かかります)。
- 蛍光標識の退色最小にするように低いかまたは最小限の光条件下での試料調製と実験を行う。
2。定常タイムコースとOGG1の活性部位滴定の測定
2.1。サンプル調製と定常状態の時間経過
- 氷の上に1.5 mlのマイクロチューブに液(50mM HEPES、pH7.5で、20mMの塩化カリウム、0.5mMのEDTA、および0.1%ウシ血清アルブミン)反応バッファーで個別にDNA基質とOGG1ソリューションを準備します。 DNAの濃度は400 nMであり、OGG1の見かけの濃度は、ブラッドフォードタンパク質アッセイによって決定される30、60、90、または120 nMである。 37に設定したヒートブロック、℃で反応チューブを置き37℃でプレインキュベートし、酵素とは別にDNA基質ソリューション℃で1分間。
- ピペッティングによりOGG1とDNA基質溶液の等量を混合することにより反応を開始します。これらのソリューションを混合した後tions 1:1(体積/体積)は、最終濃度は、200nMのDNAを、15、30、45、または60 nMのOGG1、それぞれである。時間間隔でアリコート(10μlの)を取り外し、1MのNaOHを1μlと混合することにより反応を停止。タイムコースの対照として、酵素なしの反応混合物を10μlの1MのNaOHを1μlと混合される。
- 得られた生成物AP-サイトを切断する5分間90℃のヒートブロックに反応サンプルを置きます。加熱後、各サンプルを中和するために1 M HClを1μLを加える。
- 各反応試料にゲルローディング緩衝液を等量(12μl)を(95%ホルムアミド、20mMのEDTA、0.02%ブロモフェノール、および0.02%キシレンシアノール)を加え、次いで95℃に設定したヒートブロックに混合物を配置2分間、その後、すぐにチューブを氷上に置きます。
- 89 mMのトリス-HCl、pHが8.8、89mMのホウ酸、および2mM EDTA 8 M尿素を含む15%変性ポリアクリルアミドゲル上にサンプル(5μl)を読み込む。基板と開裂の製品はよく後に分離されているゲルを実行している。
2.2。ゲルのイメージング
- 蛍光標識されたDNAを検出し、基板と製品のバンドを可視化することができイメージャを用いたゲルをスキャンします。
- イメージングゲルの後にバンドを定量化する。いくつかの背景切断をNaOH( 図2A)を有する基板自体の治療後に観察されてもよいことに留意されたい。各反応生成物の測定された量からのバックグラウンドを差し引く。
2.3。データ分析
- 各反応時間(t)( 図2B)に形成された生成物の量をプロットする。バースト(0、y切片)と線形定常相(V SS)の傾きの大きさを決定するために、式1を用いて生データを分析する。
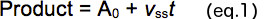
- プロット、酵素の活性画分の測定値を提供するステップとy切片総タンパク質濃度( 図2C すなわち見かけの酵素濃度)に対して。活性酵素の割合を決定するための補正係数を提供するために、ゼロ切片を持つ線形近似を使用します。
- (近似直線の傾きすなわち )製品解離速度定数を提供し、活性酵素濃度( 図2D)に定常状態速度相対をプロットします。
3。プリ定常タイムコースの測定
図2、バースト位相の間に生成物の形成に示すと、手動混合焼入れによって測定することが速すぎる。したがって、急冷流器具は、ミリ秒の時間スケール( 図3)5で発生する急激な反応を測定するための強力な方法を提供することができる。器具は、急速に混合するためにコンピュータ制御の駆動モータを使用してndは、指定された反応時間後に反応をクエンチ。例えば、初期バースト位相とOGG1によって触媒生成物形成のその後の定常位相を測定するKintek RQF -3急冷流量用音源を使用しています。急冷·フロー·インスツルメンツは、いくつかのメーカーから入手できます。
3.1。サンプルの調製
2-1で説明したように別々に1.5 mlのチューブにDNA基質とOGG1溶液を調製する。 DNAおよび活性OGG1の濃度は、それぞれ、400 nmおよび80 nMである。 1:1(容量/容量)混合した後、活性OGG1 200 nMのDNAおよび40nMの最終濃度が得られる。
3.2。急速クエンチフロー楽器の調製
- 温度制御のための迅速なクエンチフロー音源(37°C)に循環水浴を接続します。
- 機器パラメータを調整し、製造者の命令に応じて所望の時間ポイントの適切な反応ループを選択するtions。反応ループは、代替の反応時間を提供するために、可変の長さである。
- 反応バッファーを準備し、NaOHを10ミリリットルルアーロック使い捨て注射器で解決策をクエンチし、ドライブ·ポートにこれらの注射器を接続し、反応バッファー(シリンジB)および142 mMのNaOHを(シリンジQ)(LOAD位置にシリンジロードバルブ)でドライブの貯水池を読み込む。シリンジから気泡を除去するために、数回前後に溶液を働かせる。
- ステッピングモータを下げ、それが接触する注射器の上部(FIRE位置にシリンジロードバルブとサンプルのLoadバルブ)まで。
- サンプルループ、反応ループ、水とメタノールで出口ラインをフラッシュします。完全にフラッシュされた領域を(FLUSH位置にバルブ)乾燥させます。
3.3。プレ定常状態の時間経過
- 16ゲージの針を使用してかぶった1.5mlチューブの上部に穴を作る。急冷反応を収集するために出口ラインでのチューブを取り付けます。
- 希望reactioを設定キーパッドを使用してn個の時間(秒)。ステッピングモータは、一定のクエンチボリュームを維持するように、ループの音量を調整するためにバックアップされます。ドライブ·ポートに接続されている使い捨て注射器とバッファを追加することにより、ステッピングモータのプラットフォームに対してシリンジBの位置プランジャ。
- DNA基質とOGG1ソリューションを1ミリリットルルアーロック使い捨て注射器、それぞれ(LOAD位置のサンプルバルブ)記入してください。サンプルロードポートのそれぞれにDNA基質とOGG1ソリューションと注射器を取り付けた後、(LOAD位置のサンプルのLoadバルブ)は、それぞれ、DNA基質とOGG1ソリューションをサンプルループを埋める。
- FIRE位置にすべてのシリンジのロードバルブとサンプルロードバルブを設定します。キーストロークによる反応( 例えば 、 "G"を押すか、 "START"キー)を起動します。 DNA基質とOGG1ソリューション(18μlの各々)が直ちに反応ループ内で混合される。
- 反応は自動的にRの36μLを混合することにより、所望の反応時に急冷されるまで待機142 mMのNaOHを86μLでeaction混合物。クエンチング反応後、試料を出口ラインから排出される。
- LOAD位置とFLUSHの位置にサンプルのLoadバルブにシリンジロードバルブを設定します。サンプルループ、反応ループ、水とメタノールで出口ラインをフラッシュし、3.2で説明したように乾燥させます。各時点については、上記の手順を繰り返します。
- 手動または急速クエンチ楽器で行うことができますバックグラウンド補正を決定する酵素なしの対照実験を行います。急速クエンチ計器と制御を行うためには、DNA基質とのDNAのサンプルループを埋めるけどOGG1空のサンプルループを保つ。反応時間を設定して、ミックスを行い、よう3.3.4-3.3.6で説明急冷。
- 2 M NaOHを、2 M HCl、水、メタノールでサンプルループと反応ループを洗った後、洗浄したラインを乾燥させます。ホームポジションにステッピングモータを設定した後、LOADのPOSITIOにシリンジロードバルブを切り替えるn個と水でドライブ貯水池を洗う。
- その後急冷反応熱のサンプル、および2.1で説明したようにポリアクリルアミドゲル電気泳動、変性15%別基板と製品のDNAを扱う。
- 2.2で説明したようにゲル上のバンドを可視化し、定量化する。
3.4。データ分析
一定の一次速度(K obsは )、バースト(0)の振幅を設ける立ち上がり指数と線形項(式2)式に非線形回帰分析により、生成物形成の経時変化に適合し、かつ線形速度(V SS)。
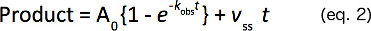
4。シングルターンオーバータイムコース
- 2-1で説明したように別の1.5mlチューブにDNA基質とOGG1を準備します。濃度DNAおよび活性OGG1のtionsは、それぞれ、100及び500nmであり、この収混合1:1(v / v)の後の50 nMのDNAおよび250 nMの最終濃度OGG1。
- 急速クエンチフロー測定器を準備し、3.2と3.3で説明したように反応サンプルを準備します。
- 熱でクエンチした反応サンプルを処理し、2.1で説明したように15%の別々の基板と、製品に変性ポリアクリルアミドゲル電気泳動にそれらの対象。
- 2.2で説明したようにゲル上のバンドを可視化し、定量化する。
- 式(3)で与えられたように(K OBS)定数一次速度を決定するために、単一の指数関数に製品形成の時間コースを合わせる。
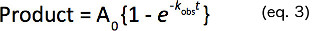
結果
定常状態動態解析は、ブラッドフォードタンパク質アッセイ2によって決定される200nMのDNAの基板とOGG1 4つの異なる見掛け濃度(15、30、45、および60 nM)を用いて行った。生成物形成の経時変化は、それぞれのタンパク質濃度( 図2B)に対して、それぞれ、2.2、11であったy切片、15、および26 nMのを決定するために、線形方程式に適合させた。 y軸切片、さらに実際の各タン?...
ディスカッション
運動アプローチは小学校速度定数を定義する方法を概説し、ここで説明。生成物形成の経時変化が急激に発生した第二相酵素の売上高がある場合、化学反応後の工程は、その後の触媒ターンオーバーの際の律速である。 OGG1の場合には、第一の売上高のいずれか(S E)DNA濃度を制限することで、高い酵素濃度を用いて測定することができる。前者の場合、反応は、 '単売?...
開示事項
著者らは、開示することは何もありません。
謝辞
我々は有益な提案や議論のための原稿とラジェンドラ·プラサドの重要な読書のための博士ジュリーK.ホートンに感謝します。本研究の一部は、もともと生物化学 、サッサA らのジャーナルに掲載された。ら 、 "ヒト8 -オキソグアニンDNAグリコシラーゼのグリコシラーゼ活性上のDNAシーケンス文脈効果。" J BIOLケム 。287、36702から36710(2012)2。この作品は、学内研究プログラムにおける保健研究プロジェクトグラントZ01-ES050158の国立研究所、NIEHSにより、全部または一部を、サポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 5’-6-FAM labeled oligonucleotides containing a single 8-oxoG | Eurofins MWG Operon | 5’-P32 radiolabeled oligonucleotides can be used as well. Polyacrylamide gel purification grade is recommended. | |
| Unlabeled oligonucleotides (complementary strand) | Eurofins MWG Operon | Polyacrylamide gel purification grade is recommended. | |
| 1 ml BD Luer Lock disposable syringe | BD Medical | 309628 | Lue Lock disposable syringe from other vendors can be used as well. |
| 10 ml BD Luer Lock disposable syringe | BE Medical | 309604 | Luer Lock disposable syringe from other vendors can be used as well. |
| Equipment | |||
| Circulating water bath | Any vender | ||
| RQF-3 Rapid Quench-Flow Instrument | KinTek Corporation | Rapid Quench-Flow Instrument from other vendors can be used as well. | |
| Typhoon Phosphorimager 8600 | GE Healthcare Life Sciences | Imager from other vendors can be used as well. | |
| KaleidaGraph | Synergy Software | ||
参考文献
- Porello, S. L., Leyes, A. E., David, S. S. Single-turnover and pre-steady-state kinetics of the reaction of the adenine glycosylase MutY with mismatch-containing DNA substrates. Biochemistry. 37, 14756-14764 (1998).
- Sassa, A., Beard, W. A., Prasad, R., Wilson, S. H. DNA sequence context effects on the glycosylase activity of human 8-oxoguanine DNA glycosylase. The Journal of Biological Chemistry. 287, 36702-36710 (2012).
- Wong, I., Lundquist, A. J., Bernards, A. S., Mosbaugh, D. W. Presteady-state analysis of a single catalytic turnover by Escherichia coli uracil-DNA glycosylase reveals a "pinch-pull-push" mechanism. The Journal of Biological Chemistry. 277, 19424-19432 (1074).
- Johnson, K. A. Advances in transient-state kinetics. Current Opinion in Biotechnology. 9, 87-89 (1998).
- Johnson, K. A. Rapid kinetic analysis of mechanochemical adenosinetriphosphatases. Methods in Enzymology. 134, 677-705 (1986).
- Kovtun, I. V., et al. OGG1 initiates age-dependent CAG trinucleotide expansion in somatic cells. Nature. 447, 447-452 (2007).
- Hill, J. W., Hazra, T. K., Izumi, T., Mitra, S. Stimulation of human 8-oxoguanine-DNA glycosylase by AP-endonuclease: potential coordination of the initial steps in base excision repair. Nucleic Acids Research. 29, 430-438 (2001).
- Zharkov, D. O., Rosenquist, T. A., Gerchman, S. E., Grollman, A. P. Substrate specificity and reaction mechanism of murine 8-oxoguanine-DNA glycosylase. The Journal of Biological Chemistry. 275, 28607-28617 (2000).
- Waters, T. R., Swann, P. F. Kinetics of the action of thymine DNA glycosylase. The Journal of Biological Chemistry. 273, 20007-20014 (1998).
- Nilsen, H., et al. Excision of deaminated cytosine from the vertebrate genome: role of the SMUG1 uracil-DNA glycosylase. The EMBO Journal. 20, 4278-4286 (2001).
- Gill, S. C., von Hippel, P. H. Calculation of protein extinction coefficients from amino acid sequence data. Analytical Biochemistry. 182, 319-326 (1989).
- Van de Berg, ., Beard, B. J., A, W., Wilson, S. H. DNA structure and aspartate 276 influence nucleotide binding to human DNA polymerase beta. Implication for the identity of the rate-limiting conformational change. The Journal of Biological Chemistry. 276, 3408-3416 (2001).
- Krishnamurthy, N., Haraguchi, K., Greenberg, M. M., David, S. S. Efficient removal of formamidopyrimidines by 8-oxoguanine glycosylases. Biochemistry. 47, 1043-1050 (2008).
- Leipold, M. D., Muller, J. G., Burrows, C. J., David, S. S. Removal of hydantoin products of 8-oxoguanine oxidation by the Escherichia coli DNA repair enzyme, FPG. Biochemistry. 39, 14984-14992 (2000).
- Zhao, X., Krishnamurthy, N., Burrows, C. J., David, S. S. Mutation versus repair: NEIL1 removal of hydantoin lesions in single-stranded, bulge, bubble, and duplex DNA contexts. Biochemistry. 49, 1658-1666 (2010).
- Leipold, M. D., Workman, H., Muller, J. G., Burrows, C. J., David, S. S. Recognition and removal of oxidized guanines in duplex DNA by the base excision repair enzymes hOGG1, yOGG1, and yOGG2. Biochemistry. 42, 11373-11381 (2003).
- Robey-Bond, S. M., Barrantes-Reynolds, R., Bond, J. P., Wallace, S. S., Bandaru, V. Clostridium acetobutylicum 8-oxoguanine DNA glycosylase (Ogg) differs from eukaryotic Oggs with respect to opposite base discrimination. Biochemistry. 47, 7626-7636 (2008).
- Jarem, D. A., Wilson, N. R., Delaney, S. Structure-dependent DNA damage and repair in a trinucleotide repeat sequence. Biochemistry. 48, 6655-6663 (2009).
- Livingston, A. L., Kundu, S., Henderson Pozzi, M., Anderson, W. D., David, S. S. Insight into the roles of tyrosine 82 and glycine 253 in the Escherichia coli adenine glycosylase MutY. Biochemistry. 44, 14179-14190 (2005).
- Kundu, S., Brinkmeyer, M. K., Livingston, A. L., David, S. S. Adenine removal activity and bacterial complementation with the human MutY homologue (MUTYH) and Y165C, G382D, P391L and Q324R variants associated with colorectal cancer. DNA Repair. 8, 1400-1410 (2009).
- Zharkov, D. O., Rosenquist, T. A. Inactivation of mammalian 8-oxoguanine-DNA glycosylase by cadmium(II): implications for cadmium genotoxicity. DNA Repair. 1, 661-670 (2002).
- Jarem, D. A., Wilson, N. R., Schermerhorn, K. M., Delaney, S. Incidence and persistence of 8-oxo-7,8-dihydroguanine within a hairpin intermediate exacerbates a toxic oxidation cycle associated with trinucleotide repeat expansion. DNA Repair. 10, 887-896 (2011).
- Mokkapati, S. K., Wiederhold, L., Hazra, T. K., Mitra, S. Stimulation of DNA glycosylase activity of OGG1 by NEIL1: functional collaboration between two human DNA glycosylases. Biochemistry. 43, 11596-11604 (2004).
- Sidorenko, V. S., Nevinsky, G. A., Zharkov, D. O. Mechanism of interaction between human 8-oxoguanine-DNA glycosylase and AP endonuclease. DNA Repair. 6, 317-328 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved