Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
L'état d'équilibre des mesures cinétiques, pré-état stationnaire, et un seul chiffre d'affaires de l'activité de ADNglycosylase
Dans cet article
Résumé
des plages de temps pour l'activité glycosylase de 8-oxoguanine ADN glycosylase sont biphasique présentant une salve de formation de produit et une phase de régime permanent linéaire. Utilisant des techniques de trempe de trésorerie, l'éclatement et les taux de régime permanent peut être mesurée, qui correspondent à l'excision de 8 oxoguanine et la libération de la glycosylase de l'ADN du produit, respectivement.
Résumé
Human 8-oxoguanine ADN glycosylase (OGG1) excise le mutagène oxydatif lésion de l'ADN 8-oxo-7 ,8-dihydroguanine (8-oxoG) à partir de l'ADN. Caractérisation cinétique des OGG1 est entrepris pour mesurer les taux d'excision 8 oxoG et version du produit. Lorsque la concentration est inférieure à OGG1 substrat d'ADN, les temps de formation de produit sont biphasique; une phase exponentielle rapide (c. salve) de formation du produit est suivie par une phase de régime permanent linéaire. La salve initial de la formation du produit correspond à la concentration de l'enzyme correctement en prise sur le substrat, et l'amplitude de rafale dépend de la concentration de l'enzyme. La constante de l'éclatement de vitesse de premier ordre correspond à la fréquence intrinsèque de l'excision 8 oxoG et le taux de régime permanent lent mesure le taux de libération du produit (Product DNA dissociation constante de vitesse, k off). Ici, nous décrivons les approches état d'équilibre, pré-état stationnaire, et un seul chiffre d'affaires d'isoler et de mesurer spétapes SPÉCIFIQUE pendant OGG1 cyclisme catalytique. A fluorescent oligonucléotide marqué lésion contenant et OGG1 purifiés sont utilisés pour faciliter les mesures cinétiques précises. Depuis faibles concentrations d'enzymes sont utilisés pour effectuer des mesures en régime permanent, le mélange des réactifs manuel et extinction de la réaction peut être effectuée pour déterminer le taux de régime permanent (k off). En outre, l'extrapolation de la vitesse de régime permanent à un point situé sur l'ordonnée à l'instant zéro indique qu'une salve de formation de produit est survenu au cours de la première rotation (c. ordonnée à l'origine est positif). La constante de temps de la phase de salve exponentiel de vitesse de premier ordre peut être mesurée à l'aide d'une technique de mélange et de refroidissement rapide, qui examine la quantité de produit formé à des intervalles de temps courts (<1 s) avant la phase de régime permanent, et correspond à la vitesse de 8 -oxoG excision (c.-à-chimie). L'étape chimique peut également être mesurée en utilisant une approche unique chiffre d'affaires où le cyclisme catalytique estempêché par l'ADN de substrat de saturation avec l'enzyme (E> S). Ces approches permettent de mesurer les constantes de vitesse élémentaires qui influencent l'efficacité d'élimination d'une lésion de l'ADN.
Introduction
Un milieu aérobie accélère l'instabilité génomique. Une importante lésion de l'ADN promutagène résultant du stress oxydatif est la 7,8-dihydro-8-oxoguanine (8 oxoG). Cela est dû au potentiel de codage ambigu de 8 oxoG. Human 8 oxoguanine ADN glycosylase (OGG1) est chargé d'initier la réparation par excision de base de 8 oxoG. L'activité glycosylase de OGG1 excise la base de 8 oxoG résultant dans l'ADN de produit avec un site apurinique (AP-site). Une activité de lyase faible de OGG1 peut inciser le site de AP dans certains cas.
Caractérisation cinétique des ADN glycosylases trouve généralement qu'ils présentent des cours à temps biphasique. Après une phase initiale rapide de la formation du produit (c.-à-burst), une phase de régime permanent linéaire est observée 3.1. Ce comportement est révélateur d'une étape à la suite chimie (ie changement de conformation ou la libération du produit) étant de limitation de vitesse au cours de la partie linéaire de l'évolution dans le temps, alors que la fraiseère phase, souvent désigné comme la phase transitoire, correspond à la formation du produit sur le site actif de l'enzyme au cours du premier cycle de la réaction. Lorsque la libération du produit est la limitation du débit au cours de la phase stationnaire, mesures d'activité fournissent une mesure qualitative du produit ADN affinité de liaison, mais ne fournissent pas d'informations concernant la cinétique des événements sur le site actif de l'enzyme (c.-à-chimie). En conséquence, les méthodes d'isoler et de mesurer la phase de la salve de pré-état stationnaire exponentielle sont nécessaires pour sonder événements au cours de la première rotation enzymatique au niveau du site actif 4 de l'enzyme.
Il existe trois approches cinétiques classiques pour caractériser le comportement catalytique de OGG1, (1) l'état d'équilibre, (2) pré-état stationnaire, et (3) un seul chiffre d'affaires. Ces approches se distinguent les unes des autres par la concentration d'enzyme dans le milieu réactionnel et le rapport enzyme à substrat utilisé dans chaque approche. Dans une approche typique d'équilibre,parfois appelé plusieurs cinétique de rotation, de faibles concentrations d'enzyme sont utilisés pour suivre la formation du produit. La concentration du substrat dépasse largement la concentration de l'enzyme de sorte que plusieurs revirements enzymatiques n'affectent pas significativement la concentration du substrat. Dans cette situation, les temps devraient être linéaire et il est souvent difficile de discerner si une explosion a eu lieu au cours du premier chiffre d'affaires en raison de la faible concentration d'enzyme utilisée dans cette approche; noter que l'amplitude de la rafale est équivalente à la concentration de l'enzyme. Cela peut être surmonté en utilisant une concentration de l'enzyme supérieur et en extrapolant l'évolution temporelle linéaire au temps zéro pour détecter si le premier chiffre d'affaires s'est rapidement enzymatique. L'interception sur l'axe des ordonnées (axe des y) doit être proportionnelle à la concentration de l'enzyme et fournit une mesure de l'enzyme activement engagé avec le substrat. Bien que cette approche ne peut en principe fournir des preuves de l'existence d'une phase de la salve, une difrents approche est nécessaire pour mesurer la cinétique de la phase de la salve. Dans de nombreux cas, la phase d'éclatement est trop rapide pour mesurer par des techniques de mélange et d'extinction manuelles. Dans cette situation, cinétique pré-état stable et unique chiffre d'affaires (c.-à-cinétique transitoire) approches nécessitent souvent un instrument de mélange rapide et la trempe de suivre les points de temps au début d'une réaction 5. Dans une approche pré-état d'équilibre des concentrations élevées d'enzyme sont utilisés de telle sorte qu'une quantité importante de produit est formé au cours de la première rotation. Depuis plusieurs revirements sont suivis pour observer cyclisme catalytique (ie la phase linéaire qui suit l'éclatement), la concentration en substrat est supérieure à la concentration de l'enzyme ([enzyme] <[substrat]). Pour isoler des événements sur le site actif de l'enzyme sans vélo catalytique, conditions unique chiffre d'affaires sont utilisés. Dans ce cas, le substrat est saturé avec l'enzyme (E S >>) de telle sorte que la totalité du substrat participeront in le 'chiffre d'affaires unique »et présenteront typiquement un cours à temps simple exponentielle.
Comme indiqué ci-dessus pour les enzymes qui présentent une phase d'éclatement, la libération du produit (k off) limite souvent le taux de la phase stationnaire de l'évolution dans le temps. Le taux de libération du produit (. / Heure V SS, conc) peut être déterminée à partir de la pente de la phase stationnaire linéaire. La concentration d'enzyme active (E) est nécessaire pour convertir le taux de libération du produit à un rythme intrinsèque constante k off = v ss / [E]. Fait important, la concentration de l'enzyme active est généralement inférieure à la concentration mesurée en protéines due aux impuretés, enzyme inactive, enzyme non-productive liés au substrat, et la méthode utilisée pour déterminer la concentration en protéines. La concentration de l'enzyme active puisse être déterminée à partir de l'amplitude de salve lorsque la libération du produit est lente. Ainsi, en extrapolant à un cours à temps l'état d'équilibre àtemps zéro fournit une estimation de l'enzyme active nécessaire pour calculer k off (sortie du produit) du taux d'équilibre observé.
Pour mesurer la cinétique de l'explosion, une approche pré-état d'équilibre est nécessaire de suivre la formation du produit au cours du premier chiffre d'affaires qui se produit avant la phase stationnaire linéaire. La cinétique d'éclatement suit la formation de l'enzyme produit intermédiaire. Une fois que la réaction est initiée par l'enzyme de mélange avec le substrat, la quantité de l'enzyme produit augmente rapidement jusqu'à ce que la réaction atteigne une phase de régime permanent. Si la catalyse est beaucoup plus rapide que la libération du produit, l'amplitude de la rafale est égale à enzyme engagé activement et de l'approche exponentielle observée à l'équilibre (k obs) correspond à la vitesse de conversion chimique du substrat en produit, en supposant que la vitesse inverse de chimie est négligeable.
Dans certains cas, cy catalytiques'accrocher interfère avec une analyse pré-état stationnaire, comme lorsque l'ampleur des taux de la chimie et de la libération du produit ne sont pas significativement différentes. Dans ce cas, employant excès relatif de l'enzyme de substrat empêche les cycles catalytiques et les limites substrat lié avec l'enzyme pour un chiffre d'affaires unique. Par conséquent, la première étape chimique de la réaction peut être isolé et déterminé avec précision que la constante de vitesse de premier ordre (k obs). Cette constante de vitesse devrait être similaire à kobs déterminée à partir de l'approche pré-état stationnaire décrit ci-dessus.
Ici, nous décrivons comment ces approches cinétiques peuvent être utilisés pour analyser l'activité glycosylase de OGG1.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparation de l'enzyme et substrat d'ADN
- Surexpriment OGG1 comme une protéine de fusion GST dans E. coli, utilisent la GST-tag pour la purification, puis supprimer la TPS-tag par clivage avec HRV-3C protéase (Figure 1) 6.
- Achetez la synthèse chimique 5'-6-carboxyfluorescéine (6-FAM) oligonucléotide marqué contenant un seul résidu de 8 oxoG et son brin complémentaire d'oligonucléotides non marqués. Les oligonucléotides 34-mères contiennent un 8-oxoG à la position 17 à partir de l'extrémité 5 '. Purifier ces oligonucléotides par électrophorèse sur gel de polyacrylamide.
- Préparer substrat d'ADN bicaténaire contenant 8-oxoG par mélange de 5'-6-FAM oligonucléotide marqué avec son brin complémentaire dans un rapport molaire de 1:1,2 à tampon d'hybridation (tampon Tris-HCl, pH 7,5, NaCl 10 mM, 50, 1 mM EDTA) dans un tube à centrifuger de 1,5 ml. Placer le tube dans un flotteur dans l'eau bouillante pendant 5 min. Laisser le tube en place, puis de laisser l'eau refroidir lentement à rtempérature oom (cela prend environ 2 heures).
- Effectuer la préparation d'échantillon et de l'expérience dans des conditions de lumière faible ou minimal afin de minimiser photoblanchiment du marqueur fluorescent.
2. Mesure de la Course de l'heure Steady-State et Active site titrage de OGG1
2.1. La préparation des échantillons et évolution dans le temps l'état d'équilibre
- Préparer substrat d'ADN et Ogg1 solutions séparément dans un tampon de réaction (50 mM HEPES, pH 7,5, 20 mM KCl, 0,5 mM EDTA et 0,1% de sérum albumine bovine) dans des tubes de 1,5 ml microtubes sur la glace. La concentration en ADN est de 400 nM et la concentration apparente de OGG1 est de 30, 60, 90, ou 120 nm comme déterminé par un dosage des protéines de Bradford. Placer les tubes de réaction dans un ensemble de blocs de la chaleur à 37 ° C. Enzyme pré-incuber des solutions de substrat d'ADN séparément à 37 ° C pendant 1 min.
- Démarrer la réaction en mélangeant des volumes égaux des solutions de substrat Ogg1 et de l'ADN par pipetage. Après avoir mélangé ces solutionstions 1:1 (v / v), les concentrations finales sont 200 ADN nM et 15, 30, 45, ou 60 nM OGG1, respectivement. Retirer aliquotes (10 pi) à des intervalles de temps et stopper la réaction en mélangeant avec 1 pi de 1 M NaOH. Comme la commande pour les cours à temps partiel, 10 pl du mélange de réaction sans enzyme est mélangée avec 1 pi de 1 M NaOH.
- Placer les échantillons de réaction dans un bloc ° de chaleur 90 ° C pendant 5 min à cliver le produit résultant AP-site. Après le chauffage, ajouter 1 l de HCl 1 M pour neutraliser chaque échantillon.
- Ajouter un volume égal (12 pi) de tampon de chargement de gel (95% de formamide, 20 mM EDTA, 0,02% bromophénol et 0,02% cyanol de xylène) à chaque échantillon de réaction, puis placez le mélange dans un bloc chauffant réglé à 95 ° C pendant 2 min, puis placez immédiatement le tube sur la glace.
- Charger les échantillons (5 pi) sur un gel de polyacrylamide dénaturant à 15% contenant 8 M d'urée dans 89 mM Tris-HCl, pH 8,8, acide borique 89 mM et 2 mM EDTA. Le substrat et les produits clivés sont bien séparés aprèsexécuter le gel.
2.2. L'imagerie du gel
- Scannez le gel en utilisant un imageur qui permet de détecter l'ADN marqué par fluorescence et de visualiser le substrat et les bandes de produit.
- Quantifier les bandes après l'imagerie du gel. Notez que certains clivage de fond peut être observée après traitement du substrat lui-même avec NaOH (figure 2A). Soustraire ce contexte de la quantité mesurée de chaque produit de la réaction.
2.3. L'analyse des données
- Tracer la quantité du produit formé à chaque temps de réaction (t) (Figure 2B). Analyser les données brutes à l'aide de l'équation 1 pour déterminer l'amplitude de la salve (A 0, ordonnée à l'origine) et de la pente de la phase de régime permanent linéaire (V SS).
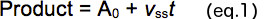
- Terrainl'ordonnée à l'origine par rapport à la concentration totale de protéine (par exemple une concentration de l'enzyme apparent, la figure 2C), fournissant une mesure de la fraction active de l'enzyme. Utilisation d'un ajustement linéaire avec une ordonnée à zéro pour fournir le facteur de correction pour la détermination de la fraction d'enzyme active.
- Tracer la vitesse de régime permanent par rapport à la concentration en enzyme active (figure 2D), fournissant la vitesse de dissociation de produit constante (soit la pente de la droite de régression).
3. Mesure de la Course de temps pré-état stationnaire
Comme le montre la figure 2, la formation du produit au cours de la phase de la salve est trop rapide pour mesurer par mélange manuel et de trempe. Par conséquent, un instrument trempe écoulement rapide peut fournir une méthode puissante pour mesurer les réactions rapides qui se produisent à l'échelle de la milliseconde (Figure 3) 5. L'appareil utilise un moteur d'entraînement commandé par ordinateur pour mélanger rapidement unnd étancher réactions après le temps de réaction spécifiées. Par exemple, utiliser un RQF-3 Rapid Instrument Quench-Flow Kintek pour mesurer la phase de la salve initiale et ultérieure phase de l'état d'équilibre de formation du produit catalysée par OGG1. Instruments Quench-écoulement rapide sont disponibles auprès de plusieurs fabricants.
3.1. Préparation de l'échantillon
Préparez séparément substrat d'ADN et Ogg1 solutions dans des tubes de 1,5 ml comme décrit dans 2-1. Les concentrations d'ADN et OGG1 actifs sont 400 nm et 80 nm, respectivement. Cela se traduit par des concentrations finales de l'ADN de 200 nM et 40 nM de Ogg1 actif après mélange 1:1 (v / v).
3.2. Préparation de l 'instrument de trempe à écoulement rapide
- Connexion d'un bain d'eau en circulation à l'instrument de trempe à écoulement rapide pour le contrôle de la température (37 ° C).
- Réglez les paramètres de l'appareil et sélectionner la boucle de réaction appropriée pour le temps désiré points selon le instructions du fabricanttions. Les boucles de réaction sont de longueurs variables pour fournir d'autres durées de réaction.
- Préparer le tampon de réaction et la soude étancher solutions dans 10 ml luer lock seringues jetables et attacher ces seringues aux ports d'unité et de charger les réservoirs d'entraînement avec un tampon de réaction (seringues B) et 142 mM NaOH (seringue Q) (seringues charge Vannes en position de chargement) . Pour éliminer les bulles d'air de la seringue, fonctionnent de la solution d'avant en arrière plusieurs fois.
- Abaisser le moteur pas à pas jusqu'à ce qu'il touche le haut de la seringue (vannes de charge de seringues et vannes de charge de l'échantillon dans la position de FEU).
- Rincer les boucles d'échantillonnage, la boucle de réaction et la sortie ligne avec de l'eau et du méthanol. Sécher les zones rougies complètement (soupapes en position affleurante).
3.3. Évolution dans le temps de pré-état stationnaire
- Faire un trou dans la partie supérieure d'un tube fermé de 1,5 ml à l'aide d'une aiguille de calibre 16. Fixer un tube à la sortie ligne pour recueillir la réaction trempé.
- Réglez le reactio désirén temps (en secondes) à l'aide du clavier. Le Stepper Motor va sauvegarder pour ajuster le volume de la boucle de manière à maintenir le volume de refroidissement constant. plongeurs de position pour seringue B contre la plate-forme du moteur pas à pas par addition de tampon avec les seringues jetables connectés aux ports d'entraînement.
- Remplir 1 ml luer lock seringues jetables avec substrat d'ADN et Ogg1 solutions, respectivement (valves de l'échantillon en position de charge). Fixez seringues avec le substrat de l'ADN et Ogg1 solutions à chacun des ports de charge de l'échantillon, puis remplissez les boucles d'échantillon avec un substrat d'ADN et Ogg1 solutions, respectivement (exemples charge Vannes en position de charge).
- Réglez tous Charger Vannes seringue et vannes de charge de l'échantillon à la position de FEU. Déclencher la réaction par un coup de clavier (par exemple, appuyez sur la touche "START" "G" ou). substrat d'ADN et Ogg1 solutions (18 pi chacun) sont immédiatement mélangés dans la boucle de réaction.
- Attendez jusqu'à ce que la réaction est automatiquement désactivé au moment de réaction souhaitée en mélangeant 36 pl de la rmélange eaction avec 86 pl de 142 mM NaOH. Après arrêt de la réaction, l'échantillon est déchargé de la sortie de ligne.
- Régler la charge Vannes seringue à la position de la charge et les exemples charge Vannes à la position affleurante. Rincer les boucles d'échantillonnage, la boucle de réaction et la sortie ligne avec de l'eau et du méthanol et sécher comme décrit au paragraphe 3.2. Répétez la procédure ci-dessus pour chaque point de temps.
- Effectuer un essai témoin sans enzyme pour déterminer une correction de fond, qui peut être fait manuellement ou avec l'instrument rapide trempe. Pour effectuer un contrôle avec l'instrument rapide trempe, remplissez la boucle d'échantillonnage de l'ADN avec le substrat d'ADN, mais garder la boucle d'échantillonnage pour OGG1 vide. Réglez le temps de réaction, effectuer le mélange et trempe tel que décrit dans 3.3.4-3.3.6.
- Laver les boucles d'échantillonnage et la boucle de réaction avec NaOH 2 M, 2 M HCl, de l'eau et du méthanol, puis sécher les lignes lavés. Après avoir réglé le moteur pas à pas à la position d'origine, commutation de la charge Vannes seringue à la positio LOADn et laver les réservoirs d'entraînement avec l'eau.
- Traiter les échantillons trempés de réaction avec la chaleur, et ensuite substrat séparé et de l'ADN du produit de 15% par électrophorèse dénaturante en gel de polyacrylamide, comme décrit en 2.1.
- Visualiser et de quantifier les bandes sur le gel comme décrit au paragraphe 2.2.
3.4. L'analyse des données
Monter les durées de la formation du produit par l'analyse de régression non linéaire à une équation à une hausse des termes exponentiels et linéaires (équation 2) qui fournissent la constante de vitesse de premier ordre (Kobs), l'amplitude de la salve (A 0), et une vitesse linéaire (V SS).
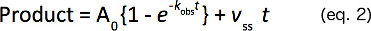
4. Cours de temps seul chiffre d'affaires
- Préparer substrat d'ADN et OGG1 dans des tubes de 1,5 ml séparés comme décrit dans 2-1. La concentrationtion de l'ADN et OGG1 actifs sont 100 nm et 500 nm, respectivement, ce qui donne des concentrations finales de 50 d'ADN nM et 250 nM OGG1 après mélange 1:1 (v / v).
- Préparation de l'instrument trempe écoulement rapide et préparer les échantillons de réaction comme décrit dans 3.2 et 3.3.
- Traiter les échantillons de réaction stoppée avec la chaleur et les soumettent à 15% électrophorèse sur gel dénaturant de polyacrylamide de substrat et de produits séparé comme décrit au paragraphe 2.1.
- Visualiser et de quantifier les bandes sur le gel comme décrit au paragraphe 2.2.
- Monter les durées de formation du produit à une seule exponentielle pour déterminer la constante de vitesse de premier ordre (k obs) comme indiqué dans l'équation 3.
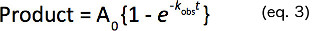
Access restricted. Please log in or start a trial to view this content.
Résultats
Analyse cinétique état d'équilibre a été effectuée en utilisant 200 substrat d'ADN nM et quatre concentrations apparentes différentes de OGG1 (15, 30, 45, et 60 nM) telle que déterminée par un dosage de protéines Bradford 2. Les cours du temps de la formation du produit ont été ajustées à une équation linéaire pour déterminer l'ordonnée à l'origine, qui étaient de 2,2, 11, 15 et 26 nm, respectivement, par rapport à chaque concentration de la protéine (figure...
Access restricted. Please log in or start a trial to view this content.
Discussion
Les approches cinétiques décrites ici un aperçu des méthodes pour définir des constantes cinétiques élémentaires. Si une évolution dans le temps de formation du produit est biphasique avec le premier chiffre d'affaires enzymatique produisant rapidement, puis une étape après la chimie est limitante pendant revirements catalytiques ultérieurs. Dans le cas de OGG1, le premier chiffre peut être mesurée à l'aide de fortes concentrations d'enzymes avec soit de limitation (S E)...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous remercions le Dr Julie K. Horton pour la lecture critique du manuscrit et le Dr Rajendra Prasad des suggestions utiles et des discussions. Des portions de cette recherche ont été publiés à l'origine dans The Journal of Biological Chemistry, Sassa A et. al., "séquence d'ADN effets de contexte sur l'activité de l'homme Glycosylase ADNglycosylase 8 oxoguanine." J Biol Chem. 287, 36702-36710 (2012) 2. Ce travail a été financé, en tout ou en partie, par les National Institutes of Health Research Project Grant Z01-ES050158 dans le programme de recherche intra-muros, NIEHS.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 5’-6-FAM labeled oligonucleotides containing a single 8-oxoG | Eurofins MWG Operon | 5’-P32 radiolabeled oligonucleotides can be used as well. Polyacrylamide gel purification grade is recommended. | |
| Unlabeled oligonucleotides (complementary strand) | Eurofins MWG Operon | Polyacrylamide gel purification grade is recommended. | |
| 1 ml BD Luer Lock disposable syringe | BD Medical | 309628 | Lue Lock disposable syringe from other vendors can be used as well. |
| 10 ml BD Luer Lock disposable syringe | BE Medical | 309604 | Luer Lock disposable syringe from other vendors can be used as well. |
| Equipment | |||
| Circulating water bath | Any vender | ||
| RQF-3 Rapid Quench-Flow Instrument | KinTek Corporation | Rapid Quench-Flow Instrument from other vendors can be used as well. | |
| Typhoon Phosphorimager 8600 | GE Healthcare Life Sciences | Imager from other vendors can be used as well. | |
| KaleidaGraph | Synergy Software | ||
Références
- Porello, S. L., Leyes, A. E., David, S. S. Single-turnover and pre-steady-state kinetics of the reaction of the adenine glycosylase MutY with mismatch-containing DNA substrates. Biochemistry. 37, 14756-14764 (1998).
- Sassa, A., Beard, W. A., Prasad, R., Wilson, S. H. DNA sequence context effects on the glycosylase activity of human 8-oxoguanine DNA glycosylase. The Journal of Biological Chemistry. 287, 36702-36710 (2012).
- Wong, I., Lundquist, A. J., Bernards, A. S., Mosbaugh, D. W. Presteady-state analysis of a single catalytic turnover by Escherichia coli uracil-DNA glycosylase reveals a "pinch-pull-push" mechanism. The Journal of Biological Chemistry. 277, 19424-19432 (1074).
- Johnson, K. A. Advances in transient-state kinetics. Current Opinion in Biotechnology. 9, 87-89 (1998).
- Johnson, K. A. Rapid kinetic analysis of mechanochemical adenosinetriphosphatases. Methods in Enzymology. 134, 677-705 (1986).
- Kovtun, I. V., et al. OGG1 initiates age-dependent CAG trinucleotide expansion in somatic cells. Nature. 447, 447-452 (2007).
- Hill, J. W., Hazra, T. K., Izumi, T., Mitra, S. Stimulation of human 8-oxoguanine-DNA glycosylase by AP-endonuclease: potential coordination of the initial steps in base excision repair. Nucleic Acids Research. 29, 430-438 (2001).
- Zharkov, D. O., Rosenquist, T. A., Gerchman, S. E., Grollman, A. P. Substrate specificity and reaction mechanism of murine 8-oxoguanine-DNA glycosylase. The Journal of Biological Chemistry. 275, 28607-28617 (2000).
- Waters, T. R., Swann, P. F. Kinetics of the action of thymine DNA glycosylase. The Journal of Biological Chemistry. 273, 20007-20014 (1998).
- Nilsen, H., et al. Excision of deaminated cytosine from the vertebrate genome: role of the SMUG1 uracil-DNA glycosylase. The EMBO Journal. 20, 4278-4286 (2001).
- Gill, S. C., von Hippel, P. H. Calculation of protein extinction coefficients from amino acid sequence data. Analytical Biochemistry. 182, 319-326 (1989).
- Van de Berg,, Beard, B. J., A, W., Wilson, S. H. DNA structure and aspartate 276 influence nucleotide binding to human DNA polymerase beta. Implication for the identity of the rate-limiting conformational change. The Journal of Biological Chemistry. 276, 3408-3416 (2001).
- Krishnamurthy, N., Haraguchi, K., Greenberg, M. M., David, S. S. Efficient removal of formamidopyrimidines by 8-oxoguanine glycosylases. Biochemistry. 47, 1043-1050 (2008).
- Leipold, M. D., Muller, J. G., Burrows, C. J., David, S. S. Removal of hydantoin products of 8-oxoguanine oxidation by the Escherichia coli DNA repair enzyme, FPG. Biochemistry. 39, 14984-14992 (2000).
- Zhao, X., Krishnamurthy, N., Burrows, C. J., David, S. S. Mutation versus repair: NEIL1 removal of hydantoin lesions in single-stranded, bulge, bubble, and duplex DNA contexts. Biochemistry. 49, 1658-1666 (2010).
- Leipold, M. D., Workman, H., Muller, J. G., Burrows, C. J., David, S. S. Recognition and removal of oxidized guanines in duplex DNA by the base excision repair enzymes hOGG1, yOGG1, and yOGG2. Biochemistry. 42, 11373-11381 (2003).
- Robey-Bond, S. M., Barrantes-Reynolds, R., Bond, J. P., Wallace, S. S., Bandaru, V. Clostridium acetobutylicum 8-oxoguanine DNA glycosylase (Ogg) differs from eukaryotic Oggs with respect to opposite base discrimination. Biochemistry. 47, 7626-7636 (2008).
- Jarem, D. A., Wilson, N. R., Delaney, S. Structure-dependent DNA damage and repair in a trinucleotide repeat sequence. Biochemistry. 48, 6655-6663 (2009).
- Livingston, A. L., Kundu, S., Henderson Pozzi, M., Anderson, W. D., David, S. S. Insight into the roles of tyrosine 82 and glycine 253 in the Escherichia coli adenine glycosylase MutY. Biochemistry. 44, 14179-14190 (2005).
- Kundu, S., Brinkmeyer, M. K., Livingston, A. L., David, S. S. Adenine removal activity and bacterial complementation with the human MutY homologue (MUTYH) and Y165C, G382D, P391L and Q324R variants associated with colorectal cancer. DNA Repair. 8, 1400-1410 (2009).
- Zharkov, D. O., Rosenquist, T. A. Inactivation of mammalian 8-oxoguanine-DNA glycosylase by cadmium(II): implications for cadmium genotoxicity. DNA Repair. 1, 661-670 (2002).
- Jarem, D. A., Wilson, N. R., Schermerhorn, K. M., Delaney, S. Incidence and persistence of 8-oxo-7,8-dihydroguanine within a hairpin intermediate exacerbates a toxic oxidation cycle associated with trinucleotide repeat expansion. DNA Repair. 10, 887-896 (2011).
- Mokkapati, S. K., Wiederhold, L., Hazra, T. K., Mitra, S. Stimulation of DNA glycosylase activity of OGG1 by NEIL1: functional collaboration between two human DNA glycosylases. Biochemistry. 43, 11596-11604 (2004).
- Sidorenko, V. S., Nevinsky, G. A., Zharkov, D. O. Mechanism of interaction between human 8-oxoguanine-DNA glycosylase and AP endonuclease. DNA Repair. 6, 317-328 (2007).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon