Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Steady-State-, Pre-Steady-State-und Einzel-Umsatz Kinetic Measurement für DNA Glycosylase Aktivität
In diesem Artikel
Zusammenfassung
Zeit Kurse für die glycosylase Aktivität von 8-oxoguanine DNA glycosylase sind zweiphasige ausstellenden ein Platzen der Produkt-Bildung und einer linearen steady-state-Phase. Verwendung Quench-Flow-Techniken, die platzen und die Steady-State-Raten gemessen werden können, die Exzision von 8-oxoguanine und Freisetzung des glycosylase DNA aus dem Produkt entsprechen.
Zusammenfassung
Menschliche 8-Oxoguanin-DNA-Glycosylase (OGG1) schneidet das mutagene oxidative DNA Läsion 8-oxo-7 ,8-dihydroguanine (8-oxoG) von DNA. Kinetische Charakterisierung OGG1 vorgenommen wird, um die Raten von 8-oxoG Exzision und Produkt-Release zu messen. Wenn die Konzentration niedriger als OGG1 Substrat-DNA ist, sind Zeitverläufe der Produktbildung zweiphasige, eine schnelle exponentielle Phase (dh Burst) der Produktbildung durch einen linearen stationären Phase. Anfängliches Platzen der Produktbildung entspricht der Konzentration des Enzyms auf das Substrat ordnungsgemäß in Eingriff, und der Burst-Amplitude abhängig von der Konzentration des Enzyms. Die ersten Ordnung Geschwindigkeitskonstante des Bursts entspricht der intrinsischen Rate von 8-oxoG Exzision und die langsamere steady-state Inflationsrate misst die Rate der Freisetzung (Produkt DNA Dissoziationsgeschwindigkeitskonstante, k off). Hier beschreiben wir Steady-State-, pre-steady-state und Single-Turnover-Ansätze zu isolieren und zu messen specific Schritte während OGG1 katalytische Radfahren. A fluoreszenzmarkierte Läsion enthaltenden Oligonukleotid und gereinigt OGG1 werden verwendet, um präzise kinetische Messungen zu erleichtern. Seit niedrig Enzymkonzentrationen verwendet werden, um steady-state Messungen, manuelles Mischen von Reagenzien und Abschrecken der Reaktion machen kann durchgeführt werden, um die steady-state (k off) zu ermitteln. Zusätzlich zeigt Extrapolation der stabilen Frequenz bis zu einem Punkt auf der Ordinate auf Null Mal, dass ein Platzen der Produktbildung in der ersten Umsatz (dh der y-Achse positiv ist) eingetreten ist. Die ersten Ordnung Geschwindigkeitskonstante der exponentiellen Burst-Phase kann unter Verwendung eines schnellen Vermischung und Abschrecken Technik, die die Menge des Produkts, in kurzen Zeitabständen (<1 s) vor der stationären Phase gebildet untersucht und entspricht der Höhe von 8 werden oxoG-Exzision (dh Chemie). Die chemische Schritt kann auch unter Verwendung eines Single-Turnover-Ansatz, wo katalytische Radfahren istverhindert durch Sättigen Substrat-DNA mit dem Enzym (E> S). Diese Ansätze lassen sich messen elementare Geschwindigkeitskonstanten, die die Effizienz der Entfernung von einer DNA-Läsion beeinflussen.
Einleitung
Einer aeroben Umgebung beeilt genomische Instabilität. Ein wesentlicher promutagene DNA Läsion aus oxidativem Stress ist 7,8-dihydro-8-oxoguanine (8-oxoG). Dies ist aufgrund der mehrdeutigen Codierung Potential 8-oxoG. Menschliche 8-oxoguanine DNA glycosylase (OGG1) ist verantwortlich für die Initiierung Basenexzisionsreparatur von 8-oxoG. Die glycosylase Aktivität OGG1 schneidet das 8-oxoG Basis resultierende Produkt in DNA mit einer Apurin Website (AP-site). Eine schwache Lyaseaktivität von OGG1 können die AP-Website in einigen Fällen einzuschneiden.
Kinetische Charakterisierung von DNA Glycosylasen allgemein fest, dass sie zweiphasige zeitliche Verläufe aufweisen. Nach einer anfänglichen schnellen Phase der Produktbildung (dh Burst) wird eine lineare stationären Phase beobachtet 1-3. Dieses Verhalten ist, das einen Schritt nach Chemie (dh Konformationsänderung oder Produkt release) ist geschwindigkeitsbestimmend während des linearen Teils der Zeitverlauf, während der Bohrerst Phase, die oft als die transiente Phase bezeichnet, entspricht die Produktbildung in der aktiven Stelle des Enzyms während des ersten Zyklus der Reaktion. Bei Produkt-Release Rate Limiting während der Steady-State-Phase ist, bieten Aktivitätsmessungen eine qualitative Messung der Produkt-DNA Bindungsaffinität, aber keine kinetische Informationen über Veranstaltungen im aktiven Zentrum des Enzyms (dh Chemie). Dementsprechend sind Methoden zu isolieren und messen die exponentielle pre-steady-state Burst-Phase benötigt, um Ereignisse während der ersten enzymatischen Umsatz an das Enzym aktive Zentrum 4 sondieren.
Es gibt drei Standard kinetische Ansätze, um die katalytische Verhalten OGG1 charakterisieren, (1) Steady-State, (2) pre-steady-state, und (3) Single-Turnover. Diese Ansätze unterscheiden sich durch die Konzentration des Enzyms in dem Reaktionsgemisch und dem Enzym zu Substrat-Verhältnis in jedem Ansatz verwendet. In einem typischen Steady-State-Ansatz,manchmal als mehrere Umsatzkinetik bezeichnet werden, sind geringe Konzentrationen von Enzym zur Bildung Produkt folgen. Die Substratkonzentration übersteigt bei weitem die Enzymkonzentration so dass mehrere enzymatische Umsätze nicht wesentlich beeinträchtigen Substratkonzentration. In dieser Situation sollte Zeitverläufe linear sein, und es ist oft schwierig zu erkennen, ob ein Burst in der ersten Umsatz aufgrund der geringen Enzymkonzentration in diesem Ansatz trat; beachten Burstamplitude entspricht der Enzymkonzentration. Dies kann durch Verwendung einer höheren Enzymkonzentration und Extrapolieren der linearen Zeitverlauf auf den Zeitpunkt Null zu erfassen, ob die erste enzymatischen Umsetzung schnell aufgetreten überwunden werden. Der Achsenabschnitt auf der Ordinate (y-Achse) sollte proportional zur Enzymkonzentration und stellt ein Maß für das Enzym aktiv mit dem Substrat in Eingriff. Obwohl dieser Ansatz im Prinzip den Nachweis für die Existenz eines Burst-Phase, und ein DIF-schiedenen Ansatz erforderlich, um die Kinetik der Burst-Phase messen. In vielen Fällen ist der Burst-Phase ist zu schnell, um durch manuelles Mischen und Abschrecken Techniken messen. In dieser Situation nähert sich pre-steady-state und Single-Turnover-kinetische (dh transient kinetische) erfordern oft einen schnellen-mixing und Abschrecken Instrument zu frühen Zeitpunkten einer Reaktion 5 folgen. In einer vorher stationären Ansatz hohe Konzentrationen des Enzyms, so dass eine erhebliche Menge an Produkt in der ersten Umsatzes gebildet werden. Da mehrere Umsätze gefolgt sind katalytische Radfahren (dh die lineare Phase, die die Burst folgt) zu beobachten, ist Substratkonzentration größer als die Enzymkonzentration ([Enzym] <[Substrat]). Um Ereignisse im aktiven Zentrum des Enzyms zu isolieren ohne Katalysator Radfahren, sind Single-Turnover-Bedingungen verwendet. In diesem Fall wird das Substrat mit dem Enzym gesättigt (E >> S), so dass alle von dem Substrat wird i teilnehmenn der "einzigen Umsatz" und wird typischerweise eine Single-exponentiellen Zeitverlauf.
Wie oben für Enzyme, die eine Burst-Phase, Produkt-Release (k off) weisen darauf hingewiesen, begrenzt oft die Geschwindigkeit der Steady-State-Phase des zeitlichen Verlaufs. Die Rate der Freisetzung (v ss, Konz. / Zeit) aus der Steigung der linearen steady-state-Phase bestimmt werden. Die aktive Enzymkonzentration (E) benötigt wird, um die Rate der Produkt-Release einer intrinsischen Geschwindigkeitskonstante konvertieren wo k off = v ss / [E]. Wichtig ist, dass das aktive Enzym-Konzentration in der Regel niedriger als der gemessene Proteinkonzentration durch Verunreinigungen, inaktives Enzym, Enzym nicht-produktiv Substrat gebunden, und die Methode zur Protein-Konzentration bestimmen. Die aktive Enzymkonzentration kann aus dem Burstamplitude bestimmt werden, wenn Produkt-Release ist langsam. So Extrapolation eine steady-state zeitlichen Verlauf zuNull Zeit liefert eine Schätzung der aktiven Enzym benötigt, um k off (Produktversion) aus dem beobachteten steady-state zu berechnen.
Um die Kinetik des Bursts messen, ist ein Pre-Steady-State-Ansatz notwendig, um Produkt-Bildung während der ersten Umsätze, die vor dem linearen stationären Phase tritt folgen. Die Burst-Kinetik folgt die Bildung von Enzym-Zwischenprodukts. Sobald die Reaktion durch Mischen von Enzym mit dem Substrat eingeleitet wird, die Menge an Enzym-Produkt schnell erhöht, bis die Reaktion einen stationären Zustand erreicht Phase. Wenn Katalyse ist viel schneller als Produkt-Release ist die Amplitude des Bursts gleich aktiv engagiert Enzym und dem beobachteten exponentiellen Annäherung an das Gleichgewicht (k obs) entspricht der Geschwindigkeit der chemischen Umwandlung von Substrat zu Produkt, vorausgesetzt, dass das Gegenteil von Chemie vernachlässigbar.
In einigen Fällen katalytischen cyklammern stört mit einem pre-steady-state-Analyse, z. B. wenn die Größen der Preise für Chemie und Produkt-Release sind nicht signifikant unterschiedlich. In diesem Fall, unter Verwendung überschüssigen Enzym relativ zu dem Substrat verhindert katalytische Rad-und Grenzwerte Substrat mit dem Enzym in einem Umsatz gebunden. Dementsprechend kann die erste chemische Schritt der Reaktion isoliert und genau bestimmt als die Geschwindigkeitskonstante erster Ordnung (k obs). Diese Geschwindigkeitskonstante sollte ähnlich k obs ermittelt aus der pre-steady-state oben beschriebenen Ansatz.
Hier beschreiben wir, wie diese kinetische Ansätze verwendet werden, um die Aktivität von glycosylase OGG1 analysieren.
Protokoll
1. Herstellung des Enzyms DNA-Substrat und
- Over-express OGG1 als GST-Fusionsprotein in E. coli, nutzen die GST-tag für die Reinigung, und entfernen Sie dann die GST-tag durch Spaltung mit HRV-3C Protease (Abbildung 1) 6.
- Kaufen Sie das chemisch synthetisiert 5'-6-Carboxy-(6-FAM) markierte Oligonukleotid mit einem einzelnen 8-oxoG Rückstand und ihre komplementäre unmarkiertem Oligonukleotidstrang. Die 34-mer-Oligonucleotide enthalten ein 8-oxoG an Position 17 vom 5'-Ende. Reinigen dieser Oligonucleotide durch Polyacrylamidgelelektrophorese.
- Bereiten doppelsträngigen DNA-Substrats, das 8-oxoG durch Mischen von 5'-6-FAM markierte Oligonukleotid mit ihren komplementären Strang in einem Molverhältnis von 1:1,2 in Hybridisierungspuffer (10 mM Tris-HCl, pH 7,5, 50 mM NaCl, 1 mM EDTA) in einem 1,5 ml Mikrozentrifugenröhrchen. Das Röhrchen in einen Schwimmer in kochendem Wasser 5 min. Lassen Sie den Schlauch an Ort und Stelle und dann lassen Sie das Wasser langsam abkühlen zu room Temperatur (dies dauert ca. 2 h).
- Führen Probenvorbereitung und das Experiment unter niedrigen oder minimalen Lichtverhältnissen so zu minimieren Bleichen der Fluoreszenz-Markierung.
2. Messung der Beharrungszustand Zeit Course und Active-Site Titration von OGG1
2.1. Probenvorbereitung und steady-state Zeitverlauf
- Bereiten DNA Substrat und OGG1 Lösungen getrennt in Reaktionspuffer (50 mM HEPES, pH 7,5, 20 mM KCl, 0,5 mM EDTA und 0,1% Rinderserumalbumin) in 1,5 ml Mikrozentrifugenröhrchen auf Eis. Die Konzentration der DNA ist 400 nm und die scheinbare Konzentration OGG1 30, 60, 90, oder 120 nm, wie durch einen Bradford-Protein-Assay bestimmt. Legen Sie die Reaktionsgefäße in einem Heizblock Satz bei 37 ° C Pre-Inkubation Enzym und DNA-Substrat-Lösungen separat bei 37 ° C für 1 min.
- Starten Sie die Reaktion durch Mischen gleicher Volumina der OGG1 und DNA-Substrat-Lösungen durch Pipettieren. Nach dem Mischen diese Lösunggen 1:1 (v / v), sind die Endkonzentrationen 200 nM DNA und 15, 30, 45 oder 60 nM OGG1 sind. Entfernen Aliquote (10 ul) in zeitlichen Abständen und wird die Reaktion durch Mischen mit 1 ul 1 M NaOH. Da die Steuerung für die Zeit-Verläufe, 10 ul des Reaktionsgemisches ohne Enzym wird mit 1 ul 1 M NaOH gemischt.
- Die Reaktionsröhrchen Proben in einem 90 ° C Heizblock 5 min zu spalten, das resultierende Produkt AP-Website. Nach dem Erhitzen, fügen Sie 1 ul 1 M HCl, um jede Probe zu neutralisieren.
- In das gleiche Volumen (12 ul) Gelladepuffer (95% Formamid, 20 mM EDTA, 0,02% Bromphenolblau und 0,02% Xylencyanol) zu jeder Reaktion Probe, und legen Sie dann die Mischung in einem Heizblock bei 95 ° C eingestellt für 2 min, dann sofort den Schlauch auf Eis.
- Laden der Proben (5 ul) auf einem 15% igen denaturierenden Polyacrylamidgel mit 8 M Harnstoff in 89 mM Tris-HCl, pH 8,8, 89 mM Borsäure, 2 mM EDTA und. Das Substrat und die gespaltenen Produkte sind nach abgetrenntAusführen des Gels.
2.2. Imaging des Gels
- Scannen Sie das Gel mit einem Imager, der die Fluoreszenz-markierten DNA erkennen und visualisieren das Substrat und Produkt-Bands.
- Quantifizierung der Banden nach der Bebilderung das Gel. Beachten Sie, dass einige Hintergrund Spaltung nach der Behandlung des Substrats selbst mit NaOH (2A) beobachtet werden. Subtrahieren diesem Hintergrund aus der gemessenen Menge jedes Reaktionsprodukt.
2.3. Datenanalyse
- Zur Erstellung der Menge des Produkts an jedem Reaktionszeit (t) (Fig. 2B) ausgebildet ist. Analysieren der Rohdaten unter Verwendung der Gleichung 1, um die Amplitude des Burst (A 0, y-Achse) und der Steigung der linearen stationären Phase (V ss) zu bestimmen.
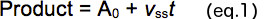
- Grundstückdie y-Achse relativ zu der Gesamt-Protein-Konzentration (dh ersichtlich Enzymkonzentration, 2C), der ein Maß der aktiven Fraktion des Enzyms. Verwenden Sie eine lineare Anpassung mit Nullachsenabschnitt um den Korrekturfaktor für die Bestimmung des Anteils an aktivem Enzym bereitzustellen.
- Zeichnen Sie die Steady-State-Rate relativ zum aktiven Enzym-Konzentration (Abbildung 2D) und bietet das Produkt Dissoziationsgeschwindigkeitskonstante (dh Steigung der angepassten Linie).
3. Messung der Pre-steady-state Zeitverlauf
Wie in 2, Produktbildung während der Burst-Phase gezeigt zu schnell ist, um durch manuelles Mischen und Abschrecken zu messen. Somit kann eine schnelle Quench-Flow-Gerät bieten eine leistungsfähige Methode, um schnelle Reaktionen, die auf die Millisekunde Zeitskala (Abbildung 3) 5 auftreten messen. Das Gerät verwendet einen Computer-gesteuerten Antriebsmotor schnell mischen einnd löschen Reaktionen nach angegebenen Reaktionszeiten. Verwenden Sie zum Beispiel eine Kintek RQF-3 Schnelle Quench-Flow-Instrument, um die anfängliche Burst-Phase und anschließender Steady-State-Phase des Produkt-Bildung durch OGG1 katalysiert messen. Schnelle Quench-Flow Instruments sind von verschiedenen Herstellern erhältlich.
3.1. Vorbereitung der Probe
Gesondert vorbereiten DNA Substrat und OGG1 Lösungen in 1,5-ml-Röhrchen wie in 2-1 beschrieben. Die Konzentrationen von DNA und aktive OGG1 sind 400 nM und 80 nM. Dies führt zu einer Endkonzentration von 200 nM und 40 nM DNA aktiv OGG1 nach dem Mischen 1:1 (v / v).
3.2. Vorbereitung der schnellen Quench-Flow-Gerät
- Schließen Sie ein zirkulierendes Wasserbad zur schnellen Quench-Flow-Instrument für Kontrolle der Temperatur (37 ° C).
- Stellen Sie die Parameter des Gerätes und wählen die entsprechende Reaktion Schleife für den gewünschten Zeitpunkten nach den Anweisungen des Herstellerstionen. Die Reaktion Schleifen sind von variablen Längen, alternative Reaktionszeiten bieten.
- Bereiten Reaktionspuffer und NaOH stillen Lösungen in 10 ml Luer Lock Einmalspritzen und befestigen diese Spritzen, um das Laufwerk Ports und laden Sie die Antrieb Reservoirs mit Reaktionspuffer (Spritzen B) und 142 mM NaOH (Spritze Q) (Spritze Ladeventile in LOAD-Position) . Um die Luftblasen aus der Spritze zu entfernen, funktioniert die Lösung hin und her mehrmals.
- Senken Sie den Schrittmotor, bis sie die Spitze der Spritze (Spritze Ladeventile Sample laden Ventile in Position FIRE).
- Spülen Sie die Loops, die Reaktion Loop, und die Exit-Linie mit Wasser und Methanol. Trocknen Sie die geröteten Bereiche komplett (Ventile in FLUSH Position).
3.3. Pre-steady-state Zeitverlauf
- Machen Sie ein Loch in der Spitze eines verkappten 1,5-ml-Röhrchen mit einer 16-Gauge-Nadel. Bringen Sie ein Rohr an der Ausfahrt Linie um den gestoppten Reaktion zu sammeln.
- Stellen Sie die gewünschte reaction Zeit (in Sekunden) über die Tastatur ein. Der Schrittmotor wird eine Sicherungskopie zu for-Schleife Lautstärke so einzustellen, wie konstant Quench Volumen zu halten. Position Kolben für Spritze B gegen den Schrittmotor-Plattform durch Zugabe von Puffer mit den Einwegspritzen, die an den Antrieb Ports.
- Füllen Sie 1 ml Luer Lock Einmalspritzen mit DNA-Substrat und OGG1 Lösungen, bzw. (Probenventile in LOAD-Position). Bringen Spritzen mit DNA-Substrat und OGG1 Lösungen für jede der Probenmenge Ports, und füllen Sie dann die Probe Loops mit DNA-Substrat und OGG1 Lösungen bzw. (Sample Load-Ventile in LOAD-Position).
- Stellen Sie alle Spritze Ladeventile Sample Ladeventile der FIRE Position. Starten Sie die Reaktion durch eine Tastatur Hub (zB Presse von "G" oder "START"-Taste). DNA-Substrat und OGG1 Lösungen (18 ul pro Stück) werden sofort in der Reaktionsschleife gemischt.
- Warten Sie, bis die Reaktion automatisch an der gewünschten Reaktionszeit durch Mischen 36 ul der r abgeschreckteaktion Mischung mit 86 ul 142 mM NaOH. Nach Quenchen der Reaktion wird die Probe aus der Auslassleitung abgeführt.
- Legen Sie die Spritze Ladeventile der LOAD-Position und die Probe Ladeventile dem FLUSH Position. Spülen Sie die Loops, die Reaktion Loop, und die Exit-Linie mit Wasser und Methanol und trocknet wie in 3.2 beschrieben. Wiederholen Sie diesen Vorgang für jeden Zeitpunkt.
- Durchführen einer Kontrollexperiment ohne Enzym, um einen Hintergrund-Korrektur, die von Hand oder mit dem schnellen Abschrecken Instrument durchgeführt werden soll. Um ein Steuerelement mit der S-Quench Instrument durchzuführen, füllen Sie die Sample-Loop für die DNA mit DNA-Substrat aber halten Sie die Sample-Loop für OGG1 leer. Stellen Sie die Reaktionszeit, führen Sie die Mischung und Abschrecken in 3.3.4-3.3.6 beschrieben.
- Waschen Sie die Probe Loops und die Reaktion Schleife mit 2 M NaOH, 2 M HCl, Wasser und Methanol und dann trocknen Sie die gewaschene Linien. Nach der Einstellung der Schrittmotor zur Ausgangsposition, schalten Sie die Spritze Ladeventile der LOAD Positionierungn und waschen Sie die Antrieb Stauseen mit Wasser.
- Behandeln der abgeschreckten Proben Reaktion mit Hitze und dann getrennten Substrat und Produkt-DNA von 15% denaturierende Polyacrylamid-Gelelektrophorese, wie in 2.1 beschrieben.
- Visualisierung und Quantifizierung der Banden auf dem Gel, wie in 2.2 beschrieben.
3.4. Datenanalyse
Montieren Sie die zeitlichen Verläufe der Produktbildung durch nicht-lineare Regressionsanalyse, um eine Gleichung mit einer steigenden exponentiellen und linearen Terme (Gleichung 2) die Bereitstellung der ersten Ordnung Geschwindigkeitskonstante (k obs), die Amplitude des Bursts (A 0) und eine lineare Geschwindigkeit (v ss).
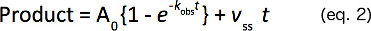
4. Einzel-Umsatz Zeitverlauf
- Bereiten DNA Substrat und OGG1 in separaten 1,5-ml-Röhrchen wie in 2-1 beschrieben. Die Konzentrationgen von DNA und aktive OGG1 sind 100 nm und 500 nM; ergibt dies Endkonzentrationen von 50 nM und 250 nM DNA OGG1 nach dem Mischen 1:1 (v / v).
- Bereiten Sie den schnellen Quench-Flow-Gerät und bereiten die Reaktion Proben wie in 3.2 und 3.3 beschrieben.
- Behandeln Sie die Proben abgeschreckt Reaktion mit Wärme und unterziehen sie 15% denaturierende Polyacrylamid-Gelelektrophorese auf separaten Substrat und Produkte wie in 2.1 beschrieben.
- Visualisierung und Quantifizierung der Banden auf dem Gel, wie in 2.2 beschrieben.
- Montieren Sie die zeitlichen Verläufe der Produktbildung zu einem einzigen exponentiellen die erster Ordnung (k obs) zu bestimmen, wie in Gleichung 3 gegeben.
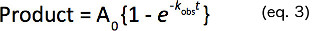
Ergebnisse
Die Steady-State kinetischen Analyse wurde unter Verwendung von 200 nM DNA Substrat und vier verschiedenen scheinbaren Konzentrationen OGG1 (15, 30, 45 und 60 nm), wie durch einen Bradford-Protein-Assay bestimmt 2 durchgeführt. Die Zeitverläufe der Produktbildung von einer linearen Gleichung, um die y-Achse, die 2,2 waren, 11, 15 und 26 nm, relativ zueinander Proteinkonzentration (2B) zu bestimmen, zu passen. Die y-fängt wurden weiter aufgetragen relativ zueinander tatsächlichen Proteinko...
Diskussion
Die kinetischen Ansätze hier beschriebenen Methoden, um einen Überblick über elementare kinetischen Konstanten definieren. Wenn ein Zeitverlauf der Produktbildung ist biphasisch mit der ersten enzymatischen Umsatz auftretenden schnell, dann ein Schritt nach Chemie geschwindigkeitsbestimmenden während anschließende katalytische Umsätze. Im Falle OGG1 kann die erste Umsatz unter Verwendung hoher Konzentrationen Enzym entweder mit Begrenzung (S E) DNA-Konzentrationen werden. Im ersten Fall wi...
Offenlegungen
Autoren haben nichts zu offenbaren.
Danksagungen
Wir danken Dr. Julie K. Horton für die kritische Durchsicht des Manuskripts und Dr. Rajendra Prasad für Anregungen und Diskussionen. Teile dieser Forschung wurden ursprünglich in The Journal of Biological Chemistry, Sassa A et veröffentlicht. al., "DNA-Sequenz Context Auswirkungen auf die Aktivität von humanem Glycosylase 8-Oxoguanine DNA Glycosylase." J Biol Chem. 287, 36702-36710 (2012) 2. Diese Arbeit wurde unterstützt, im Ganzen oder in Teilen, durch National Institutes of Health Research Project Grants Z01-ES050158 im Interne Research Program, NIEHS.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 5’-6-FAM labeled oligonucleotides containing a single 8-oxoG | Eurofins MWG Operon | 5’-P32 radiolabeled oligonucleotides can be used as well. Polyacrylamide gel purification grade is recommended. | |
| Unlabeled oligonucleotides (complementary strand) | Eurofins MWG Operon | Polyacrylamide gel purification grade is recommended. | |
| 1 ml BD Luer Lock disposable syringe | BD Medical | 309628 | Lue Lock disposable syringe from other vendors can be used as well. |
| 10 ml BD Luer Lock disposable syringe | BE Medical | 309604 | Luer Lock disposable syringe from other vendors can be used as well. |
| Equipment | |||
| Circulating water bath | Any vender | ||
| RQF-3 Rapid Quench-Flow Instrument | KinTek Corporation | Rapid Quench-Flow Instrument from other vendors can be used as well. | |
| Typhoon Phosphorimager 8600 | GE Healthcare Life Sciences | Imager from other vendors can be used as well. | |
| KaleidaGraph | Synergy Software | ||
Referenzen
- Porello, S. L., Leyes, A. E., David, S. S. Single-turnover and pre-steady-state kinetics of the reaction of the adenine glycosylase MutY with mismatch-containing DNA substrates. Biochemistry. 37, 14756-14764 (1998).
- Sassa, A., Beard, W. A., Prasad, R., Wilson, S. H. DNA sequence context effects on the glycosylase activity of human 8-oxoguanine DNA glycosylase. The Journal of Biological Chemistry. 287, 36702-36710 (2012).
- Wong, I., Lundquist, A. J., Bernards, A. S., Mosbaugh, D. W. Presteady-state analysis of a single catalytic turnover by Escherichia coli uracil-DNA glycosylase reveals a "pinch-pull-push" mechanism. The Journal of Biological Chemistry. 277, 19424-19432 (1074).
- Johnson, K. A. Advances in transient-state kinetics. Current Opinion in Biotechnology. 9, 87-89 (1998).
- Johnson, K. A. Rapid kinetic analysis of mechanochemical adenosinetriphosphatases. Methods in Enzymology. 134, 677-705 (1986).
- Kovtun, I. V., et al. OGG1 initiates age-dependent CAG trinucleotide expansion in somatic cells. Nature. 447, 447-452 (2007).
- Hill, J. W., Hazra, T. K., Izumi, T., Mitra, S. Stimulation of human 8-oxoguanine-DNA glycosylase by AP-endonuclease: potential coordination of the initial steps in base excision repair. Nucleic Acids Research. 29, 430-438 (2001).
- Zharkov, D. O., Rosenquist, T. A., Gerchman, S. E., Grollman, A. P. Substrate specificity and reaction mechanism of murine 8-oxoguanine-DNA glycosylase. The Journal of Biological Chemistry. 275, 28607-28617 (2000).
- Waters, T. R., Swann, P. F. Kinetics of the action of thymine DNA glycosylase. The Journal of Biological Chemistry. 273, 20007-20014 (1998).
- Nilsen, H., et al. Excision of deaminated cytosine from the vertebrate genome: role of the SMUG1 uracil-DNA glycosylase. The EMBO Journal. 20, 4278-4286 (2001).
- Gill, S. C., von Hippel, P. H. Calculation of protein extinction coefficients from amino acid sequence data. Analytical Biochemistry. 182, 319-326 (1989).
- Van de Berg, ., Beard, B. J., A, W., Wilson, S. H. DNA structure and aspartate 276 influence nucleotide binding to human DNA polymerase beta. Implication for the identity of the rate-limiting conformational change. The Journal of Biological Chemistry. 276, 3408-3416 (2001).
- Krishnamurthy, N., Haraguchi, K., Greenberg, M. M., David, S. S. Efficient removal of formamidopyrimidines by 8-oxoguanine glycosylases. Biochemistry. 47, 1043-1050 (2008).
- Leipold, M. D., Muller, J. G., Burrows, C. J., David, S. S. Removal of hydantoin products of 8-oxoguanine oxidation by the Escherichia coli DNA repair enzyme, FPG. Biochemistry. 39, 14984-14992 (2000).
- Zhao, X., Krishnamurthy, N., Burrows, C. J., David, S. S. Mutation versus repair: NEIL1 removal of hydantoin lesions in single-stranded, bulge, bubble, and duplex DNA contexts. Biochemistry. 49, 1658-1666 (2010).
- Leipold, M. D., Workman, H., Muller, J. G., Burrows, C. J., David, S. S. Recognition and removal of oxidized guanines in duplex DNA by the base excision repair enzymes hOGG1, yOGG1, and yOGG2. Biochemistry. 42, 11373-11381 (2003).
- Robey-Bond, S. M., Barrantes-Reynolds, R., Bond, J. P., Wallace, S. S., Bandaru, V. Clostridium acetobutylicum 8-oxoguanine DNA glycosylase (Ogg) differs from eukaryotic Oggs with respect to opposite base discrimination. Biochemistry. 47, 7626-7636 (2008).
- Jarem, D. A., Wilson, N. R., Delaney, S. Structure-dependent DNA damage and repair in a trinucleotide repeat sequence. Biochemistry. 48, 6655-6663 (2009).
- Livingston, A. L., Kundu, S., Henderson Pozzi, M., Anderson, W. D., David, S. S. Insight into the roles of tyrosine 82 and glycine 253 in the Escherichia coli adenine glycosylase MutY. Biochemistry. 44, 14179-14190 (2005).
- Kundu, S., Brinkmeyer, M. K., Livingston, A. L., David, S. S. Adenine removal activity and bacterial complementation with the human MutY homologue (MUTYH) and Y165C, G382D, P391L and Q324R variants associated with colorectal cancer. DNA Repair. 8, 1400-1410 (2009).
- Zharkov, D. O., Rosenquist, T. A. Inactivation of mammalian 8-oxoguanine-DNA glycosylase by cadmium(II): implications for cadmium genotoxicity. DNA Repair. 1, 661-670 (2002).
- Jarem, D. A., Wilson, N. R., Schermerhorn, K. M., Delaney, S. Incidence and persistence of 8-oxo-7,8-dihydroguanine within a hairpin intermediate exacerbates a toxic oxidation cycle associated with trinucleotide repeat expansion. DNA Repair. 10, 887-896 (2011).
- Mokkapati, S. K., Wiederhold, L., Hazra, T. K., Mitra, S. Stimulation of DNA glycosylase activity of OGG1 by NEIL1: functional collaboration between two human DNA glycosylases. Biochemistry. 43, 11596-11604 (2004).
- Sidorenko, V. S., Nevinsky, G. A., Zharkov, D. O. Mechanism of interaction between human 8-oxoguanine-DNA glycosylase and AP endonuclease. DNA Repair. 6, 317-328 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten