Method Article
発生とのマルチ表現型ハイコンテンツスクリーニング
要約
Q熱リケッチアは人獣共通感染症のQ熱の責任偏性細胞内グラム陰性細菌です。ここでは、 コクシエラ蛍光トランスポゾン変異体の生成と同様に、得られた内部移行、複製、および細胞傷害性表現型の自動識別と分析のための方法を説明します。
要約
細菌性病原体による宿主細胞の浸潤およびコロニー形成が重要なホスト機能を破壊して操作することができる病原因子として定義された原核生物の多数のタンパク質、の活性に依存します。ホスト/病原体相互作用の研究は、細菌感染症を理解し、感染症に対抗する代替戦略を開発することが極めて重要です。このアプローチは、しかし、細菌の病原性決定因子の公正な、自動化された同定および特徴付けのための新たなハイスループットアッセイの開発を必要とします。ここでは、トランスポゾン突然変異誘発によりGFPタグ付き変異体ライブラリーを生成するための方法を説明し、ハイコンテントスクリーニングの開発には、複数のトランスポゾンに関連する表現型の同時同定のために接近します。私たちの作業モデルは、SEと関連している細胞内細菌性病原体のQ熱リケッチア 、人獣共通感染症のQ熱の病原体であり、その結果としての健康と経済的負担との発生をヴィア。この病原体の偏性細胞内性質は、最近まで、厳しくコクシエラのハイスループット/高含量のアプローチを実現するための理想的なモデルを作り、ホスト病原体の相互作用に関与する細菌因子の同定を妨げています。
概要
新興、流行細菌コクシエラバーネッティは、Q熱、重度の健康と経済的影響1と衰弱インフルエンザ様人獣共通感染症の大規模な集団発生の原因です。 コクシエラの主な貯水池は、国内および農場の動物であり、それは米国の乳牛の90%以上がCを運ぶと推定されています2 バーネッティ 。人間は汚染されたエアロゾルの吸入によって感染している偶然の宿主です。ヒトQ熱は65% に達し、1,3-死亡率、致命的な合併症を有していてもよい、いずれかの急性または慢性疾患として現れます。 1の感染量で- 10生物、 コクシエラが知られている最も感染性病原体であり、これは潜在的なバイオ兵器4として研究されてきました。オランダ(2007 - 2010)におけるQ熱の最近の爆発的な流行は、年間2,000人以上に182からエスカレートケースで、この病原体の深刻な毒性の例として立っ5。
コクシエラ感染の顕著な効率は、おそらく細胞をホストする独自の適応と合わせ、環境ストレスに対する抵抗性と関連しています。実際、 コクシエラは、いくつかの厳しい条件(乾燥、温度など )に対して非常に抵抗性である代謝的に不活性小細胞変異体(SCV)の形で環境中に存在します。非食細胞の浸潤がコクシエラ接着 /浸潤のOmpA 7および未同定の受容体によって媒介されている間のSCVは、最大3インテグリン6βαのVを介して食細胞によって取り込まれます。取り込み後、 コクシエラは Rab5のとEEA1 8早期エンドソームマーカーに陽性、タイトフィットの液胞に存在します。細菌は代謝的に活性な大細胞変異体(小型商用車)に変換し、ドット/ ICMの4型分泌系(T4SS)9を活性化することにより、エンドソーム酸性化への対応します、 レジオネラ·ニューモフィラ 10のそれに非常に相同です。ドット/ ICMのエフェクターの分泌がコクシエラは細菌が繁栄し、積極的にアポトーシス11から感染細胞を保護することができるアクティブなリソソーム酵素を含む大きな、LAMP1陽性酸性コンパートメントを生成することができます。したがって、 コクシエラの細胞内周期は細菌エフェクター12のドット/ ICM-媒介転座によって制御されているが、宿主細胞の浸潤、細菌の複製および感染の普及に関与する微生物の要因はほとんどわかっていません。
宿主細胞内の1)内在化、2)細胞内複製、3)細胞から細胞への広がり:トランスポゾン突然変異誘発および蛍光ベースのアッセイを組み合わせることで、我々は、 コクシエラ感染の主なステップに関与する細菌の要因の同時同定のための公平なアプローチを開発しています、および4)持続。これまでに、我々は千メートルにわたってスクリーニングしコクシエラの病因7を調節する宿主-病原体相互作用への前例のない洞察力を提供してくれ500 コクシエラコード配列、でutations。注目すべきことに、このアプローチは、 コクシエラと細胞生物学の特徴を共有する他の細胞内病原体の研究に適用することができます。
プロトコル
GFPタグコクシエラトランスポゾン変異体のライブラリーの1世代
ローカルルールに準拠して、微生物安全キャビネット(MSC)にバイオセイフティ封じ込めレベル2(BSL-2)でコクシエラバーネッティ RSA439 NMIIを操作します。使用細菌のモデルと互換性場合、クローン変異体を得る確率を高めるために1.4.1から1.4.4への手順を繰り返します。典型的な変異体のライブラリーを使用し、生物のゲノムに注釈付きコード配列の3倍の数に等しい変異体の数の(少なくとも)で構成されています。
- エレクトロコクシエラRSA439 NMIIの調製:
- 13.4 mMのクエン酸、16.1 mMのクエン酸ナトリウム、3.67 mMリン酸カリウム、1mMの塩化マグネシウム、0.02mMの塩化カルシウム、0.01mMの硫酸鉄、125.4 mM塩化ナトリウム、1.5mMのL-システイン0.1 G:1X ACCM-2 13を準備します/ Lバクトネオペプトン、2.5グラム/ Lカザミノ酸、を1g / Lのメチルβシクロデキストリン125ミリリットル/ L RPMI。 4.75とフィルター滅菌し(オートクレーブしない)にpHを調整します。注:液体ACCM-2は、約1ヶ月間、4℃で安定です。
- -80℃のストックから(以前にステップ1.5のように生成し、定量細菌ストックから) コクシエラ RSA439 NMIIの2×10 6ゲノム当量(GE)/ mlのACCM-2の100ミリリットルに接種し、75で細菌懸濁液を配布( -フラスコ当たりの細菌懸濁液の15 mlの10)通気キャップ付きcm 2の細胞培養フラスコ。 5%CO 2、2.5%O 2の加湿雰囲気中、37℃で7日間増殖。
- プール4°Cで1時間3,900×gで50ミリリットルチューブおよび遠心機で得られた細菌懸濁液。
- 上清を捨て、10%グリセロール30mlにペレットを再懸濁します。 4°Cで1時間3,900×gで遠心分離します。
- 10%グリセロール(典型的には2 ml)および500μlのアリコートをチューブ中の50μlの適切な量でペレットを再懸濁します。再懸濁したBを保ちます全体のプロセスの間に氷上でacteria。注:この段階では、細菌は、エレクトロであり、1つのアリコートは、一エレクトロポレーションを行うのに十分です。細菌懸濁液を、6ヶ月間-80℃で保存することができ、または直接のプラスミドDNAのエレクトロポレーションのために使用することができます。
- transposon-とトランスポザーゼをコードするプラスミドで有能コクシエラのエレクトロポレーション:
注:以下のプロトコルのために、転移因子およびトランスポザーゼは(それぞれ、PITR-CAT-GFPとのpUC19-Himar1C9)二つの異なるプラスミドによりコードされている7。両方のプラスミドは、コクシエラでエレクトロときにそれら自殺プラスミド作り、コクシエラ固有の複製起点を欠いています。これは、安定したトランスポゾン挿入を保証します。転移因子は、選択のためのコクシエラプロモーターp1169とGFPと生成された変異体をタグ付けするコクシエラプロモーターP311の調節下にGFP遺伝子の調節下にクロラムフェニコール耐性カセットを含みます。- PR氷上で10分間、0.1センチエレクトロポレーションキュベットを電子冷却。 10μgのトランスポゾンプラスミドおよびトランスポザーゼプラスミド710μgのエレクトロコクシエラ 50μlのを混ぜます。そのプラスミド濃度はグリセロールの希釈を最小限にするよりも高い500 / mlのあることを確認してください。
- 次のセットアップを使用して、エレクトロポレーション:18 kVで、500Ω、25μF。得られた時定数は9と13ミリ秒の間に含まれていることを確認。
- すぐに、RPMIの950μlを添加する電気穿孔細菌を再懸濁し、スクリューキャップチューブに移し、室温で保管してください。
- エレクトロポレーション、細菌の200μLを取り、6ウェルプレートに1%の熱不活性化ウシ胎児血清(FBS)を補充したACCM-2の3ミリリットルに追加。エレクトロポレーション、細菌の残量にDMSOを88μl加え、-80℃で、店舗(10%DMSOの最終濃度に達するまで)。
- トランスポゾン突然変異体の選択。
- Incuba5%CO 2、2.5%O 2の加湿雰囲気中で37℃で一晩(1.2.4)上記のように接種した6ウェルプレートTE。適切な抗生物質(375 / mlのカナマイシン、3 / mlのクロラムフェニコール)を追加します。上記の条件でさらに3日間細菌培養物をインキュベートします。
- 個々の変異体の単離:
- コクシエラトランスポゾン変異体の固体ACCM-2のプレートの作製とめっき
注:以下の手順は、1ペトリ皿のために、細菌培養物のいくつかの希釈は、コロニーを単離するための最適な接種量を評価するためにテストする必要があります。- 電子レンジで0.5%アガロース10.5 mlで加熱し、55℃の水浴中で冷まします。 37℃で2×ACCM-2(pHは4.75)11.25 mlで加熱します。
- 底部アガロースを準備します。
- 2X ACCM-2 10mlで溶融し、0.5%アガロースを10mlを混合し、適切な抗生物質(375 / mlのカナマイシン又は3を追加/ mlのクロラムフェニコール)。
- ペトリ皿にすぐに注ぎます。ペトリ皿unliddedを維持し、20分間、30分間、空気乾燥するための媒体を涼しくしましょう。
- トップアガロースを準備します。
- 適切な抗生物質(375 / mlのカナマイシン、3 / mlのクロラムフェニコール)を追加し、37℃でインキュベート5mlのポリスチレンチューブ内の水の0.75ミリリットルで2回ACCM-2の1.25ミリリットルを混ぜます。
- 5秒間細菌培養(典型的には1〜100μL)と渦を追加します。
- 、溶融アガロース0.5 mlを加え混ぜ、すぐに底アガロースに注ぎます。
- 、20分間冷却ペトリ皿に蓋を交換し、アガロース凝固を促進するために、4℃で20分間インキュベートすることができます。
- MSCにunlidded 20分間空気乾燥。 6~7日間、5%CO 2および2.5%O 2の加湿雰囲気中、37℃でプレートを成長します。
- 残りの細菌にDMSOを追加-80℃で10%のDMSO、店舗の最終濃度を達成するために、L培養。
- 次のように最適希釈を評価:コロニーは直径0.5〜1ミリメートルであり、適切に交差汚染を避けるために隔離されていることを確認してください。 1.4.1.2と1.4.1.3で説明したようにACCM-2寒天上の適切な希釈でポイント1.4.2およびプレートから残りの細菌培養物を解凍します。 1.4.1.3.5に記載されているように6〜7日間インキュベートします。
- コロニーが検出されたら、/適切な抗生物質(375 / mlのカナマイシンまたは3μgのを含むACCM-2の1.5ミリリットルで、1ミリリットルチップの先端を切断分離されたコロニーを含むプラグをピッキングし、ピペッティングによりコロニーを分散させることによってそれらを収集24ウェルプレート中のmlのクロラムフェニコール)。 1.3.1に記載した条件で6日間、個々のコロニーを増幅します。インキュベーションの3日目に、それぞれの文化をピペットで細菌塊を分散させます。
- 10で96ウェルプレート中の2Dバーコードスクリューキャップチューブ中の各変異体の懸濁液を保存します-80℃での%のDMSO。
- コクシエラトランスポゾン変異体の固体ACCM-2のプレートの作製とめっき
- 細菌濃度の評価:
注:以下の手順は、無菌媒体に複製する細菌の突然変異体(1.4.4を参照)の成長曲線を得るために適用することができます。- 標準曲線の準備:
- 二本鎖DNAを2μg/ mlのストック溶液を1×トリスEDTA(TE)中の(既知のサイズと濃度の典型的ランダムプラスミド)を準備します。 2 / mlのから2 ngの/ mlの範囲の濃度を得るために、原液から10倍連続希釈液を調製します。 ( 材料の表を参照)、黒壁や底部を有する96ウェルマイクロプレートの単一ウェルに各濃度の50μLを分注します。
- 1×TE緩衝液に200と96ウェルマイクロプレートに各サンプルに希釈された試薬の55μlを添加する:二本鎖DNAの定量試薬1を希釈します。プレートシェーカーを用いて十分に混合し、暗所で、室温で2〜5分間インキュベートします。
- 蛍光を発するを使用したサンプルの蛍光を測定標準的なフルオレセインの波長(励起〜480nmで、発光〜520 nm)のためのNCEマイクロプレートリーダーおよびフィルタ。
- 蛍光強度の測定値に対するプラスミド濃度範囲をプロットします。
- 細菌懸濁液の定量:
- 黒壁と底面( 材料の表を参照のこと)で96ウェルマイクロプレートにウェルあたり10%トリトンX-100の5μLを分注します。プレートシェーカー上で、各ウェルに細菌懸濁液の50μLを加え、室温で10分間インキュベートします。
- 1×TE緩衝液に200と96ウェルマイクロプレートに各サンプルに希釈された試薬の55μlを添加する:二本鎖DNAの定量試薬1を希釈します。プレートシェーカーを用いて十分に混合し、暗所で、室温で2〜5分間インキュベートします。
- 標準的なフルオレセインの波長(励起〜480nmで、発光〜520 nm)のための蛍光マイクロプレートリーダーおよびフィルタを使用して、サンプルの蛍光を測定します。
- 細菌DNを取得するには濃度は、点1.5.1.4で得られたチャートの蛍光測定値をプロットします。細菌の濃度を得るために、 コクシエラゲノム (2.2 FG)の質量によってDNA濃度を分割します。ゲノム当量/ mlの中で結果を表現します。
- ACCM-2での大きな成長欠陥を示す変異体を捨てます。
- 標準曲線の準備:
2.単一のプライマーコロニーPCR、シーケンシング、および注釈
注:以下のプロトコルは、96サンプルのDNA増幅のためのものである、マルチチャンネルピペットは、次の手順をお勧めします。トランスポゾン特異的プライマー(2.3)を有する磁気ビーズ及びDNA配列決定を使用して、PCR産物をカラム精製は、外部の会社に委託されています。
- 増幅プライマーはCoxiellにトランスポゾン挿入部位をカバーするPCR産物を得るために、反転タンデムリピート(ITR)の上流100および200塩基対の間にハイブリダイズするように設計されていることを確認ゲノム。 3 PCRミックスのミリリットル(1×高忠実度緩衝液、200μMのdNTPを、1μMの増幅プライマー、20 U / mlの高忠実度DNAポリメラーゼ)を準備し、氷の上に設定され、96ウェルPCRプレートにウェルあたり29μLを分注します。 PCRミックスへの転送ACCM-2における固定相の各変異体の1μLを。
- 最初の変性で実行PCR(98°C、1分)、20高ストサイクル(98°C、10秒; 50℃、30秒; 72℃、90秒)、30低ストサイクル(98°C、 10秒; 30℃、30秒; 72℃、90秒)、30高ストサイクル(98°C、10秒; 50℃、30秒; 72℃、90秒)72での最終伸長によって実施されました7分間°C。
- トランスポゾン特異的プライマーを用いて、磁気ビーズおよび配列DNAを用いてPCR産物を精製します。 50°Cと75°C、40%、60%、18から25ヌクレオチドの間の長さおよびアニーリングSiのGC含量との間に含まれる予測融解温度を有するトランスポゾン特異的プライマーを設計トランスポゾンITRの最初の塩基対の上流TE増幅プライマーのハイブリダイゼーション部位の下流に少なくとも100塩基対。
- 配列解析ソフトウェアを使用して、Q熱リケッチア493 NMIの完全な、注釈付きのゲノムをロードします。ロードして(BLASTN)配列決定の結果を合わせ、転位の部位を決定するために、「位置合わせ基準に "関数を使用します。非マッチングおよび/または二重reads.Toは変異体ライブラリーの飽和を監視表示を有する変異体を捨て、同じサイトに複数のトランスポゾン挿入の発生を記録しておきます。
3.真核細胞コクシエラ変異体と細胞内増殖のモニタリングとチャレンジ
注:マルチチャンネルピペットは、次の手順をお勧めします。感染症は、黒壁や平らな透明底部を有するマイクロ滅菌96ウェル中で三連で行いました。 GFP 14ワットを表現重量Q熱リケッチアロバートHeinzenによって提供されます。
- (RPMI培地を完了)、抗生物質の非存在下で、10%ウシ胎児血清(FBS)を補充したフェノールレッドを含まないRPMI中でVero細胞を成長させます。
- 感染の前日には、PBS 10mlでコンフルエントまたはサブコンフルエント細胞培養フラスコからVero細胞を洗浄します。
- 細胞培養フラスコにトリプシンEDTA溶液1mlを添加することによって、ベロ細胞を剥離し、5%CO 2の加湿雰囲気中、37℃で3〜5分間インキュベートします。
- 完全RPMI培地10ml中の細胞を再懸濁し。細胞をカウントし、1mlあたり10 5細胞の細胞懸濁液を準備します。
- 平坦な透明底部を有する黒色96ウェルプレートの各ウェルに100μlの細胞懸濁液を分注します。
- 室温で400×gで5分間遠心分離し、ウェルの底部での細胞接着を促進し、5%CO 2の加湿雰囲気中で37℃で一晩インキュベートします。
- Coを含む96ウェルプレートを解凍RTでxiella変異体とは、深い96ウェルプレートにフェノールレッド及びFBSを含まないRPMI300μlの中の細菌懸濁液150μLを希釈します。
- Vero細胞を含むマイクロプレートから培地を除去し、100μl/ウェルの希釈コクシエラ変異体(100のMOI)を分注します。 (100と200の重量コクシエラに感染した細胞は、感染の多重度でGFP を発現している14(MOI))を陽性対照として陰性(非感染細胞)コントロール、ウェルA2およびA3とA1をよく使用します。
- エアロゾルタイト遠心プレートホルダーを使用して、室温で400×gで10分間プレートを遠心。
- その後、100μl/ウェルの新鮮な完全RPMI培地で細菌を含む培地を交換して2時間5%CO 2の加湿雰囲気中で37℃でインキュベートします。
- 標準的なフルオレセインの波長(励起〜480nmで、発光〜520 nm)のための蛍光マイクロプレートリーダーとフィルタを使用して、7日間のGFP蛍光を毎日測定します。回避するために、干渉培地中で凝縮及び信号分散液に、マイクロプレートリーダーで底励起および発光記録を使用するため。
自動画像取得のための試料の調製4。
注:手順は、1枚の96ウェルプレートのためであり、それに応じてボリュームをスケールアップ。 4.2からのステップは、プレートウォッシャーを利用することができます。
- 日の感染後第 7に、プレートから培地を除去し、50μL/ウェルの適切な希釈(通常1で細胞透過性蛍光色素を含有する新鮮な、完全培地に置き換え:1,000、使用する細胞株に応じて最適化することが可能に)。 5%CO 2の加湿雰囲気中で37℃で60分間- 30細胞をインキュベートします。
- 、PBS中の4%パラホルムアルデヒド(PFA)を50μl/ウエルで培地を交換し、室温(RT)で30分間インキュベートし、次いでPFAを含有する緩衝液を除去し、PBSで3回洗浄します。
- PBSおよびdispensを削除Eを50μl/ウェルの0.05%サポニンで補充液(0.5%ウシ血清アルブミン、PBS、pH7.4中の50mM NH 4 Clで)をブロックします。室温で30分間インキュベートします。
- :500希釈の40μl/ウェルの(上記)サポニンとし、1で抗LAMP1抗体を補充した新鮮なブロッキング溶液でブロッキング液を交換してください。室温で30分間静置します。
- ブロッキング溶液を削除し、100μlのPBS /ウェルで96ウェルプレートを5回洗浄します。
- 5μgのでステップ4.4で適用される抗LAMP1抗体を明らかにし、ヘキスト33258で:40μL/ウェル(上記)サポニンを補った溶液、(千1の希釈で)適切な蛍光標識した二次抗体をブロックするのを分配/ mlです。室温で30分間静置します。
- ブロッキング溶液を削除し、100μlのPBS /ウェルで96ウェルプレートを5回洗浄します。固定された細胞が乾燥しないでくださいように、96ウェルプレート内の最後の洗浄に対応するPBSの体積を残します。
- 画像その後の分析のため、光から保護し、4℃で直ちにプレートまたはストアプレート、。
5.画像取得
- 装備落射蛍光自動顕微鏡を用いてGFP(488nmで、細菌)、ヘキスト33258(350nmで、宿主細胞核)、赤(〜555nmで、細胞膜マーカー)および遠赤色(〜615ナノメートル、LAMP1)チャンネルの画像を取得20Xを目的としました。画像化するために、サンプルあたり5,000個の細胞の最小値を、ウェル当たり21の独立したフィールドを取得します。参照として、宿主細胞核チャネルを使用してオートフォーカスを適用します。細菌性病原体は、宿主細胞の低い割合に感染して作業する場合、ユーザーが分析するために500に感染した細胞の最小値を得るために、ウェルあたり撮像された独立したフィールドの数を調整することができます。
6.画像処理
注:次の手順は、画像解析ソフトウェアCellProfilerの使用に特異的です。すべての場合において、最適なalgoritセグメント化のためのHMは、実験的に定義する必要があり、画像の境界に触れるオブジェクトは、適切な機能を排除する必要があります。
- CellProfiler内のすべての画像をロードします。
- 次の手順で、宿主細胞核として(もヘキストによって標識) コクシエラコロニーの検出を回避するために、ヘキストチャネルからGFPチャネルを減算するモジュール「ImageMath "を使用します。
- ステップ6.2の結果として得られる画像からセグメント宿主細胞核にモジュール「IdentifyPrimaryObjects」を使用してください。セグメント化されたオブジェクトの「核」という名前を付けます。
- シードとしてステップ6.3で検出された核を使用して、555nmの画像からセグメントの宿主細胞へのモジュール「IdentifySecondaryObjects」を使用してください。セグメント化されたオブジェクトの「セル」という名前を付けます。
- (オプション)ステップ6.4で同定された細胞から、ステップ6.3で特定された核を減算するモジュール「IdentifyTertiaryObjects "を使用します。セグメント化されたオブジェクトに名前を付け、「細胞質221 ;.
- 背景を削除し、LAMP1陽性区画の次の識別を容易にするために、615 nmの画像上のモジュール「EnhanceOrSuppressFeatures」を使用してください。
- LAMP1陽性区画を識別するためにステップ6.6で得られた画像上のモジュール「IdentifyPrimaryObjects」を使用してください。セグメント化されたオブジェクト「リソソーム」という名前を付けます。
- コクシエラコロニーを識別するために、488 nmの画像上のモジュール「IdentifyPrimaryObjects」を使用してください。セグメント化されたオブジェクトの「コロニー」という名前を付けます。
- シードとしてステップ6.8で検出コクシエラコロニーを使用して、コクシエラ含有液胞を識別するために、615 nmの画像上のモジュール「IdentifySecondaryObjects」を使用してください。セグメント化されたオブジェクト「CCVS」という名前を付けます。
- 細胞上で検出CCVSを選択するために、モジュール「MaskObjects」を使用します(画像の境界に触れる細胞が除去されているように、いくつかのCCVSは「外側」の細胞を検出することができます)。解像度に名前を付けますultingは「フィルタ処理CCVSを "オブジェクト。
- (画像の境界に触れる細胞が排除されているように、いくつかのコロニーを「外側」の細胞を検出することができる)の細胞で検出されたコロニーを選択するにはモジュール「MaskObjects」を使用してください。結果のオブジェクト "フィルタ済みコロニー」という名前を付けます。
- 細胞にリソソームを関連付けるために、モジュール「MaskObjects "を使用します。結果のオブジェクト "フィルタ済みリソソーム」という名前を付けます。
- コクシエラコロニーを含有する細胞を選択するために、モジュール「MaskObjects」を使用してください。結果のオブジェクト「感染細胞」という名前を付けます。
- 「オブジェクトを関連付ける "オブジェクトを親オブジェクト「セル」の子として「フィルタさCCVS」を設定するには、モジュールを使用してください。これはCCVS /セルの数をカウントできるようになります。
- モジュールを使用して親オブジェクト「セル」の子としてオブジェクト」フィルタを適用したコロニーを「設定する」オブジェクトを関連付けます」。この意志コロニー/セルの数をカウントすることができます。
- 親オブジェクト「セル」の子としてオブジェクト「フィルタ済みリソソーム "に設定するには、「オブジェクトを関連付ける」モジュールを使用してください。これは、リソソーム/セルの数をカウントすることを可能にします。
- 核、細胞ろ過CCVS、濾過したコロニーそして濾過リソソームの形態素解析を得るために、モジュール「MeasureObjectSizeShape "を使用してください
- フィルタ済みCCVSに関連するGFP蛍光を定量化し、CCVS内コクシエラ複製の効率を推定するために、モジュール「MeasureObjectIntensity "を使用します。
- モジュール「OverlayOutlines」と「SaveImages」を使用して分割結果と品質管理のため、元の画像を重ねます。
- 全部または画像解析結果の選択をエクスポートするモジュール「ExportToSpreadsheet "を使用します。
- (オプション)結果を分析する「ExportToDatabase」モジュールを使用しますソフトウェアCellProfilerアナリストを使用して。
7.データ解析
- 得られた各パラメータについて、次に変異体あたりの平均値を計算する(画像分割におけるエラーに起因する)外れ値を識別し、排除します。
- 重要な表現型を識別するために、Zスコアを使用してください。 Zスコア>と表現型を考えてみましょう-2間-2と-4 Zスコアのように穏やかな表現型Zスコアと有意でない、表現型として≤-4のような強いです。
- 実験のニーズに応じたパラメータの組み合わせをプロットします。
結果
トランスポゾン突然変異体を単離すると、単一プライマーコロニーPCRは、各変異体のためのトランスポゾン挿入部位を同定するための強固な、ハイスループット法です。このアプローチは、典型的なネストされたPCRプロトコルから派生が、ここでは単一のプライマーは、アニーリング温度( 図1A)のストリンジェンシーに依存して鋳型DNAに特異的および/ または非特異的にハイブリダイズします。典型的なPCR産物は、特異的であり、そのほとんどが、複数のDNA断片( 図1B)から成ります。右トランスポゾンITRの上流、および増幅プライマーにより認識される配列の下流にアニールする異なる配列決定プライマーの使用は、配列決定工程( 図1C)に対する特異性を提供します。配列解析のための自動化ソフトウェアは、トランスポゾン挿入( 図1C)の正確な部位を提供するコクシエラゲノムに得られた配列を整列させます。すべてのトランスポゾン挿入は、その後、アンすることができますコクシエラゲノム ( 図1D)にotated。
各コクシエラ変異体が単離され、貯蔵またはスクリーニングのいずれかの前にACCM-2培地に無菌的に増幅される。2、16 ドット/ ICMコクシエラ遺伝子 ( 図2A)38のトランスポゾン変異体の例を示す図 。 コクシエラ突然変異体の生存率を評価するために、純粋な成長曲線を1.5(Figurie 2B)に記載の細菌濃度アッセイを、接種後7日間の細菌培養物をサンプリングして適用することによって得られます。増幅された変異体は、その後7日間三重96ウェルプレート中で、上皮細胞とインキュベートします。 GFPタグ化されて生成されたすべてのコクシエラ変異体は、細胞内の増殖曲線は各24時間、すべてのウェルのGFPの蛍光強度を測定した、時間の関数( 図2C)として測定値をプロットしています。
INTR無細胞成長曲線は、 コクシエラゲノム内のすべてのトランスポゾン挿入に関連する表現型の定量的な分析を提供します。同じトランスポゾン突然変異体についての定性的な情報を追加するために、我々は、自動画像収集と分析のために選びました。七日感染後、プレートは、固定された4に記載のように免疫蛍光のために処理され、そのようなCellProfiler(ブロード研究所5.自動画像解析ソフトウェアに記載されるように自動化され、落射蛍光顕微鏡を用いて分析www.cellprofiler.com )、取得したチャネルを処理します独立セグメント( 図3)の比較分析のためのオブジェクトを特定しました。これは、宿主細胞核、細胞輪郭、リソソームおよびコクシエラコロニー ( 図3上パネル)の同定および形態学的特徴付けを可能にします。細胞およびリソソームとコクシエラコロニーの相関identificatiを可能にします上と(正LAMP1、 図3左下のパネルである) コクシエラ含有液胞の具体的な形態学的分析。宿主細胞の輪郭とコクシエラコロニーを相関させること、感染細胞( 図3下部の中央パネル)の同定および特定の形態学的分析を可能にします。最後に、4チャネルは、例示および品質管理の目的( 図3右下のパネル)のためにマージされます。
自動画像解析から得られたデータは、「マルチ表現型の散布図」を得るために、互いに対してプロットすることができます。一例として、 図4Aにコクシエラコロニー (μM2)平均面積は、コクシエラ(複製表現型)の細胞内複製に影響を与える突然変異を同定するために、細胞当たりのコロニー数( 図4A)に対してプロットされ、および/ または宿主細胞に侵入する細菌の能力(INTERNAlization表現型)。統計解析は、軽度(-4 図4A、ピンクと赤のドット)の最も左の部分に分類しました。細胞内のコクシエラ内在に影響を与え突然変異がプロットの底部にグループ分けした( 図4(a)は 、光とダークブルードット)、そして最後に、プロットの一番右の領域では、緑のドットは(非有意な表現型をもたらす変異に対応しますZスコア> -2)。重要なのは、複製に失敗するが、それでも、宿主細胞に侵入することができる変異体は、細胞核( 図4C、第二パネル)をホストするために、隣接する単一の細菌、または小さなコロニーとして、感染の7日後に検出されています。したがって、 コクシエラのサイズ」コロコロ国立環境研究所は「大幅に影響を受けることになりますが、感染した細胞の数は、WT コクシエラに感染した細胞と比較して変化しません。逆に、宿主細胞に侵入するコクシエラの能力に影響を与える変異は、コロニー/細胞の数の減少をもたらします。この数が大幅に1未満である場合には、平均して、感染した細胞の全体数の減少があることを示します。あるいは、 コクシエラコロニーの (ミクロン2)平均面積は、 コクシエラ (細胞傷害性表現型)の細胞傷害性を付与する突然変異を同定するために、感染( 図4B)が生存した宿主細胞の数に対してプロットすることができます。上記のように、統計的分析は、軽度(-4 図3B緑の点)。 37変異軽度影響を受けた宿主細胞の生存( 図3B、光赤ドット)、および7の変異は、細胞の生存( 図3B、暗赤色のドット)をホストするために特に有害でした。実験的なニーズに応じて、自動化された画像解析処理により得られた追加のパラメータは、他のチャートを導出するために使用することができることに注意してください。
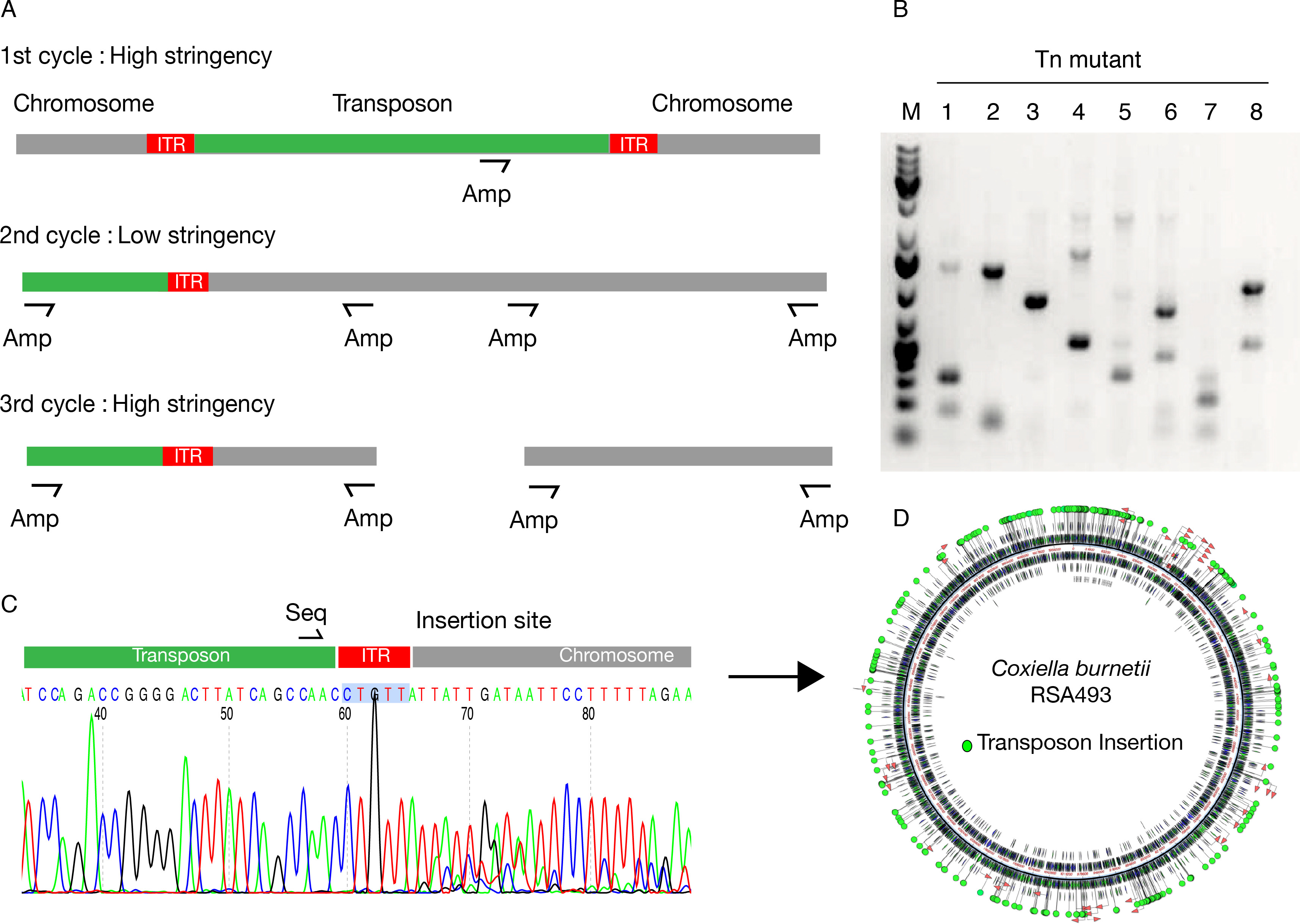
図1:配列決定及びコクシエラトランスポゾン変異体の注釈(A)単一コロニーPCRプライマーは、トランスポゾン挿入の部位を含むDNA断片を増幅するために使用されます。増幅プライマー(アンプ)はアニール温度のストリンジェンシーに依存して特異的および非特異的プライマーの両方として使用されます。 (B)単一のプライマーコロニーPCRの典型的な結果。各をReacti上のトランスポゾン挿入部位を含むいくつかの可変サイズのフラグメントの数を生成します。他のいくつかは、ランダムに副産物低ストPCRサイクルのように増幅されます。 (C)トランスポゾン配列にハイブリダイズする配列決定プライマー(配列)の使用は、目的の断片の配列決定を可能にします。 (D)配列分析ソフトウェアは、細菌ゲノムのトランスポゾン挿入の自動アノテーションを可能にする。 この図の拡大版を表示するには、こちらをクリックしてください。

図2:無菌とコクシエラトランスポゾン変異体の細胞内増殖(A)のパイロットの画面では、我々は、単離し、配列決定し、16コアグラムで38トランスポゾン突然変異体をスクリーニングしていますコクシエラドット/ ICMの分泌系のエンは(赤で示しました)。 (B)各トランスポゾン突然変異体の生存率を評価するために、それぞれの成長は無菌培地中で単離は、蛍光タグ化DNAの挿入剤を使用して、8日間にわたってモニタリングされます。 (C)それぞれの変異体は、その後、上皮細胞に感染するために使用されます。トランスポゾンは、GFPカセットを有しているように、細胞内の細菌の増殖を、マイクロプレートリーダーを使用して、 コクシエラ·レプリケーションに関連するGFP蛍光の変化を追跡することによって、感染の7日にわたって監視されている。 この図の拡大版を表示するには、こちらをクリックしてください。
図3: コクシエラ感染の自動画像解析オート。落射蛍光顕微鏡をトリプリケート96ウェルプレートのウェルあたり画像21の位置に使用されて嵌合さ。画像解析ソフトセグメントは、定量および分析のために取得された各チャネル内のオブジェクト。すべての場合において、画像の境界に触れるのオブジェクトが除外されます。 (A)ヘキストチャネル(緑の円内)は、宿主細胞の核を識別するために使用されます。 (B)これらは(核の位置は青丸で囲まれている、細胞の輪郭が緑色である)をCy3チャネルでの宿主細胞の輪郭を識別するためのシードとして使用されます。 (C)のCy5チャネル(緑の円内)LAMP1陽性区画を識別するために使用されます。以前に同定された細胞の輪郭(赤)に含まれるオブジェクトのみが画像解析のために保持されています。 (D)GFPチャネルが(緑の円内) コクシエラコロニーを同定するために使用されます。 (E)LAMP1陽性コンパートメントとコクシエラコロニーの相関コクシエラの同定を可能にします含有液胞(CCVS)。以前に同定された細胞の輪郭(緑色)に含まれるオブジェクトのみが画像解析のために保持されています。 (F)は、細胞の輪郭とコクシエラコロニーの相関は、感染細胞(疑似カラー)の同定を可能にします。 4蛍光チャネルで取得した(G)画像( コクシエラコロニー (緑)、宿主細胞の核(青)、宿主細胞原形質膜(灰色)、LAMP1陽性区画(赤)に対応する)はマージされ、例示および質のために使用されコントロール。スケールは10μmのバー。 この図の拡大版を表示するには、こちらをクリックしてください。
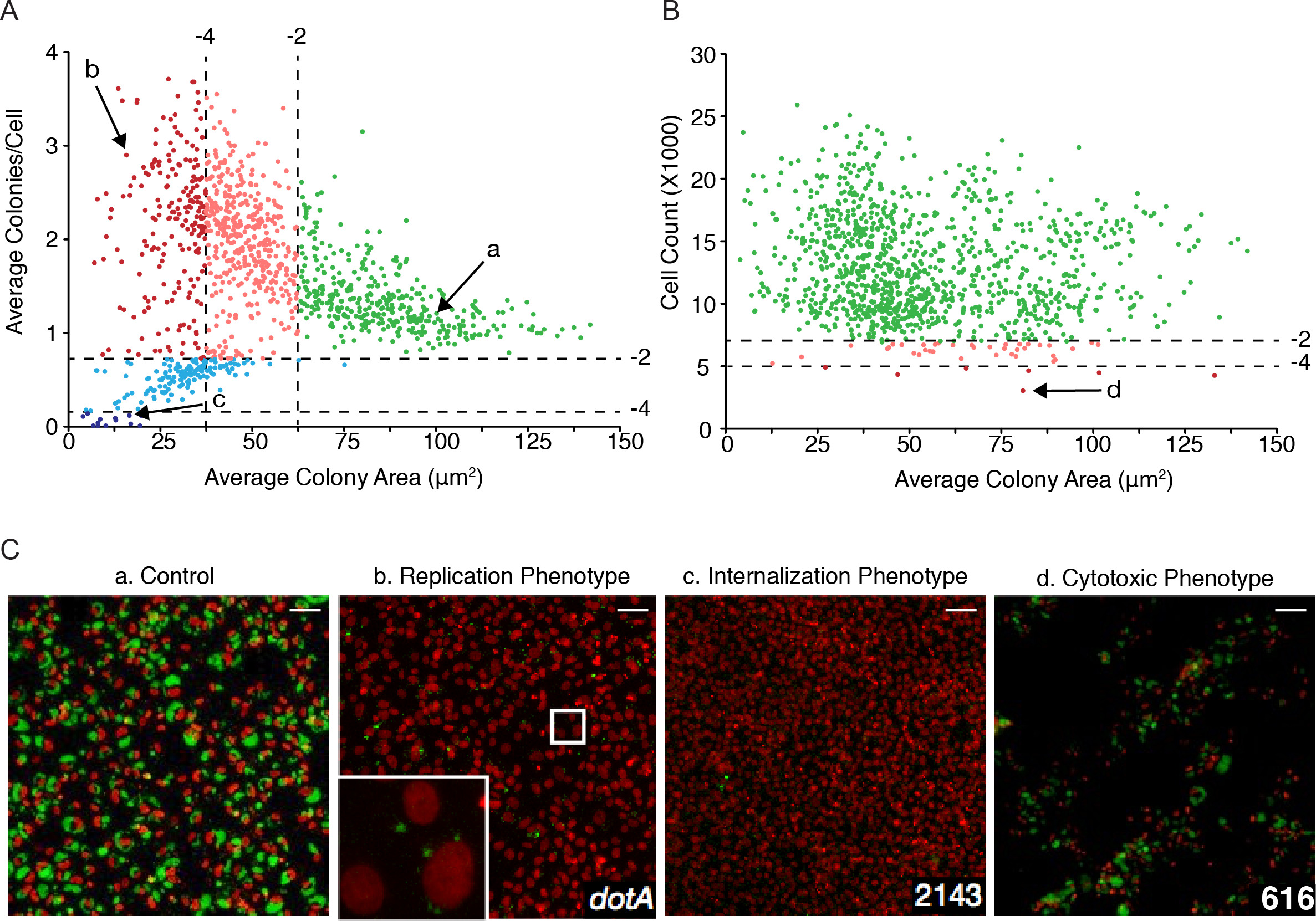
図4:ホスト/病原体相互作用に関与コクシエラ因子の大規模同定イオン(A)は、 コクシエラコロニーの (ミクロン2)平均面積は、関心の複製および内在化の表現型を同定するために、細胞当たりのコロニーの相対数に対してプロットされています。緑の点は、Zスコア> -2(有意ではない)により、WT コクシエラから逸脱した表現型を表します。ピンクと水色のドットが-2、-4(軽度の表現型)との間のZスコアで、それぞれ複製および内在化表現型を表します。赤とダークブルードットは、Zスコア≤-4(強い表現型)との表現型を表します。 コクシエラコロニーの (ミクロン2)(B)の平均面積は、トランスポゾン挿入に起因する細胞毒性効果を推定するために、感染の7日を生き延びた細胞(感染し、感染していない)の合計数に対してプロットしました。緑の点は、Zスコア> -2(有意ではない)により、WT コクシエラから逸脱した表現型を表します。ピンクのドットは、細胞傷害性のpHを表します-2、-4(軽度の表現型)との間のZスコアとenotypes。レッドドットは、Zスコア≤-4(強い表現型)を用いて細胞傷害性表現型を表します。矢印はC.(C)に対応する小文字によって示される変異体複製、内在化および細胞傷害性表現型の代表的な画像を示します。全ての場合において、宿主細胞の核は赤色であり、 コクシエラコロニーは緑色です。スケールは50μmでバー。 この図の拡大版を表示するには、こちらをクリックしてください。
ディスカッション
宿主/病原体相互作用の研究は、細菌感染症を理解し、感染症に対抗するための代替戦略を開発するために注目すべき方法であることが証明されています。しかし、別の細菌性病原体によって精緻化戦略の多様性のために、細菌の毒性因子および感染中に標的にされているホストのシグナル伝達経路の同定および特徴は、本当の挑戦を表します。これは重要な宿主/病原体相互作用のハブの大規模同定のための新たなアプローチの開発を求めています。革新的な、ハイスループットおよびハイコンテントスクリーニング技術の最近の開発は、細胞内細菌病原体15の研究に適合させることができる貴重なリソースを表します。ここでは、トランスポゾン突然変異誘発および蛍光ベースのアッセイを組み合わせたスクリーニング手法を開発するためのモデルとして、人獣共通細菌性病原体のQ熱リケッチアを使用しています。 ImportanTLY、このスクリーニング方法は、侵入複製し、感染した細胞内で永続化するために、この細菌によって開発された戦略のグローバルな概要を提供し、 コクシエラ細胞内サイクルの複数のステップを同時に監視することができます。
ここで説明するアプローチは、2つのよく確立された技術、トランスポゾン突然変異誘発し、正常細菌性病原体の研究に適用された蛍光に基づいたアッセイに基づいています。高スループット/高含量スクリーンのコンテキストでこれらの技術を組み合わせることで、私たちはイベントの非常に高い数(典型的には、細菌の変異15,000感染した細胞は、画像化され、分析される)を分析することによって、細菌の変異の数が多いの効果を評価することができます。これは、このような、自然に、高い変動の対象である宿主細胞と細胞内複製、細菌の侵入などのイベントの重要な統計的分析を提供します。これは、EP以外のその細胞株に注目することが重要ですithelialスクリーニングのこのタイプのために使用することができます。宿主細胞の細胞小器官を検出することが容易であるしかし、平らで大きな上皮細胞画像解析のために最適です。自動化された顕微鏡の大部分が自動的にプレートを大量に処理することができるので、同時にスクリーニングすることができる変異体の数に事実上制限はありません。病原体に応じて、ユーザー缶特権落射蛍光または共焦点顕微鏡を使用します。画像取得の時間は、主にウェルあたりと視野当たりの取得チャネル数、取得フィールドの数に、顕微鏡カメラの感度に依存することになります。ユーザーは、スクリーニングプロトコルを最適化するために、これらの要因を調整する方法を決めることができます。例として、点5.1で示される条件を使用して1つの96ウェルプレート/時間をイメージ。画像分析は、主に使用されるマシン(またはマシンのクラスタ)に依存します。我々は、12コア(2×3.06 GHzの6コア)、48ギガバイトのRAMのワークステーションを使用しています。このマシンはapproxiが必要です一枚の板から取得した画像を解析するmately 40分。
これらのアッセイを開発する際に考慮すべき重要な側面は、多数のサンプルの操作および処理を可能にする新規のセットアップ(または既存の最適化)のプロトコルです。典型的な例は、私たちは急速に非常に少量のサンプルから、各トランスポゾンの挿入部位を含むコクシエラ DNA断片を増幅し、配列することができ、単一のプライマーコロニーPCR法の開発です。我々の経験に基づいて、高忠実度ポリメラーゼを注意深く選択し、再現性のある結果を得るために試験されなければなりません。このアプローチの唯一の制限は、ほとんどの場合、処理されたサンプルの約30%が悪用可能でない、観察にPCRまたは配列決定の段階に起因するいずれかを非表示にすることができます。しかし、新しいコクシエラトランスポゾン突然変異体の単離が律速段階ではないことを考えると、これはrepreしません大きな問題を送りました。同様に、変異型株式の細菌濃度を定量化するための信頼性の高いアッセイの開発は、このアプローチのための鍵となっています。懸濁液中の凝集するコクシエラの傾向に起因して、光学密度の測定値を使用することはあったコクシエラの文化や、既存の代替の濃度定量PCR(定量PCR)を計算するためには適用されません。ここで、蛍光タグ化DNAの挿入剤の使用は、細菌の定量化を高速化。
このアプローチは、使用される病原体に応じて、いくつかの細胞内区画のための蛍光マーカーを発現する安定な細胞株の使用を利用することができます。もう一つの重要な側面は、フェノールレッドを欠く細胞培養培地の使用です。我々は、このpH指示薬は、自動化された蛍光リーダーに記録された信号を飽和赤と緑のスペクトルにまたがる自然蛍光を有していることを観察しました。
Tここで紹介する彼の戦略は、ランダムトランスポゾン突然変異誘発に依存しています。興味のある変異体のために、私たちは、サザンブロットおよびトランスポゾン挿入部位のPCR増幅を使用してユニークな入れ換えを検証(およびクローン)をお勧めします。
プロトコルセクションに記載された装置に加えて、ここに提示スクリーニングアプローチを使用して興味のチームは、データ収集、データ格納用のサーバと迅速な画像解析のためのワークステーションのためにリレーショナルデータベースのセットアップに非常に有利になります。
重要なことは、ここで説明する方法は、ランダム突然変異誘発法は、病原体のために存在する提供他の細胞内細菌性病原体の研究に適している、細胞株は、病原体に感染することができ、これは、感染の間に、特定の表現型を表示します。
開示事項
The authors declare that they have no competing financial interests.
謝辞
This work was supported by an Avenir/ATIP grant to Matteo Bonazzi and a Marie Curie CIG (N° 293731) to Eric Martinez. The authors would like to thank Dr. Robert Heinzen for sharing C. burnetii strains, antibodies and tools, Virginie Georget and Sylvain DeRossi (Montpellier Rio Imaging-MRI, 1919 route de Mende, 34293 Montpellier) for their technical assistance and data analysis.
資料
| Name | Company | Catalog Number | Comments |
| Citric acid | Sigma | C0759-500G | ACCM-2 medium component |
| Sodium citrate | Sigma | S4641-500G | ACCM-2 medium component |
| Potassium phosphate | Sigma | 60218-100G | ACCM-2 medium component |
| Magnesium chloride | Sigma | M2670-100G | ACCM-2 medium component |
| Calcium chloride | Sigma | C5080-500G | ACCM-2 medium component |
| Iron sulfate | Sigma | F8633-250G | ACCM-2 medium component |
| Sodium chloride | Sigma | S9625-500G | ACCM-2 medium component |
| L-cysteine | Sigma | C6852-25G | ACCM-2 medium component |
| Bacto Neopeptone | BD (Beckton-Dickinson) | 211681 | ACCM-2 medium component |
| Casamino acids | BD (Beckton-Dickinson) | 223050 | ACCM-2 medium component |
| Methyl-B-Cyclodextrin | Sigma | C4555-10G | ACCM-2 medium component |
| RPMI w/glutamax | Gibco | 61870-010 | ACCM-2 medium component |
| RPMI w/glutamax, without phenol red | Gibco | 32404-014 | For cell culture and infection |
| Fetal Bovine Serum | GE healthcare | SH30071 | For cell culture |
| Trypsin EDTA (0.25%) | Life Technologies | 25200-056 | For cell culture |
| Saponin | Sigma | 47036 | For immunofluorescence staining |
| Ammonium chloride | Sigma | A9434 | For immunofluorescence staining |
| Bovine Serum Albumine | Sigma | A2153 | For immunofluorescence staining |
| Electroporation cuvette 0.1 cm | Eurogentec | ce0001-50 | For Coxiella transformation |
| Trackmates screw top tubes with caps | Thermo Scientific | 3741 | 2D barcoded screwcap |
| 96-well microplate with black walls and bottom | Greiner | 655076 | Flat dark bottom, for dsDNA quantitation |
| 96-well microplate, ��clear, black walls | Greiner | 655090 | Flat transparent bottom, for cell culture and imaging |
| 96-well PCR microplate | Biorad | 2239441 | DNAse/RNAse free |
| 96-well plate, Deepwell | Labcon | 949481 | For Coxiella mutants infections |
| PicoGreen | Life Technologies | P7581 | For dsDNA quantitation |
| Triton X-100 | Sigma | T9284-500ML | For dsDNA quantitation |
| Phusion High fidelity DNA Polymerase | New England Biolabs | M0530L | For single primer colony PCR |
| Celltracker Red CMTPX | Life Technologies | C34552 | For imaging |
| Anti-LAMP1 antibody | Sigma | 94403-1ML | For immunofluorescence staining |
| Hoechst 33258 | Sigma | L1418 | For imaging |
| Fluorescence microplate reader Infinite 200 Pro | Tecan | ||
| Epifluorescence automated microscope Cellomics | Thermo Scientific |
参考文献
- Maurin, M., Raoult, D. Q fever. Clinical microbiology reviews. 12 (4), 518-553 (1999).
- Kim, S. G., Kim, E. H., Lafferty, C. J., Dubovi, E. Coxiella burnetii. in bulk tank milk samples, United States. Emerging infectious diseases. 11, 619-621 (2005).
- Kazar, J. Coxiella burnetii. infection. Annals of the New York Academy of Sciences. 1063, 105-114 (2005).
- Madariaga, M. G., Rezai, K., Trenholme, G. M., Weinstein, R. A. Q fever: a biological weapon in your backyard. The Lancet infectious diseases. 3, 709-721 (2003).
- Der Hoek, W. V. a. n., et al. Epidemic Q fever in humans in the Netherlands. Advances in Experimental Medicine and Biology. 984, 329-364 (2012).
- Capo, C., et al. Subversion of monocyte functions by Coxiella burnetii.: impairment of the cross-talk between alphavbeta3 integrin and CR3. Journal of immunology. 163 (11), 6078-6085 (1999).
- Martinez, E., Cantet, F., Fava, L., Norville, I., Bonazzi, M. Identification of OmpA, a Coxiella burnetii. protein involved in host cell invasion, by multi-phenotypic high-content screening. PLoS pathogens. 10 (3), e1004013 (2014).
- Romano, P. S., Gutierrez, M. G., Berón, W., Rabinovitch, M., Colombo, M. I. The autophagic pathway is actively modulated by phase II Coxiella burnetii. to efficiently replicate in the host cell. Cellular microbiology. 9 (4), 891-909 (2007).
- Newton, H. J., Mcdonough, J. a., Roy, C. R. Effector protein translocation by the Coxiella burnetii. Dot/Icm type IV secretion system requires endocytic maturation of the pathogen-occupied vacuole. PloS one. 8 (1), e54566 (2013).
- Vogel, J. P. Turning a tiger into a house cat: using Legionella pneumophila. to study Coxiella burnetii.. Trends in microbiology. 12 (3), 103-105 (2004).
- Van Schaik, E. J., Chen, C., Mertens, K., Weber, M. M., Samuel, J. E. Molecular pathogenesis of the obligate intracellular bacterium Coxiella burnetii. Nature reviews. Microbiology. 11, 561-573 (2013).
- Beare, P. A., et al. Dot/Icm type IVB secretion system requirements for Coxiella burnetii growth in human macrophages. mBio. 2, (2011).
- Omsland, A., et al. Isolation from animal tissue and genetic transformation of Coxiella burnetii. are facilitated by an improved axenic growth medium. Applied and environmental microbiology. 77 (11), 3720-3725 (2011).
- Beare, P. a., Sandoz, K. M., Omsland, A., Rockey, D. D., Heinzen, R. a Advances in genetic manipulation of obligate intracellular bacterial pathogens. Frontiers in microbiology. 2 (May), 97 (2011).
- Brodin, P., Christophe, T. High-content screening in infectious diseases. Current opinion in chemical biology. 15 (4), 534-539 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved