Method Article
Génération et multi-phénotypique criblage à haut contenu de
Dans cet article
Résumé
Coxiella burnetii est une bactérie intracellulaire Gram négatif obligatoire responsable de la maladie zoonotique la fièvre Q. Nous décrivons ici des procédés pour la génération de mutants de transposon Coxiella fluorescent ainsi que l'identification et l'analyse automatisée de l'internalisation, la réplication et cytotoxiques phénotypes résultants.
Résumé
Invasion et la colonisation des cellules hôtes de bactéries pathogènes dépendent de l'activité d'un grand nombre de protéines procaryotes, défini comme étant des facteurs de virulence, qui peut renverser et manipuler des fonctions essentielles de l'hôte. L'étude des interactions hôte / pathogène est donc extrêmement important de comprendre les infections bactériennes et développer des stratégies alternatives pour contrer les maladies infectieuses. Cependant, cette approche nécessite le développement de nouveaux tests à haut débit pour la impartiale, l'identification automatisée et la caractérisation des facteurs de virulence bactériens. Ici, nous décrivons un procédé pour la production d'une banque de mutants GFP-étiqueté par mutagenèse par transposon et le développement d'un criblage à haut contenu approches pour l'identification simultanée de plusieurs phénotypes de transposon associé. Notre modèle de travail est le bactérienne pathogène Coxiella burnetii intracellulaire, l'agent étiologique de la fièvre Q zoonose, qui est associé à soivere les épidémies de la santé avec une conséquente et le fardeau économique. La nature intracellulaire obligatoire de cet agent pathogène a, jusqu'à récemment, gravement entravé l'identification des facteurs bactériens impliqués dans les interactions hôte-pathogène, faisant de Coxiella le modèle idéal pour la mise en œuvre d'approches à haut débit / haute-contenu.
Introduction
La bactérie Coxiella burnetii émergente endémique est responsable de grandes épidémies de fièvre Q, une zoonose débilitante pseudo-grippal avec la santé et l'impact économique grave 1. Les principaux réservoirs de Coxiella sont des animaux domestiques et d'élevage, et on estime que plus de 90% des bovins laitiers aux États-Unis portent C. burnetii 2. Les humains sont des hôtes accidentels qui sont infectés par inhalation d'aérosols contaminés. La fièvre Q humaine manifeste soit comme une maladie aiguë ou chronique, qui peut avoir des complications mortelles avec un taux de mortalité atteignant 65% 1,3. Avec une dose infectieuse de 1 - 10 organismes, Coxiella est le pathogène le plus infectieux connu et il a été étudié comme un bio arme potentielle 4. L'épidémie récente de explosive fièvre Q aux Pays-Bas (2007 - 2010), avec des cas escalade de 182 à plus de 2000 par an, est un exemple de la virulence de cet agent pathogène grave5.
L'efficacité remarquable des infections Coxiella est probablement associée à sa résistance au stress environnemental, combiné avec son adaptation unique de cellules hôtes. En effet, Coxiella est présent dans l'environnement sous forme de variantes métaboliquement inactifs à petites cellules (SCV), qui sont remarquablement résistante à plusieurs conditions difficiles (dessiccation, température, etc.). VCS sont absorbé par des cellules phagocytaires via α V β 3 intégrines 6 tandis que l'invasion des cellules non-phagocytaires est médiée par la Coxiella adhérence / invasion OmpA 7 et un récepteur non encore identifié. Après absorption, Coxiella réside dans les vacuoles moulants, positifs pour les marqueurs endosomes précoces et Rab5 EEA1 8. Les bactéries répondent aux acidification endosomique en convertissant des variantes de cellules grandes métaboliquement actifs (VUL) et l'activation d'un type 4 système de sécrétion Dot / Icm (T4SS) 9, Fortement homologue à celle de 10 Legionella pneumophila. La sécrétion d'effecteurs Dot / ICM permet Coxiella pour générer un grand compartiment acide, LAMP1 positif contenant des enzymes lysosomales actifs où les bactéries peuvent se développer et protéger activement les cellules infectées de l'apoptose 11. Par conséquent, le cycle intracellulaire de Coxiella est contrôlée par la translocation Dot / Icm d'effecteurs à médiation par 12 bactériennes, cependant, les facteurs impliqués dans l'invasion microbienne de la cellule hôte, la replication bactérienne et la diffusion de l'infection restent en grande partie inconnus.
Combinant mutagenèse de transposon et des essais basés sur la fluorescence, nous développons des approches impartiales pour l'identification simultanée de facteurs bactériens impliqués dans les principales étapes d'infections Coxiella: 1) l'internalisation dans les cellules hôtes, 2) la réplication intracellulaire, 3) la propagation de cellule à cellule et 4) la persistance. À ce jour, nous avons passé au crible plus de 1000 mutations dans 500 séquences codantes Coxiella, qui nous ont fourni des perspectives sans précédent dans les interactions hôte-pathogène qui régulent Coxiella pathogenèse 7. Fait à noter, cette approche peut être appliquée à l'étude d'autres pathogènes intracellulaires qui partagent des caractéristiques de biologie cellulaire avec Coxiella.
Protocole
1. La génération d'une banque de mutants de transposon Coxiella GFP étiqueté
Manipuler Coxiella burnetii RSA439 NMII dans un niveau de confinement 2 de biosécurité (BSL-2) dans une enceinte de sécurité microbienne (MSC) en conformité avec les règles locales. Si compatible avec le modèle bactérienne utilisée, répétez les étapes à partir de 1.4.1 à 1.4.4 pour augmenter la probabilité d'obtention de mutants clonales. Une banque de mutants typique est composé (au moins) d'un certain nombre de mutants qui est égale à trois fois le nombre de séquences annotées dans le génome de l'organisme utilisé codage.
- Préparation de électrocompétente Coxiella RSA439 NMII:
- Préparer 1x ACCM-2 13: 13,4 mM d'acide citrique, le citrate de sodium 16,1 mM, phosphate de potassium 3,67 mM, chlorure de magnésium 1 mM, 0,02 mM de chlorure de calcium, mM sulfate 0,01 de fer, le chlorure de sodium 125,4 mM, 1,5 mM de L-cystéine, 0,1 g / L Bacto Neopeptone, 2,5 g / l de casaminoacides, 1 g / L méthyle bêta cyclodextrine, 125 ml / L RPMI. Ajuster le pH à 4,75 et stériliser par filtration (ne pas autoclaver). Remarque: Liquide ACCM-2 est stable à 4 ° C pendant environ 1 mois.
- Inoculer 100 ml d'ACCM-2 avec 2 x 10 6 génome équivalent (GE) / ml de Coxiella RSA439 NMII (à partir d'un stock bactérienne préalablement généré et quantifiés comme dans l'étape 1.5) à partir des stocks C -80 ° et distribuer la suspension bactérienne dans 75 cm 2 cellules des flacons de culture avec des bouchons ventilés (10 - 15 ml de suspension bactérienne par flacon). Croître pendant 7 jours à 37 ° C dans une atmosphère humidifiée de 5% de CO 2 et de 2,5% d'O 2.
- Piscine La suspension bactérienne résultant en tubes de 50 ml et centrifuger à 3900 g pendant 1 h à 4 ° C.
- Jeter le surnageant et remettre le culot dans 30 ml de 10% de glycérol. Centrifuger à 3900 g pendant 1 h à 4 ° C.
- Remettre en suspension le culot dans un volume suffisant de glycerol à 10% (typiquement 2 ml) et 50 aliquote de 500 pl dans des tubes ul. Gardez b remis en suspensionacteria sur la glace pendant tout le processus. Remarque: A ce stade, les bactéries sont électrocompétente et une aliquote est suffisante pour effectuer une électroporation. Les suspensions bactériennes peuvent être conservés à -80 ° C pendant 6 mois ou être utilisées directement pour l'électroporation de l'ADN plasmidique.
- Électroporation de Coxiella compétente transposon- et les plasmides de transposase codant:
Remarque: pour le protocole suivant, l'élément transposable et la transposase sont codées par deux plasmides différents (PITR-CAT-GFP et pUC19-Himar1C9, respectivement) 7. Les deux plasmides ont pas une origine de réplication spécifique à Coxiella, ce qui en fait des plasmides suicides lorsque électroporé dans Coxiella. Cela garantit insertions de transposons stables. L'élément transposable contient une cassette de résistance au chloramphénicol sous la régulation du promoteur p1169 Coxiella pour la sélection et le gène de la GFP sous la régulation du promoteur p311 Coxiella pour marquer les mutants générés avec GFP.- Pre-refroidir une cuvette d'électroporation de 0,1 cm pendant 10 min sur de la glace. Mélanger 50 pi de électrocompétente Coxiella avec 10 ug transposon plasmide et 10 ug de plasmide transposase 7. Assurez-vous que la concentration de plasmide est supérieure à 500 pg / ml afin de minimiser la dilution du glycérol.
- Électroporation en utilisant la configuration suivante: 18 kV, 500 Ω, 25 pF. Assurez-vous que la constante de temps résultante est comprise entre 9 et 13 msec.
- Immédiatement ajouter 950 pi de RPMI, remettre en suspension les bactéries électroporation et les transférer dans un tube à bouchon à vis et conserver à la température ambiante.
- Prendre 200 ul des bactéries électroporées et à ajouter 3 ml d'ACCM-2 supplémenté avec 1% inactivé par la chaleur de sérum bovin fœtal (FBS) dans des plaques à 6 puits. Ajouter 88 pi de DMSO pour le volume restant de bactéries électroporées (pour atteindre une concentration finale de 10% de DMSO) et conserver à -80 ° C.
- La sélection des mutants de transposon:
- Incubate des plaques à 6 puits inoculés comme décrit ci-dessus (1.2.4) pendant une nuit à 37 ° C dans une atmosphère humidifiée de 5% de CO 2 et de 2,5% d'O 2. Ajouter les antibiotiques appropriés (375 ug / ml de kanamycine ou 3 ug / ml de chloramphenicol). Incuber la culture bactérienne pendant 3 jours supplémentaires dans les conditions décrites ci-dessus.
- Isolement de mutants individuels:
- Préparation de solides ACCM-2 plaques et le placage de mutants de transposons Coxiella
Remarque: Les instructions suivantes sont pour une boîte de Pétri, plusieurs dilutions de cultures bactériennes doivent être testés pour évaluer le volume d'inoculation optimale pour l'isolement de la colonie.- Chauffer 10,5 ml de 0,5% d'agarose dans un micro-ondes et laisser refroidir dans un bain d'eau à 55 °. Chauffer 11,25 ml de 2x ACCM-2 (pH 4,75) à 37 ° C.
- Préparer agarose bas:
- Mélanger 10 ml de 0,5% d'agarose fondu avec 10 ml de 2x-2 ACCM et ajouter les antibiotiques appropriés (375 ug / ml de kanamycine ou 3ug / ml de chloramphenicol).
- Verser immédiatement dans la boîte de Pétri. Gardez le plat unlidded Petri, laisser refroidir moyen pendant 30 min et sécher à l'air pendant 20 min.
- Préparer top agarose:
- Mélanger 1,25 ml de 2x ACCM-2 avec 0,75 ml d'eau dans un tube 5 ml de polystyrène, ajouter les antibiotiques appropriés (375 ug / ml de kanamycine ou 3 ug / ml de chloramphenicol) et incuber à 37 ° C.
- Ajouter la culture bactérienne (typiquement de 1 à 100 ul) et vortex pendant 5 s.
- Ajouter 0,5 ml d'agarose fondu, mélanger et verser immédiatement sur le agarose bas.
- Laisser refroidir pendant 20 min, remettre le couvercle sur la boîte de Pétri et incuber à 4 ° C pendant 20 min pour faciliter la solidification agarose.
- Sécher à l'air pendant 20 min dans un unlidded MSC. Cultiver les plaques à 37 ° C dans une atmosphère humidifiée de 5% de CO 2 et de 2,5% d'O 2 pendant 6 à 7 jours.
- Ajouter du DMSO pour les bactéries restantesl cultures afin de parvenir à une concentration finale de 10% de DMSO et conserver à -80 ° C.
- Évaluer la dilution optimale de la façon suivante: faire en sorte que les colonies sont de 0,5 à 1 mm de diamètre et sont convenablement isolées pour éviter la contamination croisée. Décongeler restant cultures bactériennes à partir du point 1.4.2 et la plaque à la dilution appropriée sur ACCM-2 agar comme décrit dans 1.4.1.2 et 1.4.1.3. Incuber pendant 6 à 7 jours comme décrit dans 1.4.1.3.5.
- Une fois que les colonies sont détectables, les recueillir en coupant l'extrémité d'une pointe de 1 ml, la cueillette de la fiche contenant des colonies isolées et la dispersion de la colonie par pipetage dans 1,5 ml d'ACCM-2 contenant les antibiotiques appropriés (375 pg / ml de kanamycine ou 3 pg / ml de chloramphénicol) dans une plaque de 24 puits. Amplifier colonies individuelles pendant 6 jours dans les conditions décrites au paragraphe 1.3.1. Au jour 3 de l'incubation, disperser les amas bactériens par pipetage chaque culture.
- Stocker chaque suspension mutant dans 2D tubes BOUCHON code à barres dans des plaques à 96 puits dans 10% De DMSO à -80 ° C.
- Préparation de solides ACCM-2 plaques et le placage de mutants de transposons Coxiella
- L'évaluation de la concentration bactérienne:
Remarque: le protocole suivant peut être appliqué pour obtenir les courbes des mutants à replication bactériennes dans un milieu axénique (voir 1.4.4) de croissance.- Norme préparation de la courbe:
- Préparer un / stock ml solution à 2 pg d'ADN double brin (typiquement un plasmide aléatoire de taille connue et la concentration) en 1x Tris-EDTA (TE). Préparer 10 dilutions successives de la solution mère pour obtenir des concentrations allant de 2 pg / ml à 2 ng / ml. Distribuer 50 pi de chaque concentration de puits individuels d'une microplaque de 96 puits avec des murs noirs et le fond (voir le tableau des matériaux).
- On dilue le réactif quantification ADNdb 1: 200 dans du tampon 1 x TE et ajouter 55 ul du réactif dilué dans chaque échantillon dans la microplaque à 96 puits. Mélangez bien à l'aide d'un agitateur de plaque et incuber pendant 2 à 5 min à température ambiante, dans l'obscurité.
- Mesurer la fluorescence des échantillons en utilisant une fluorescence unence lecteur et filtres microplaque pour des longueurs d'onde standards de fluorescéine (~ excitation 480 nm, émission ~ 520 nm).
- Tracer la gamme de concentration de plasmide contre les lectures d'intensité de fluorescence.
- Suspension bactérienne quantification:
- Distribuer 5 pi de 10% de Triton X-100 par puits dans une microplaque de 96 puits avec des murs noirs et le fond (voir le tableau des matériaux). Ajouter 50 pi des suspensions bactériennes dans chaque puits et incuber 10 min à température ambiante, sur un agitateur de plaque.
- On dilue le réactif quantification ADNdb 1: 200 dans du tampon 1 x TE et ajouter 55 ul du réactif dilué dans chaque échantillon dans la microplaque à 96 puits. Mélangez bien à l'aide d'un agitateur de plaque et incuber pendant 2 à 5 min à température ambiante, dans l'obscurité.
- Mesurer la fluorescence des échantillons en utilisant un lecteur et des filtres de fluorescence pour microplaques longueurs d'onde standards de fluorescéine (~ excitation 480 nm, émission ~ 520 nm).
- Pour obtenir le DN bactérienneUne concentration, tracer les lectures de fluorescence dans le tableau obtenu au point 1.5.1.4. Diviser la concentration d'ADN par la masse du génome de Coxiella (2,2 fg) pour obtenir des concentrations bactériennes. Exprimez résultats de génome / ml équivalentes.
- Jeter les mutants présentant un défaut de croissance significative dans ACCM-2.
- Norme préparation de la courbe:
2. Primer seule colonie PCR, séquençage et d'annotation
Remarque: le protocole suivant est pour l'amplification d'ADN de 96 échantillons, une pipette multicanaux est recommandé pour les étapes suivantes. purification de la colonne de produits de PCR utilisant des billes magnétiques et séquençage de l'ADN avec une amorce spécifique-transposon (2.3) sont sous-traitée à une société externe.
- Assurez-vous que l'amorce d'amplification est conçue pour hybrider entre 100 et 200 paires de bases en amont de l'unité de répétition en tandem inverse (ITR), pour obtenir des produits de PCR couvrant le site d'insertion du transposon sur Coxiellun génome. Préparer 3 ml de mélange de PCR (1x tampon de haute fidélité, 200 uM de dNTP, 1 uM amorce d'amplification, 20 U / ml de haute fidélité de l'ADN polymérase) et distribuer 29 pi par puits dans une plaque à 96 puits PCR mis sur la glace. Transfer 1 ul de chaque mutant en phase stationnaire dans ACCM-2 au mélange de PCR.
- Exécuter PCR avec une dénaturation initiale (98 ° C, 1 min), 20 cycles de haute stringence (98 ° C, 10 s; 50 ° C, 30 s; 72 ° C, 90 sec), 30 cycles de faible stringence (98 ° C, 10 sec; 30 ° C, 30 s; 72 ° C, 90 sec) et 30 cycles de stringence élevée (98 ° C, 10 s; 50 ° C, 30 s; 72 ° C, 90 sec) suivi d'une extension finale à 72 ° C pendant 7 min.
- Purifier produits de PCR utilisant des billes magnétiques et de l'ADN de séquence avec une amorce spécifique transposon. Concevoir l'amorce spécifique de transposon avec une température de fusion comprise entre prédite 50 ° C et 75 ° C, une teneur en GC comprise entre 40% et 60%, une longueur comprise entre 18 et 25 nucléotides et si un de recuitte en aval du site d'hybridation d'amorce et l'amplification d'au moins 100 paires de bases en amont de la première paire de l'ITR du transposon de base.
- Grâce à un logiciel d'analyse de séquence, charger le complète, génome annoté de Coxiella burnetii 493 NMI. Utilisez la fonction "aligner à la référence" pour charger et aligner (blastn) les résultats de séquençage et de déterminer le site de la transposition. Jeter mutants avec des non-appariement et / ou l'affichage à double reads.To surveiller la saturation de la bibliothèque mutant, garder une trace de la présence de multiples insertions de transposons sur le même site.
3. Les cellules eucaryotes défi avec Coxiella Mutants et la surveillance des intracellulaire croissance
Remarque: Une pipette multicanaux est recommandée pour les étapes suivantes. Les infections ont été effectuées en triple dans stérile microplaques de 96 puits avec des murs noirs et fond transparent plat. Coxiella burnetii poids exprimant la GFP 14 wtel que prévu par le Dr Robert Heinzen.
- Cultiver des cellules Vero dans du RPMI sans rouge de phénol supplémenté avec 10% de sérum fœtal bovin (FBS) en l'absence d'antibiotiques (RPMI complet médias).
- Le jour avant l'infection, se laver les cellules Vero confluentes à partir d'un sous-confluente ou ballon de culture de cellules avec 10 ml de PBS.
- Détacher les cellules Vero en ajoutant 1 ml de solution de trypsine EDTA dans le ballon de culture de cellules et incuber pendant 3 à 5 min à 37 ° C dans une atmosphère humidifiée de 5% de CO 2.
- Resuspendre les cellules dans 10 ml de milieu RPMI complet. Compter les cellules et à préparer une suspension cellulaire de 10 5 cellules par ml.
- Distribuer 100 ul de la suspension cellulaire dans chaque puits d'une plaque de 96 puits noir avec fond transparent plat.
- Centrifuger pendant 5 minutes à 400 x g à la température ambiante pour faciliter l'adhérence des cellules au fond des puits et incuber pendant une nuit à 37 ° C dans une atmosphère humidifiée de 5% de CO 2.
- Décongeler les plaques à 96 puits contenant du Comutants xiella à RT et diluer 150 pi de suspension bactérienne dans 300 pi de RPMI sans rouge de phénol et de FBS dans un puits profond plaque de 96 puits.
- Retirez le papier de la microplaque contenant des cellules Vero et distribuer 100 pl / puits de mutants Coxiella dilué (MOI de 100). Utilisez le puits A1 comme négatif (cellules non infectées) A2 de contrôle et de puits et A3 comme contrôles positifs (cellules infectées par Coxiella poids exprimant la GFP 14 à des multiplicités d'infections (MOI) de 100 et 200).
- Centrifuger la plaque pendant 10 min à 400 g à température ambiante en utilisant un support de plaque d'centrifugeuse étanche aux aérosols.
- Incuber à 37 ° C dans une atmosphère humidifiée de 5% de CO 2 pendant 2 heures puis remettez-les bactéries contenant le milieu avec 100 pl / puits de, milieu frais RPMI complet.
- Mesurer la fluorescence de la GFP tous les jours pendant 7 jours en utilisant un lecteur et des filtres de fluorescence pour microplaques longueurs d'onde standards de fluorescéine (~ excitation 480 nm, émission ~ 520 nm). Éviterperturbations dues à la condensation et à la dispersion du signal dans le milieu de culture, en utilisant une excitation de fond et enregistrement de l'émission sur le lecteur de microplaques.
4. Préparation des échantillons pour l'acquisition d'images automatisé
Remarque: La procédure est pour une plaque de 96 puits, l'échelle des volumes en conséquence. Étapes de 4.2 peuvent profiter d'un laveur de plaques.
- Le 7 après l'infection d'une journée, éliminer le milieu de la plaque et les remplacer par 50 pl / puits de milieu complet frais contenant un colorant fluorescent perméable cellulaire à la dilution appropriée (habituellement de 1: 1000, doit être optimisé en fonction de la lignée cellulaire utilisée ). Incuber les cellules pendant 30 - 60 min à 37 ° C dans une atmosphère humidifiée de 5% de CO 2.
- Remplacer milieu avec 50 pl / puits de 4% de paraformaldehyde (PFA) dans du PBS, incuber pendant 30 min à température ambiante (TA) puis retirer le tampon contenant du PFA et laver 3 fois avec du PBS.
- Retirer du PBS et Dispense 50 pl / puits de solution de blocage (0,5% d'albumine de sérum bovin, 50 mM de NH 4 Cl dans du PBS, pH 7,4) additionné de 0,05% de saponine. Incuber à température ambiante pendant 30 min.
- Remplacer solution de blocage avec 40 pl / puits de solution de blocage frais supplémenté avec de la saponine (comme ci-dessus) et avec un anticorps anti-LAMP1 à une dilution de 1: 500. Incuber la plaque pendant 30 min à température ambiante.
- Retirer la solution de blocage et laver la plaque de 96 puits 5 fois avec 100 pl / puits de PBS.
- Distribuer 40 pl / puits de solution de blocage additionné de saponine (comme ci-dessus), l'anticorps secondaire marqué par fluorescence approprié (à une dilution de 1: 1000) pour révéler l'anticorps anti-LAMP1 appliqué à l'étape 4.4, et avec Hoechst 33258 à 5 pg / ml. Incuber la plaque pendant 30 min à température ambiante.
- Retirer la solution de blocage et laver la plaque de 96 puits 5 fois avec 100 pl / puits de PBS. Laissez le volume de PBS correspondant au dernier lavage dans la plaque de 96 puits, que les cellules fixées ne doivent pas sécher.
- L'image de la plaque immédiatement ou plaque de conserver à 4 ° C, à l'abri de la lumière, pour une analyse ultérieure.
5. Image Acquisition
- Acquérir des images de la GFP (488 nm, bactéries), Hoechst 33258 (350 nm, la cellule hôte de noyaux), rouge (~ 555 nm, la membrane cellulaire de marqueur) et rouge lointain (~ 615 nm, LAMP1) canaux en utilisant une épifluorescence automatisé microscope équipé avec un objectif 20X. Acquérir 21 champs indépendants par puits pour l'image d'un minimum de 5 000 cellules par échantillon. Appliquer autofocus en utilisant le canal noyaux de la cellule hôte comme une référence. Lorsque vous travaillez avec des bactéries pathogènes infectant un faible pourcentage de cellules hôtes, les utilisateurs peuvent ajuster le nombre de champs indépendants imagées par puits, afin d'obtenir un minimum de 500 cellules infectées à analyser.
6. Traitement de l'image
Note: les étapes suivantes sont spécifiques pour l'utilisation de la CellProfiler logiciel d'analyse d'image. Dans tous les cas, la algorit optimalehm pour la segmentation doit être définie expérimentalement et les objets touchant la frontière de l'image doit être éliminée avec la fonction appropriée.
- Charger toutes les images dans CellProfiler.
- Utilisez le module "ImageMath" pour soustraire le canal de GFP du canal Hoechst, pour éviter la détection de colonies Coxiella (également marquées par Hoechst) que les noyaux des cellules de l'hôte dans les étapes suivantes.
- Utilisez le module "IdentifyPrimaryObjects" à noyaux cellulaires secteur d'accueil de l'image résultant de l'étape 6.2. Nommez les objets segmentés «noyaux».
- Utilisez le module "IdentifySecondaryObjects" à cellules segments d'accueil de 555 nm en utilisant les images des noyaux détectées à l'étape 6.3 comme semences. Nommez les objets segmentés «Cellules».
- (Facultatif) Utilisez le module "IdentifyTertiaryObjects" pour soustraire noyaux identifiés à l'étape 6.3 de cellules identifiées à l'étape 6.4. Nommez les objets segmentés cytoplasme "221 ;.
- Utilisez le module "EnhanceOrSuppressFeatures" sur les 615 images nm pour éliminer fond et de faciliter l'identification suivante de compartiments de LAMPE1 positif.
- Utilisez le module "IdentifyPrimaryObjects" sur l'image obtenue à l'étape 6.6 pour identifier les compartiments de LAMPE1 positif. Nommez les objets segmentés "lysosomes".
- Utilisez le module "IdentifyPrimaryObjects» sur l'image à 488 nm à identifier les colonies Coxiella. Nommez les objets segmentés "colonies".
- Utilisez le module "IdentifySecondaryObjects" sur les 615 images nm pour identifier les vacuoles contenant Coxiella-en utilisant les colonies Coxiella détectés à l'étape 6.8 comme semences. Nommez les objets segmentés "de CCVS".
- Utilisez le module "MaskObjects" pour sélectionner CCVs détectés sur des cellules (comme les cellules touchant la frontière de l'image ont été éliminés, certains CCV peuvent être détectés cellules "extérieurs"). Nommez les resObjets Ulting "CCVs filtrées".
- Utilisez le module "MaskObjects" pour sélectionner colonies détectées sur des cellules (comme les cellules touchant la frontière de l'image ont été éliminés, certaines colonies peuvent être détectés cellules "extérieurs"). Nommez les objets qui en résultent "des Colonies filtrées".
- Utilisez le module "MaskObjects" d'associer lysosomes de cellules. Nommez les objets qui en résultent "filtrée Lysosomes".
- Utilisez le module "MaskObjects" pour sélectionner des cellules contenant des colonies Coxiella. Nommez les objets résultant de "cellules infectées".
- Utilisez le module "Relier les objets" pour définir les objets "de CCVS filtrées" que les enfants des objets parents "Cellules". Cela permettra à compter le nombre de VCC / Cell.
- Utilisez le module "Relier les objets" pour définir les objets "des Colonies filtrées" que les enfants des objets parents "Cellules". Cette volontépermettre le comptage du nombre de colonies / Cell.
- Utilisez le module "Relier les objets" pour définir les objets "filtrée Lysosomes" comme les enfants des objets parents «Cellules». Cela permettra à compter le nombre de lysosomes / Cell.
- Utilisez le module "MeasureObjectSizeShape" pour obtenir une analyse morphologique des noyaux, les cellules, CCVS filtré, Colonies filtrée et filtrée lysosomes
- Utilisez le module "MeasureObjectIntensity" pour quantifier la fluorescence de la GFP associé à Filtrée CCV et d'estimer l'efficacité de la réplication au sein de Coxiella CCV.
- En utilisant les modules "OverlayOutlines" et "SaveImages" superposer les résultats de segmentation et l'image d'origine pour le contrôle de la qualité.
- Utilisez le module "ExportToSpreadsheet" d'exporter la totalité ou une sélection de résultats d'analyse d'image.
- (Facultatif) Utilisez le module "ExportToDatabase" pour analyser les résultatsl'aide de l'analyste de CellProfiler logiciel.
7. Analyse des données
- Pour chaque paramètre obtenu, identifier et éliminer les valeurs aberrantes (en raison d'erreurs dans la segmentation d'image), puis calculer les valeurs moyennes par mutant.
- Utilisez Z-scores pour identifier les phénotypes importantes. Considérez phénotypes avec un Z-score> -2 comme non significative, les phénotypes avec un Z-score entre -2 et -4 aussi doux et phénotypes avec un Z-score ≤ -4 aussi forte.
- combinaisons de terrain de paramètres en fonction des besoins expérimentaux.
Résultats
Après isolement de mutants de transposons, colonie unique d'amorces de PCR est une méthode à haut débit robuste pour identifier le site d'insertion du transposon pour chaque mutant. Cette approche découle d'un protocole typique PCR nichée, mais ici une seule amorce hybridant spécifiquement et / ou de manière non spécifique à l'ADN de matrice en fonction de la stringence de la température de recuit (figure 1A). Les produits de PCR typiques sont constitués de multiples fragments d'ADN, dont la plupart sont spécifiques (figure 1B). L'utilisation d'une amorce de séquençage différent qui apparie droit en amont de l'ITR du transposon, et en aval de la séquence reconnue par l'amorce d'amplification fournit une spécificité pour l'étape de mise en séquence (figure 1C). Logiciel automatisé pour l'analyse de séquence permet d'aligner les séquences obtenues dans le génome de Coxiella fournir l'emplacement exact des insertions de transposons (figure 1c). Toutes les insertions de transposons peuvent être ensuite annotated sur le génome de Coxiella (Figure 1D).
Chaque mutant Coxiella est isolé et amplifié dans des conditions axéniques-2 ACCM milieu soit avant le stockage ou le criblage. La figure 2 illustre un exemple de mutants de transposons 38 à 16 points / ICM Coxiella gènes (figure 2A). Pour évaluer la viabilité des mutants Coxiella, courbes de croissance axéniques sont obtenues par échantillonnage cultures bactériennes pendant 7 jours après l'inoculation et l'application de l'analyse de la concentration bactérienne décrit dans 1.5 (Figurie 2B). Mutants amplifiés sont ensuite incubées avec les cellules épithéliales, en triplicata plaques de 96 puits pendant 7 jours. Tous les mutants générés étant Coxiella GFP-étiqueté, les courbes de croissance intracellulaires sont obtenus en mesurant l'intensité de fluorescence de la GFP de chaque puits, chaque 24 h, et le tracé des valeurs mesurées en fonction du temps (figure 2C).
Intrles courbes de croissance acellulaires fournissent une analyse quantitative des phénotypes associés à chaque insertion de transposon dans le génome de Coxiella. Pour ajouter des informations qualitatives sur les mêmes mutants de transposons, nous avons opté pour l'acquisition d'image automatisé et d'analyse. Sept jours après l'infection, les plaques sont fixées, traitées pour immunofluorescence comme décrit à 4 et analysés en utilisant un système automatisé, tel que décrit microscope à épifluorescence en 5. logiciel d'analyse d'image automatisée comme CellProfiler (Broad Institute, www.cellprofiler.com ) traite les canaux acquis et indépendamment des segments identifiés des objets d'analyse comparative (figure 3). Ceci permet l'identification et la caractérisation morphologique des noyaux de cellules hôtes, des contours de cellules, des lysosomes et Coxiella colonies (figure 3 panneaux supérieurs). Corréler colonies Coxiella avec les cellules et permet aux lysosomes identificatiet une analyse morphologique spécifique de vacuoles contenant Coxiella (qui sont LAMP1 positif, Figure 3 du panneau en bas à gauche). Corréler colonies Coxiella avec des contours de la cellule hôte permet l'identification et l'analyse morphologique spécifique des cellules infectées (Figure 3 en bas du panneau de centre). Enfin, les 4 canaux sont fusionnés pour l'illustration et à des fins de contrôle de qualité (Figure 3 du panneau en bas à droite).
Les données obtenues par analyse d'image automatisée peuvent être tracées uns contre les autres pour obtenir des «nuages de points multi-phénotypiques". A titre d'exemple, sur la figure 4A la superficie moyenne (en um 2) de colonies Coxiella est tracée en fonction du nombre de colonies par cellule (figure 4a), afin d'identifier des mutations qui affectent la réplication intracellulaire de Coxiella (phénotype de replication) et / ou la capacité des bactéries d'envahir les cellules hôtes (internalisation phénotype). L'analyse statistique a été utilisé pour définir les régions dans le nuage de points résultant correspondant aux douces (-4 (figure 4A, rose et points rouges); mutations qui ont affecté Coxiella internalisation dans les cellules ont été regroupés dans la partie inférieure de la parcelle (figure 4A, bleu clair et bleu foncé points) et enfin, des points verts dans la région la plus à droite de la parcelle correspondent aux mutations résultant de phénotypes non significatifs ( Z-score> -2). Fait important, les mutants qui ne parviennent pas à se répliquer mais sont encore capables d'envahir les cellules hôtes, sont détectés après 7 jours d'infection que des bactéries ou des simples, de petites colonies, adjacentes à accueillir les noyaux des cellules (figure 4C, panneau secondes). Par conséquent, la taille de Coxiella "coloentre- "sera affectée de façon significative, mais le nombre de cellules infectées ne varie pas par rapport à des cellules infectées par le WT Coxiella. Au contraire, des mutations qui affectent la capacité de Coxiella à envahir les cellules hôtes se traduisent par une diminution du nombre de colonies / cellule. Lorsque ce nombre est nettement inférieur à 1, cela signifie que, en moyenne, on constate une diminution du nombre total de cellules infectées. En variante, la superficie moyenne (en um 2) de colonies Coxiella peut être tracée en fonction du nombre de cellules hôtes qui survivent infection (figure 4B), pour identifier les mutations qui confèrent une cytotoxicité à Coxiella (phénotype cytotoxique). Comme ci-dessus, l'analyse statistique a été utilisé pour définir les régions dans le nuage de points résultant correspondant aux douces (-4 (figure 3B points verts). 37 mutations survie de la cellule hôte légèrement affectée (figure 3B, les points rouges légers), et 7 mutations étaient particulièrement préjudiciable pour accueillir la survie des cellules (figure 3B, points rouges sombres). S'il vous plaît noter que les paramètres supplémentaires obtenus par l'image procédure d'analyse automatisé peuvent être utilisés pour tirer d'autres cartes, en fonction des besoins expérimentaux.
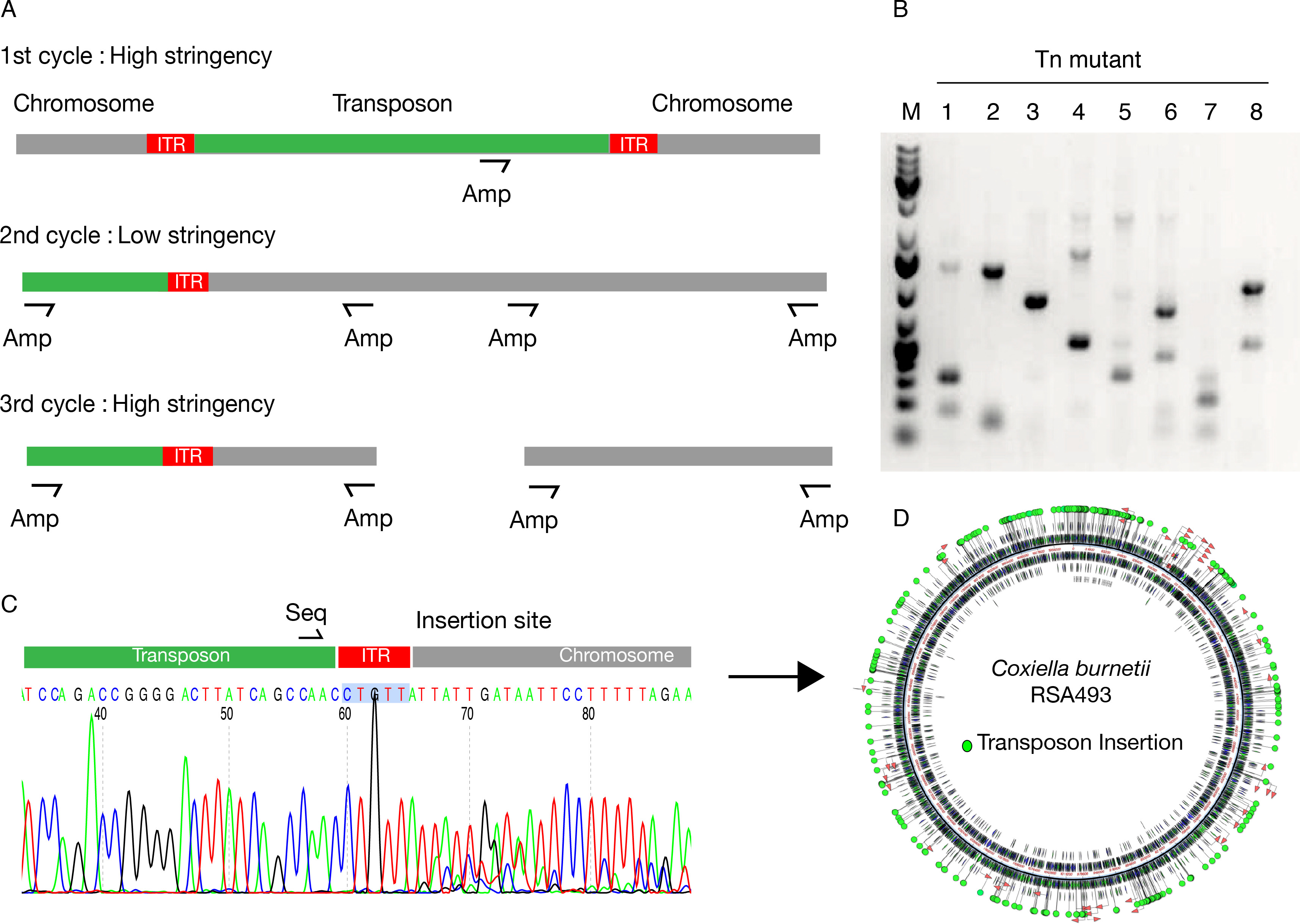
Figure 1:. Séquençage et annotation de mutants de transposon Coxiella (A) à l'unité primaire colonie PCR est utilisée pour amplifier des fragments d'ADN contenant le site d'insertion du transposon. Une amorce d'amplification (Amp) est utilisé à la fois comme amorce spécifique et non-spécifique en fonction de la stringence de la température de recuit. (B) de résultat typique de colonie seule amorce PCR. Chaque Reactile produit un certain nombre de fragments de taille variable, dont certaines contiennent le site d'insertion du transposon; certains autres sont amplifiés de manière aléatoire en tant que sous-produits du cycle de PCR à faible stringence. (C) L'utilisation d'une amorce de séquençage (SEQ) qui hybride à la séquence du transposon permet le séquençage des fragments d'intérêt. Logiciel (D) d'analyse de séquence permet l'annotation automatique des insertions de transposons sur le génome bactérien. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.

Figure 2:. Axénique et la croissance intracellulaire de mutants de transposons Coxiella (A) Dans l'écran de pilote, nous avons isolé, séquencé et projeté 38 mutants de transposons dans 16 g de baseEnes du système dot / ICM sécrétion Coxiella (indiqués en rouge). (B) Afin d'évaluer la viabilité de chaque mutant de transposon, la croissance de chaque isoler dans un milieu de culture axénique est surveillé de plus de 8 jours à l'aide d'un agent de fluorescence taggés intercalant d'ADN. (C) Pour chaque mutant est ensuite utilisé pour infecter des cellules épithéliales. Comme le transposon possède une cassette de la GFP, la croissance bactérienne intracellulaire est surveillé de plus de 7 jours de l'infection en suivant les variations de fluorescence de la GFP associé à la réplication Coxiella, en utilisant un lecteur de microplaques. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
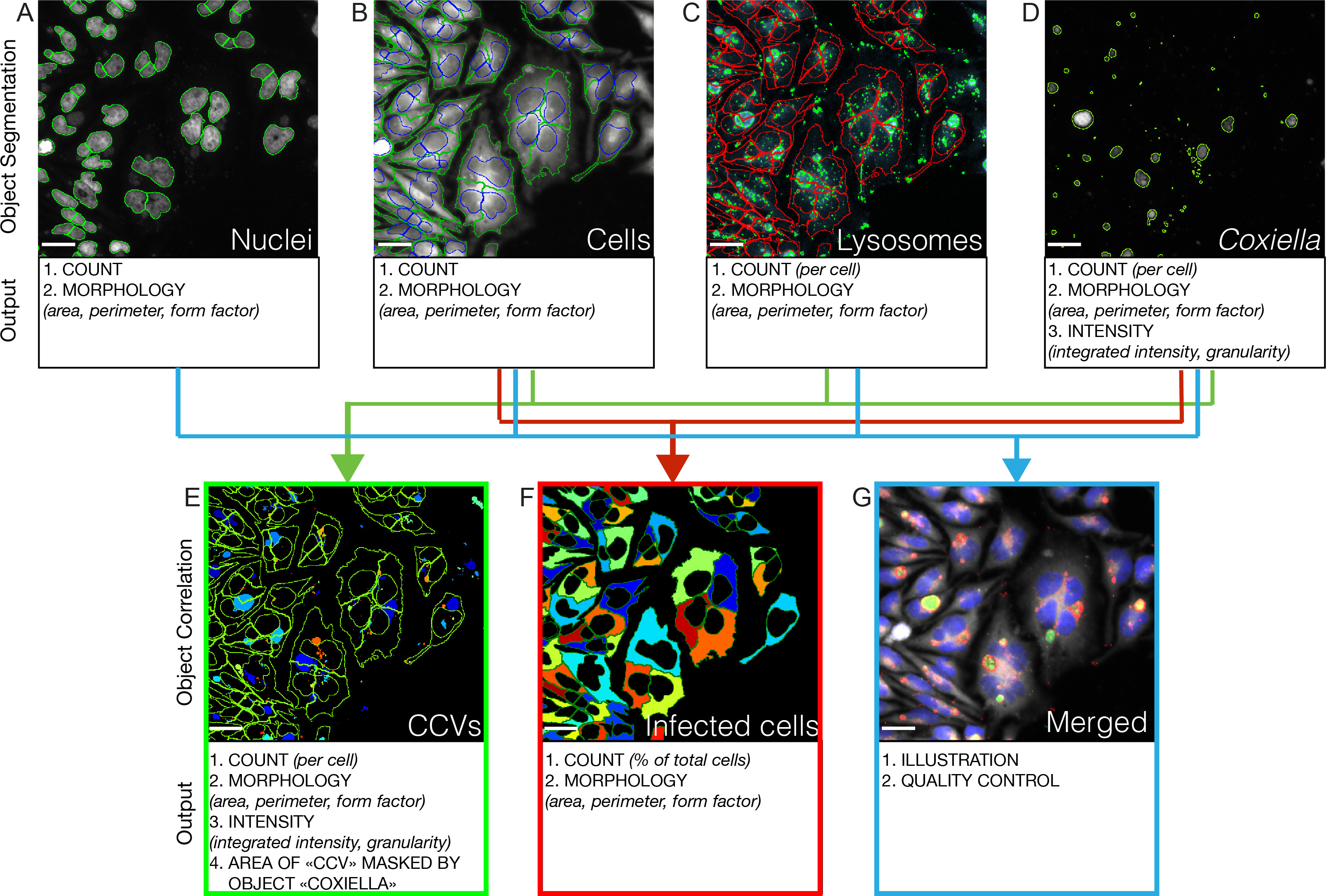
Figure 3: analyse d'image automatisée des infections Coxiella Une auto.accouplée microscope à épifluorescence est utilisé pour l'image 21 positions par puits en triple de plaques à 96 puits. Les segments de logiciels d'analyse d'images objets dans chaque canaux acquis pour la quantification et l'analyse. Dans tous les cas, les objets touchant la frontière des images sont exclus. (A) Le canal Hoechst est utilisé pour identifier les noyaux des cellules de l'hôte (entouré en vert). (B) Ceux-ci sont utilisés comme semences pour identifier les contours de la cellule hôte dans le canal Cy3 (la position des noyaux est entouré en bleu, les contours de cellules sont en vert). (C) Le canal Cy5 est utilisé pour identifier les compartiments de LAMPE1-positive (dans le cercle vert); seulement les objets inclus dans les contours de cellules identifiées précédemment (en rouge) sont retenus pour l'analyse d'image. (D) Le canal de la GFP est utilisé pour identifier les colonies de Coxiella (entouré en vert). (E) corrélation colonies Coxiella avec des compartiments de LAMPE1 positif permet l'identification de Coxiella vacuoles contenant (CCVS); seulement les objets inclus dans les contours de cellules identifiées précédemment (en vert) sont retenus pour l'analyse d'image. (F) en corrélation avec des contours Coxiella colonies de cellules permet l'identification de cellules infectées (de pseudocolored). (G) Les images acquises dans les 4 canaux de fluorescence (correspondant à colonies Coxiella (vert), des noyaux de la cellule hôte (bleu), la membrane de la cellule hôte plasma (gris), les compartiments de LAMPE1-positif (rouge)) sont fusionnés et utilisée pour l'illustration et de la qualité contrôle. Échelle barres 10 um. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
Figure 4: l'identification à grande échelle des facteurs impliqués dans Coxiella hôte / pathogène interagissentions. (A) Le Quartier moyenne (en pm 2) des colonies Coxiella est tracée contre le nombre relatif de colonies par cellule, afin d'identifier et de réplication internalisation phénotypes d'intérêt. Les points verts représentent phénotypes qui dévient de WT Coxiella par un Z-score> -2 (non significatif). Points roses et bleu clair représentent réplication et de l'internalisation des phénotypes respectivement, avec un Z-score entre -2 et -4 (phénotypes bénins). Points bleu foncé Rouge et représentent phénotypes avec un Z-score ≤ -4 (phénotypes forts). (B) La surface moyenne (en um 2) de Coxiella colonies a été tracée en fonction du nombre total de cellules (infectées et non infectées) qui ont survécu 7 jours suivant l'infection pour estimer l'effet cytotoxique résultant de la insertions de transposons. Les points verts représentent phénotypes qui dévient de WT Coxiella par un Z-score> -2 (non significatif). Points roses représentent cytotoxique phenotypes avec un Z-score entre -2 et -4 (phénotypes bénins). Les points rouges représentent les phénotypes cytotoxiques avec un Z-score ≤ -4 (phénotypes forts). Les flèches indiquent mutants illustrés par la lettre minuscule correspondante de C (C) des images représentatives de la réplication, l'internalisation et phénotypes cytotoxiques. Dans tous les cas, les noyaux de cellules hôtes sont en rouge, les colonies sont Coxiella en vert. Échelle barres 50 um. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
Discussion
L'étude des interactions hôte / pathogène a prouvé être une méthode remarquable pour comprendre les infections bactériennes et développer des stratégies alternatives pour contrer les maladies infectieuses. Toutefois, en raison de la diversité des stratégies élaborées par différents pathogènes bactériens, l'identification et la caractérisation des facteurs de virulence bactériens et des voies de signalisation hôte qui sont ciblés au cours des infections représentent un véritable défi. Cela nécessite le développement de nouvelles approches pour l'identification à grande échelle des principaux hubs d'interaction hôte / pathogène. Le développement récent de, à haut débit innovantes et des techniques de criblage à haut contenu représente une ressource inestimable qui peut être adapté à l'étude des bactéries pathogènes intracellulaires 15. Ici, nous avons utilisé la zoonose bactérienne pathogène Coxiella burnetii comme un modèle à développer des approches de dépistage qui combinent mutagenèse de transposon et des essais basés sur la fluorescence. Importantly, cette méthode de dépistage permet la surveillance simultanée de plusieurs étapes du cycle intracellulaire Coxiella, fournissant un aperçu global des stratégies développées par cette bactérie d'envahir, de reproduire et de persister dans les cellules infectées.
L'approche décrite ici est basée sur deux techniques bien établies, la mutagenèse de transposon et des essais basés sur la fluorescence, qui ont été appliquées avec succès à l'étude des agents pathogènes bactériens. La combinaison de ces techniques dans le contexte des écrans à haut débit / haute contenu nous permet d'évaluer les effets d'un nombre élevé de mutations bactériennes en analysant un nombre très élevé d'événements (typiquement 15 000 cellules infectées par des mutations bactériennes sont imagées et analysées). Ceci fournit une analyse statistique importante d'événements tels que l'invasion bactérienne des cellules hôtes et la réplication intracellulaire, qui sont, par nature, soumises à une forte variabilité. Il est important de noter que les lignées cellulaires autres que epithelial peuvent être utilisés pour ce type de dépistage. Toutefois, les cellules epitheliales plates et larges sont optimales pour l'analyse d'images comme des organelles de cellules hôtes sont plus faciles à détecter. Parce que la majorité des microscopes automatisés peut traiter automatiquement un grand nombre de plaques, il n'y a pratiquement pas de limites au nombre de mutants qui peut être projeté simultanément. En fonction de l'agent pathogène, l'utilisateur peut privilégier l'utilisation d'une épifluorescence ou un microscope confocal. Le moment de l'acquisition de l'image est essentiellement dépendre de la sensibilité de la caméra du microscope, sur le nombre de champs par puits et acquises sur le nombre de canaux acquises par le champ de vision. L'utilisateur peut décider comment ajuster ces facteurs pour optimiser le protocole de dépistage. A titre d'exemple, nous imager une plaque de 96 puits / h en utilisant les conditions indiquées au point 5.1. L'analyse d'images dépend largement de la machine (ou groupe de machines) utilisé. Nous utilisons un 12-core (2 x 3,06 GHz 6-Core), 48 Go de RAM poste de travail. Cette machine nécessite environ 40 min pour analyser les images acquises d'une plaque.
Un aspect important à prendre en compte lors de l'élaboration de ces tests est la mise en place de la nouvelle (ou l'optimisation des protocoles existants) pour permettre la manipulation et le traitement d'un grand nombre d'échantillons. Un exemple typique est le développement de l'approche PCR de l'amorce de colonie unique, qui nous a permis d'amplifier rapidement et fragments séquence Coxiella ADN contenant le site d'insertion du transposon chaque, à partir de très petits échantillons. Basé sur notre expérience, la polymérase haute fidélité doit être soigneusement sélectionnés et testés afin d'obtenir des résultats reproductibles. La seule limitation de cette approche peut se cacher dans l'observation que, dans la majorité des cas, environ 30% des échantillons traités ne sont pas exploitables, soit en raison de la PCR ou les étapes de séquençage. Cependant, étant donné que l'isolement des nouveaux mutants de transposons Coxiella est pas une étape de limitation de vitesse, cela ne repré pasenvoyé un enjeu majeur. De même, le développement d'un test fiable de quantifier la concentration bactérienne des stocks mutantes a été la clé de cette approche. En raison de la tendance de Coxiella d'agréger quand en suspension, l'utilisation des lectures de densité optique est pas applicable pour le calcul de la concentration de cultures Coxiella et la seule alternative existante était PCR quantitative (qPCR). Ici, l'utilisation d'un agent intercalant d'ADN marqués par fluorescence significativement accélérer la quantification des bactéries.
Cette approche peut également profiter de l'utilisation de lignées cellulaires stables exprimant des marqueurs fluorescents pour plusieurs compartiments intracellulaires selon l'agent pathogène utilisé. Un autre aspect important est l'utilisation de milieux de culture cellulaire dépourvu de rouge de phénol. Nous avons constaté que cet indicateur de pH a une fluorescence naturelle couvrant le spectre rouge et vert qui sature le signal enregistré sur le lecteur de fluorescence automatisé.
Ta stratégie présentée ici repose sur la mutagenèse aléatoire transposon. Pour les mutants d'intérêt, nous vous recommandons de valider transpositions uniques (et clonalité) en utilisant transfert de Southern et amplifications PCR du site d'insertion du transposon.
Outre l'équipement décrit dans la section de protocole, équipes intéressées à l'aide de l'approche de dépistage présenté ici, auront grand avantage dans la mise en place d'une base de données relationnelle pour la collecte de données, un serveur pour le stockage des données et une station de travail pour l'analyse rapide des images.
Fait important, le procédé décrit ici est approprié pour l'étude d'autres agents pathogènes bactériens intracellulaires fourni un procédé de mutagenèse aléatoire existe pour l'agent pathogène, des lignées de cellules peuvent être infectées par l'agent pathogène et celui-ci affiche un phénotype spécifique au cours de l'infection.
Déclarations de divulgation
The authors declare that they have no competing financial interests.
Remerciements
This work was supported by an Avenir/ATIP grant to Matteo Bonazzi and a Marie Curie CIG (N° 293731) to Eric Martinez. The authors would like to thank Dr. Robert Heinzen for sharing C. burnetii strains, antibodies and tools, Virginie Georget and Sylvain DeRossi (Montpellier Rio Imaging-MRI, 1919 route de Mende, 34293 Montpellier) for their technical assistance and data analysis.
matériels
| Name | Company | Catalog Number | Comments |
| Citric acid | Sigma | C0759-500G | ACCM-2 medium component |
| Sodium citrate | Sigma | S4641-500G | ACCM-2 medium component |
| Potassium phosphate | Sigma | 60218-100G | ACCM-2 medium component |
| Magnesium chloride | Sigma | M2670-100G | ACCM-2 medium component |
| Calcium chloride | Sigma | C5080-500G | ACCM-2 medium component |
| Iron sulfate | Sigma | F8633-250G | ACCM-2 medium component |
| Sodium chloride | Sigma | S9625-500G | ACCM-2 medium component |
| L-cysteine | Sigma | C6852-25G | ACCM-2 medium component |
| Bacto Neopeptone | BD (Beckton-Dickinson) | 211681 | ACCM-2 medium component |
| Casamino acids | BD (Beckton-Dickinson) | 223050 | ACCM-2 medium component |
| Methyl-B-cyclodextrin | Sigma | C4555-10G | ACCM-2 medium component |
| RPMI w/glutamax | Gibco | 61870-010 | ACCM-2 medium component |
| RPMI w/glutamax, without phenol red | Gibco | 32404-014 | For cell culture and infection |
| Fetal bovine serum | GE healthcare | SH30071 | For cell culture |
| Trypsin EDTA (0.25%) | Life Technologies | 25200-056 | For cell culture |
| Saponin | Sigma | 47036 | For immunofluorescence staining |
| Ammonium chloride | Sigma | A9434 | For immunofluorescence staining |
| Bovine serum albumin | Sigma | A2153 | For immunofluorescence staining |
| Electroporation cuvette 0.1 cm | Eurogentec | ce0001-50 | For Coxiella transformation |
| Trackmates screw top tubes with caps | Thermo Scientific | 3741 | 2D barcoded screwcap |
| 96-well microplate with black walls and bottom | Greiner | 655076 | Flat dark bottom, for dsDNA quantitation |
| 96-well PCR microplate | Biorad | 2239441 | DNAse/RNAse free |
| 96-well plate, deepwell | Labcon | 949481 | For Coxiella mutants infections |
| PicoGreen | Life Technologies | P7581 | For dsDNA quantitation |
| Triton X-100 | Sigma | T9284-500ML | For dsDNA quantitation |
| Phusion high fidelity DNA polymerase | New England Biolabs | M0530L | For single primer colony PCR |
| Celltracker Red CMTPX | Life Technologies | C34552 | For imaging |
| Anti-LAMP1 antibody | Sigma | 94403-1ML | For immunofluorescence staining |
| Hoechst 33258 | Sigma | L1418 | For imaging |
| Fluorescence microplate reader Infinite 200 Pro | Tecan | ||
| Epifluorescence automated microscope Cellomics | Thermo Scientific |
Références
- Maurin, M., Raoult, D. Q fever. Clinical microbiology reviews. 12 (4), 518-553 (1999).
- Kim, S. G., Kim, E. H., Lafferty, C. J., Dubovi, E. Coxiella burnetii. in bulk tank milk samples, United States. Emerging infectious diseases. 11, 619-621 (2005).
- Kazar, J. Coxiella burnetii. infection. Annals of the New York Academy of Sciences. 1063, 105-114 (2005).
- Madariaga, M. G., Rezai, K., Trenholme, G. M., Weinstein, R. A. Q fever: a biological weapon in your backyard. The Lancet infectious diseases. 3, 709-721 (2003).
- Der Hoek, W. V. a. n., et al. Epidemic Q fever in humans in the Netherlands. Advances in Experimental Medicine and Biology. 984, 329-364 (2012).
- Capo, C., et al. Subversion of monocyte functions by Coxiella burnetii.: impairment of the cross-talk between alphavbeta3 integrin and CR3. Journal of immunology. 163 (11), 6078-6085 (1999).
- Martinez, E., Cantet, F., Fava, L., Norville, I., Bonazzi, M. Identification of OmpA, a Coxiella burnetii. protein involved in host cell invasion, by multi-phenotypic high-content screening. PLoS pathogens. 10 (3), e1004013 (2014).
- Romano, P. S., Gutierrez, M. G., Berón, W., Rabinovitch, M., Colombo, M. I. The autophagic pathway is actively modulated by phase II Coxiella burnetii. to efficiently replicate in the host cell. Cellular microbiology. 9 (4), 891-909 (2007).
- Newton, H. J., Mcdonough, J. a., Roy, C. R. Effector protein translocation by the Coxiella burnetii. Dot/Icm type IV secretion system requires endocytic maturation of the pathogen-occupied vacuole. PloS one. 8 (1), e54566 (2013).
- Vogel, J. P. Turning a tiger into a house cat: using Legionella pneumophila. to study Coxiella burnetii.. Trends in microbiology. 12 (3), 103-105 (2004).
- Van Schaik, E. J., Chen, C., Mertens, K., Weber, M. M., Samuel, J. E. Molecular pathogenesis of the obligate intracellular bacterium Coxiella burnetii. Nature reviews. Microbiology. 11, 561-573 (2013).
- Beare, P. A., et al. Dot/Icm type IVB secretion system requirements for Coxiella burnetii growth in human macrophages. mBio. 2, (2011).
- Omsland, A., et al. Isolation from animal tissue and genetic transformation of Coxiella burnetii. are facilitated by an improved axenic growth medium. Applied and environmental microbiology. 77 (11), 3720-3725 (2011).
- Beare, P. a., Sandoz, K. M., Omsland, A., Rockey, D. D., Heinzen, R. a Advances in genetic manipulation of obligate intracellular bacterial pathogens. Frontiers in microbiology. 2 (May), 97 (2011).
- Brodin, P., Christophe, T. High-content screening in infectious diseases. Current opinion in chemical biology. 15 (4), 534-539 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon