Method Article
ヒト膵臓の開発にリネージュの決定要因を問い合わせるためのゲノム編集とhPSCsの指向性分化
要約
iCRISPRプラットフォームを使用HPSC変異株を作製し、グルコース応答性β様細胞にhPSCsを区別するためのプロトコルが記載されています。 HPSC指向分化にゲノム編集技術を組み合わせることにより、人間開発と疾患の進行中の系譜決定因子の役割の体系的な分析のための強力なプラットフォームを提供します。
要約
自己再生または分化するヒト多能性幹細胞(hPSCs)で遺伝子機能を尋問することは、人間開発を理解し、皿に病気のメカニズムを解剖に向けて貴重なプラットフォームを提供しています。この潜在的な用途に活用するには、密接にそれらのインビボの対応を再現疾患関連細胞型を生成するために、疾患関連遺伝子にHPSC変異体を生成するための効率的なゲノム編集ツールを必要とするだけでなく、in vitroでの HPSC分化プロトコル。 iCRISPRという名前hPSCsのための効率的なゲノム編集プラットフォームはAAVS1遺伝子座におけるCas9発現カセットのTALEN媒介ターゲティングによって開発されました。ここでは、化学的に定義された培地および無フィーダー条件で培養細胞を用いた誘導Cas9 HPSCラインを生成するためのプロトコルが記載されています。 nまでのいずれかの遺伝子ノックアウトまたはhPSCsで正確な遺伝的変化のためのiCRISPRシステムを使用するための詳細な手順、接合端(NHEJ)に相同オンまたは相同性指向修理(HDR)テンプレートを使用して、正確なヌクレオチド変異を経由して、それぞれ、含まれています。これらの技術的な手順は、設計、生産、およびCRISPRのガイドRNA(gRNAs)のトランスフェクションの記述を含みます。 T7E1またはRFLPアッセイによってCRISPR変異率の測定;およびクローン変異株の樹立と検証。最後に、 生体内膵臓の胚発生に模倣することによってグルコース応答性膵β細胞様細胞への我々 HPSC分化に記載の手順。有向HPSC分化にiCRISPR技術を組み合わせることにより、膵臓の開発と糖尿病のメカニズムの理解を促進するために遺伝子機能の系統的な検査を可能にします。
概要
ヒト多能性幹細胞(hPSCs)自己複製の両方三胚性生殖系列の全ての誘導体を生じる能力を有します。彼らは人間の発達のコンテキストで細胞プロセスを再現するユニークなプラットフォームとして機能することにより、細胞置換療法と病気のモデリングのための貴重なリソースを提供しています。彼らはまた、スケーラブル、高スループット分析のための実験的な細胞の供給源です。効率的な遺伝子改変ツールの欠如および培養皿に複雑な胚の発達段階を反復することの困難:しかし、進展があるため、2つの主要な課題に制限されていました。
遺伝子改変は、通常の開発と疾患における遺伝子機能を研究するための不可欠なツールです。相同組換えを介してのアプローチを標的古典的な遺伝子が証明されているがしかし、マウス胚性幹細胞(たmESC)における遺伝子機能を分析するための強力なツール1は、このアプローチであることがhPSCs 2、3に適用された場合、非常に非効率的となっています。ジンクフィンガーヌクレアーゼ(ZFNを)、転写活性化因子のようなエフェクターヌクレアーゼ(TALENs)、およびクラスタ化された定期的interspaced短いパリンドローム反復(CRISPR)/ CRISPRは関連を含む実験使用に自然からプログラマブル、サイト固有のヌクレアーゼ、最近の活発なアク(CAS)システム4は 、ゲノム工学はhPSCsを含め、生物および細胞株の広い範囲で非常に容易課題となっていることを意味します。これらの遺伝子の編集ツールは、このようなCas9エンドヌクレアーゼとキメラヌクレアーゼは、いずれかの非相同を活性化するために、正確な位置での二重鎖切断(DSB)を誘導する内因性DNA修復機構をトリガすることによって遺伝子改変の全範囲を可能にすることができるという事実を利用します結合(NHEJ)終了または相同性指向修理(HDR)。両方のメカニズムはEITHを誘導することによって遺伝子操作のために利用することができますえーランダム挿入および欠失変異(インデル; NHEJ を介した)は 、ヒト疾患のモデリングのための患者の変異を再現したり、遺伝子治療のための疾患を引き起こす変異を修正するために、遺伝子の対立遺伝子、または(HDR経由で)正確なヌクレオチド置換を無効にフレームシフト変異を作成します。
DNA標的認識を指定するDNAの切断と変数のCRISPR RNA(crRNA)およびトランス活性化(tracrRNA)二重のために必要な一定のRNA誘導型Cas9エンドヌクレアーゼ:CRISPR / Casの媒介ゲノム工学は、2つのコンポーネントが必要です。 crRNA / tracrRNA二本鎖は、より効率的に5、6、7働くことが見出されている単一のキメラガイドRNA(gRNA)で置き換えることができます。 CRISPR / Cas9システムが最も実験的な生物および細胞株に適合されているが、Cas9およびgRNAの送達および発現は有意に変化し、さらに阿智に最適化される必要がありますhPSCs 8を含む多くのシステムでの前夜効率的なゲノム編集。効率的なゲノム編集プラットフォーム、iCRISPRは、hPSCs 5に設立されました。このシステムでは、TALEN媒介アプローチは、テトラサイクリン応答エレメント(TREと逆テトラサイクリン制御トランス(M2rtTA)や他とのトランス 、一つの対立遺伝子の 「導入遺伝子セーフハーバー軌跡」AAVS1の両方の対立遺伝子を標的とするために使用されてきました)hPSCsにおけるCas9(iCas9)の発現を駆動します。確立されたクローン系(iCas9 hPSCs)では、Cas9は非常にドキシサイクリン処理で表現されています。一方、、その小さなサイズ(100ヌクレオチド)に、単一または複数のgRNAsは、簡単に高効率でiCas9 hPSCsに送達することができ、部位特異的切断のためのCas9を指示することができ、効率的なNHEJ媒介遺伝子破壊を可能にするだけでなく、HDR-媒介します短い一本鎖DNA(ssDNA)ドナーテンプレートの存在下での正確なヌクレオチド修飾。 iCRISPRシステムは、することができます成功し、9が重要な発生遺伝子5で二対立遺伝子(ホモ接合または複合ヘテロ接合)またはヘテロ接合機能喪失型変異を有する疾患模倣HPSC株のパネルを生成するために使用されます。グループの数はhPSCsにCRISPR / CASを使用して、効率的な遺伝子編集を報告しているが、成功は技術的に熟達研究室の小さな数に制限されたままです。 iCRISPRプラットフォームは、異なるスキルレベルの研究者による日常的遺伝子編集のための効率的かつシンプルなソリューションを提供しています、そして、それは既に公開された私たちのグループによる研究およびその他9、10、11、12の数で使用されています。このアプローチは、さらにdCas9-KRAB 13の発現に基づいて、誘導性サイレンシングに拡張されています。
ゲノム編集テクノの進展に伴いでれっと、大幅な改善もHPSCのメンテナンスと向かう分化に達成されています。 hPSCsのための培養条件は、定義された細胞外マトリックス成分に無フィーダー条件に、かつ化学的に培地条件14を定義したために、複雑な培地製剤から照射したマウス胚性線維芽細胞(iMEF)フィーダ依存から進化してきました。このような改善はiMEF準備とノックアウト血清代替成分中のバッチ間の差異に起因するhPSCsのばらつきを低減し、ひいてはHPSCの分化のためのより再現環境を提供しています。また、改良されたヒト胚発達を支配するシグナル伝達経路についての知識、ならびにハイスループット薬物スクリーニングの発見は、改良された分化プロトコル15、16、17、18につながっています。これらのプロトコルは、より密接VIVに模倣します発達段階oおよび密接にそれらのインビボの対応を再現細胞型を生成します。膵臓系統へのHPSCの分化のために、初期のプロトコルは比較的よく早期膵臓の開発を模倣するが、最終的に未熟な胎児の表現型のものであり、グルコース刺激に乏しい回答polyhormonalβ細胞を生成しました。最近の進歩は16、17、19、20は、このように形成し、monohormonalβ細胞のさらなる成熟として後でイベントを調査するために私達を許可して、グルコース応答性膵β細胞様細胞の生成のために許可されています。
ここでは、詳細グルコース応答pancrに向かってHPSCベースのインビトロ分化プラットフォームとiCRISPRシステムを組み合わせることにより、膵臓の開発の研究のためのゲノム修飾されたラインの適用eaticβ様細胞。改良されたHPSCの分化プロトコルを備えた強力なゲノム編集ツールのこの結合は、疾患の因果関係を検証するための需要を満たすために必要なスピードとスケールを提供していますが、また正常な発達と病気9の根底にある転写制御へのさらなるメカニズムの調査のための洗練された遺伝子操作を可能にするだけでなく、 。
プロトコル
このプロトコルはHPSCラインH1、HUES8、およびMEL-1、化学的に定義されており、無フィーダー条件で( 材料および機器の表を参照してください)と私たちの仕事に基づいています。他のHPSCラインまたは異なる培養条件下で維持hPSCsのために、さらなる最適化が推奨されます。
化学的に定義されており、フィーダーフリーの条件で1 HPSC文化
- 無フィーダー条件にiMEFフィーダー上HPSC文化を適応させます。直接無フィーダー条件で回収時に凍結した細胞が十分に生存しない例では、最初のiMEF状態で細胞を回収した後、無フィーダー条件に適応します。
注:一般的には、無フィーダー条件に適合させるiMEFフィーダー上で培養hPSCsのための2の通路を取ります。 - 培地を毎日継代細胞は〜80%コンフルエントに達したhPSCsを変更します。 〜1で一般的には、通路hPSCs:6 - 1時15比率ごとに4から6日間。 10μMのROCK阻害剤Y-27632 WHを追加解凍または細胞を継代アン。
- hPSCsを播種する前に、5μg/ mlの(1ミリリットル/ 10 cm 2)を有するプレコート培養皿を室温(RT)で少なくとも1時間、ビトロネクチンの組換えヒト形態(VTN)を切り捨て。また、基礎培地にサプリメントを追加することにより、完全な化学的に規定された培地を準備します。
- 、培養培地を除去した Ca 2+およびMg 2+を含まないPBSで細胞を1回洗浄し、〜2、0.5 mMのEDTAで細胞を処理する- RTで5分。
- コロニーが切り離されている前に、EDTAを吸引します。穏やかにピペッティングして、小片にHPSCコロニーを分散させ、完全培地中で細胞を再懸濁。
- 解離hPSCsを収集し、5分間、200×gで細胞をスピンダウン。完全培地中でペレット化しhPSCsを再懸濁し、VTN被覆プレート上の細胞を播種します。
iCas9 HPSC行の2世代
- AAVS1-TALEN-L、AAVS1-T:順序は、以下のプラスミドを増幅しますALEN-R、AAVS1-ネオM2rtTA、およびAAVS1-Puroを-iCas9。
注:30°Cでのプラスミドの形質転換および増幅のための組換え欠損Stbl3コンピテント細胞を使用し、予期しない組換え事象を回避するために。 - 典型的には、1つのターゲティング実験のために1 10 cmディッシュ(〜1×10 7細胞の場合〜80%コンフルエント)にhPSCsを準備します。
注:エレクトロポレーションは、通常、有意な細胞死を引き起こし、AAVS1遺伝子座にターゲティングTALEN媒介遺伝子は、抗生物質選択を必要とするので、細胞の比較的大きな数は正確に標的細胞を同定するために播種される必要があります。薬物濃度の最適化は、各細胞株および各培養条件のために推奨されます。 - 1日目では、(エレクトロポレーションの前日)、メディアの変更中に10μMのROCK阻害剤を追加します。
- 0日目 、(エレクトロポレーションの日)には、事前にVTN-コーティングしたプレートを準備します。
- USI単一細胞にhPSCsを解離ngの1×解離試薬(参照してください材料および機器の表 )。簡単に言えば、培養液を除去した Ca 2+およびMg 2+を含まないPBSで細胞を1回洗浄し、2〜3分間、37℃で1×解離試薬で細胞を処理します。細胞が剥離している前に、解離試薬を吸引。穏やかにピペッティングして、完全培地の10.5 mLの単一細胞懸濁液にhPSCsを分散させます。
- 自動細胞カウンターを用いて細胞数をカウントし、0.5 mLの細胞懸濁液を取ります。 5分間、200×gでhPSCsをペレット化し、12.5×10 6細胞/ mLでの冷(4℃)、PBSで細胞を再懸濁します。
- 800-μLHPSC懸濁液(12.5×10 6細胞/ mL)にプラスミド( 表1参照 ) を加え、よく混ぜます。 0.4 cmのエレクトロポレーションキュベットに混合物を移し、約5分間氷上に保ちます。
- 250 Vと500μFで電気穿孔システムを用いて細胞をエレクトロポ。時定数OBS13ミリ - エレクトロポレーション後erved一般的に9です。
- エレクトロポレーション後、予め温めた完全培地5mLで15 mLコニカルチューブに細胞を移します。 成功した標的化のためのクリティカル:使用健康に増殖hPSCsと、転送再懸濁し、エレクトロポレーション後に細胞をプレーティングする際に非常に穏やかに細胞を処理します。
- 5分間、200×gで細胞をペレット化。 3 VTN-コーティングされた、10cmディッシュの各々に10μMのROCK阻害剤とプレート1、2.5、および5×10 6個の細胞を完全培地10mLで細胞を再懸濁し、これは、プレートの少なくとも一つは、コロニーピッキングのための単一細胞クローン密度で十分な植民地を持っていることを保証します。
- 1日目 (エレクトロポレーション後の日)に培地を変更してください。
- 日2日-細胞が〜60%コンフルエントである場合5、ネオマイシン選択を開始。 500 / mlのG418硫酸塩で毎日培地を変更します。 SelectIOによる有意な細胞死nは典型的には2日間のG418選択の後に観察されます。
- 6日目 、抗生物質選択を含まない培地を変更します。
- 日7-9で、ピューロマイシン選択を開始します。 1μg/ mLのピューロマイシン二塩酸塩で毎日培地を変更します。有意な細胞死は、次の日に観察する必要があります。
- 直径2ミリメートル- HPSC単一細胞コロニーが1に達するまで、10日目に、抗生物質選択なしで毎日培地交換を開始します。
注:一般的に、10 cmディッシュ中の50個のコロニーを0日目にメッキ2.5×10 6 hPSCsで観察されています。 - 実体顕微鏡下で24個のコロニー - 12を選択してください。機械的に23-G針を使用して小片(コロニーあたり〜10個)にHPSCコロニーを脱凝集(200-μLピペットチップでも結構です)と直接VTNでコーティングした24ウェルプレートに細胞を移します。
- 細胞がコンフルエントになるまで毎日培地を変更します。通路24ウェルプレートの各ウェル内の細胞に6ウェルプレートのウェルを複製します。
- 細胞を6ウェルプレートでコンフルエントになる場合には、さらなる特徴付けのためのゲノムDNA抽出のために凍結保存し、他のよくするための1つのウェルを使用します。
- PCR遺伝子型決定、サザンブロッティング、RT-qPCR分析、染色体分析、および多能性アッセイによって確立iCas9ラインを特徴付けるし、検証します。 Zhu らを参照してください。詳細な実験手順については、21。
iCRISPRシステムを使用したHPSC突然変異系統の3世代
- HPSCノックアウト株の作製
- gRNAの設計と製造
- 野生型タンパク質の機能を破壊する可能性を最大にするために、目的の遺伝子中の標的領域を選択します。よく注釈付きの遺伝子について、本質的な機能ドメインの上流の標的領域を選択します。また、設計gRNAsは、開始コドンの下流領域を標的にします。少なくとも2つの異なるを選択してください目的の遺伝子のための領域。
- オンラインCRISPR設計ツール(http://crispr.mit.edu)を使用して設計gRNAs。各対象領域については、低電位のターゲットオフおよびクローン変異体のライン22を生成するための最高のターゲティング効率を有するものを使用したデザイン3 gRNAs。
注:高ゲノム編集効率を達成するためには、RNAオリゴとして代わりのために過去の経験でプラスミドに比べて低分子RNAの高いトランスフェクション効率のプラスミドDNAとしてgRNAをお届けすることをお勧めします。 - T7プロモーター配列、可変20-ntのcrRNA認識配列(N)20(PAMシーケンスを含まない)、および一定のキメラガイド配列を含む120ヌクレオチド(nt)のDNAオリゴを注文。 ddH 2 Oに100μMの原液にオリゴを希釈し、作業溶液として250 nMのを準備します。
注:TAATACGACTCACTATAGGG(N)20 GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTT - gRNA インビトロ転写(IVT)用の二本鎖DNA(dsDNA)テンプレートを生成する( 表2参照)T7FとTracrRプライマーを用いたオリゴをPCR増幅します。 PCR反応混合物( 表3を参照)、PCRサイクル条件の50μLを使用した ( 表4参照)。
- 20μLにPCR増幅テンプレートとインビトロ gRNA転写用の高収量T7転写キットを使用し、製造元の指示に従って、 インビトロ gRNA転写ミックス( 表5参照)。製造元の指示に従って転写クリーンアップキットを使用してgRNA製品を精製します。
- 溶出緩衝液100μL中 - (100μgの典型的には〜50)ごとに、製造業者の指示としてハイスループット精製プロトコールを以下の溶出gRNAs。 -80で320 ngの/μL(10μM)可能な場合は、店舗に濃度を調整° C使用時まで。
- PCRおよびサンガー配列決定プライマーの設計
- 千塩基対 - 製品のサイズは通常、〜500の範囲で、標的領域を増幅するPCRプライマーを設計および検証します。
- 設計サンガー配列決定プライマーは、精製せずにPCR産物の直接配列決定を可能にするために、PCR産物の内部結合します。
- iCas9 hPSCsでgRNAトランスフェクション
- 1日目に、24時間gRNAトランスフェクションの前に2μg/ mLのドキシサイクリンでiCas9細胞を扱います。
- gRNAトランスフェクションの0日目に、事前にVTN-コーティングしたプレートを準備します。
- ステップ2.5で説明したように、1×解離試薬を使用して単一細胞にiCas9細胞を解離します。
- 5分間、200×gでhPSCsをペレットおよび2μg/ mLのドキシサイクリンおよび10μMのROCK阻害剤を補充した完全培地中で〜0.5×10 6細胞/ mLで細胞を再懸濁。
- 24ウェルプレートの個々のウェル中に再懸濁した細胞のプレートを0.5mL。非トランスフェクト対照として機能するように、追加の井戸を準備します。
- 各gRNAについては、以下のトランスフェクション混合物を作る:低血清培地で、50μLをミックス+ 1 gRNA(10μM)のμL;ミックスB、μLトランスフェクション試薬の減少血清培地+ 3の50μL。
- 100μLの混合物を作るためにミックスAとBを結合します。 RTで5分間インキュベートします。 24ウェルプレートの複製ウェル中の細胞に混合物の50μLを加え、よく混ぜます。
- さらにターゲティング効率を高めるために、必要に応じて1日目に、第二のトランスフェクションを行います。それ以外の場合は、ドキシサイクリンを含まない培地を変更します。
- 日2日- 3、毎日培地を変更してください。
- 4日目に、DNA抽出キットを使用して、各トランスフェクトし、非トランスフェクト対照細胞の1つのウェルからのゲノムDNAを抽出します。 50 ngの/μに濃度を調整しますL.
- gRNAターゲティング配列に隣接する標的領域をPCR増幅すると、以前に5説明したように、T7エンドヌクレアーゼI(T7EI)消化または制限酵素断片長多型(RFLP)アッセイを使用して、編集効率を推定します。
- T7EIアッセイ
- ステップ3.1で設計および検証プライマーを用いて標的領域をPCR増幅します。
- 混合物を調製した ( 表6を参照)、 表7に概説した条件を用いてPCR産物のDNA変性およびハイブリダイゼーションを行います。
注:我々の経験に基づいて、我々のPCR条件を使用した場合T7EIアッセイのためにPCR産物を精製するために一般に必要ではありません。しかし、精製は、他の条件で有益であり得ます。 - 変性の10μLを使用して、37℃で30分間T7EI消化を行い、PCR産物とT7E1の0.2μL(2 U)(10 U /μL)をハイブリダイズさせました。
- RESOLVゲル電気泳動によってT7E1消化PCRサンプルを電子。カットノーカットDNAの相対的なバンド強度を決定するためのImageJを使用してください。式を用いてインデル頻度を計算する:(1 - (√(1-(B + C))/(A + B + C))が)未消化PCR産物の強度であり、b及びcは100とx T7E1-切断された製品の強度。
- RFLPアッセイ
注:Cas9切断部位(PAM配列の3塩基対5 ')に制限部位が近接している場合(<5塩基対)は、RFLPアッセイはインデル頻度を定量化するために行うことができます。- ステップ3.1.4.1で説明したのと同じPCR産物を使用してください。
- Cas9切断部位に近接する制限部位を含む制限酵素でPCR産物を消化。
- ゲル電気泳動により消化したPCRサンプルを解決します。カットノーカットDNAの相対的なバンド強度を決定するためのImageJを使用してください。インデルを計算式を用いて、周波数:A /(A + B + C)は、未消化PCR産物とbとcの強度であり、消化産物の強度がある場合、×100。
- クローン変異株の樹立
注:gRNAトランスフェクションでiCRISPRシステムを使用してhPSCsにおけるゲノム編集は非常に効率的であり、何の抗生物質の選択は必要ありません。クローン株を確立するためには、単一細胞由来のコロニーの形成を確実にするために、比較的低密度で細胞を播種することが必要です。- (T7EIまたはRFLPアッセイを使用して)最高の編集効率を有すると良好な細胞生存とgRNAsを識別します。クローン変異体のラインを確立するためによく対応する重複を使用してください。
- ステップ2.5で説明したように、1×解離試薬を使用して、単一細胞懸濁液にhPSCsを解離させます。 3 VTN-コーティングされた、10cmディッシュの各々に500、1,000及び2,000細胞をReplate。
- メディア毎日のuを変更単一細胞コロニーは直径〜2ミリメートルに達しntil。
- T7EIおよび/またはRFLPアッセイによってターゲティング効率の推定に応じて、各gRNAのための48個のコロニー - 24を選択してください。機械的に23-G針を使用して小片(コロニーあたり〜10個)に各コロニーを脱凝集(200-μLピペットチップでも結構です)と重複VTN-コーティングし、96ウェルプレート中の細胞をreplate。ゲノムDNAの抽出およびサンガー配列決定およびさらなる拡大のために他のプレートのための1つのプレートを使用してください。
- 96ウェルプレート中の細胞がコンフルエントになっている場合には、以下に概説するように、簡単なプロトコルを使用して(フェノール/クロロホルム抽出なし)のゲノムDNAを抽出します。
- 培地を除去およびCa 2+およびMg 2+を含まないPBSで細胞を1回洗浄します。 96ウェルプレートの各ウェルに溶解緩衝液50μL(プロテイナーゼK(10 mg / mlで)、PCR緩衝液10倍の5μL、とのddH 2 Oの40μLの5μL)を追加します。接着フィルムとincubaを使用してプレートをシールテ一晩で55℃。
- 翌日、96ウェルPCRプレートに細胞溶解物を転送し、プロテイナーゼKを不活性化するためにサーモサイクラー中で99℃で10分間インキュベート
- テンプレートとして細胞溶解物の1μLを使用して、T7EIまたはRFLPアッセイと同じプライマーを用いて標的領域をPCR増幅します。
- PCR産物の内部のプライマー結合でサンガー配列決定のためのPCR産物の1μLを使用してください。
- 凍結ストックのためのフレームシフトインデル変異を有するクローンを増幅します。また、さらなる実験のための同質遺伝子の制御線として機能するように、同じ標的と実験から、野生型クローンのカップルを増幅します。
- gRNAの設計と製造
- 正確なヌクレオチド変異を有する変異株の作製
注:(NHEJ)の非相同末端結合により生成されたノックアウト変異体と比較して、正確なヌクレオチド変化は、DNA repaiの存在下での相同性指向性修復(HDR)を介して達成することができますrのテンプレート。このような正確なヌクレオチド変異は、野生型hPSCsにおける患者特異的変異の生成のためにと患者由来のiPS細胞における変異の補正を可能にします。- HDRテンプレートとして一本鎖DNAの設計
- ステップ3.1.1で説明したように、患者特異的変異に近接した3 gRNAs - デザインとは、2を生産します。
- HDRテンプレートとしてそれぞれの側にホモロジーの80塩基 - 〜40によって挟まれた患者特異的変異を含む一本鎖DNA(一本鎖DNA)を設計します。
- 可能な場合は、正しい修理後の追加の切断を低減するために、PAM配列に対して、またはPAM配列自体にgRNA認識配列内および近接した地域のssDNA鋳型にサイレント変異を導入。
- 可能な場合は、RFLPアッセイを用いて救済効率を推定するために使用することができるように、同様に新規な制限消化部位を導入するためにサイレント変異を設計します。
- gRNA / ssDNAを同時トランスフェクションおよびクローン株の樹立
- gRNAおよび非トランスフェクト対照それぞれについて、トランスフェクション混合物A及びBと、ステップ3.1.3で説明したように、iCas9細胞にgRNA /一本鎖DNAの同時トランスフェクションを行う24ウェルプレートの複製ウェルで細胞をトランスフェクトします。ミックス:低血清培地の50μL+ 1 gRNAのμL(10μM)+ 2本鎖DNAのμL(10μM)。 Bを混ぜる:μLトランスフェクション試薬の減少血清培地+ 3の50μLを。
- トランスフェクションの後、各トランスフェクトし、非トランスフェクト対照細胞の1つのウェルからのゲノムDNAを抽出し、T7EI及び/又はRFLPアッセイを用いて救済効率を推定します。
- 最高の修復効率と良好な細胞生存とgRNA / ssDNAの混合物を特定します。クローン変異体のラインを確立するためによく対応する重複を使用してください。
- T7EIによってターゲティング効率の推定および/またはRFLPのお尻に応じて、96個のコロニー - 48ピックAY。一般に、HDR媒介突然変異の効率は、変異ノックアウトよりも低く、したがって、より多くのコロニーを採取する必要があります。
- シーケンス、展開して、ステップ3.1.6で説明したように、クローン株を検証します。
- HDRテンプレートとして一本鎖DNAの設計
グルコース応答性膵β細胞へのインビトロ HPSC分化4.
注:HPSC突然変異体のin vitroでの分化に疾患関連細胞型に皿の中の病気のモデリングのためのプラットフォームを提供します。以下のプロトコルは、膵臓の発達および糖尿病の研究9、16、17のためのグルコース応答性膵β細胞へhPSCsのin vitroでの分化に焦点を当てています。
- 胚体内胚葉への分化HPSC
- 説明するように、化学的に定義され、無フィーダー条件でHPSC変異体と野生型のコントロールラインを維持しますステップ1中のd。
- 、分化にhPSCsを準備するために、1×解離試薬を用いhPSCsを解離し、完全培地中で単一細胞懸濁液にそれらを分散させます。
- 5分間、200×gで細胞をペレット化し、10μMのROCK阻害剤との完全培地中で細胞を再懸濁。細胞数をカウントし、VTN被覆プレート上/ cm 2の〜1.4×10 5細胞で細胞を播種します。
- 播種後培地の24時間を変更します。
- 0日目に、細胞は、~80%コンフルエントに達したとき、48時間後に分化を開始します。
注:48時間密集度の播種密度とレベルを最適化し、高膵臓分化効率を達成するためには、個々のラインのために推奨されます。 - HPSC培地を吸引およびCa 2+およびMg 2+なしで一度PBSで細胞を洗浄します。
- 分化0日目(D0)を培地に培地を変更します。
- 1日目- 2、ジを変更表8のレシピによると、中毎日fferentiation。
- 3日目に、免疫染色による胚体内胚葉マーカーSOX17、FOXA2、およびCXCR4を検討し、フローサイトメトリー分析。
- 膵臓前駆への胚体内胚葉分化
- 3日目- 9、 表9のレシピによると、毎日培地を交換することにより、膵臓細胞系統に向けて胚体内胚葉分化を続けます。
- 7日目に、免疫蛍光染色により、早期膵臓前駆(PP1)マーカーPDX1を調べ、フローサイトメトリー分析。
- 10日目には、それ以降の膵臓前駆(PP2)マーカーPDX1およびNKX6.1を調べます。また、膵臓内分泌細胞へのさらなる分化のための気液界面にPP2細胞を転送するために準備します。
- 膵内分泌が異なります気液界面でentiation
- 4時間解離前に10μMのROCK阻害剤PP2細胞を扱います。
- 培地を除去およびCa 2+およびMg 2+なしで一度PBSで細胞を洗浄します。
- 1つの10 cmのディッシュにPP2細胞に1×解離試薬を2mLを加え、2、37℃でインキュベートする - 3分。
- 細胞が剥離している前に、解離試薬を吸引。 BLAR培地10 mLを加え、穏やかにピペッティングにより単一細胞へのPP2細胞を分散させます。
- 単一細胞懸濁液を収集します。 5分間、200×gで細胞数およびペレットをカウントします。
- S5の分化培地中で〜0.5×10 5細胞/μLで細胞ペレットを再懸濁し、5スポット-トランスウェルインサートフィルターにスポット当たりの細胞の10μLを。 10 cmの挿入で1 6ウェルインサート中の15スポットや〜100スポット - 10を配置します。
- 、〜1.5 6ウェルインサート用mLおよび〜8 mLのF各トランスウェルインサートの底にS5媒体を追加します。または10 cmの挿入。
- 表10のレシピで毎日培地を変更します。
- 免疫蛍光染色によって、34日目に膵臓内分泌マーカーPDX1、NKX6.1 NEUROD1、NKX2.2、インスリン、およびグルカゴンを調べ、分析17フローサイトメトリー。
- グルコース刺激インスリン分泌(GSIS)アッセイ16、17とHPSC由来のβ様細胞の機能を調べます。
結果
HPSC化学的に定義されており、フィーダーフリー文化適応とメンテナンス
iMEFフィーダー上で培養hPSCsは急速に無フィーダー培養条件でVTNコートプレートに適合させることができます。通常iMEFフィーダー培養と同じ分割比は、適応の間に使用することができます。 図1Aは、KSRベースの培地中のフィーダーを含まない培地中でVTN被覆表面上iMEFフィーダー上hPSCsの典型的な形態を示しています。 図1Bは、典型的な形態変化と適応(4日)の最初の通過中HPSCコロニーの成長を示しています。細胞は、さらに将来の実験のために継代または凍結することができます。分化実験を開始する前に、適応文化の3継代 - 2を行います。私たちは適応段階の間に核型異常を認められていないが染色体分析はまた、適応後に推奨されます。
「FO:キープtogether.within-ページ=「ターゲティングTALEN媒介AAVS1を通じてiCas9 HPSC行の1 ">ジェネレーション先に示したように、hPSCsはAAVS1 TALENプラスミドとCas9とM2rtTAプラスミド対5でエレクトロポレーションしました。 図2AおよびBは、デザインとiCas9 HPSCクローン株を生成するための手順全体をターゲットに詳細なドナーベクターを示しています。 ( - 図2C、12日エレクトロポレーション後10)抗生物質選択後、適当な大きさと典型的なHPSCの形態を示す単一細胞由来のクローンを採取する準備ができました。 ( 図2D)をサザンブロッティング5によって検証として通常〜クローンの50%が正しく、ランダム統合せずに標的にされています。ランダムな統合は、ドキシサイクリン処理の不在下でCas9の漏出性発現を引き起こす可能性があります。
効率的な遺伝MiCRISPRプラットフォームを使用したhPSCsでodification
設立iCas9 hPSCsでは、Cas9はドキシサイクリン処理で表現され、それがDSBのを生成し、トランスフェクトgRNAs、によってその標的遺伝子座に導かれます。修理テンプレートが存在しない場合には、NHEJを介して、DNA修復は、多くの場合、遺伝子破壊またはノックアウトにつながるインデルを生成します。修復テンプレート( 例えば、一本鎖DNA供与体)の存在下では、HDRは、野生型HPSC背景で患者特異的変異生成またはpatient-における疾患関連遺伝子変異の補正として、正確な遺伝子改変のために使用することができます派生iPS細胞( 図3A)。それはiCRISPRシステムを使用してクローン変異株を生成するために〜1ヶ月かかります。 iCas9誘導およびgRNAトランスフェクション後、T7EI及び/又はRFLPアッセイをCas9切断効率を評価するために使用し、そしてトランスフェクトされた細胞は、後で〜(低密度で10cmの皿の中に単一細胞として500播種した - 2、000細胞/ 10 cmディッシュ)。 10 - 12日間後、単一細胞由来のクローンを拡大し、さらなる特性( すなわち、遺伝子型決定およびウェスタンブロッティング)( 図3B)のための96ウェルプレートのウェルに入れました。 iCRISPR媒介遺伝子ノックアウトは非常に効率的であるため、何も選択プロセスが関与していない、そして通常20 - 50%二対立遺伝子変異体は、容易に( 図3C)5を達成することができます。効率的かつ正確な遺伝的変化のために、gRNAsは、(ゲノム配列の小さいが、具体的な変更のための)特定の配列変化を運ぶのssDNAドナーと同時トランスフェクトされています。多くの場合、修正された対立遺伝子で再切断を防ぐために、PAMシーケンス( 図3D)またはで近接してサイレント変異を含むことが推奨されます。このシステムでは、両方の対立遺伝子における追加変更することなく、目的のHDR-媒介ゲノム改変を有するクローンの〜10%が( 図3E)を達成しています。迅速及びpiCRISPRプラットフォームを使用してHPSCゲノムのreciseの変更は、私たちは迅速かつ効率的に人間開発や病気を研究するためのモデルとして役立つHPSCラインを作ることができます。
グルコース応答性β様細胞に向かってhPSCsの効率的な分化
HPSC膵臓分化における最近の進歩は、密接に膵臓の胚発生を再現プロトコルの開発を可能にしました。未分化のhPSCs最初の16後、PDX1 +早期膵臓前駆細胞(PP1)およびPDX1 + NKX6.1 +、後に膵臓前駆細胞(PP2)に、胚体内胚葉に分化し、最終的にグルコース応答性β様細胞への、17、19、20 、23、24。これらのprotocオールをさらに確実にPDX1 + NKX6.1 +膵臓前駆細胞およびグルコース応答性β様細胞9を生成するために最適化しました。 図4Aは、分化の各段階で使用される詳細な化学物質のサプリメントを示しています。通常、2日最初の細胞播種後〜80%の密集度はHUES8 hPSCs( 図4B)の分化を開始するのに最適です。いくつかの異なる播種密度をテストすることは非常に各特定の細胞株について最適化された条件を発見することをお勧めします。通常PP2の段階で少なくとも75%FOXA2 + SOX17 + DE段階の細胞および40%のPDX1 + NKX6.1 +細胞( 図4C)を達成することができます。 S5の段階では、細胞凝集体の周りにオーラのような形状の存在は(NKX6.1 + CPEP +グルコース応答性β細胞様細胞へのさらなる分化のために重要である、細胞の良好な生存のための指標である図4D )。このプロトコルは、ヒト膵臓dを研究するために有用です皿の中のevelopmentと病気。

図1. HPSCは、化学的に定義されており、フィーダーフリーメンテナンスとiMEFフィーダから文化適応。 hPSCsの(A)代表的な画像を分割する準備ができて、4日目にiMEFフィーダー上またはVTN上で培養しました。 (B)iMEFフィーダーからのフィーダーを含まない培養への適応の最初の通過中hPSCsの典型的な形態。 iMEFフィーダー上で培養4日目hPSCsは、ROCK阻害剤と化学的に定義された培地でVTNコートプレート上に分割し、プレーティングしました。分割比はiMEFフィーダー条件で培養するための通常の分割比と同じでした。培地は、ROCK阻害剤なしで毎日変更されました。 この図の拡大版をご覧になるにはこちらをクリックしてください。
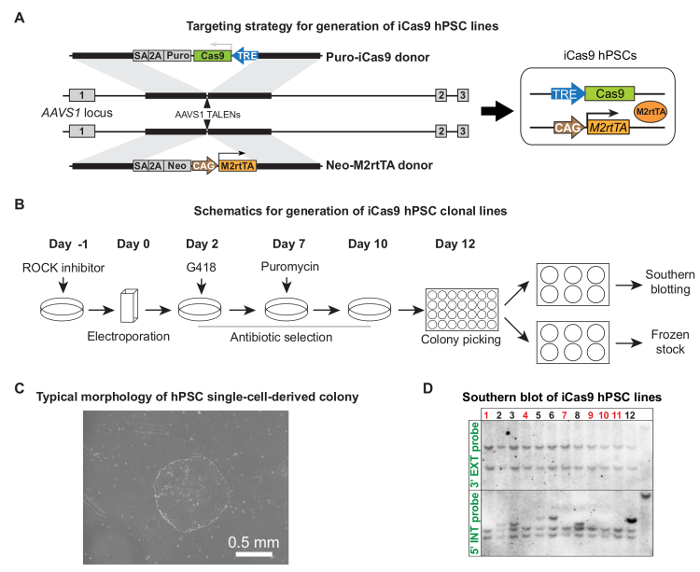
iCas9 HPSCラインの図2世代。 (A)iCas9 HPSC株の生成のための戦略をターゲットに。プーロ-Cas9ドナーとネオM2rtTAドナーがAAVS1 TALENsのペアによって、ヒトAAVS1遺伝子座に標的にされました。 (B)電気穿孔、抗生物質選択、コロニーピッキング、拡張、および細胞株の特徴付けを含むプロセスをターゲット全体の概略図。 (C)約10でピックアップする準備ができて代表単一細胞クローン-エレクトロポレーション後12日間。 (D)は、2つのドナープラスミドの追加の統合を使用せずに正確に標的クローンを同定するためのサザンブロット例。正しいクローンは赤でマークされています。 (A)及び(D)は、許可を得て、参考5から適応させました。ig2large.jpg "ターゲット=" _空白 ">この図の拡大版をご覧になるにはこちらをクリックしてください。
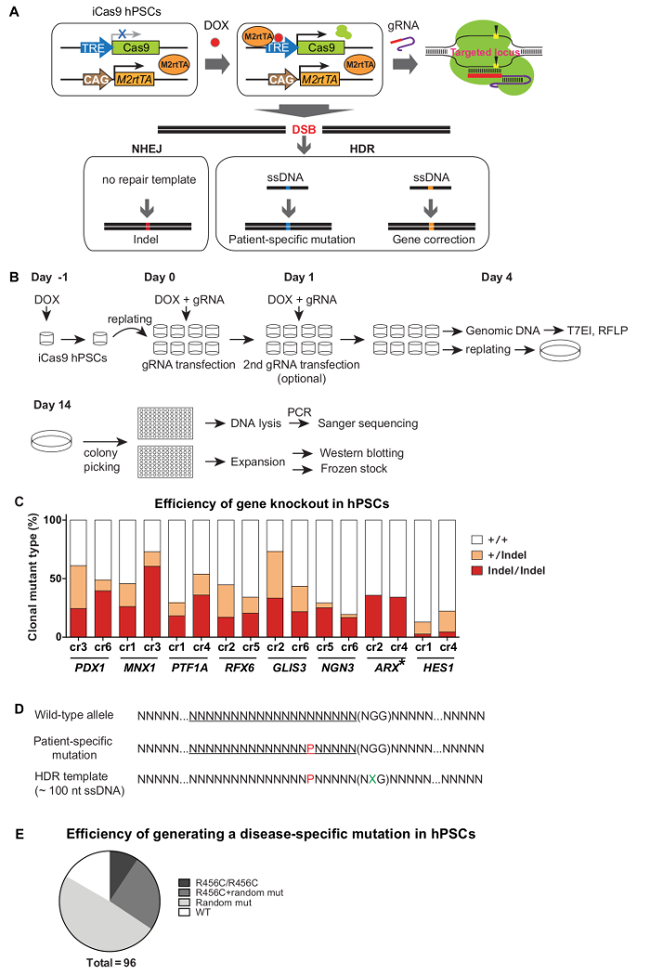
図3. iCRISPRシステムを使用したhPSCsにおける効率的な遺伝子改変。遺伝子ノックアウト変異体またはiCRISPRシステムを使用して、正確な遺伝子改変を生成するための(A)回路図。 (B)の手順とクローン株の確立をターゲットに。 hPSCs 9 NHEJを介して、(C)遺伝子ノックアウト効率。 (D)特定の塩基修飾を運ぶのssDNAの設計。 P、赤:患者特異的変異;緑の中のX、:サイレント変異。 HDR媒介正確なヌクレオチド修飾の(E)効率。 GATA6座に(特定の塩基C> T変異によって引き起こされる)正確なR456C変異はiCRISPRシステムを使用して導入しました。(詳細については5を参照を参照してください)。 (C)及び(E)はパーミッションで、それぞれ、リファレンス9およびリファレンス5から適応させました。 この図の拡大版をご覧になるにはこちらをクリックしてください。
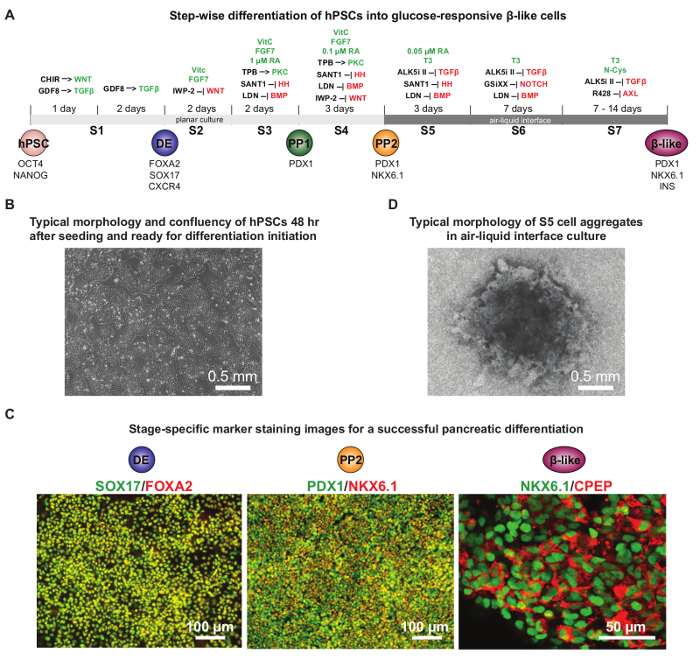
グルコース応答性膵β細胞様細胞への図4.ダイレクトHPSC分化。分化の各段階で補足した化学物質による詳細な分化プロトコルの(A)回路図、。分化の間に活性化または阻害されるシグナル伝達経路は、それぞれ、緑色または赤色で強調表示されます。 CHIR:GSK3阻害剤; GDF8:成長分化因子8またはミオスタチン、TGF-βタンパク質ファミリーのメンバー; HH:ハリネズミ。 DE:胚体内胚葉; PP1:PDX1 +早期膵臓前駆; PP2:PDX1 + NKX6.1 +、後に膵臓前駆。 (B)の典型的な密集度-時分化開始のための準備ができて播種後hPSCs 48時間の(70〜80%)。 (C)胚体内胚葉に成功した分化を示す代表的免疫蛍光染色画像(DE)ステージ(FOXA2及びSOX17の共染色)、膵臓前駆期(PP2:PDX1およびNKX6.1共染色)、およびグルコース応答性β-細胞期(NKX6.1およびc-ペプチド同時染色)などです。一般に、β様細胞段階で10%以上のNKX6.1 + CPEP +細胞を得るために、少なくとも75%FOXA2 + SOX17 + DE細胞および40%のPDX1 + NKX6.1 + PP2細胞は、対応する段階で必要とされます。 (D)気液界面培養における挿入フィルター膜上のS5の段階での細胞集合体の典型的な形態。生き残った細胞は(暗所で)集合体の中央部に移行し、エッジ上のオーラのような構造を残しています。 ( )および(D)は、権限を持つリファレンス9から適応させました。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| プラスミド | 量 |
| AAVS1-TALEN-L | 5μgの |
| AAVS1-TALEN-R | 5μgの |
| AAVS1-ピューロ-iCas9 | 40μgの |
| AAVS1-ネオM2rtTA | 40μgの |
表1
| プライマー | シーケンス |
| T7F | TAATACGACTCACTATAGGG |
| TracrR | AAAAGCACCGACTCGGTGCC |
表2
| 成分 | 量 |
| ddH 2 O | 35.5μL |
| 5X PCR反応緩衝液 | 10μL |
| dNTPミックス(25mMの) | 0.5μL |
| T7F(10μM) | 1.25μL |
| TracrR(10μM) | 1.25μL |
| T7-gRNA IVTテンプレート(250 nM)を | 1μL |
| DNAポリメラーゼ | 0.5μL |
| 全体のPCR反応ミックス | 50μL |
表3
| PCRサイクル条件 | |||
| サイクル数 | 変性 | 焼きなまし | 広がります |
| 1 | 94ºC、2分 | ||
| 2月31日 | 94ºC、20秒 | 60ºC、20秒 | 72ºC、1分 |
| 32 | 72ºC、2分 | ||
表4
| 成分 | 金額 |
| T7 ATP | 2μL |
| T7のCTP | 2μL |
| T7 GTP | 2μL |
| T7のUTP | 2μL |
| T7 10×バッファー | 2μL |
| T7酵素ミックス | 2μL |
| PCR増幅した鋳型 | 8μL |
| 総インビトロ gRNA転写ミックス | 20μL |
| 一晩に6時間、37ºCでインキュベートします |
表5
| 成分 | 量(μL) |
| 未精製PCR産物 | 8 |
| バッファ2の10倍 | 2 |
| 蒸留水(のdH 2 O) | 10 |
表6
| DNA変性およびハイブリダイゼーションサイクリング条件 | ||
| 温度 | デュレーション | サーマルサイクラー条件 |
| 95°C | 10分 | |
| 85°C | 1分 | 2°C / sで85℃に上昇 |
| 75°C | 1分 | 0.3℃/秒で75℃まで上昇 |
| 65°C | 1分 | 0.3℃/秒で65℃まで上昇 |
| 55°C | 1分 | 0.3℃/秒で55℃まで上昇 |
| 45°C | 1分 | 0.3℃/秒で45℃まで上昇 |
| 35°C | 1分 | 0.3℃/秒で35℃まで上昇 |
| 25°C | 1分 | 0.3℃/秒で25℃まで上昇 |
| 4°C | ホールド | |
表7
| ステージ | 日 | メディア | 補足 | |
| S1 | D0 | S1 | GDF8 100ng / mlの | CHIR-99021 3μM |
| D1 | S1 | GDF8 100ng / mlの | CHIR-99021 0.3μM | |
| D2 | S1 | GDF8 100ng / mlの | ||
| S1媒体:MCDB 131 + 1×L-グルタミンサプリメント+ 0.5%BSA +1.5グラム/ LのNaHCO 3 + 10mMのグルコース | ||||
表8
| ステージ | 日 | メディア | 補足 | |||||||
| S2 | D3-D4 | S1 | LAA 0.25 mMの | FGF7 50 ngの/ mLの | IWP-2 2.5μM | |||||
| S3 | D5-D6 | S3 | LAA 0.25 mMの | FGF7 50 ngの/ mLの | SANT-1 0.25μM | RA 1μM | LDN 100 nMの | TPB 200 nMの | ITS-X 1:200 | |
| S4 | D7-D9 | S3 | LAA 0.25 mMの | FGF7 2 ngの/ mLの | SANT-1 0.25μM | RA 0.1μM | LDN 200 nMの | TPB 100 nMの | ITS-X 1:200 | IWP-2 2.5μM |
| S3媒体:MCDB 131 + 1×L-グルタミンサプリメント+ 2%BSA +2.5グラム/ LのNaHCO 3 + 10mMのグルコース | ||||||||||
表9
| ステージ | 日 | メディア | 補足 | |||||||
| S5 | D10-D12 | S5 | T3 1μM | ALK5i II 10μM | SANT-1 0.25μM | RA 0.05μM | LDN 100 nMの | ITS-X 1:200 | ZnSO 4、10μM | ヘパリンを10μg/ mLの |
| S6 | d13- D19 | S5 | T3 1μM | ALK5i II 10μM | GSiXX 100 nMの | LDN 100 nMの | ITS-X 1:200 | ZnSO 4、10μM | ヘパリンを10μg/ mLの | |
| S7 | d20- D33 | S5 | T3 1μM | ALK5i II 10μM | N-Cysを1mMの | トロロックス10μM | R428 2μM | ITS-X 1:200 | ZnSO 4、10μM | ヘパリンを10μg/ mLの |
| S5媒体:BLAR + 1X L-グルタミンサプリメント+ 2%BSA +1.5グラム/ LのNaHCO 3 + 20mMのグルコース | ||||||||||
表10
ディスカッション
生成突然変異系統のための時間に関する検討事項
ゲノム編集のためのCRISPR / CASシステムに基づいて、最近のアプローチが成功した標的につながっているが、より効果的かつ普遍的なプラットフォームは、遺伝子機能の大規模解析のために好ましいであろう。 iCRISPRプラットフォームは利息5、9のいずれかの遺伝子に変異を導入するための迅速かつ効率的な方法を提供しています。まず、PCRベースのgRNA合成方法は、時間のかかるクローニングステップなしで一日に配列形式でgRNAs数百の製造を可能にします。第二に、iCas9 hPSCsにおけるドキシサイクリン誘導性Cas9発現と、gRNAトランスフェクションを伴うステップは仕事の最小限の量を必要とし、したがって、複数のgRNAターゲッティング実験を同時に行うことができます。我々のシステムの分析で達成第三に、高いターゲティングによる効率〜24 - トランスフェクトgRNAあたり48コロニーがsuffでなければなりません効率は、標的遺伝子座に依存して変化するがicientは、単一の遺伝子のための複数の1アレル及び対立遺伝子変異体系統を確立します。訓練を受けた個人は、機械的に、一気に384コロニー(4×96ウェルプレート)を選択するために解剖顕微鏡下で約4時間を取るべきであることが可能であるので、訓練を受けた個人は、内12の遺伝子に影響する変異株を作製することが期待できます1から2ヶ月。短時間でHPSC変異体の合理化された世代は発達過程9を規制する 、互いに相互作用する転写因子および/またはシグナル伝達経路成分の配列の体系的な分析を可能にします。また、効率的な多重遺伝子ターゲティングは、複雑な人間の形質の根底にある遺伝的相互作用を調査への扉を開きます。
正確な遺伝子改変の生成
簡単にアクセスできる体細胞型からの患者固有のiPS細胞の誘導 sおよび疾患関連細胞型への分化は、疾患関連変異の機能的検証のための素晴らしい機会を提供します。しかし、個体間の遺伝的背景にはかなりのばらつきに、患者からiPS細胞間および健康なドナーからの直接比較は1つが、背景効果から疾患の表現型を区別することができない場合があります。したがって、バック野生型配列へ病変異を補正して同系制御性IPSCを生成する等25,26によって提案されているように、野生型HPSC背景に患者特異的変異を導入することが必要です。機能喪失(ヌル)の変異に加えて、一つは今より正確に、hypermorphic低形質、neomorphic、またはドミナントネガティブ患者を含むhPSCsにおける内因性遺伝子座の中に患者特異的配列改変を導入することにより病気のメカニズムを分析することができます突然変異。
それはNHEJ媒介DNA修復よりもはるかに効率が低いので。修理テンプレートの存在下で、DSB修復のためのHDRを採用して正確な遺伝子改変ntent ">、患者特異的変異、薬剤選択カセット、および相同性アームを含むドナープラスミド以前に患者特異的変異の薬剤選択と検証した後、第二段階は、一般的に薬剤選択カセットを除去するために必要である。最も広く使用されているのCre-loxP配列とFLP-FRTシステムは残しながら。修理テンプレート2として使用されています内因性遺伝子座における残留シーケンス、のpiggyBacトランスポゾンの使用は、薬剤選択カセット27のシームレスな除去を可能にした。さらに最近では、短い一本鎖DNAテンプレートはまた、操作されたDNAエンドヌクレアーゼ28で効率的なHDRをサポートすることが示されている。ドナープラスミドと比較すると、一本鎖DNAを直接合成し、従って、時間のかかるクローニングステップを回避することができる。ここで、であるました同時トランスフェクションgRNAと相同性指向性修復を誘導するssDNA鋳型によって、iCRISPRが高効率で特定のヌクレオチド修飾を導入することができる、ことを示しアン。これは、タンパク質の機能的ドメイン内だけでなく、ヒト疾患変異をモデル化し、潜在的な治療的介入のためのこれらの疾患関連変異を補正するための必須のヌクレオチドの役割を解剖するだけでなくするために重要です。各糖尿病などの複雑かつ多遺伝子疾患をモデル化し、複数の配列変異体に関連付けられている感受性遺伝子座の数が多いために遺伝学者のための課題でした。 iCRISPRプラットフォームは、対立遺伝子シリーズの迅速な生成のためにまたは多重化可能遺伝子ターゲティングを許容するように、個別にまたは同質遺伝子的背景との組み合わせで、複数の疾患関連遺伝子座の調査を容易にすることができます。効率およびオフターゲット効果をターゲット
我々は持っています T7E1とRFLPアッセイの結果及び配列決定により同定された変異体のライン数との間に良好な相関関係を発見しました。これは、クローン株の確立と並行して、これらのアッセイを行うことの重要性を強調しています。得ターゲティング効率は、ゲノム遺伝子座に依存して変化し得るが、ほとんどの単一遺伝子ターゲティング実験において、20 -クローンの60%が(フレームおよびフレームシフト変異を含む)変異した対立遺伝子9の両方で発見されました。多重遺伝子ターゲティングを行った場合には、5〜10%の効率で三重二対立遺伝子突然変異体のクローンが5を得ました。 10%5 -いくつかの遺伝子の一本鎖DNA媒介HDRはまた、ホモ接合ノックイン1の範囲のクローンを得るための効率で、正確な遺伝的変化を得るために行われました。 hPSCsにCRISPR / CASでの作業、同じgRNA標的配列を共有していない潜在的なオフターゲットサイト内の任意の突然変異がまだ検出されるべきです小娘= "外部参照"> 5、9、12。最近の研究で行われる全ゲノム配列決定はまた、CRISPR / Casの29を使用して生成されたクローンHPSCラインに実質的なオフターゲット変異を同定するために失敗しました。それにもかかわらず、オフターゲット効果部位での変異によって導入表現型を交絡の潜在的な影響を最小限にするために、同じ遺伝子内の異なる配列を標的とする少なくとも2つの独立しgRNAsを使用して独立した変異株を作製することが提案されています。別gRNAsを使用して生成された複数のラインで観察された類似の表現型は、主に表現型がオフターゲット効果から来ている可能性を除外することができました。
フィーダー依存独立した文化とターゲット対
HPSC培養のための従来の方法は、動物のPRODを含み、血清または血清代替を含む培地中でフィーダー細胞上のそれらの維持・拡大を伴いますウシ血清アルブミンのようなUCTS。フィーダー細胞、血清、血清代替品、およびアルブミンすべては、複雑な、未定義の成分が含まれており、かなりのバッチ可変性を示します。無フィーダーと化学的に定義された条件への適応が大幅HPSC維持のための努力を低減し、より重要なのは、分化実験の一貫性を向上させます。現時点では、完全に定義された培養条件30で培養hPSCsのゲノム編集手順を説明し、非常に少数の研究があります。我々はgRNAトランスフェクションおよびクローン堆積は、フィーダー依存培養条件と比較して、無フィーダー条件で行ったときの効率をターゲットに高いCRISPRを発見しました。これはコロニー形成のためのトランスフェクションおよび単一細胞播種後に増加した細胞の生存のためであると考えています。また、フィーダー細胞は、以前にそれによってトランスフェクション効率を低下させる、トランスフェクション試薬を隔離することが示されています。
結合ダイレクト差別とゲノムの編集
前分化プロトコルはインスリン陽性細胞のわずかな部分のみが得られている、の大半はpolyhormonalたと胎児の内分泌細胞23に似ていました 。最近の進歩は、より成熟したグルコース応答性β様細胞16、17、19、20にhPSCsの分化を可能にしました。私たちは日常的にHUES8 hPSCs 9を用いて、少なくとも75%の胚体内胚葉細胞、40%のPDX1 + NKX6.1 +膵臓前駆細胞、および約20%のNKX6.1 + CPEP +グルコース応答性β様細胞を得ることができます。 iCRISPRゲノム編集システムと組み合わせると、このより堅牢な分化プロトコルは、膵臓分化の膵臓前駆細胞および内分泌段階に不可欠である転写因子の解析を容易にしました。これは、ようになりますそれが可能な、将来の研究では、糖尿病9の根底にあるメカニズムへの機能検証や調査のための候補疾患遺伝子の大規模な数を調べました。
将来のアプリケーションや行き方
我々のiCRISPRシステムは長いドナーDNAテンプレートをコードするタンパク質タグまたは蛍光レポーター12を使用して標的とHDR媒介遺伝子を通じて、このようなレポーター対立遺伝子の作成など、より複雑なゲノムの改変、の生成を容易にすることができます。我々は、システムにおいて達成効率を標的ハイCRISPRに、このプロセスはさらに、薬剤選択12を必要とせず、hPSCsに行うことができることを示しています。さらに、標的化多重遺伝子は、我々は、このようなワーク31の第一の例であると考えて我々の最近の研究で示されているように、複雑なヒト疾患の根底にある遺伝的相互作用を調査するために使用することができます。 iCRISPRはまた、プロモーターおよびエンハンサーなどの調節領域の欠失を作成することにより、いずれかの非コードRNAまたは遺伝子における遺伝子調節制御を理解するために使用することができます。効率iCRISPRを使用して調節変異体を生成するために、gRNAsを含む、DNA結合タンパク質の結合部位を破壊するように設計されたが、基本転写機構または組織特異的な転写因子に限定されないことができます。 ssDNA鋳型媒介HDRは、特定のタンパク質結合部位を突然変異させるために使用することができます。最後に、我々は、in vitroでの分化プロトコルと組み合わせた場合、さらに最適化が多能性表現型または疾患の表現型のより高いスループット遺伝子解析のためのhPSCsでiCRISPRプラットフォームの使用を可能にするであろうことを想定しています。これらのiCRISPR媒介の研究は、候補疾患関連遺伝子およびそれらの機能的関連性の研究のより迅速な同定を可能にすることができます。
開示事項
著者らは、開示することは何もありません。
謝辞
この作品は、NIH / NIDDK(R01DK096239)とニューヨーク州の幹細胞科学(NYSTEM C029156)によって部分的に資金を供給されました。 ZZはスローンケタリング研究所の幹細胞生物学のためのセンターからNYSTEMポスドクフェローシップによってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| Chemically defined medium (E8) | Thermo Fisher Scientific | A1517001 | Essential 8 basal medium and Essential 8 supplement included |
| Truncated recombinant human form of vitronectin | Thermo Fisher Scientific | A14700 | |
| ROCK inhibitor Y-27632 | Selleck Chemicals | S1049 | |
| Dissociation reagent (TrypLE Select enzyme) | Thermo Fisher Scientific | 12563029 | 1x, animal origin free, recombinant enzyme |
| G418 Sulfate | Thermo Fisher Scientific | 10131035 | Geneticin Selective Antibiotic |
| Puromycin dihydrochloride | Sigma-Aldrich | P8833 | Puromycin Selective Antibiotic |
| DNA Polymerase (Herculase II Fusion) | Agilent Technologies | 600679 | PCR kit |
| MEGAshortscript T7 Transcription kit | Thermo Fisher Scientific | AM1354 | |
| MEGAclear Transcription Clean-Up Kit | Thermo Fisher Scientific | AM1908 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| Opti-MEM medium | Thermo Fisher Scientific | 31985062 | Reduced Serum Medium |
| Lipofectamine RNAiMAX Transfection Reagent | Thermo Fisher Scientific | 13778150 | |
| DNeasy Blood & Tissue Kit | QIAGEN | 69504 | Genomic DNA extraction kit |
| Proteinase K | Roche | 3115879001 | |
| MCDB 131 medium | Thermo Fisher Scientific | 10372-019 | |
| BLAR medium | Thermo Fisher Scientific | Custom-made with a published formulation (Rezania et al., 2014) | |
| L-glutamine supplement (GlutaMAX) | Thermo Fisher Scientific | 35050061 | |
| NaHCO3 | Thermo Fisher Scientific | 144-55-8 | |
| Glucose | Sigma-Aldrich | G8769 | |
| BSA | LAMPIRE Biological Laboratories | 7500855 | Fatty acid free |
| GDF8 | PeproTech | 120-00 | |
| CHIR-99021 | Stemgent | 04-0004 | GSK-3 inhibitor |
| L-Ascorbic acid | Sigma-Aldrich | A4544 | Vitamin C |
| FGF7 | R&D Systems | 251-KG | |
| SANT1 | Tocris Bioscience | 1974 | Hedgehog inhibitor |
| RA | Sigma-Aldrich | R2625 | Retinoic acid |
| LDN | Stemgent | 04-0019 | BMP inhibitor |
| IWP-2 | Tocris Bioscience | 3533 | Wnt antagonist |
| ITS-X | Thermo Fisher Scientific | 51500-056 | |
| TPB | EMD Millipore | 565740-1MG | PKC activator |
| 3,3’,5-Triiodo-L-thyronine (T3) | Sigma-Aldrich | T6397 | |
| ALK5i II | Enzo Life Sciences | ALX-270-445 | ALK5 inhibitor II |
| ZnSO4 | Sigma-Aldrich | Z0251 | |
| Heparin | Sigma-Aldrich | H3149 | |
| GSiXX | EMD Millipore | 565789 | Gamma secretase inhibitor XX, NOTCH signaling inhibitor |
| N-Cys (N-acetyl cysteine) | Sigma-Aldrich | A9165 | |
| Trolox | EMD Millipore | 648471 | Vitamin E analogue |
| R428 | Selleck Chemicals | S2841 | AXL receptor tyrosine kinase inhibitor |
| 24 mm Transwell with insert | Corning Life Sciences | 3414 | |
| Gene Pulser Xcell Electroporation System | Bio-Rad | 1652660 | |
| 0.4 cm Electroporation Cuvettes | Bio-Rad | 1652081 | |
| AAVS1-TALEN-L | Addgene | 59025 | |
| AAVS1-TALEN-R | Addgene | 59026 | |
| AAVS1-Neo-M2rtTA | Addgene | 60843 | |
| AAVS1-Puro-iCas9 | Addgene | 58409 | |
| T7 Endonuclease I | NEB | M0302L | |
| Buffer 2 | NEB | B7002S | NEBuffer 2 |
| Long oligonucleotide | Eton Bioscience or IDT | ||
| SOX17 antibody | R&D Systems | AF1924 | 1:500 |
| FOXA2 antibody | Millipore | 07-633 | 1:100 |
| CXCR4-APC antibody | R&D Systems | FAB170A | 1:25 |
| PDX1 antibody | R&D Systems | AF2419 | 1:500 |
| NKX6.1 antibody | DSHB | F55A12 | 1:500 |
| NKX2.2 antibody | DSHB | 74.5A5 | 1:100 |
| NEUROD1 antibody | Santa Cruz Biotechnology | sc-1084 | 1:100 |
| Insulin antibody | Dako | A0564 | 1:2,000 |
| C-peptide antibody | DSHB | GN-ID4-c | 1:2,000 |
| Glucagon antibody | Sigma-Aldrich | G2654 | 1:1,000 |
参考文献
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Eiges, R., et al. Establishment of human embryonic stem cell-transfected clones carrying a marker for undifferentiated cells. Curr Biol. 11 (7), 514-518 (2001).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- González, F., et al. An iCRISPR Platform for Rapid, Multiplexable, and Inducible Genome Editing in Human Pluripotent Stem Cells. Cell Stem Cell. 15 (2), 215-226 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Braam, S. R., et al. Improved genetic manipulation of human embryonic stem cells. Nat Methods. 5 (5), 389-392 (2008).
- Zhu, Z., et al. Genome Editing of Lineage Determinants in Human Pluripotent Stem Cells Reveals Mechanisms of Pancreatic Development and Diabetes. Cell Stem Cell. 18 (6), 755-768 (2016).
- Kotini, A. G., et al. Functional analysis of a chromosomal deletion associated with myelodysplastic syndromes using isogenic human induced pluripotent stem cells. Nat Biotechnol. 33 (6), 646-655 (2015).
- Carlson-Stevermer, J., et al. High-Content Analysis of CRISPR-Cas9 Gene-Edited Human Embryonic Stem Cells. Stem Cell Reports. 6 (1), 109-120 (2016).
- Zhu, Z., Verma, N., Gonzalez, F., Shi, Z. D., Huangfu, D. A CRISPR/Cas-Mediated Selection-free Knockin Strategy in Human Embryonic Stem Cells. Stem Cell Reports. 4 (6), 1103-1111 (2015).
- Mandegar, M. A., et al. CRISPR Interference Efficiently Induces Specific and Reversible Gene Silencing in Human iPSCs. Cell Stem Cell. 18 (4), 541-553 (2016).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Methods. 8 (5), 424-429 (2011).
- Rezania, A., et al. Production of functional glucagon-secreting alpha-cells from human embryonic stem cells. Diabetes. 60 (1), 239-247 (2011).
- Pagliuca, F. W., et al. Generation of functional human pancreatic beta cells in vitro. Cell. 159 (2), 428-439 (2014).
- Rezania, A., et al. Reversal of diabetes with insulin-producing cells derived in vitro from human pluripotent stem cells. Nat Biotechnol. 32 (11), 1121-1133 (2014).
- Chen, S., et al. A small molecule that directs differentiation of human ESCs into the pancreatic lineage. Nat Chem Biol. 5 (4), 258-265 (2009).
- Nostro, M. C., et al. Efficient generation of NKX6-1+ pancreatic progenitors from multiple human pluripotent stem cell lines. Stem Cell Reports. 4 (4), 591-604 (2015).
- Russ, H. A., et al. Controlled induction of human pancreatic progenitors produces functional beta-like cells in vitro. EMBO J. 34 (13), 1759-1772 (2015).
- Zhu, Z., Gonzalez, F., Huangfu, D. The iCRISPR platform for rapid genome editing in human pluripotent stem cells. Methods Enzymol. 546, 215-250 (2014).
- Soh, C. L., Huangfu, D. CRISPR/Cas9-Mediated Mutagenesis of Human Pluripotent Stem Cells in Defined Xeno-Free E8 Medium. Methods in Molecular Biology. 1498, (2017).
- D'Amour, K. A., et al. Production of pancreatic hormone-expressing endocrine cells from human embryonic stem cells. Nat Biotechnol. 24 (11), 1392-1401 (2006).
- Kelly, O. G., et al. Cell-surface markers for the isolation of pancreatic cell types derived from human embryonic stem cells. Nat Biotechnol. 29 (8), 750-756 (2011).
- Musunuru, K. Genome editing of human pluripotent stem cells to generate human cellular disease models. Dis Model Mech. 6 (4), 896-904 (2013).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell Stem Cell. 18 (5), 573-586 (2016).
- Yusa, K., et al. Targeted gene correction of alpha1-antitrypsin deficiency in induced pluripotent stem cells. Nature. 478 (7369), 391-394 (2011).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Huang, X., et al. Production of Gene-Corrected Adult Beta Globin Protein in Human Erythrocytes Differentiated from Patient iPSCs After Genome Editing of the Sickle Point Mutation. Stem Cells. 33 (5), 1470-1479 (2015).
- Shi, Z. D., et al. Genome Editing in hPSCs Reveals GATA6 Haploinsufficiency and a Genetic Interaction with GATA4 in Human Pancreatic Development. Cell Stem Cell. , (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved