Method Article
인간의 췌장 개발에 리니지의 결정 요인을 들여다보기위한 게놈 편집 및 hPSCs의 감독 차별화
요약
프로토콜은 iCRISPR 플랫폼을 사용 hPSC 돌연변이 라인을 생성하고 설명하는 글루코스 응답 형 β 세포로 hPSCs 차별화. hPSC 지시 분화와 게놈 편집 기술을 결합하면 인간 발달 및 질병 진행의 계보 결정의 역할의 체계적인 분석을위한 강력한 플랫폼을 제공합니다.
초록
자기 갱신 또는 인간 다 능성 줄기 세포를 분화 (hPSCs)에 질의 유전자 기능은 인간 발달을 이해하고 접시에 질병 메커니즘을 해부으로 가치있는 플랫폼을 제공합니다. 이 잠재적 인 응용 프로그램을 활용하는 것은 밀접하게 자신의 생체 대응 요점을 되풀이 질환 관련 세포 유형을 생산하기 위해 질병 관련 유전자 hPSC 돌연변이뿐만 아니라 체외 hPSC 차별화 프로토콜을 생성하는 효율적인 게놈 편집 도구가 필요합니다. iCRISPR라는 hPSCs위한 효율적인 유전자 편집 플랫폼은 AAVS1 궤적에 Cas9 발현 카세트의 TALEN 매개 타겟팅을 통해 개발되고있다. 여기서, 화학적으로 정의 된 배지 및 공급없는 상태에서 배양 된 세포를 사용하여 유도 Cas9 hPSC 라인의 생성을위한 프로토콜을 설명한다. N을 통해 하나의 유전자 녹아웃 또는 hPSCs의 정확한 유전 적 변화에 대한 iCRISPR 시스템 사용에 대한 자세한 절차,온 - 동종 (NHEJ)에 가입 종료 또는 동성 지시 수리 (HDR) 템플릿을 사용하여 정확한 염기 변화를 통해 각각 포함되어 있습니다. 이러한 기술적 인 절차는 설계, 생산의 설명 및 CRISPR 가이드 RNA를 (gRNAs)의 형질을 포함한다; T7E1 또는 RFLP 분석에 의한 CRISPR 돌연변이 속도의 측정; 설립과 클론 돌연변이 라인의 검증 및. 마지막으로, 생체 췌장 배아 발달에 모방에 의한 포도당 응답 췌장 β와 같은 세포로 hPSC 차별화 우리 연대기 절차. 감독 hPSC 차별화와 iCRISPR 기술을 결합하면 췌장 개발과 당뇨병 질병 메커니즘에 대한 우리의 이해를 촉진하는 유전자 기능의 체계적인 검사를 할 수 있습니다.
서문
인간 만능 줄기 세포 (hPSCs는) 할 수있는 기능이 모두 자기 갱신과 세 개의 배아 생식 계통의 모든 파생 상품에 상승을 제공합니다. 이들은 인간 개발 환경에서 세포 과정 요점을 되풀이하기위한 고유 한 플랫폼으로서 작용하여 세포 대체 요법, 질병 모델링 귀중한 자원을 제공한다. 또한 스케일 러블 고 처리량 분석에 대한 실험 세포의 공급원이다. 효율적인 유전자 조작 도구의 부족 및 배양 접시에 배아 복잡한 발육 단계 recapitulating 어렵다는 : 그러나, 진보로 인해 두 가지 문제를 한정하고있다.
유전 적 변형은 정상 개발 및 질병의 유전자 기능을 연구하는 필수적인 도구입니다. 상동 재조합을 통해 접근을 대상으로 고전적인 유전자를 입증하면서 그러나, 마우스 배아 줄기 세포 (mESCs)에서 유전자 기능을 해부하는 강력한 도구 하나,이 방법이 될 수 있습니다hPSCs 2, 3에 적용 할 때 매우 비효율적이었다. 징크 핑거 뉴 클레아 제 (ZFNs), 전사 활성제와 같은 이펙터 뉴 클레아 제 (TALENs) 및 클러스터 정기적으로 interspaced 짧은 회문 반복 (CRISPR) / CRISPR은 관련을 포함하여 실험실 사용에 자연 프로그램, 사이트 별 클레아의 최근 활발한 가입 (CAS) 시스템 (4)은 유전체 공학 hPSCs 포함한 유기체 및 세포주의 넓은 범위에서 훨씬 쉽게 작업이 될 것을 의미한다. 이러한 유전자 편집 도구 등 Cas9 효소로서 키메라 클레아 어느 비 - 상동 활성화 정확한 위치에서 이중 가닥 나누기 (DSBs)을 유발하는 내인성 DNA 수리 장치를 트리거하여 유전 적 변형의 전체 범위를 허용 할 수 있다는 사실을 활용 (NHEJ)에 가입 종료하거나 상동 - 감독 수리 (HDR). 두기구는 eith을 유도하여 유전자 조작을 위해 이용 될 수있다어 임의 삽입과 삭제 돌연변이 (삽입과 삭제, NHEJ을 통해)은 인간의 질병 모델링을위한 환자 돌연변이 요점을 되풀이하거나 유전자 치료를위한 질병을 유발하는 돌연변이를 해결하기 위해, 유전자 대립 유전자, 또는 (HDR 통해) 정확한 염기 치환을 무효화 프레임 이동 돌연변이를 만드는 방법 .
DNA 대상 인식을 지정 DNA 절단 및 가변 CRISPR의 RNA (crRNA) 및 트랜스 활성화 (tracrRNA) 양면 인쇄에 필요한 일정 RNA 유도 Cas9 효소 : CRISPR / 카스 매개 게놈 엔지니어링은 두 가지 구성 요소가 필요합니다. crRNA / tracrRNA 듀플렉스보다 효율적 5, 6, 7 일 밝혀졌다 단일 키메라 가이드 RNA (gRNA)로 대체 될 수있다. CRISPR / Cas9 시스템은 대부분의 유기체 실험 세포주 Cas9 및 gRNA 크게 변동의 전달 및 발현에 적응해야되었지만 상기 아치하도록 최적화 될hPSCs 8 등 많은 시스템에서 이브 효율적인 게놈 편집. 효율적인 게놈 편집 플랫폼, iCRISPR는 hPSCs 5 년에 설립되었습니다. 이 시스템에서, TALEN 중재 접근 방식은 (TRE를 역 테트라 사이클린 제어 transactivator (M2rtTA)과 테트라 사이클린 응답 요소와 다른와 트랜스, 하나의 대립 유전자의 "유전자 안전한 항구의 궤적"AAVS1의 두 대립 유전자를 대상으로 사용되어왔다 )이 hPSCs에서 Cas9 (iCas9의 표현)를 구동. 설립 클론 라인 (iCas9 hPSCs)에서 Cas9은 매우 독시사이클린 처리로 표현된다. 한편, 인해 작은 크기 (100 NT), 하나 또는 여러 개의 gRNAs 쉽게 높은 효율로 iCas9 hPSCs에 전달 될 수 있으며, HDR-매개뿐만 아니라, 효율적인 NHEJ 매개 유전자 중단을 가능하게, 사이트 별 분열에 대한 Cas9을 안내 할 수 있습니다 짧은 단일 가닥 DNA (ssDNA를) 기증자 템플릿의 존재 정확한 염기 수정. iCRISPR 시스템이 될 수 있습니다성공적 9 중요한 발달 유전자 5 biallelic (동형 접합성 또는 이형 화합물) 또는 이형 기능 상실 돌연변이 질병 모방 hPSC 라인의 패널을 생성하는 데 사용된다. 그룹의 개수에 hPSCs CRISPR / CAS를 이용한 효율적인 유전자 편집보고되었지만, 성공은 기술적으로 숙련 실험실 소수의 제한 남아있다. iCRISPR 플랫폼은 상이한 기술 수준의 연구자 루틴 유전자 편집 효율적인하면서도 간단한 해결책을 제공하며, 이는 이미 기 등 9, 10, 11, 12에 의해 출판 다수의 연구에 사용되었다. 이 방법은 또한 상기 dCas9-KRAB (13)의 식에 기초하여 유도 침묵으로 확장되었다.
게놈 편집 테크노의 진행과 함께말의 뜻, 상당한 개선도 hPSC 유지 관리 및 감독 차별화를 달성했다. hPSCs을위한 배양 조건은 조사 된 마우스 배아 섬유 아세포 (IMEF) 공급에 의존하는 정의 된 세포 외 기질 구성 요소에 대한 공급이없는 상태로, 복잡한 미디어 제제에서이 화학적으로하는 매체 조건 (14)로부터 진화했다. 이러한 개선은 IMEF 준비와 녹아웃 혈청 교체 부품의 배치 별 차이로 인해 hPSCs의 변동성을 감소하고, 따라서 hPSC 차별화에 대한 더 많은 재생 가능한 환경을 제공하고있다. 한편, 높은 처리량 약물 스크리닝에서 인간 배아 발달에 적용 경로뿐 아니라 발견 시그널링 향상된 기술이 향상 분화 프로토콜 15, 16, 17, 18을 이끌어왔다. 이러한 프로토콜은 더 밀접하게 비브에서 모방오 발달 단계는 밀접하게 자신의 생체 대응 요점을 되풀이 세포 유형을 생성합니다. 췌장 혈통에 hPSC 차별화를 들어, 초기 프로토콜은 비교적 잘하지만 결국 미성숙 한 태아의 표현형의했다 포도당 자극에 제대로 반응 polyhormonal β 세포를 발생 초기에 췌장 개발을 모방. 최근의 진보 16, 17, 19, 20 등의 형성 및 monohormonal β 세포의 더 성숙 후술 사건을 조사 할 수있게된다 글루코스 응답 췌장 β 형 세포의 생성을 허용하고있다.
여기서는 상세히 글루코스 응답 pancr 향해 hPSC 기반 체외 분화 플랫폼으로 iCRISPR 시스템을 조합함으로써 췌장 발달의 연구 게놈 변성 라인의인가eatic β와 같은 세포. 개선 된 hPSC 차별화 프로토콜과 강력한 게놈 편집 도구의 결합은 질병의 인과 관계 검증에 대한 증가하는 수요를 충족하는 데 필요한 속도와 규모를 제공 할뿐만 아니라 정상적인 개발 및 질병 (9)을 기본 전사 컨트롤에 더 기계적인 조사에 대한 정교한 유전자 조작을 할 수 있습니다뿐만 아니라, .
프로토콜
이 프로토콜은 hPSC 라인 H1, HUES8 및 MEL-1 화학적 및 공급이없는 상태에서 (자재 및 장비의 표를 참조하십시오)와 함께 우리의 작업을 기반으로합니다. 다른 hPSC 라인 또는 다른 배양 조건에서 유지 hPSCs를 들어, 더 최적화하는 것이 좋습니다.
화학적으로 정의하고 공급이없는 상태에서 1 hPSC 문화
- 공급 장치가없는 상태로 IMEF 피더에 hPSC 문화를 적응. 직접 공급이없는 상태로 복구 할 때 냉동 세포가 잘 생존하지 않는 경우, 첫째 IMEF 상태에서 세포를 복구하고 공급이없는 상태에 적응.
참고 : 일반적으로, 피더없는 조건에 적응 할 수 IMEF 피더에 배양 hPSCs 2 구절을합니다. - 매체를 매일 통로 세포 ~ 80 % 포화 상태에 도달 한 hPSCs을 변경합니다. 일반적으로, 1 ~에서 통과 hPSCs : 6 - 1시 15분 비율 모든 4-6일. 10 μM ROCK 저해제 Y-27632 WH 추가해동 또는 세포를 계대 EN.
- 실온에서 적어도 1 시간 (RT) 5 μg의 / ㎖ (1 ㎖ / 10cm 2) 비트로 넥틴 (VTN)의 절단 된 재조합 인간의 형태로 hPSCs, 프리 코트 문화 요리를 파종하기 전에. 또한, 기초 배지로 보충을 추가하여 완전한 화학적 매체를 준비합니다.
- , 배지를 제거 칼슘과 마그네슘 2 +없이 PBS로 한 번 세포를 씻어 2 ~ 0.5 mM의 EDTA로 세포를 치료 - RT에서 5 분.
- 식민지가 분리 한 전에 EDTA를 대기음. 부드럽게 피펫으로 작은 조각으로 hPSC 콜로니를 분산하고 완전 배지에서 세포를 재현 탁.
- 해리 hPSCs를 수집하고 5 분 동안 200 XG에서 세포를 스핀 다운. 완전한 매체에 펠릿을 재현 탁하고 hPSCs VTN - 코팅 된 플레이트에서 세포를 종자.
iCas9 hPSC 라인의 2 세대
- AAVS1-TALEN-L, AAVS1-T : 주문은 다음과 같은 플라스미드를 증폭ALEN-R, AAVS1 - 신 M2rtTA 및 AAVS1 - 순수하지 않아-iCas9.
참고 : 예기치 않은 재조합 이벤트를 피하기 30 ° C에서 플라스미드의 변환 및 증폭을위한 재조합 결핍 Stbl3 유능한 세포를 사용하십시오. - 일반적으로 하나의 대상으로 실험 한 10 cm 접시 (~ 1 × 10 7 세포 ~ 경우 80 % 합류)에 hPSCs을 준비합니다.
주 : 일렉트로 보통 중요한 세포 죽음과 AAVS1 궤적 타겟팅 TALEN 매개 유전자 항생제의 선택을 필요로 발생하기 때문에, 전지가 비교적 다수 올바르게 표적 세포를 식별하기 위해 시드 할 필요가있다. 약물 농도에 대한 최적화는 각 셀 라인과 각각의 배양 조건을 권장합니다. - 일 -1에서 (전기 전날), 미디어 변화 동안 10 μM의 ROCK 억제제를 추가합니다.
- 0 일, (전기의 날)에 사전에 VTN 코팅 된 플레이트를 준비합니다.
- USI 단일 세포로 hPSCs을 떼어 놓다NG 1 배 해리 시약 (참조하시기 바랍니다 재료 및 장비 표). 요약하면, 배양 배지를 제거하고 칼슘 마그네슘 2+없이 PBS로 한번 세포를 세척하고, 2 ~ 3 분 동안 37 ℃에서 1X 해리 시약 세포 치료. 세포를 분리하기 전에 해리 시약 대기음. 부드럽게 피펫으로 완전 배지 중 10.5 mL의 단일 세포 현탁액으로 분산 hPSCs.
- 자동 세포 계수기를 사용하여 세포 수를 계산하는 0.5 ㎖의 세포 현탁액을 가지고. 5 분 200 XG에 hPSCs 펠렛 12.5 × 10 6 세포 / ㎖에서 감기 (4 ° C) PBS에 세포를 재현 탁.
- 800 μL hPSC 현탁액 (12.5 × 106 세포 / mL)에 플라스미드 (표 1 참조)를 첨가하고 잘 혼합한다. 0.4-cm의 전기 큐벳에 혼합물을 전송하고 ~ 5 분 동안 얼음에 보관하십시오.
- 250 V, 500 μF에서 전기 시스템을 이용하여 세포를 Electroporate; 시정 OBS13 밀리 - 전기 후 erved 일반적으로 9입니다.
- 전기 천공 후, 예열 된 완전 배지 5 ㎖과 함께 15 mL의 원뿔형 튜브에 세포를 옮긴다. 사용 건강한 증식 hPSCs 및 전송 재현 탁하고, 전기 후 세포를 도금 할 때 매우 부드럽게 세포를 처리 : 성공적인 타겟팅 중요.
- 5 분 동안 200 XG에서 세포 펠렛. 세 VTN 코팅, 10 cm 요리의 각각에 10 μM의 ROCK 저해제 및 판 1, 2.5, 5 × 10 6 세포 완전 배지 10 ㎖의 세포를 재현 탁; 이 판 중 적어도 하나는 식민지 따기위한 단일 세포 클론 밀도에 충분한 식민지있을 것이라는 점을 보장한다.
- 1 일 (전기 다음날)에, 매체를 변경합니다.
- 일 2 일 - 세포 ~ 60 %의 합류 때 5, 네오 마이신 선택을 시작합니다. 500 μg의 / mL의 G418 황산 매일 매체를 변경; 중요한 세포 사멸 selectio 예정n은 일반적으로 이일 G418 선택 후 관찰된다.
- 주 6 일, 항생제 선택하지 않고 매체를 변경합니다.
- 일 7-9에서 퓨로 마이신 선택을 시작합니다. 1 μg의 / ML의 퓨로 마이신 염산염 매일 매체를 변경; 중요한 세포 사멸은 다음 날 관찰해야한다.
- 직경 2mm - 10 일째에, hPSC 단일 셀의 식민지가 1에 도달 할 때까지 항생제 선택하지 않고 매일 매체를 변경 시작합니다.
참고 : 일반적으로 10 cm 접시에 50 식민지가 2.5 × 10 6 hPSCs와 관찰이 날 0에 도금. - 실체 현미경 하에서 24 식민지 - (12)을 선택합니다. 기계적으로 작은 조각으로 hPSC 식민지를 해리하는 (~ 식민지 당 10 개) 23-G 바늘을 사용하여가 (200 μL 피펫 팁도 괜찮)과 VTN 코팅 된 24 웰 플레이트에 직접 세포를 전송합니다.
- 세포가 합류 될 때까지 매일 매체를 변경합니다. 통로 24 웰 플레이트의 각 웰에 세포 내로6 웰 플레이트의 우물을 복제.
- 세포가 6 웰 플레이트에 합류 될 때, 냉동 재고 및 추가 특성화 게놈 DNA 추출을위한 아니라, 다른 하나 잘 사용합니다.
- 특성화 및 PCR의 유전자형, 남부 블로 팅, RT-qPCR에 분석, 염색체, 그리고 만능 분석에 의해 설립 iCas9 라인을 확인합니다. 주홍 등의 알을 참조하십시오. 자세한 실험 절차 21.
iCRISPR 시스템을 사용하여 hPSC 돌연변이 라인의 3 세대
- hPSC 녹아웃 라인의 세대
- gRNA 설계 및 생산
- 야생형 단백질의 기능을 방해 할 가능성을 최대화 할 목적 유전자의 타겟 영역을 선택한다. 잘 주석 유전자를 들어, 필수적인 기능 도메인의 상류 대상 영역을 선택합니다. 또한, 디자인 gRNAs는 개시 코돈의 하류 지역을 대상으로한다. 적어도 2 개의 다른 선택관심의 유전자 영역.
- 온라인 CRISPR 설계 도구 (http://crispr.mit.edu)를 사용하여 디자인 gRNAs. 각 대상 영역에 대해 낮은 전위 표적 클론 돌연변이 오프 라인 (22)을 생성하기위한 가장 효율적으로 타겟팅을 사용하여 디자인 3 gRNAs.
주 : 하이 게놈 편집 효율을 달성하기 위하여, RNA 올리고으로 대신하기 때문에 과거의 경험에서 플라스미드에 비하여 소형의 RNA의 높은 형질 전환 효율이 플라스미드 DNA로 gRNA을 제공하도록 추천된다. - T7 프로모터 서열을 함유하는 (NT) DNA 올리고는 가변 20 NT crRNA 인식 서열 (N) 20 (PAM에 서열을 포함하지 않음) 120 뉴클레오티드 주문하고 일정한 키메라 가이드 서열. DDH 2 O에 100 μM 원액에 올리고 희석 및 솔루션 작업으로 250 나노를 준비합니다.
참고 : TAATACGACTCACTATAGGG (N) (20) GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTT - T7F TracrR 및 프라이머를 사용하여 PCR을 올리고이-증폭 gRNA 시험 관내 전사 (IVT)에 대한 이중 가닥 DNA (dsDNA) 템플릿을 생성하기 (표 2 참조). PCR 반응 혼합물 (표 3 참조), PCR 사이클 조건의 50 μL를 사용하여 (표 4 참조).
- 20 μL의 PCR 증폭 템플릿으로 시험 관내 gRNA 전사에 대한 높은 수율 T7 전사 키트를 사용, 제조업체의 지침에 따라 체외 gRNA 전사 믹스 (표 5 참조). 제조업체의 지침에 따라 전사 청소 키트를 사용하여 gRNA 제품을 정화.
- 용출 버퍼 100 μL에 - (100 μg의 전형적 ~ 50) 용출 gRNAs는 제조업체의 지침에 따라, 높은 처리량 정화 프로토콜을 다음과 같습니다. -80에서 320 NG / μL (10 μM) 가능하면 저장에 농도를 조정# 176; 사용할 때까지 C.
- PCR과 생어 시퀀싱 프라이머 디자인
- 디자인은 제품의 크기는 일반적으로 범위에 타겟 영역을 PCR 증폭 프라이머를 검증 ~ 500 - 1000 BP.
- PCR 산물에 대해 내부적으로 결합 디자인 생거 시퀀싱 프라이머 정제없이 PCR 산물을 직접 서열 분석을 허용한다.
- iCas9 hPSCs에서 gRNA 형질 전환
- 일 -1에서 24 시간 gRNA 형질 전환 전에 2 μg의 / ML의 독시 싸이클린과 iCas9 세포를 취급합니다.
- gRNA 형질의 날 0에서 미리 VTN 코팅 된 플레이트를 준비합니다.
- 단계 2.5에 기재된 바와 같이, 1X 해리 시약을 사용하여 단일 세포로 iCas9 세포 해리.
- 5 분 200 XG에 hPSCs 펠렛과에서 세포를 재현 탁 ~ 0.5 × 10 6 세포 / mL의 2 μg의 / ML의 독시 싸이클린 10 μM의 ROCK 저해제로 보충 완전 배지입니다.
- 24 웰 플레이트의 각 웰에 재현 탁 세포의 플레이트 0.5 mL를. 비 형질 전환 된 컨트롤을 제공하기 위해 추가 우물을 준비합니다.
- 각 gRNA 들어, 형질 감염 혼합물을 : 감소 된 혈청 배지를 50 μL 믹스 gRNA + 1 (10 μM)의 μL; 믹스 B, μL 형질 전환 시약의 환원 혈청 배지 + 3 50 μL.
- 섞어을 결합하고 B는 100 μL 혼합물을 만들기 위해. RT에서 5 분 동안 인큐베이션. 24 웰 플레이트의 중복 우물에 세포 혼합물의 50 μL를 넣고 잘 섞는다.
- 상기 타겟팅 효율을 증가시키기 위해 필요하다면 제 1 일에서 제 형질 전환을 수행한다. 그렇지 않으면, 독시사이클린없이 매체를 변경합니다.
- 일 2 - 3, 매일 매체를 변경합니다.
- 4 일째에, DNA 추출 키트를 사용하여 각 형질 전환 및 비 형질 제어 셀의 한 웰로부터 게놈 DNA를 추출한다. 50 NG / μ로 농도를 조절합니다엘.
- gRNA 표적 서열을 플 랭킹 타겟 영역을 PCR은-증폭 이전 5 바와 같이 T7 엔도 뉴 클레아 제 I (T7EI) 분해 또는 제한 단편 길이 다형성 (RFLP) 분석을 이용하여 편집 효율을 추정한다.
- T7EI 분석
- 설계 단계 3.1에서 검증 된 프라이머를 사용하여 대상 영역을 PCR은-증폭.
- 혼합물 (표 6 참조)를 준비하고, 표 7에 설명 된 조건을 사용하여 PCR 산물의 DNA 변성 및 혼성화를 수행한다.
주 : 우리의 경험을 바탕으로, 우리 PCR 조건을 사용하는 경우 T7EI 분석을 위해 PCR 산물을 정제하기 위해, 일반적으로 필요하지 않다. 그러나 정제는 다른 조건에서 도움이 될 수 있습니다. - 변성의 10 μL를 사용하여 30 분 동안 37 ° C에서 T7EI 소화을 수행하고 PCR 제품 및 T7E1 0.2 μL (2 U) (10 U / μL)를 하이브리드.
- 알아낼겔 전기 영동에 의해 T7E1 소화 PCR 샘플을 전자. 잘라 내기 및 포경 DNA의 상대 밴드 강도를 결정하기 위해 ImageJ에 사용합니다. 수식을 이용하여 INDEL 주파수를 계산한다 ((1) - (+ √ (1- (b + c)) / (A + B + C))) (A)가 소화되지 않은 PCR 생성물되고, B와 C의 강도 × 100, T7E1 절단 된 제품의 강도.
- RFLP 분석
주 : 제한 부위가 근접하게되는 경우에 (<5 BP)를 Cas9 분열 사이트 (PAM에 서열 3 bp의 5 ')하기 위해, RFLP 분석이 INDEL 주파수를 정량화하기 위해 수행 될 수있다.- 단계 3.1.4.1에서 설명한 것과 같은 PCR 제품을 사용한다.
- Cas9 절단 위치 부근에서 제한 부위를 포함하는 제한 효소로 PCR 생성물을 소화.
- 겔 전기 영동에 의해 분해 된 PCR 샘플을 해결. 잘라 내기 및 포경 DNA의 상대 밴드 강도를 결정하기 위해 ImageJ에 사용합니다. INDEL을 계산화학식 사용 주파수 : A / (A + B + C)는 (A)가 소화되지 않은 PCR 생성물 및 (B)와 (C)의 강도를 분해 된 생성물의 강도는 여기서 × 100.
- 클론 돌연변이 라인 구축
참고 : gRNA 형질을 가진 iCRISPR 시스템을 사용 hPSCs의 게놈 편집이 매우 효율적이며, 항생제 선택이 필요하지 않습니다. 클론 라인을 확립하기 위해, 단일 세포 유래 콜로니의 형성을 보장하기 위해 상대적으로 낮은 농도로 세포를 배정 할 필요가있다.- 합니다 (T7EI 또는 RFLP 분석을 사용하여) 가장 높은 편집 효율이 좋은 세포 생존과 gRNAs을 확인합니다. 클론 돌연변이 라인 구축을 위해 잘 대응하는 중복을 사용합니다.
- 단계 2.5에 기재된 바와 같이, 1X 해리 시약을 사용하여 단일 세포 현탁액 내로 해리를 hPSCs. 세 VTN 코팅, 10 cm 요리의 각각에 500, 1,000, 2,000 세포를 Replate.
- 매체 매일 U 변경단일 셀 ntil 콜로니 지름 ~ 2mm 도달한다.
- T7EI 및 / 또는 RFLP 분석에 의해 효율성을 대상의 추정에 따라, 각 gRNA 48 식민지 - (24)을 선택합니다. 기계적으로 작은 조각으로 각 식민지를 해리하는 (~ 식민지 당 10 개) 23-G 바늘을 사용하여가 (200 μL 피펫 팁도 괜찮습니다)와 중복 VTN 코팅, 96 웰 플레이트에 세포를 replate. 게놈 DNA 추출 및 생거 시퀀싱 및 추가 확장에 대한 다른 플레이트에 대해 하나의 플레이트를 사용합니다.
- 96 웰 플레이트의 세포가 합류하게되면 아래에 설명 된대로, 간단한 프로토콜을 사용하여 (페놀 / 클로로포름 추출 제외) 게놈 DNA를 추출합니다.
- 매체를 제거하고 칼슘과 마그네슘 2 +없이 PBS로 한 번 세포를 씻으십시오. 96 웰 플레이트의 각 웰에 용해 완충액 50 μL (5 μL의 테 K (10 ㎎ / ㎖), 5 μL PCR 완충액의 10 배 및 DDH 2 O 40 μL)을 추가한다. 접착제 필름 및 incuba를 사용하여 플레이트를 밀봉테 하룻밤에 55 ° C.
- 다음 날, 테 K.를 실활 열 순환기에서 99 ° C에서 10 분 동안 96 웰 PCR 플레이트에 세포 용 해물을 전송하고 부화
- 주형으로서 세포 용 해물 1 μL를 사용 T7EI 또는 RFLP 분석과 동일한 프라이머를 사용하여 타겟 영역을 PCR 증폭은 -.
- PCR 산물에 내부적으로 결합 프라이머와 생거 시퀀싱을위한 PCR 제품의 1 μL를 사용합니다.
- 냉동 주식에 대한 프레임 이동 INDEL 변이와 클론을 증폭. 또한, 추가 실험으로 동종 제어 라인을 제공하기 위해 동일한 타겟팅 실험에서 야생형 클론의 몇 가지를 증폭.
- gRNA 설계 및 생산
- 정확한 염기 변화와 돌연변이 라인의 세대
주 : (NHEJ)를 결합 비 상동 단부 생성 녹아웃 돌연변이와 비교는 정확한 뉴클레오티드 변화는 DNA의 repai의 존재 상동 관한 수리 (HDR)를 통해 달성 될 수있다R 템플릿. 이러한 정확한 염기 변형은 야생형 hPSCs에서 환자 - 특정 돌연변이 생성을 위해 환자 유래 iPSCs 변이의 보정을 허용한다.- HDR 서식 파일로 ssDNA를 디자인
- 단계 3.1.1에 기재된 바와 같이, 환자 특이 적 돌연변이에 근접 3 gRNAs - 디자인 및 2를 생성한다.
- HDR 템플릿 등의 각 측면에 동성 80 NT - ~ (40)에 의해 측면 환자의 특정 변이를 포함하는 단일 가닥 DNA (ssDNA를)을 디자인합니다.
- 가능하면 정확한 복구 후에 추가 절단을 줄이기 위해, 자체 gRNA 인식 서열 내에 상기 PAM 시퀀스에 근접 또는 PAM 시퀀스 영역에서 ssDNA를 템플릿 침묵 돌연변이를 도입.
- 가능한 경우뿐만 아니라 RFLP 분석법을 사용하여 보수 효율을 추정하기 위해 사용될 수 있도록하는 신규 한 제한 효소 부위를 도입하기 위해 침묵 돌연변이를 설계한다.
- gRNA / ssDNA를 공동 형질 전환 및 클론 라인의 설립
- gRNA 비 형질 제어 각각에 대해 형질 전환 혼합물 A 및 B에, 단계 3.1.3에서 설명 된 바와 같이, iCas9 세포 내로 gRNA / ssDNA를의 공동 트랜 스펙 션을 수행하는 24 웰 플레이트의 중복 웰에서 세포를 형질 감염. 감소 된 혈청 배지 + 1 μL gRNA의 (10 μM) + 2 μL ssDNA를의 (10 μM)의 50 μL하십시오를 섞는다. B 믹스 : μL 형질 전환 시약의 감소 혈청 배지 + 3의 50 μL를.
- 형질 전환 후, 각각의 형질 전환 및 비 형질 전환 제어 셀의 한 우물에서 게놈 DNA를 추출하여 T7EI을 사용하여 수리 효율 및 / 또는 RFLP 분석을 추정한다.
- 가장 높은 보수의 효율성과 좋은 세포 생존과 gRNA / ssDNA를 혼합물을 확인합니다. 클론 돌연변이 라인 구축을 위해 잘 대응하는 중복을 사용합니다.
- T7EI 및 / 또는 RFLP 엉덩이로 타겟팅 효율성의 추정에 따라, 96 식민지 - 48 선택찬성. 일반적으로, HDR 매개 돌연변이 효율이 녹아웃 돌연변이 미만인하여보다 콜로니 촬상 할 필요가있다.
- 단계 3.1.6에 설명 된대로 순서, 확장 한 클론 라인을 확인합니다.
- HDR 서식 파일로 ssDNA를 디자인
포도당 응답 췌장 β 세포로 체외 hPSC 차별화 4.
참고 : 질병 관련 세포 유형으로 hPSC 돌연변이의 체외 분화가 접시에 질병 모델링을위한 플랫폼을 제공합니다. 다음 프로토콜은 췌장 발달과 당뇨병 연구 9, 16, 17 포도당 응답 췌장 β 세포로 hPSCs의 체외 분화에 초점을 맞추고 있습니다.
- 최종 내배엽으로 hPSC 차별화
- 설명하는 바와 같이, 화학적 및 피더가없는 상태 hPSC 돌연변이 및 야생형 제어 라인 유지1 단계 라.
- 분화에 대한 hPSCs을 제조 1X 해리 시약을 사용 hPSCs를 해리 완전 배지에서 단일 세포 현탁액을 분산.
- 5 분 동안 200 XG에서 세포 펠렛 및 10 μM의 ROCK 저해제와 완전 배지에서 세포를 다시 일시 중지합니다. 세포 수를 카운트하고있는 세포를 시드 ~ VTN - 코팅 된 플레이트에 1.4 × 105 세포 / ㎠.
- 파종 후 매체 24 시간을 변경합니다.
- 0 일에, 세포 ~ 80 % 포화 상태에 도달 한 경우, 48 시간 후 분화를 시작합니다.
참고 : 높은 췌장 분화 효율을 달성 48 시간에서 포화 상태의 파종 밀도와 수준을 최적화하려면 각각의 라인을 권장합니다. - hPSC 매체를 기음과 칼슘 및 마그네슘 2+없이 한 번 PBS로 세포를 씻어.
- 차별화 날 0 (D0) 매체에 매체를 변경합니다.
- 일 1 일 - 2 디 변경표 8의 조리법에 따라, 매체 매일 fferentiation.
- 3 일에, 면역 형광 염색법에 의해 최종 내배엽 마커 SOX17, FOXA2 및 CXCR4를 조사 및 분석 유동 세포 계측법.
- 췌장 전구에 확실한 내배엽 분화
- 일 3 일 - 9, 표 9의 조리법에 따라, 매일 매체를 변경하여 췌장의 혈통을 향한 최종 내배엽 분화를 계속합니다.
- 주 7 일, 면역 형광 염색법에 의한 초기 췌장 전구 (PP1) 마커 PDX1을 조사 및 분석 유동 세포 계측법.
- 10 일째에, 나중에 췌장 전구 (PP2) 마커 PDX1 및 NKX6.1을 확인합니다. 한편, 췌장 내분비 세포로 더욱 분화 공기 - 액체 계면에 PP2 세포를 전송할 준비한다.
- 췌장 내분비 다를공기 - 액체 인터페이스 entiation
- 4 시간 분리하기 전에 10 μM의 ROCK 저해제와 PP2 세포 치료.
- 매체를 제거하고 칼슘과 마그네슘 2+없이 한 번 PBS로 세포를 씻어.
- 한 10 cm 접시에 PP2 셀에 1 배 해리 시약 2 mL를 넣고 2 37 ° C에서 부화 - 3 분.
- 세포를 분리하기 전에 해리 시약 대기음. BLAR 매체의 10 ㎖를 추가하고 부드럽게 피펫 팅 아래에 의해 하나의 세포에 PP2 세포를 분산.
- 단일 세포 현탁액을 수집한다. 5 분 동안 200 XG에서 세포 수 및 펠릿 카운트.
- ~ 0.5 × 10 5 세포 / μL에서 S5 분화 배지에서 세포 펠렛을 재현 탁하고 5 자리 - 트랜스 웰 삽입 필터에 자리 당 세포의 10 μL를. 10 cm 삽입 한 6 잘 삽입 15 반점 ~ 100 점 - (10)를 놓습니다.
- ~ 1.5 6 웰 인서트 ~ 8 mL의 용액 F 각 트랜스 웰 인서트의 바닥 S5 매체 추가또는 10 cm 삽입.
- 표 10의 조리법 매일 매체를 변경합니다.
- 면역 형광 염색법에 의해 하루 34 췌장 내분비 마커 PDX1, NKX6.1 NEUROD1, NKX2.2, 인슐린 및 글루카곤을 검토 및 분석 (17) 유동 세포 계측법.
- 포도당 자극 인슐린 분비 (GSIS) 분석 16, 17 hPSC 파생 β와 같은 세포의 기능을 검사합니다.
결과
hPSC 화학적 정의 및 피더없는 문화 적응 및 유지 관리
IMEF 모이 배양 hPSCs 신속 피더가없는 배양 조건 VTN - 코팅 된 플레이트로 구성 될 수있다. 통상 피더 IMEF 배양과 같은 분할 비 적응 기간 동안 사용될 수있다. 도 1a는 KSR 기반 매체와 공급이없는 매체에서 VTN 코팅 표면에 IMEF 피더에 hPSCs의 전형적인 형태학을 보여줍니다. 도 1b는 전형적인 형태 변화 및 적응 (4 일)의 제 1 통로 중 hPSC 콜로니의 성장을 나타낸다. 세포는 더 계대 또는 미래의 실험에 고정 될 수 있습니다. 분화 실험을 시작하기 전에 적응 문화의 3 통로 - 2를 수행합니다. 우리는 적응 단계에서 핵형 이상을 관찰하지 않은 있지만, 핵형 또한, 적응 후 좋습니다.
"fo를하기 : 유지-together.within 페이지를 ="1 ">를 통해 iCas9 hPSC 라인의 세대 TALEN 매개 AAVS1 타겟팅이전에 나타낸 바와 같이,이 hPSCs AAVS1 TALEN 플라스미드 및 플라스미드 M2rtTA Cas9 및 (5)의 한 쌍의 전기 천공 하였다. 도 2A 및 B는 디자인 iCas9 hPSC 클론 라인을 생성하는 전체 과정을 상세 타겟팅 도너 벡터를 나타낸다. (- 그림 2C, 십이일 전기 후 10) 항생제 선택 후, 적절한 크기와 일반 hPSC 형태를 표시하는 단일 세포 유래 클론 뽑 준비가되어 있었다. 남부 (그림 2D) 5 블로 팅에 의해 확인으로 보통 ~ 클론의 50 %가 제대로 임의의 통합없이 대상이된다. 랜덤 통합 독시사이클린 치료 부재시 Cas9 누설의 발현을 야기 할 수있다.
효율적인 유전 MiCRISPR 플랫폼을 사용 hPSCs에서 odification
설립 iCas9 hPSCs에서 Cas9는 독시사이클린 처리로 표현하고 DSBs을 생성하는 형질 gRNAs에 의해 목표 궤적 안내. 수리 템플릿이없는 경우, NHEJ 통해 DNA 수리 종종 유전자 붕괴 또는 넉 아웃 될 삽입과 삭제를 발생시킨다. 수리 템플릿 (예 ssDNA를 공여체)의 존재하에 HDR 그러한 야생형 hPSC 배경에서 환자 - 특정 돌연변이 생성 또는 론 환자의 질병 관련 유전자 변이를 보정하는 등의 정확한 유전 적 변형을 위해 이용할 수있다 파생 된 iPSCs (그림 3A). 그것은 iCRISPR 시스템을 이용한 클론 돌연변이 라인을 생성하는 1개월 ~ 걸린다. iCas9 유도 및 gRNA 형질 후에 T7EI 및 / 또는 RFLP 분석법 Cas9 절단 효율을 평가하는데 사용하고, 형질 감염된 세포는 나중에 ~ 저밀도 (10-cm 접시에 단셀 500 시딩 - 2 000세포 / 10 cm 요리). 10-12일 후 단일 세포 유래 클론 확장 및 상기 특성 (즉, 유전자형 및 웨스턴 블롯) (도 3B)에 대한 96 웰 플레이트의 웰에 넣었다. iCRISPR 매개 유전자 녹아웃 용이성 때문에, 어떤 선택 처리가 포함되지 않으며, 일반적 20-50% biallelic 돌연변이 쉽게 (도 3C) (5)를 얻을 수있다. 효율적이고 정확한 유전자 변형 들어 gRNAs은 (게놈 서열 작지만 특정 변경에 대한) 특정 순서 변경을 나르는 ssDNA를 공여체와 공동 - 형질 감염된다. 종종, 수정 대립 유전자에 다시 절단을 방지하기 위해,에 또는 PAM 순서 (그림 3D)에 근접 자동 돌연변이를 포함하는 것이 좋습니다. 이 시스템으로, ~ 두 대립 유전자의 추가 변경없이 원하는 HDR-매개 게놈 수정을 들고 클론의 10 % (도 3E) 달성된다. 급속 및 p-iCRISPR 플랫폼을 사용하여 hPSC 게놈의 recise 수정은 우리가 신속하고 효율적으로 인간 발달 및 질병 연구를위한 모델 역할 hPSC 라인을 만들 수 있습니다.
포도당 응답 β와 같은 세포를 향해 hPSCs의 효율적인 분화
hPSC 췌장 분화 최근 진행 밀접 췌장 배아 개발 요점을 되풀이 프로토콜의 개발을 허용했다. 미분화 hPSCs는 먼저 PDX1 + 초기 췌장 전구 세포 (PP1) 및 PDX1 + NKX6.1 + 나중에 췌장 전구 세포 (PP2)로, 최종 내배엽으로, 그리고 마지막으로, 20 포도당 응답 β와 같은 세포 16, 17, 19로 분화 , 23, 24. 이 protocOLS 더 한층 확실 PDX1 NKX6.1 + + 췌장 전구 세포와 글루코오스 반응 β 형 셀 (9)를 생성하기 위해 최적화되었다. 도 4a는 분화의 각 단계에서 사용되는 상세한 화학 첨가제를 보여줍니다. 일반적으로 2 일 초기 셀 도금 후 ~ 80 % 전면 생장을 HUES8 hPSCs (도 4b)의 분화를 시작하기에 적합하다. 여러 파종 밀도를 테스트하는 것은 매우 각 특정 세포주에 대한 최적 조건을 발견하도록 권장한다. 통상적으로 75 % 이상 FOXA2 +를 PP2 단계에서 DE 단계 40 % PDX1 NKX6.1 + + + 세포에서 세포를 얻을 수있다 SOX17 (도 4C). S5 단계에서, 셀 집합체 주위 분위기 형상의 존재는 NKX6.1 + CPEP + 글루코스 응답 β 유사 세포 (도 4D에있어서 분화에 중요한 세포의 우수한 생존율에 대한 지표이다 ). 이 프로토콜은 인간의 췌장 d를 공부하는 데 유용합니다 접시에 evelopment과 질병.

그림 1. hPSC가 화학적으로 정의하고 공급이없는 유지 관리 및 IMEF 공급기에서 문화 적응. (A) hPSCs의 대표 이미지는, 4 일에 VTN IMEF 공급 장치 나에 분할 할 준비가 배양. (B)는 IMEF 공급 장치에서 공급이없는 문화에 대한 적응의 제 1 통로 동안 hPSCs의 전형적인 형태. IMEF 피더에서 배양 4 일 hPSCs는 ROCK 저해제와 화학적 매체에 VTN 코팅 된 플레이트에 분할 도금했다. 분할 비율은 IMEF 공급 조건에서 배양을위한 통상의 분할 비율과 동일하다. 매체는 ROCK 저해제없이 매일 변경되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
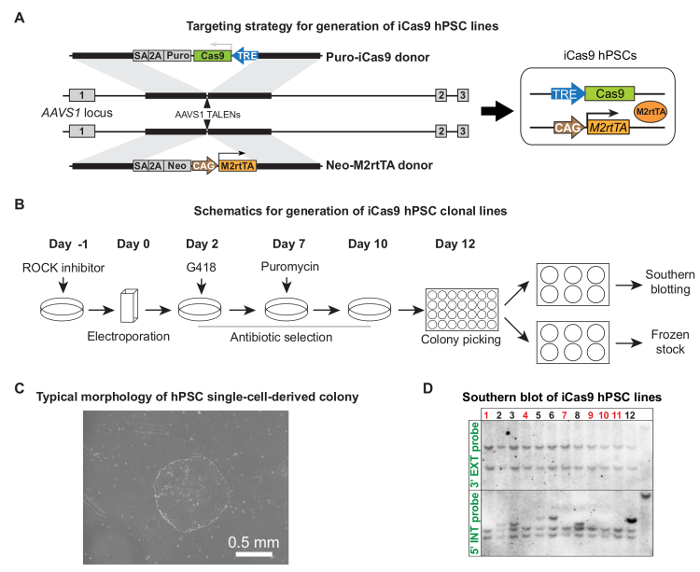
iCas9 hPSC 라인의 그림 2 세대. (A) iCas9 hPSC 라인의 생성을위한 전략을 타겟팅. 순수하지 않아-Cas9 공여체 및 네오 M2rtTA 공여체 AAVS1 TALENs 한 쌍의 인간 AAVS1 궤적로 타겟팅 하였다. 전기, 항생제 선택 콜로니 피킹 및 확장 및 세포주 특성 포함한 전체 타겟팅 프로세스의 (B) 회로도. 전기가 12 일 - (C) 대표 단일 세포 클론 준비가 약 10에서 포착 할 수 있습니다. (D)이 기증자 플라스미드의 추가 통합하지 않고 올바르게 대상으로 클론을 식별하기위한 남부 오 예. 올바른 클론은 빨간색으로 표시됩니다. (A)와 (D)는 허가, 참조 5에서 적응되었다.ig2large.jpg "대상 ="_ 빈 ">이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
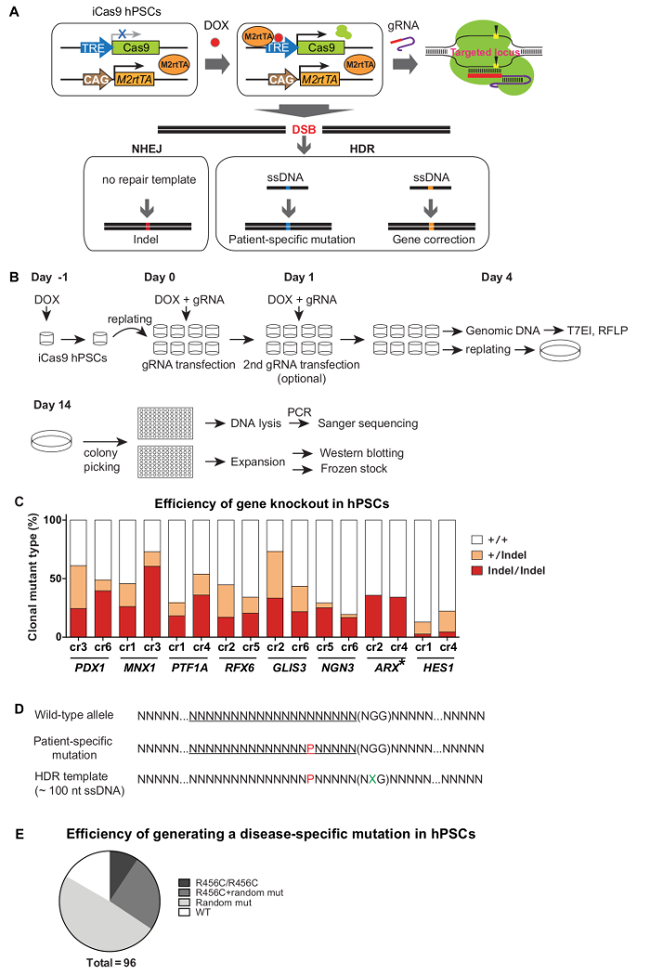
iCRISPR 시스템을 사용 hPSCs 그림 3. 효율적인 유전자 변형. 유전자 녹아웃 돌연변이 또는 iCRISPR 시스템을 이용하여 정밀한 유전자 변형의 발생에 대한 (A) 회로도. (B) 타겟팅 절차 및 클론 라인 설립. hPSCs 9 NHEJ을 통해 (C) 유전자 녹아웃 효율. 특정 염기 변형을 나르는 ssDNA를의 (D) 디자인. P, 빨간색 : 환자 맞춤형 돌연변이; 녹색 X : 침묵 돌연변이. (E)를 매개 HDR 정확한 염기 변형 효율. GATA6 궤적에서 (특정 염기 C> T 돌연변이로 인한)는 정확한 R456C 돌연변이는 iCRISPR 시스템을 사용하여 도입 하였다.(자세한 내용은 5 참조 참조). (C)와 (E)는 권한을 각각 참조 9 참조 5에서 적응되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
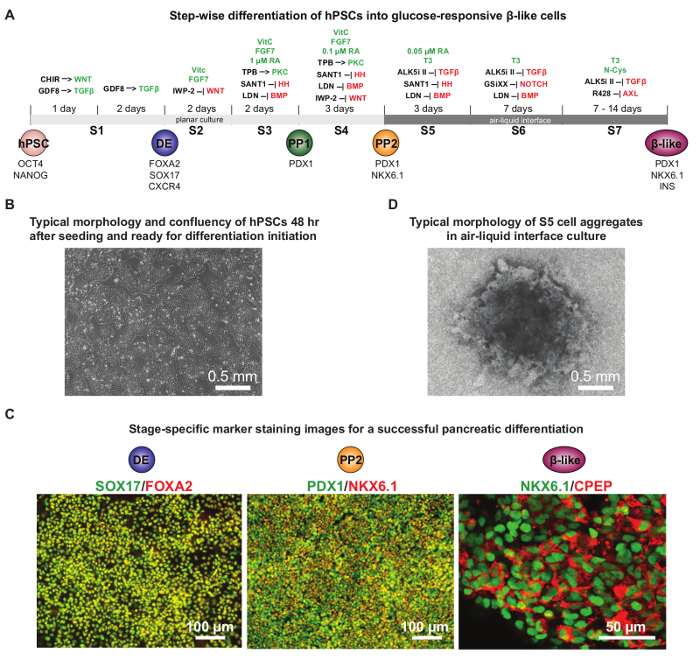
포도당 응답 췌장 β와 같은 세포로 그림 4. 감독 hPSC 차별화. 분화의 각 단계에서 보충 된 화학 물질 상세한 분화 프로토콜 (A) 회로도. 차별화하는 동안 활성화 또는 억제하는 신호 전달 경로는 각각 녹색 또는 빨간색으로 강조 표시됩니다. CHIR : GSK3 억제제; GDF8 : 성장 분화 인자 8 미오 스타틴하는 TGF 베타 단백질 가족; HH : 고슴도치; DE : 확실한 내배엽; PP1 :PDX1 + 초기 췌장 전구; PP2 : PDX1 + NKX6.1 + 나중에 췌장 전구. (B) 일반 컨 플루 - hPSCs 48 (70 ~ 80 %) 시간 준비가되면 차별화 개시를위한 파종 후. (C) 최종 내배엽에서 성공적으로 차별화를 보여주는 대표적인 면역 염색 이미지 (DE) 단계 (FOXA2 및 SOX17 공동 염색), 췌장 전구 단계 (PP2 : PDX1 및 NKX6.1 공동 염색), 포도당 응답 β- 세포 단계 (NKX6.1와 C 펩타이드 공동 염색) 등을들 수있다. 일반적으로, 상기 β 형 세포 단계에서 10 % 이상 NKX6.1 + CPEP + 세포를 달성 적어도 75 % FOXA2 + SOX17 + DE 세포 및 40 % PDX1 + NKX6.1 + PP2 셀은 해당 단계에서 요구한다. (D) 공기 - 액체 계면 배양 인서트 여과막의 S5 단계에서 세포 집합체의 전형적인 형태. 살아남은 세포 (어둠) 집합체의 중심으로 마이그레이션 및 에지 분위기 형 구조를 떠났다. (A )와 (D)는 권한 참조 9에서 적응되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 플라스미드 | 양 |
| AAVS1-TALEN-L | 5 μg의 |
| AAVS1-TALEN-R | 5 μg의 |
| AAVS1 - 순수하지 않아-iCas9 | 40 μg의 |
| AAVS1 - 네오 M2rtTA | 40 μg의 |
1 번 테이블
| 뇌관 | 순서 |
| T7F | TAATACGACTCACTATAGGG |
| TracrR | AAAAGCACCGACTCGGTGCC |
표 2
| 구성 요소 | 양 |
| DDH 2 O | 35.5 μL |
| 배 PCR 반응 완충액 | 10 μL |
| 의 dNTP 믹스 (25 mM)을 | 0.5 μL |
| T7F (10 μM) | 1.25 μL |
| TracrR (10 μM) | 1.25 μL |
| T7-gRNA IVT 템플릿 (250 ㎚) | 1 μL |
| DNA 중합 효소 | 0.5 μL |
| 전체 PCR 반응 믹스 | 50 μL |
표 3
| PCR 사이클링 조건 | |||
| 사이클 수 | 변성하다 | 어닐링 | 넓히다 |
| 1 | 94 ºC 2 분 | ||
| 2-31 | 94 ºC, 20 초 | 60 ºC, 20 초 | 72 ºC, 1 분 |
| (32) | 72 ºC 2 분 | ||
표 4
| 구성 요소 | 금액 |
| T7 ATP | 2 μL |
| T7의 CTP | 2 μL |
| T7 GTP | 2 μL |
| T7의 UTP | 2 μL |
| T7 10 배 버퍼 | 2 μL |
| T7 효소 믹스 | 2 μL |
| PCR 증폭 템플릿 | 8 μL |
| 총 체외 gRNA 전사 믹스 | 20 μL |
| 하룻밤에 6 시간 동안 37 ºC에서 품어 |
표 5
| 구성 요소 | 금액 (μL) |
| 정제되지 않은 PCR 제품 | 8 |
| 버퍼 2 배 | 이 |
| 증류수 (DH 2 O) | (10) |
표 6
| DNA 변성과 하이브리드 자전거 조건 | ||
| 온도 | 지속 | 열 순환기 조건 |
| 95 ° C | 10 분 | |
| 85 ° C | 1 분 | 2 ° C / s로 85 ° C에 램프 |
| 75 ° C | 1 분 | 0.3 ° C / s로 75 ° C에 램프 |
| 65 ° C | 1 분 | 0.3 ° C / s로 65 ° C에 램프 |
| 55 ° C | 1 분 | 0.3 ° C / s로 55 ° C에 램프 |
| 45 ° C | 1 분 | 0.3 ° C / s로 45 ° C에 램프 |
| 35 ° C | 1 분 | 0.3 ° C / s로 35 ° C에 램프 |
| 25 ° C | 1 분 | 0.3 ° C / s로 25 ° C에 램프 |
| 4 ° C | 보류 | |
표 7
| 단계 | 일 | 미디어 | 보충 | |
| S1 | D0 | S1 | GDF8 100 NG / mL로 | CHIR-99021 3 μM |
| (D1) | S1 | GDF8 100 NG / mL로 | CHIR-99021 0.3 μM | |
| (D2) | S1 | GDF8 100 NG / mL로 | ||
| S1 미디어 : MCDB 131 + 1 배 L 글루타민 보충 + 0.5 % BSA + 1.5 g / L의 NaHCO3 + 10 mM의 포도당 | ||||
표 8
| 단계 | 일 | 미디어 | 보충 | |||||||
| S2 | D3-D4 | S1 | LAA 0.25 mM의 | FGF7 50 NG / mL로 | IWP-2 2.5 μM | |||||
| S3 | D5-D6 | S3 | LAA 0.25 mM의 | FGF7 50 NG / mL로 | SANT-1 0.25 μM | RA 1 μM | LDN 100 nm의 | TPB 200 nm의 | ITS-X 1 : 200 | |
| S4 | D7-D9 | S3 | LAA 0.25 mM의 | FGF7 2 NG / mL로 | SANT-1 0.25 μM | RA 0.1 μM | LDN 200 nm의 | TPB 100 nm의 | ITS-X 1 : 200 | IWP-2 2.5 μM |
| S3 미디어 : MCDB 131 + 1 배 L 글루타민 보충 + 2 % BSA + 2.5 g / L의 NaHCO3 + 10 mM의 포도당 | ||||||||||
표 9
| 단계 | 일 | 미디어 | 보충 | |||||||
| S5 | D10-D12 | S5 | T3 1 μM | ALK5i II 10 μM | SANT-1 0.25 μM | RA는 0.05 μM | LDN 100 nm의 | ITS-X 1 : 200 | 의 ZnSO4 10 μM | 헤파린 10 μg의 / mL의 |
| S6 | d13- D19 | S5 | T3 1 μM | ALK5i II 10 μM | GSiXX 100 nm의 | LDN 100 nm의 | ITS-X 1 : 200 | 의 ZnSO4 10 μM | 헤파린 10 μg의 / mL의 | |
| S7 | d20- D33 | S5 | T3 1 μM | ALK5i II 10 μM | N-시스테인를 1mM | 트롤 록스 10 μM | R428 2 μM | ITS-X 1 : 200 | 의 ZnSO4 10 μM | 헤파린 10 μg의 / mL의 |
| S5 미디어 : BLAR + 1 배 L 글루타민 보충 + 2 % BSA + 1.5 g / L의 NaHCO3 + 20 mM의 포도당 | ||||||||||
표 10
토론
생성 돌연변이 라인을위한 시간 고려 사항
게놈 편집 CRISPR / 카스 시스템을 기반으로 최근의 접근 방식이 성공적으로 타겟팅 주도하고 있지만,보다 효과적인 보편적 인 플랫폼은 유전자 기능 분석 대규모 바람직 할 것이다. iCRISPR 플랫폼은 관심 5, 9의 유전자에 돌연변이를 소개하는 신속하고 효율적인 방법을 제공합니다. 우선, PCR 기반 gRNA 합성법은 시간 소모적 클로닝 단계없이 하루에 배열 형식 gRNAs 수백의 생산을 허용한다. 둘째, iCas9 hPSCs에서 독시사이클린 - 유도 Cas9 식으로 gRNA 형질 관련된 공정 작업의 최소량을 필요로하며, 따라서, 여러 gRNA 타겟팅 실험을 동시에 수행 될 수있다. 우리의 시스템의 분석을 달성 셋째, 높은 타겟팅으로 인해 효율성 ~ 24 - 형질 전환 gRNA 당 48 콜로니 suff해야한다효율이 목표 궤적에 따라 달라질 수 있지만 icient는 단일 유전자에 대해 여러 monoallelic 및 biallelic 돌연변이 라인을 설정합니다. 훈련 된 개인 기계적 단번에 384 콜로니 (4 × 96 웰 플레이트)을 선택하는 해부 현미경 ~ 4 시간이 소요되는 것이 가능하기 때문에, 숙련 된 개인은 내 12 유전자에 영향을 미치는 돌연변이 라인을 생성하는 것으로 할 수있다 12 개월. 단시간 hPSC 돌연변이 유선형 생성 발달 과정 (9)을 조절, 서로 상호 작용하는 전사 인자 및 / 또는 신호 경로 성분의 배열을 체계적으로 분석 할 수있다. 또한, 효율적인 다중 유전자 타겟팅은 유전 적 상호 작용을 기본 복잡한 인간의 특성을 조사에 문을 엽니 다.
정확한 유전 수정의 생성
쉽게 접근 할 수 체세포 유형에서 환자 맞춤형 iPSCs의 유도 의 질병 관련 세포 유형으로 분화 질병 관련 돌연변이의 기능 검증을위한 좋은 기회를 제공합니다. 그러나, 개인 간의 유전 적 배경에서 상당한 변화로, 환자의 iPSCs 사이의 건강한 기증자로부터 직접 비교 한 배경 효과에서 질병 표현형을 구별 할 수 없습니다 수 있습니다. 따라서, 위로 야생형 서열 질환 돌연변이를 정정하여 동질 제어 iPSCs를 생성하거나 타인 25, 26에 의해 제안 된 바와 같이, 야생형 hPSC 배경으로 환자의 특정 돌연변이를 도입하는 것이 필요하다. 기능 상실 (NULL) 돌연변이에 더하여, 하나는 더욱 정확하게 hypermorphic hypomorphic, neomorphic 또는 우성 음성 환자 포함 hPSCs에서 내인성 궤적으로 환자 별 서열 변화의 도입을 통해 질병 메커니즘을 해부 수도 돌연변이.
이 NHEJ 매개 DNA 수선보다 훨씬 덜 효율적 때문이다. 수리 템플릿의 존재 DSB 수리를 위해 HDR 고용 정확한 유전자 변형 ntent "> 환자 특이 돌연변이 약물 선택 카세트와 상 동성 암을 포함하는 도너 플라스미드 가장 널리 사용되는 Cre 호텔-에 loxP 및 FLP-FRT 시스템 남겨진 동안 이전 수리 서식이 사용되고있다. 약물 선택 및 환자 특이적인 변이의 확인 후, 제 2 단계는 일반적. 약물 선택 카세트를 제거하는 데 필요한 내인성 유전자좌 잔류 시퀀스는 피기 백 트랜스포존의 사용은 약물의 선택, 카세트 (27)의 완벽한 제거를 위해 사용할 수있다.보다 최근에, 짧은 ssDNA를 템플릿이 또한 설계 DNA 엔도 뉴 클레아 28 효율적인 HDR을 지원하기 위해 도시되어있다. 도너 플라스미드에 비해 , ssDNA를 직접 합성하고, 따라서 많은 시간이 소요되는 복제 단계를 회피 할 수있다. 여기서, 보유 될도시 욕실, 그 공동 형질 상동 - 감독 수리를 유도하는 gRNA 및 ssDNA를 템플릿을 iCRISPR 높은 효율로 특정 염기 변형을 도입하는 데 사용할 수 있습니다로. 이것은뿐만 아니라 인간 질환 돌연변이를 모델링하고 잠재적으로 치료 학적 개입이 질환 - 관련된 돌연변이를 보정하기위한 단백질의 기능적 도메인 내 필수적인 뉴클레오티드의 역할을 절개하지 중요하다. 때문에 각 당뇨병과 같은 복잡하고 multigenic 질병 모델링, 여러 시퀀스 변형과 관련된 감수성 유전자좌의 많은 수의 유전에 대한 도전이었다. iCRISPR 플랫폼은 대립 시리즈의 급속한 발전 또는 multiplexable 유전자 타겟팅 관대 같이, 개별적으로 또는 동종의 배경과 함께, 다수의 질병 - 관련 궤적의 조사를 용이하게 할 수있다.효율성 오프 대상 효과를 타겟팅
우리는이 T7E1 및 RFLP 분석 결과 및 시퀀싱에 의해 식별 돌연변이 라인의 수 사이의 상관 관계를 발견 하였다. 이 클론 라인의 설립과 평행이 분석을 수행의 중요성을 강조한다. 대부분 단일 유전자 타겟팅 실험에서 유전자 좌위에 따라 달라질 수 달성 타겟팅 효율이 20 동안 - 클론의 60 %가 두 대립 유전자가 돌연변이가 발견되었다 (프레임 이동 돌연변이를 포함하고 프레임 내의) 9. 다중 유전자 타겟팅이 수행 된 경우, 5 ~ 10 %의 효율 트리플 biallelic 돌연변이 클론 5를 얻었다. 10 % - 5 여러 유전자 ssDNA를 매개 HDR은 노크 1에서부터 클론 동형 획득 효율과 정확한 유전 적 변형을 수득 하였다. 같은 gRNA 대상 시퀀스를 공유하지 않는 잠재적 인 오프 대상 사이트에 hPSCs에 CRISPR / 카스 어떤 돌연변이를 작업 아직 검출한다아가씨 = "외부 참조"> 5, 9, 12. 최근의 연구에서 수행 전체 게놈 시퀀싱은 CRISPR / 카스 (29)를 사용하여 생성 된 클론 hPSC 라인에 상당한 오프 대상 돌연변이를 식별하는 데 실패했습니다. 그러나 오프 - 타겟 효과 부위에서 돌연변이에 의해 도입 된 표현형을 교란의 잠재적 효과를 최소화하기 위해, 동일한 유전자 내에서 상이한 서열을 타겟팅 적어도 두 개의 독립적 gRNAs 독립된 돌연변이 라인을 생성하기 위해 제안된다. 다열 관찰 유사한 표현형은 주로 표현형 오프 - 타겟 효과에서 유래한다는 가능성을 배제 할 수있는 다른 gRNAs을 이용하여 생성.
공급 장치에 의존하는 독립적 인 문화 타겟팅 대
hPSC 문화에 대한 전통적인 방법은 동물의 자극을 포함 혈청 또는 혈청 교체를 포함하는 배지에서 피더 세포에 자신의 유지 보수 및 확장을 포함예컨대 소 혈청 알부민 ucts을. 피더 세포, 혈청, 혈청 교체 및 모든 알부민은 복잡하고 정의되지 않은 성분을 함유하고 상당한 배치 변동성을 보여줍니다. 피더 - 무료 및 화학적 조건에 적응하는 것은 크게, 더 중요한 것은, 분화 실험의 일관성을 증가 hPSC의 유지 보수를 위해 노력을 줄일 수 있습니다. 현재, 완전히 정의 배양 조건 (30)에 배양 hPSCs에 게놈 편집 절차를 설명 거의 연구가있다. 우리는 gRNA 형질 전환 및 클론 증착 장치에 의존하는 배양 조건에 비해 공급이없는 조건에서 수행했을 때 효율성을 대상으로 높은 CRISPR을 발견했다. 우리는이 식민지 형성을위한 형질 전환 및 단일 세포 파종 후 증가 된 세포 생존에 기인한다고 믿는다. 또, 피더 세포는 이전시켜 형질 전환 효율을 저하 형질 전환 시약을 격리시키는 것으로 나타났다.
결합감독 차별화와 게놈 편집
이전 분화 프로토콜 인슐린 양성 세포의 작은 분획을 수득 한 대다수의 polyhormonal 있었다 태아 내분비 세포 (23)를 닮았다. 최근의 진보는 더 성숙 글루코스 응답 베타 형 셀 16, 17, 19, 20로 hPSCs의 분화를 허용하고있다. 우리는 일상적으로 HUES8 hPSCs (9)를 사용하여 75 % 이상 최종 내배엽 세포, 40 % PDX1 + NKX6.1 + 췌장 전구 세포, 그리고 20 %의 NKX6.1 + CPEP + 포도당 응답 베타 - 유사 세포를 얻을 수 있습니다. iCRISPR 게놈 편집 시스템과 결합이보다 견고한 분화 프로토콜 췌장 분화 췌장 전구 세포와 내분비 단계에 중요하다 전사 인자의 분석을 용이하게하고있다. 이 것그것은 가능한 미래 연구에서 당뇨병 9 기초 메커니즘으로 기능 검증 및 조사 후보 질환 유전자의 다수를 조사.
미래 응용 프로그램 또는 방향
등을 통해 기자 대립 유전자의 생성과 같은보다 복잡한 유전자 변형의 발생을 용이하게 할 수있다 우리의 iCRISPR 시스템 긴 기증자의 DNA 템플릿 인코딩 단백질 태그 또는 형광 기자 (12)를 사용하여 표적 유전자를 HDR은 중재. 우리 인해 시스템 효율을 달성 타겟팅 높은 CRISPR에 그렇게 도시 한이 프로세스는 또한 약물의 선택 (12) 없이도 hPSCs 수행 될 수있다. 또한, 표적 유전자는 다중화 우리는 직장 (31)의 첫번째 예를 판단 우리의 최근의 연구에서와 같이, 기본 복합 인간 질병 유전자의 상호 작용을 조사 할 수있다. ICRISPR 또한 프로모터 및 인핸서와 같은 조절 영역, 결실을 생성하여 하나의 RNA 비 암호화 유전자 또는 유전자 규제 제어를 이해하는데 사용될 수있다. 효율적 iCRISPR를 사용 규제 돌연변이를 생성하기 gRNAs을 포함하는 DNA 결합 단백질의 결합 부위를 방해하도록 설계되지만 기저 전사 기계 또는 조직 - 특이 적 전사 인자로 제한되지 않을 수있다. ssDNA를 템플릿 매개 HDR은 특정 단백질 - 결합 부위를 돌연변이를 위해 사용될 수있다. 마지막으로, 체외 분화 프로토콜과 결합 될 때 더 최적화 능성 표현형 또는 질환 표현형의 높은 처리량에 대한 유전자 분석 hPSCs에서 iCRISPR 플랫폼의 사용을 가능하게 할 것으로 예상 할. 이 iCRISPR 중재 연구는 후보의 질병 관련 유전자 및 기능 관련 연구의보다 신속한 식별을 위해 허용 할 수있다.
공개
저자는 공개 아무것도 없어.
감사의 말
이 작품은 NIH / NIDDK (R01DK096239) 및 뉴욕 주 줄기 세포 과학 (NYSTEM C029156)에 의해 부분적으로 투자되었다. ZZ는 슬로안 케터링 연구소의 줄기 세포 생물학 센터에서 NYSTEM 박사 친교에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| Chemically defined medium (E8) | Thermo Fisher Scientific | A1517001 | Essential 8 basal medium and Essential 8 supplement included |
| Truncated recombinant human form of vitronectin | Thermo Fisher Scientific | A14700 | |
| ROCK inhibitor Y-27632 | Selleck Chemicals | S1049 | |
| Dissociation reagent (TrypLE Select enzyme) | Thermo Fisher Scientific | 12563029 | 1x, animal origin free, recombinant enzyme |
| G418 Sulfate | Thermo Fisher Scientific | 10131035 | Geneticin Selective Antibiotic |
| Puromycin dihydrochloride | Sigma-Aldrich | P8833 | Puromycin Selective Antibiotic |
| DNA Polymerase (Herculase II Fusion) | Agilent Technologies | 600679 | PCR kit |
| MEGAshortscript T7 Transcription kit | Thermo Fisher Scientific | AM1354 | |
| MEGAclear Transcription Clean-Up Kit | Thermo Fisher Scientific | AM1908 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| Opti-MEM medium | Thermo Fisher Scientific | 31985062 | Reduced Serum Medium |
| Lipofectamine RNAiMAX Transfection Reagent | Thermo Fisher Scientific | 13778150 | |
| DNeasy Blood & Tissue Kit | QIAGEN | 69504 | Genomic DNA extraction kit |
| Proteinase K | Roche | 3115879001 | |
| MCDB 131 medium | Thermo Fisher Scientific | 10372-019 | |
| BLAR medium | Thermo Fisher Scientific | Custom-made with a published formulation (Rezania et al., 2014) | |
| L-glutamine supplement (GlutaMAX) | Thermo Fisher Scientific | 35050061 | |
| NaHCO3 | Thermo Fisher Scientific | 144-55-8 | |
| Glucose | Sigma-Aldrich | G8769 | |
| BSA | LAMPIRE Biological Laboratories | 7500855 | Fatty acid free |
| GDF8 | PeproTech | 120-00 | |
| CHIR-99021 | Stemgent | 04-0004 | GSK-3 inhibitor |
| L-Ascorbic acid | Sigma-Aldrich | A4544 | Vitamin C |
| FGF7 | R&D Systems | 251-KG | |
| SANT1 | Tocris Bioscience | 1974 | Hedgehog inhibitor |
| RA | Sigma-Aldrich | R2625 | Retinoic acid |
| LDN | Stemgent | 04-0019 | BMP inhibitor |
| IWP-2 | Tocris Bioscience | 3533 | Wnt antagonist |
| ITS-X | Thermo Fisher Scientific | 51500-056 | |
| TPB | EMD Millipore | 565740-1MG | PKC activator |
| 3,3’,5-Triiodo-L-thyronine (T3) | Sigma-Aldrich | T6397 | |
| ALK5i II | Enzo Life Sciences | ALX-270-445 | ALK5 inhibitor II |
| ZnSO4 | Sigma-Aldrich | Z0251 | |
| Heparin | Sigma-Aldrich | H3149 | |
| GSiXX | EMD Millipore | 565789 | Gamma secretase inhibitor XX, NOTCH signaling inhibitor |
| N-Cys (N-acetyl cysteine) | Sigma-Aldrich | A9165 | |
| Trolox | EMD Millipore | 648471 | Vitamin E analogue |
| R428 | Selleck Chemicals | S2841 | AXL receptor tyrosine kinase inhibitor |
| 24 mm Transwell with insert | Corning Life Sciences | 3414 | |
| Gene Pulser Xcell Electroporation System | Bio-Rad | 1652660 | |
| 0.4 cm Electroporation Cuvettes | Bio-Rad | 1652081 | |
| AAVS1-TALEN-L | Addgene | 59025 | |
| AAVS1-TALEN-R | Addgene | 59026 | |
| AAVS1-Neo-M2rtTA | Addgene | 60843 | |
| AAVS1-Puro-iCas9 | Addgene | 58409 | |
| T7 Endonuclease I | NEB | M0302L | |
| Buffer 2 | NEB | B7002S | NEBuffer 2 |
| Long oligonucleotide | Eton Bioscience or IDT | ||
| SOX17 antibody | R&D Systems | AF1924 | 1:500 |
| FOXA2 antibody | Millipore | 07-633 | 1:100 |
| CXCR4-APC antibody | R&D Systems | FAB170A | 1:25 |
| PDX1 antibody | R&D Systems | AF2419 | 1:500 |
| NKX6.1 antibody | DSHB | F55A12 | 1:500 |
| NKX2.2 antibody | DSHB | 74.5A5 | 1:100 |
| NEUROD1 antibody | Santa Cruz Biotechnology | sc-1084 | 1:100 |
| Insulin antibody | Dako | A0564 | 1:2,000 |
| C-peptide antibody | DSHB | GN-ID4-c | 1:2,000 |
| Glucagon antibody | Sigma-Aldrich | G2654 | 1:1,000 |
참고문헌
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Eiges, R., et al. Establishment of human embryonic stem cell-transfected clones carrying a marker for undifferentiated cells. Curr Biol. 11 (7), 514-518 (2001).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- González, F., et al. An iCRISPR Platform for Rapid, Multiplexable, and Inducible Genome Editing in Human Pluripotent Stem Cells. Cell Stem Cell. 15 (2), 215-226 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Braam, S. R., et al. Improved genetic manipulation of human embryonic stem cells. Nat Methods. 5 (5), 389-392 (2008).
- Zhu, Z., et al. Genome Editing of Lineage Determinants in Human Pluripotent Stem Cells Reveals Mechanisms of Pancreatic Development and Diabetes. Cell Stem Cell. 18 (6), 755-768 (2016).
- Kotini, A. G., et al. Functional analysis of a chromosomal deletion associated with myelodysplastic syndromes using isogenic human induced pluripotent stem cells. Nat Biotechnol. 33 (6), 646-655 (2015).
- Carlson-Stevermer, J., et al. High-Content Analysis of CRISPR-Cas9 Gene-Edited Human Embryonic Stem Cells. Stem Cell Reports. 6 (1), 109-120 (2016).
- Zhu, Z., Verma, N., Gonzalez, F., Shi, Z. D., Huangfu, D. A CRISPR/Cas-Mediated Selection-free Knockin Strategy in Human Embryonic Stem Cells. Stem Cell Reports. 4 (6), 1103-1111 (2015).
- Mandegar, M. A., et al. CRISPR Interference Efficiently Induces Specific and Reversible Gene Silencing in Human iPSCs. Cell Stem Cell. 18 (4), 541-553 (2016).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Methods. 8 (5), 424-429 (2011).
- Rezania, A., et al. Production of functional glucagon-secreting alpha-cells from human embryonic stem cells. Diabetes. 60 (1), 239-247 (2011).
- Pagliuca, F. W., et al. Generation of functional human pancreatic beta cells in vitro. Cell. 159 (2), 428-439 (2014).
- Rezania, A., et al. Reversal of diabetes with insulin-producing cells derived in vitro from human pluripotent stem cells. Nat Biotechnol. 32 (11), 1121-1133 (2014).
- Chen, S., et al. A small molecule that directs differentiation of human ESCs into the pancreatic lineage. Nat Chem Biol. 5 (4), 258-265 (2009).
- Nostro, M. C., et al. Efficient generation of NKX6-1+ pancreatic progenitors from multiple human pluripotent stem cell lines. Stem Cell Reports. 4 (4), 591-604 (2015).
- Russ, H. A., et al. Controlled induction of human pancreatic progenitors produces functional beta-like cells in vitro. EMBO J. 34 (13), 1759-1772 (2015).
- Zhu, Z., Gonzalez, F., Huangfu, D. The iCRISPR platform for rapid genome editing in human pluripotent stem cells. Methods Enzymol. 546, 215-250 (2014).
- Soh, C. L., Huangfu, D. CRISPR/Cas9-Mediated Mutagenesis of Human Pluripotent Stem Cells in Defined Xeno-Free E8 Medium. Methods in Molecular Biology. 1498, (2017).
- D'Amour, K. A., et al. Production of pancreatic hormone-expressing endocrine cells from human embryonic stem cells. Nat Biotechnol. 24 (11), 1392-1401 (2006).
- Kelly, O. G., et al. Cell-surface markers for the isolation of pancreatic cell types derived from human embryonic stem cells. Nat Biotechnol. 29 (8), 750-756 (2011).
- Musunuru, K. Genome editing of human pluripotent stem cells to generate human cellular disease models. Dis Model Mech. 6 (4), 896-904 (2013).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell Stem Cell. 18 (5), 573-586 (2016).
- Yusa, K., et al. Targeted gene correction of alpha1-antitrypsin deficiency in induced pluripotent stem cells. Nature. 478 (7369), 391-394 (2011).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Huang, X., et al. Production of Gene-Corrected Adult Beta Globin Protein in Human Erythrocytes Differentiated from Patient iPSCs After Genome Editing of the Sickle Point Mutation. Stem Cells. 33 (5), 1470-1479 (2015).
- Shi, Z. D., et al. Genome Editing in hPSCs Reveals GATA6 Haploinsufficiency and a Genetic Interaction with GATA4 in Human Pancreatic Development. Cell Stem Cell. , in press (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유